Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Fluorescencia de células activadas por la clasificación de los protoplastos de la planta
En este artículo
Resumen
Un método para el aislamiento de determinados tipos de células a partir de material vegetal se demuestra. Esta técnica emplea las líneas transgénicas que expresan proteínas fluorescentes marcador en los tipos de célula en particular, la disociación y la fluorescencia celular selección de células activadas. Además, un crecimiento de configuración se ha establecido aquí que facilita el tratamiento de Arabidopsis thaliana Plantones antes de la clasificación de células.
Resumen
De alta resolución, el tipo específico de célula de análisis de la expresión génica mejora considerablemente la comprensión de la regulación del desarrollo y las respuestas a los estímulos del medio ambiente en cualquier organismo multicelular. Hibridación in situ y la visualización del gen reportero puede hasta cierto punto se utiliza para este fin, sino para alta resolución cuantitativa RT-PCR o de alto rendimiento en todo el análisis del transcriptoma el aislamiento de ARN a partir de tipos de células es un requisito. Disociación celular de los tejidos que expresan un marcador de la proteína fluorescente en un tipo específico de célula y la posterior clasificación de células activadas fluorescencia (FACS) hace que sea posible reunir una cantidad suficiente de material para la extracción de ARN, el análisis de cDNA síntesis / amplificación y microarrays.
Un amplio conjunto de tipo de células específicas líneas reportero fluorescente está disponible para la comunidad de investigación de la planta. En este caso, dos líneas de marcador de la raíz de Arabidopsis thaliana se utilizan: P SCR:: GFP (endodermis y el centro de reposo) y P WOX5:: GFP (centro de reposo). Un gran número (miles) de las plántulas se cultivan hidropónicos o en placas de agar y se cosechan para obtener material suficiente raíz para su posterior análisis. Disociación celular de material vegetal se obtiene por digestión enzimática de la pared celular. Este procedimiento hace uso de alta osmolaridad inducida plasmólisis y celulasas disponibles en el mercado, pectinasas y hemicelulasas para liberar protoplastos en la solución.
FACS de las buenas prácticas agrarias de células positivas hace uso de la visualización de los verdes contra los espectros de emisión de color rojo de protoplastos excitado por un láser de 488 nm. GFP-positivas protoplastos se pueden distinguir por su relación cada vez mayor de verde a la emisión de color rojo. Protoplastos son típicamente ordenados directamente en tampón de extracción de ARN y se almacena para su posterior procesamiento en un momento posterior.
Esta técnica se revela que es sencillo y práctico. Además, se demuestra que se puede utilizar sin dificultad de aislar un número suficiente de células para el análisis de transcriptoma, incluso para los tipos de células muy escasos (por ejemplo, las células del centro de reposo). Por último, una configuración de crecimiento de plántulas de Arabidopsis se ha demostrado que permite el tratamiento sin complicaciones de las plantas antes de la clasificación de células (por ejemplo, para el tipo específico de célula de análisis de las respuestas al estrés biótico o abiótico). Complementarios usos potenciales de FACS de protoplastos de plantas se discuten.
Protocolo
1) Preparación del material vegetal
- Protoplastos se puede derivar de muchas especies de plantas y tejidos, siempre que la mezcla correcta de la pared celular enzimas digestivas se utiliza 1. Antes de que un experimento a gran escala se lleva a cabo una digestión de pequeña escala de la materia es aconsejable con el fin de evaluar la eficiencia protoplasting de los tejidos, enzimas, etc, y para estimar el porcentaje de células positivas para la clasificación de la célula. Aquí, los protoplastos derivados de las raíces de las plántulas de Arabidopsis thaliana que tipo de célula específica expresan la proteína verde fluorescente (GFP) se utilizan. La endodermis y el centro de reposo se caracterizan por P SCR:: GFP y el centro de reposo por P WOX5:: GFP 2,3 (Figura 1).
- Las plántulas se cultivan en hidroponia phytatrays (Sigma; Figura 2a) en un filtro de nylon (250 micras de malla; Nitex) que permite que las raíces crecen a través de en el medio de crecimiento (0,22% w / v medio de Murashige y Skoog [Sigma], el 1% w / v de sacarosa, 0,05% w / v MES [2 - (N-morfolino) etanosulfónico ácido], pH 5,7 con KOH). Por otra parte, las plantas pueden ser cultivadas en la parte superior de un filtro de nylon (malla 100 micras) en posición vertical placas de agar al 1% (figura 2b).
- El uso de los filtros antes mencionados no sólo ayuda en la cosecha de las raíces, sino que también facilita el tratamiento fácil adicionales de las plantas, si se desea. Los filtros permiten a las plantas para ser transferido en masa a phytatrays nuevas o las placas de agar complementado con un catalizador de interés. Por ejemplo, el conjunto phytatray hasta se ha utilizado para el tipo de células específicas de análisis de la respuesta transcripcional a un tratamiento de nitrógeno en plantas de Arabidopsis 4.
- Asegúrese de que su microscopio marcador fluorescente se expresa correctamente (especialmente cuando se utiliza la opción de tratamiento, como el tipo de células específicas de marcadores se puede estar influida por el tratamiento). En este caso, las plántulas son inspeccionados bajo un microscopio de fluorescencia (Nikon, Figura 1). Tenga en cuenta que tipo de células específicas líneas de marcador fluorescente que se caracteriza inicialmente con un microscopio confocal para determinar exactamente qué tipos de células se marcan y determinan la variabilidad en la expresión.
2) Preparación de la solución protoplasting
- Disolver 1,25% w / v celulasa (Yakult), un 0,3% w / v Macerozyme (Yakult), 0,4 M D-manitol, 20 mM MES y 20 mM KCl (de una reserva de 1 M) en agua desmineralizada y ajustar el pH a 5.7 con 1 M Tris / HCl pH 7,5. Esta solución será ligeramente turbio.
- Calentar la solución a 55 ° C durante 10 minutos (la solución se volverá claro) y dejar que se enfríe a temperatura ambiente antes de la adición de 0,1% w / v BSA (albúmina de suero bovino), 10 mM CaCl 2, y 5 mM β-mercaptoetanol .
3) La cosecha y protoplasting del material vegetal
- Las raíces se cosechan por raspado de ellos fuera de la malla de nylon con un bisturí y se deposita en un frasco que contiene la solución protoplasting. En general, 10 ml de solución protoplasting se utiliza por cada 1.500 raíces de las plántulas.
- Agitar los frascos con cuidado (75 rpm) a temperatura ambiente durante una hora. Un tiempo de incubación más largo puede aumentar el rendimiento de protoplastos, pero también se sumará a los efectos de protoplasting sí mismo en la expresión génica.
- Filtrar la solución de protoplastos con un filtro de 40 micras de células (BD Falcon) y dividir la solución en forma cónica tubos de 15 ml (BD Falcon).
- Girar los tubos en una centrifugadora basculante durante 10 minutos a 500 G. Nota que la velocidad de centrifugación dependerán del tipo de protoplastos utilizados, su fragilidad y la cantidad de restos celulares producidos durante el tratamiento enzimático.
- Eliminar la mayor parte del sobrenadante, resuspender los protoplastos en el resto de la solución e inspeccionarlos microscopio (Figura 3).
- Hacer uso de un hemocitómetro para estimar el número y la densidad de protoplastos. La densidad celular se determinará la velocidad de inyección de la muestra, los eventos por segundo y por lo tanto el tiempo total necesario para ordenar las células en la FACS (ver sección 4.3).
- 3.7) Cualquier proceder directamente a FACS o lavar y volver a suspender los protoplastos en una solución de incubación, como W5 (154 mM NaCl, 125 mM CaCl2, KCl 5 mM, 5 mM MES, ajustar el pH a 5,7 con KOH) o una solución protoplasting sin la añadido enzimas. Sobre todo cuando se observan los cambios transcripcional, es de suma importancia para minimizar la exposición de las muestras a las condiciones que pueden influir en la expresión de genes, como el cambio del buffer los protoplastos se mantienen in Por ello se recomienda mantener los protoplastos en una solución de protoplasting y proceder FACS a la mayor brevedad posible.
4) fluorescencia de células activadas por la clasificación de protoplastos
- Encender y preparar el clasificador de células. Aquí, un FACSAria (BD) se utiliza.
- Establecer una corriente de flujo con un 100 y mu, m de la boquilla y una presión de 20 psi vaina.
- La densidad de las células y la velocidad de inyección de la muestra se puede ajustar a una experiencia particular en función de si un rendimiento mejor o mayor velocidad alcanzable es deseado. Hemos clasificado con éxito con densidades de hasta 10 millones células / ml.
- Use la opción de la agitación de la muestra en la FACS para prevenir la sedimentación de los protoplastos. Si la obstrucción de la FACS es un problema, hay tres pasos para solucionar problemas que: 1. Realizar una inversión de flujo de la muestra-line. 2. Diluir la suspensión de protoplastos para reducir la densidad. 3. Limpieza de la solución de protoplastos mediante la repetición de la etapa de filtración (3.3) después de la centrifugación y resuspensión.
- Prepare el aparato para medir la dispersión frontal (FSC), dispersión lateral (SSC) y la emisión de 530/30 nm para GFP y 610/20 nm para el espectro de color rojo con autofluorescencia (RSA) después de la excitación por un láser de 488 nm. Estos son, en esencia, los únicos parámetros que utilizan para aislar la GFP positivos protoplastos. En este caso, los valores de voltaje son los siguientes: FSC - 60 V, 250 V SSC, 350V y 335V GFP RSA. Tenga en cuenta que los valores de voltaje óptimo será diferente para cada FACS e incluso tendrá que ser ajustado a lo largo de la vida del clasificador de células.
- Comience por la creación de un diagrama de puntos de dispersión frontal versus dispersión lateral. Aplicar los valores de voltaje de modo que los eventos medidos se centran en la parcela.
- A continuación, cree un gráfico de puntos de señales de fluorescencia verde frente a roja. Aplicar los valores de voltaje de modo que los acontecimientos se cuantificó el rendimiento de una población centrada en diagonal en la trama cuando se mira en una suspensión de protoplastos de tipo salvaje (no-GFP). Una suspensión de protoplastos derivados de una línea de marcador GFP se produce una población libre de color verde fluorescente eventos nunca antes vista en las muestras de tipo salvaje.
- Establecer restricciones para ajustar la compensación de solapamiento espectral entre las buenas prácticas agrarias y la RSA. Correcta configuración de restricción de la compensación permitirá una mejor separación de los protoplastos GFP-positivas de los protoplastos no las buenas prácticas agrarias y los escombros. Las restricciones se utiliza aquí es el siguiente: RSA, menos 17,91% GFP.
- Configurar una puerta para identificar las buenas prácticas agrarias-positivas eventos, un control negativo de la no-GFP protoplastos se debe utilizar para ayudar a definir los límites de la puerta (Figura 4).
- Implementar un corte de dispersión hacia adelante para dejar pequeños restos fuera del análisis. Visualizar los eventos de GFP-positivas en el FSC frente a SSC complot para ayudar a determinar la ubicación de la corte. Aquí, el punto de corte se estableció en 5000. Tenga en cuenta que la FACS contará escombros ordenar eventos y una muestra con altos niveles de residuos puede tener una serie de eventos por ciento diferente GFP positivos de lo esperado. Esto no es necesariamente un problema. Sin embargo, los restos más en la muestra, mayor será el tipo va a tomar.
- Dependiendo del experimento y la abundancia del tipo de células a analizar, establecer el modo de precisión, FACS, ya sea para un rendimiento óptimo o la pureza óptima de las células ordenados.
- Para la extracción de RNA, preparar los tubos de recogida (1,5 ml tubos de microcentrífuga) con la cantidad adecuada de tampón de extracción de RNA. Con esta configuración, 20.000 eventos de clasificación dará un volumen total de aproximadamente 100 l, que se clasificaron en 350 l de tampón de extracción (micro RNeasy kit, QIAGEN). Mezcla de las muestras después de la finalización de la especie como la suspensión de células pueden reunir en la parte superior.
- Almacenar las muestras o proceder directamente a la extracción de RNA. Análisis de microarrays exitosos se han preformado con el ARN extraído de tan poco como 500 eventos ordenados. En este caso, se utilizó un kit de extracción RNeasy micro (QIAGEN), el WT-Ovation Pico Sistema de amplificación de ARN y FL-Ovation cDNA biotina módulo V2 (NuGEN).
Resultados representante
Una phytatray de aproximadamente 1.500 SCR de una semana de edad, P:: GFP plántulas produjo unos 60.000 protoplastos (medida por el hemocitómetro). 2,6% de 65.000 eventos FACS-procesado se define como GFP-positivas y fueron ordenados (Figura 4b).
Ocho placas de aproximadamente 1500 de cuatro días de edad, P WOX5:: GFP plantas cada uno (12.000 en total) producido por 30.000.000 protoplastos (medida por el hemocitómetro). 0,063% de los 16.000.000 eventos FACS-procesado se define como GFP-positivas y fueron ordenados (Figura 4c).
10.000 eventos ordenados se utilizan normalmente para la extracción de ARN y puede producir 20 a 140 ng de ARN total (Figura 5).
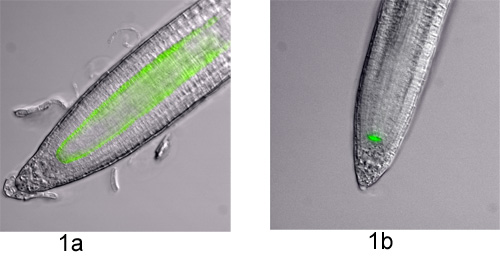
Figura 1. Tipo de células específicas líneas marcador GFP en la raíz de Arabidopsis. Imágenes de microscopía de fluorescencia fueron tomadas con contraste de interferencia diferencial (DIC) y un filtro de buenas prácticas agrarias en un microscopio Eclipse 90i (Nikon) que se ejecutan en el software Metamorph (Molecular Devices). Las imágenes DIC y GFP se superpusieron para fines de visualización. Las dos líneas de marcador utilizado en este experimento visual se muestran;a) P SCR:: GFP y b) P WOX5:: GFP.

Figura 2. Plántulas condiciones de crecimiento. Crecieron en un controlador del medio ambiente hidropónico en phytatrays (a) o en placas de agar en posición vertical (b).
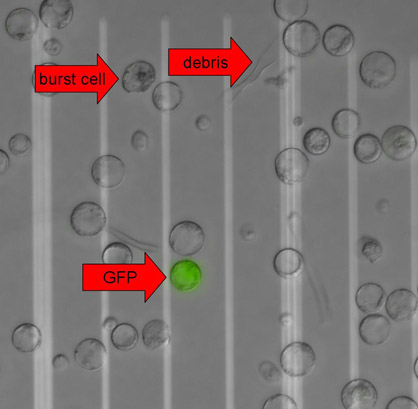
Figura 3. Protoplastos de plantas que expresan GFP. Imágenes de microscopía de fluorescencia fueron tomadas con contraste de interferencia diferencial (DIC) y un filtro de buenas prácticas agrarias en un microscopio Eclipse 90i (Nikon) que se ejecutan en el software Metamorph (Molecular Devices). Las imágenes DIC y GFP se superpusieron para fines de visualización. Las flechas indican el estallido de una célula, restos celulares y protoplastos GFP-positivas. La distancia entre dos líneas blancas es de 50 micras.
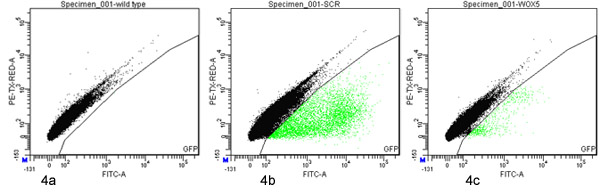
Figura 4. Fluorescencia de células activadas por la clasificación de la GFP-positivas protoplastos protoplastos derivados de tipo salvaje (a) P SCR.:: GFP (b) o P WOX5:: GFP (c) líneas de marcadores fueron analizados y ordenados con una FACSAria (BD) con puertas definido en una gráfica de puntos verdes (530/30 nm; eje x) frente roja (610/20 nm; eje y) de fluorescencia. 100.000 eventos se presentan en cada parcela. Los eventos incluidos en la puerta de la clasificación se destacan las buenas prácticas agrarias verde.
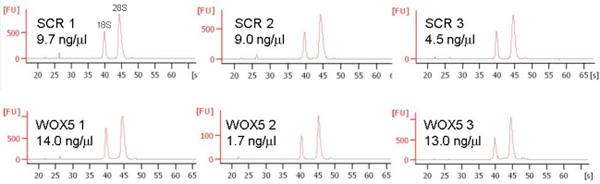
Figura 5. Representante de las extracciones de ARN de 10.000 células seleccionadas. Las células fueron ordenados directamente en la extracción del ARN de amortiguación (QIAGEN), el ARN fue purificado y se analiza la concentración, la pureza y la integridad en un Bioanalyzer 2100 (Agilent). Tres réplicas de las dos líneas marcador se muestran.
Discusión
Protoplastos puede, en principio, se derivan de una variedad de tejidos de la planta, optimización de las condiciones favorables en gran medida mejorar la calidad y la cantidad de ARN. Tanto la solución protoplasting y el tampón de incubación electiva utilizado influirá en este aspecto.
Muchas proteínas fluorescentes se pueden utilizar diferentes, dependiendo de las capacidades de la FACS utiliza, por ejemplo, GFP, RFP, YFP, PPC o sus muchas variantes y derivados. La expresió...
Agradecimientos
Este trabajo fue apoyado por la National Science Foundation (subvención no. DBI 0519984) y los Institutos Nacionales de Salud (concesión no. 5R01GM078279) ..
Materiales
| Name | Company | Catalog Number | Comments |
| 250 μm nylon mesh | Sefar Filtration | NITEX 03-250/50 | |
| 100 μm nylon mesh | Sefar Filtration | NITEX 03-100/47 | |
| Square petri dishes | Fisher Scientific | 08-757-10k | |
| Phytatrays | Sigma-Aldrich | P1552 | |
| Murashige and Skoog Basal Medium (MS) | Sigma-Aldrich | M5519 | |
| sucrose | Fisher Scientific | S5-3 | |
| MES | Sigma-Aldrich | M2933 | |
| KOH | Sigma-Aldrich | P1767 | 10 M stock |
| Eclipse 90i microscope | Nikon Instruments | ||
| Cellulase R-10 | Yakult Pharmaceutical | ||
| Macerozyme R-10 | Yakult Pharmaceutical | ||
| D-mannitol | Sigma-Aldrich | M9546 | |
| KCl | Sigma-Aldrich | P8041 | 1 M stock |
| BSA | Sigma-Aldrich | A3912 | |
| β-mercapt–thanol | Calbiochem | 444203 | |
| CaCl2 | Sigma-Aldrich | C2536 | 1 M stock |
| orbital shaker | Labline Instruments | ||
| 40 μm cell strainer | BD Biosciences | 352340 | |
| conical 15 ml tubes | BD Biosciences | 352196 | |
| table centrifuge | Sorvall, Thermo Scientific | Legend RT | |
| NaCl | Sigma-Aldrich | S3014 | |
| FACSAria | BD Biosciences | ||
| 1.5 ml microfuge tubes | VWR international | 20170-38 | |
| RNeasy micro kit | Qiagen | 74004 | |
| WT-Ovation Pico RNA Amplification System | NuGEN | 3300_12 | |
| FL-Ovation cDNA Biotin Module V2 | NuGEN | 4200_12 |
Referencias
- Sheen, J. Signal transduction in maize and Arabidopsis mesophyll protoplasts. Plant Physiol. 127, 1466-1475 (2001).
- Wysocka-Diller, J. W., Helariutta, Y., Fukaki, H., Malamy, J. E., Benfey, P. N. Molecular analysis of SCARECROW function reveals a radial patterning mechanism common to root and shoot. Development. 127, 595-603 (2000).
- Blilou, I., Xu, J., Wildwater, M., Willemsen, V., Paponov, I., Friml, J., Heidstra, R., Aida, M., Palme, K., Scheres, B. The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots. Nature. 433, 39-44 (2005).
- Gifford, M. L., Dean, A., Gutierrez, R. A., Coruzzi, G. M., Birnbaum, K. D. Cell-specific nitrogen responses mediate developmental plasticity. Proc Natl Acad Sci U S A. 105, 803-808 (2008).
- Bargmann, B. O. R., Birnbaum, K. D. Positive fluorescent selection permits precise, rapid, and in-depth overexpression analysis in plant protoplasts. Plant Physiol. 149, 1231-1239 (2009).
- Petersson, S. V., Johansson, A. I., Kowalczyk, M., Makoveychuk, A., Wang, J. Y., Moritz, T., Grebe, M., Benfey, P. N., Sandberg, G., Ljung, K. An Auxin Gradient and Maximum in the Arabidopsis Root Apex Shown by High-Resolution Cell-Specific Analysis of IAA Distribution and Synthesis. Plant Cell. 21, 1659-1668 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados