Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Microscopía óptica de dispersión basado en dos dimensiones filtros Gabor
En este artículo
Resumen
Demostramos un método de microscopía de campo oscuro, sobre la base de Gabor-como el filtrado para medir la dinámica subcelulares dentro de células vivas individuales. La técnica es sensible a las alteraciones en la estructura de los orgánulos, como la fragmentación de las mitocondrias.
Resumen
Demostramos un instrumento microscópico que puede medir la textura subcelular derivados de la morfología orgánulo y la organización dentro de células vivas sin teñir. El instrumento propuesto se extiende la sensibilidad de la etiqueta sin microscopía óptica a nanoescala cambios en el tamaño y la forma de orgánulos y se puede utilizar para acelerar el estudio de la relación estructura-función relativa a la dinámica de los procesos biológicos subyacentes orgánulos fundamentales, como la muerte celular programada o celular diferenciación. El microscopio se puede implementar fácilmente en las plataformas de microscopía existentes, y por lo tanto se puede difundir a los distintos laboratorios, donde los científicos pueden implementar y utilizar los métodos propuestos con un acceso sin restricciones.
La técnica propuesta es capaz de caracterizar la estructura subcelular mediante la observación de la célula a través de dos dimensiones ópticas filtros de Gabor. Estos filtros se puede ajustar a nanoescala con sentido (10 de nm) la sensibilidad, los atributos específicos morfológicas relacionadas con el tamaño y la orientación de la no-esférica organelas subcelulares. Aunque basado en el contraste generado por la dispersión elástica, la técnica no se basa en un modelo de dispersión inversa detallado o en la teoría de Mie para extraer medidas morfométricas. Esta técnica es aplicable a los no-esférica orgánulos que una descripción precisa de dispersión teórica no es fácil dado, y proporciona distintivo parámetros morfométricos que se pueden obtener dentro de células vivas sin teñir para evaluar su función. La técnica es ventajosa en comparación con el procesamiento digital de imágenes en que opera directamente en el campo del objeto transformar en vez de la intensidad del objeto discretizado es. No se basa en altas tasas de muestreo de la imagen y por lo tanto se puede utilizar para detectar rápidamente la actividad morfológica dentro de cientos de células a la vez, por lo que facilita en gran medida el estudio de la estructura del orgánulo más allá de la segmentación de cada orgánulo y la reconstrucción mediante microscopía de fluorescencia confocal de gran aumento imágenes digitales de los campos de visión limitado.
En esta demostración se muestran los datos de una diatomea marina para ilustrar la metodología. También muestran los datos preliminares recogidos de las células vivas para dar una idea de cómo el método puede ser aplicado en un contexto biológico relevante.
Protocolo
1. Cómo las células listo
- Las células que se cultivaron el día anterior necesita ser etiquetado con el verde Mitotracker para imágenes de fluorescencia de las mitocondrias.
- Quitar una solución de 100 M de acciones verde Mitotracker en DMSO hecho antes de los 4 ° C congelador, y la temperatura ambiente con la mano. También sacan células endoteliales de aorta bovina (BAEC) medio de cultivo celular también previamente preparado y caliente a 37 ° C en el baño de agua de mesa.
- Una vez que el Mitotracker y medio de cultivo se calientan, los sitúan en el capó y asegúrese de esterilizar sus manos con guantes y todas las superficies externas de los recipientes con una solución de etanol al 70%. No encender la luz de campana, como la etiqueta fluorescente es sensible a la luz y rápidamente photobleach a la luz ambiente de la habitación.
- Hacer la concentración adecuada para el etiquetado de las mitocondrias es muy importante. Muy poco no la etiqueta de la eficacia mitocondria, mientras que Mitotracker en exceso puede tener efectos tóxicos. Una concentración de 100 nM de Mitotracker incubaron durante 45 minutos con las células funciona bien. Prepare esta concentración mediante la adición de 100 L de acciones Mitotracker a 10 ml medio de cultivo en un tubo de 15 ml. Esto hará que un montón por lo menos durante un experimento.
- Sustituya el medio existente con el medio marcado por la succión de la media de edad con una pipeta Pasteur conectada a la línea de vacío. Inmediatamente después, agregar 2 ml del medio de la etiqueta de cada cultura y ocupado en la placa de 6 pocillos.
- Debido a que la etiqueta fluorescente es sensible a la luz, reemplazan a las células en la incubadora con rapidez, sin exponer a la luz de la habitación directo. Cubrir la placa de 6 y con las manos funciona bien para esto. Las células permanecerán en la incubadora durante 45 minutos.
2. Obtención de la configuración óptica listo
- Mientras que las células de espera en la incubadora, tenemos que activar la configuración óptica. En la sala de la óptica, la primera vez en la lámpara de vapor de mercurio, seguido por el ordenador, el microscopio, las cámaras, y el láser. Luego, conecte el dispositivo digital de microespejos (DMD) y el difusor de spinning.
- Asegúrese de que el lanzamiento óptico está alineada mirando a través del ocular del microscopio para asegurarse de que el campo de visión está iluminado por la luz del láser.
- Limpie el objetivo de doblar un pedazo de papel para lentes en un cuadrado apretado y captar firmemente con una pinza hemostática. Sumerja el papel de la lente en amoníaco libre de solución de limpieza de vidrio para absorber una pequeña cantidad en el papel. Rap a la pinza hemostática en su mano libre varias veces para eliminar cualquier exceso. Limpie el objetivo con firmeza la aplicación de un golpe limpio y continua a través del objetivo de un extremo al otro, va sobre la lente en el centro. No vuelva a deslizar o matorrales. Deseche el papel usado.
- Para cargar la muestra, el lugar de la retícula sobre el objetivo de inmersión 63x dejando caer 1-2 gotas de aceite de inmersión sobre el objetivo, mientras que el objetivo es todo el camino. A continuación, coloque la retícula en el escenario. A continuación, aumentar el objetivo para que el aceite "agarra" la muestra. El enfoque de la muestra en el ocular.
- Para alinear el condensador, ajuste la altura del condensador de forma que se alinea en el centro de la iluminación Kohler, centrándose el borde hexagonal de la parada del campo del condensador. El centro del campo del condensador parada en el campo de visión si es necesario girar los mandos de centrado del condensador. La parada de apertura del condensador debe ser cerrada.
- Iniciar el programa de IPLab y la entrada de la configuración para operar la Cascada RoperScientific 512 de la cámara. Confirme que la cámara está ajustada a modo de cuadro de transferencia. Iniciar la vista previa en vivo mediante la ejecución de la "Adquisición de enfoque" de comandos. Ajuste el prefijo para el índice y la ubicación del archivo que las imágenes se guardarán.
- Iniciar el programa de RSImage y la entrada de la configuración para operar el programa CoolSnap. El modo de reloj debe ser ajustado a la normalidad.
- Inicie el software de DMD y el lugar del iris de campo oscuro en el menú de comandos, seguido por la opción "Cargar y Reset" de comandos y ejecutar el script.
- Enviar la luz a la DMD y la cascada 512 cámara mediante el establecimiento de un microscopio optovar y la vista a LSM. Esto va a enviar la imagen a través de la DMD y la óptica alineada, proyectando la imagen del DF en el CCD. El campo oscuro (DF) la imagen debe aparecer en la vista previa en vivo ya en marcha en IPLab. Ajustar el enfoque fino del microscopio, si es necesario para enfocar la imagen en la vista previa en vivo.
- Tomar una instantánea de la esfera de la vista con la "adquisición de un solo comando". Establecer el tiempo de exposición lo suficientemente alto como para garantizar al menos 10.000 cuentas de la señal de la imagen. Después de la adquisición, la opción "Grabar como indexados", para guardar la imagen en el disco. Esta imagen de la retícula mide el tamaño del campo de visión (FOV).
- Ahora, mueva la muestra cuadrícula de modo que la retícula está más allá del campo de visión para que el fondo sólo es visible. Adquirir una imagen de fondo del campo a latiempo de exposición suficiente para garantizar que al menos 5.000 cuentas de la señal que se adquiere. Esta imagen ayudará a la sustracción de fondo de las imágenes sin filtrar.
3. Carga de la Filterbank y el uso de la configuración de filtrado para adquirir imágenes de fondo-
- Ahora, tenemos que adquirir Gabor filtrado las imágenes del fondo. Cargar el filtro de Gabor guión banco para el software de control de DMD. Ejecute el script completo para amortiguar los filtros de la memoria interna de la DMD, lo que podría tardar unos minutos.
- Una vez que el guión se almacenan, que ahora pueden adquirir las imágenes filtradas del fondo. Utilice el arranque y parada marcadores en el software de DMD para instruir a la DMD para cargar sólo un conjunto de filtros que corresponde a un filtro de Gabor-como a la vez, y ejecutar el script. La vista previa de imágenes en vivo debe cambiar de campo oscuro de la imagen filtrada por el filtro.
- Abra el script de adquisición en IPLab del disco. Ajustar el tiempo de exposición para asegurar que por lo menos 2000 puntos de la señal se adquieren. Que el script se ejecuta DMD, cancelar la vista previa en vivo en IPLab y ejecutar el script de adquisición. Este adquirirá automáticamente, índice y guardar la imagen filtrada en el disco.
- Una vez que la primera imagen que se adquiere, detener el script que se ejecuta en el software de DMD y eliminar los comandos utilizados por el script. Vuelva a colocar el inicio y marcadores de parada en el comienzo y el final del set siguiente filtro. Repetir la adquisición de IPLab.
- Repita el paso hasta el 3,4 Filterbank todo se ha utilizado y todas las imágenes filtradas han sido adquiridos y se guarda.
4. Revestimiento de las células
- Por ahora, las células pronto estará listo hasta el plato para el experimento. Enchufe el soldador en la mesa de trabajo de laboratorio. Eliminar el medio de visualización L15 y el calor a 37 ° C. Hacer una estación de trabajo con una toalla de papel y un Kimwipe. Hacer mechas de varias roturas y girando Kimwipes. Las mechas le ayudará en la transferencia de fluidos desde y hacia la placa de la célula.
- Después de esto, tenemos que la placa de la muestra. Nosotros usamos los titulares de metal mecanizado de la muestra a la placa de nuestras células, haciendo un "sandwich cubreobjetos" con la placa de metal en el medio. Aplique un cordón delgado de grasa de vacío con una jeringa en la periferia superior del agujero de la placa de metal se extiende alrededor de la mitad de los extremos de las ranuras en cada lado. Presione suavemente uno no limpia. Un cubreobjetos sobre la grasa. Da la vuelta al plato una y aplicar grasa alrededor del agujero. Apague las luces del cuarto.
- Ahora tenemos las células de la incubadora, la manipulación de los contenidos incubadora con guantes de examen de nitrilo esterilizado con etanol al 70%. Retire la placa de la célula de la incubadora, mantenga la respiración mientras la puerta de la incubadora está abierta. Tenga cuidado para reducir al mínimo la exposición a la luz de la habitación.
- Quitar los cubreobjetos que se utilizará para el experimento de la placa de seis pozos, y señaló que el lado que estaba boca arriba en el pozo es el lado con células unidas. Secar cuidadosamente el portaobjetos a ambos lados hasta que esté casi completamente seco, mientras que el seguimiento de qué lado del cubreobjetos tiene las células. A continuación, pulse el cubreobjetos, lado celda hacia abajo, en la placa de metal untada sobre el agujero de la visión, asegurándose de que no quedan espacios de aire dentro de la capa de grasa. La grasa debe formar un sello hermético para permitir para que la carga de las células con el medio L15. Una vez que esté seguro de ello, dar la vuelta a la placa posterior.
- Pipeta medio L15 a las células chapada forzando el líquido a través de la ranura entre el cubreobjetos superior y la placa de metal. Pipetear 200 l en un momento funciona bien. La pipeta de volumen debe completar el espacio situado entre el cubreobjetos con el líquido que se extiende casi a la ranura en el otro lado.
- Pipeta de otro medio de 200 l en las células plateado, pero esta vez, mantenga la mecha en la arboleda se oponen de modo que el fluido circula de un lado a otro. Este se lava las células y elimina cualquier rastro de la media de edad. Tenga cuidado para evitar que se formen burbujas dentro del líquido durante este paso. Repita este proceso 2-3 veces usando una mecha nueva por cada lavado.
- Recuerde que el soldador que está conectado? Ahora es el tiempo que se acostumbra. Da la vuelta al plato boca abajo, una vez más, el apoyo a la placa de los bordes para que el líquido queda atrapado en el reservorio de células y no puede gotear. Sumerja la punta del soldador en el vaso valap. Esto rápidamente se derrite algunos de los valap que luego se aferran a la punta del soldador. Aplique cuidadosamente el valap fundido alrededor de los bordes del cubreobjetos inferior (que ahora está mirando hacia arriba) con la punta del soldador como un aplicador. Continúe el hundimiento y la aplicación hasta que se vaya todo el camino alrededor del perímetro cubreobjetos, sellar el cubreobjetos a la placa de metal.
- El cubreobjetos inferior tiene células que crecen en ella, y pueden tener residuos del medio de secado encima de la cara expuesta. Limpie cualquier residuo de la superficie cubreobjetos por bolas y un Kimwipe Cleaning cubreobjetos en un solo movimiento de deslizamiento muy similar a la limpieza del objetivo. Esto asegura que el cubreobjetos es el más limpio en el centro donde se pueden ver.
- Desconecte el soldador y devolver la placa de 6 pocillos a la incubadora de la observación de la contención misma y los procedimientos de esterilidad. Tome las células plateado para el laboratorio de óptica y de montaje en el objetivo como se describe en los pasos 2.4 y 2.5.
5. Llevar a cabo el experimento
- Encontrar un campo agradable de aspecto saludable células.
- Adquirir una imagen de campo oscuro del campo de visión. Alinear el microscopio de contraste de interferencia diferencial (DIC) y adquirir una imagen DIC. Asegúrese de que los tiempos de exposición son lo suficientemente largos para asegurar que la señal es adecuada.
- Ahora tenemos que obtener las imágenes fluorescentes en la otra cámara. Para obtener imágenes de DIC en el CoolSnap, se utiliza un LED de color azul unido al condensador, en sustitución de él y sacarlo como sea necesario. Mientras que el microscopio sigue alineado en la DIC, enviar la luz a la CoolSnap mediante el establecimiento de la optovar microscopio de 1,0 x y la vista al 100% binocular. Desviar la imagen del ocular de la cámara. Coloque el LED en el condensador para iluminar el campo y ver el campo de visión en RSimage y ajuste de enfoque fino si es necesario. Adquirir una imagen de la CID y guardarlo en el disco. Nótese cómo el campo de visión es diferente de la obtenida por la cámara de la cascada. Estas imágenes deberán ser registradas durante la fase de análisis después del experimento.
- Obtener una imagen de fluorescencia mediante el ajuste del cubo de filtro a la filtercube fluoresceína. Adquirir una imagen con una breve vuelta a la excitación de fluorescencia utilizando el microscopio y luego se apaga tan pronto como la adquisición se haya completado. Puesto que nos hemos centrado la muestra en la DIC, la imagen de fluorescencia se centra también. Esto ahorra el tiempo de exposición de la fluorescencia, afectando el ritmo photobleaching. Guardar la imagen de fluorescencia en el disco.
- Ahora tenemos que obtener las imágenes filtradas. Restablecer el microscopio de campo oscuro y vuelva a enviar la luz a través del puerto LSM como en 2.9.
- Se presentan en una secuencia de comandos de Gabor Filterbank como en 3.3-3.5. Hemos completado la adquisición de datos para un momento determinado.
6. Cambiar el medio para exponer las células a estaurosporina (STS), y mantener el medio a través del experimento
- Mientras que las células están todavía en el escenario y sin perturbar el campo de visión, cambiar el habitual medio L-15 de la misma que contiene una solución de 1 M STS a partir de una solución 4 mM de valores de STS en DMSO. Utilice el método de absorción descrito en los pasos 4.6 para cambiar los medios de comunicación.
- Ahora, que repita los pasos 5.2-5.6 para los tiempos posteriores. Repetimos este proceso hasta que el experimento se ha completado.
- Durante el transcurso del experimento, más medio que hay que añadir que la muestra no se secan. Esto se logra por medio de pipeta a la arboleda de la placa de la célula sin necesidad de retirar del escenario y sin perturbar el campo de visión.
7. Resultados representante
Al término del experimento, los datos recogidos se incluyen un gran número de imágenes filtradas que necesitan ser procesados para extraer los datos estructurales subcelulares. Se muestran dos ejemplos de un banco de filtro óptico que consta de 9 Gabor-como los filtros con filtro S = período de 0.95μm, la desviación estándar de Gauss sobre s = S / 2 = 0.45μm, y las orientaciones Φ = 0 ° a 160 ° = Φ en 20 ° incrementos. (Ver también [1] para más detalles).
Ejemplo 1: diatomeas marinas
En primer lugar, aplicamos nuestra orientación banco de filtros sensibles a una muestra de diatomeas marinas (Carolina Biological Supply Company), con características orientadas a que eran claramente visibles en campo oscuro (DF) imagen (Fig. 1). Las imágenes ópticamente filtrada se muestran junto a la imagen filtrada de la muestra para la comparación.
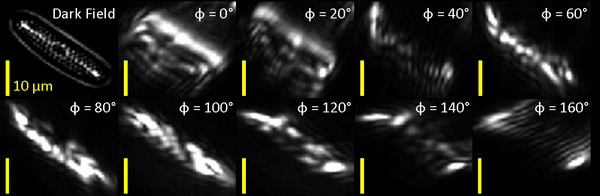
Figura 1: en campo oscuro (DF) y la imagen ópticamente filtrada de diatomeas marinas. Vamos a analizar la diatomea en la parte inferior derecha de la imagen (flecha blanca en el panel más a la izquierda).
El conjunto de nueve Gabor filtrado imágenes de la diatomea fueron procesados píxel por píxel para la orientación a objetos y la redondez. Procesamiento consistió en (1) resumir las respuestas medidas de los nueve Gabor filtrado imágenes en cada píxel para determinar la magnitud global de la respuesta de la señal por lo tanto la codificación importancia de respuesta, y (2) y detectar la orientación del filtro Gabor, Φ, en el que la respuesta es al máximo y tomando la relación de esta respuesta máxima a la respuesta promedio para todos los ángulos así que codifica la medida en que los objetos en cada píxel tiene una orientación preferente. El grado de orientación está estrechamente relacionada con la relación de aspecto geométrico de la partícula. En la figura. 2B, threspuesta por correo general del pixel para el banco de filtros (parámetro 1) y el grado de orientación o relación de aspecto (parámetro 2) están codificados en la saturación del color y la tonalidad, respectivamente. Una relación de aspecto cerca de 1 (azul) está presente en áreas en las que no hay ningún ángulo respuesta preferida, mientras que los mayores valores (rojo) indican las áreas en las que una respuesta de mayor ángulo preferido es el presente. Orientación de las partículas sub-estructura está codificada en una parcela carcaj (Fig. 2C), donde cada línea de cerca de acuerdo con la orientación a objetos subyacentes locales visibles en campo oscuro sin filtro (Fig. 2A).

Figura 2: A: imagen de campo oscuro de diatomeas. B: imagen de Orientación a Objetos. Escala de colores indica el grado de orientación (relación de aspecto) mientras que el brillo codifica significado de la respuesta total del filtro Gabor. C: La orientación de los objetos con la intensidad de la respuesta ≥ 10% del máximo. Segmento de la línea indica eje longitudinal de la estructura correspondiente.
Ejemplo 2: Las células apoptóticas
Aquí se muestran las imágenes filtradas de las células endoteliales de bovino tratado con estaurosporina (STS) que fueron procesados en la misma forma que la diatomea. Fig. 3 muestra un sin filtro de campo oscuro (DF) la imagen de las células a lo largo de las nueve imágenes filtradas en el tiempo T =- 180 min. antes del tratamiento STS.
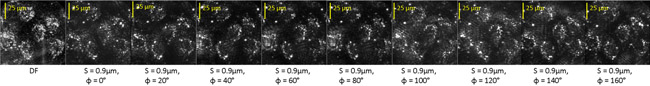
Figura 3: campo oscuro (DF) y las imágenes ópticamente filtrada de un campo que contiene varias células endoteliales de vida.
Las imágenes filtradas fueron adquiridas cada 20 minutos durante un período de tres horas después del tratamiento STS. Fig. 4a muestra un mapa de relación de aspecto de las células en función del tiempo. En este caso, el tono de color representa el grado de orientación (orientación hacia la etiqueta) como por el tono del color de la figura. 2b arriba. Sin embargo, el brillo de la relación de aspecto no fue ponderado por el filtro de respuesta promedio. Al registrarse en nuestra relación de aspecto de los mapas con imágenes de fluorescencia de las mitocondrias etiqueta en estas células (Fig. 4b), se determinó que la caída de la relación de aspecto medida se limita a las regiones que contienen mitocondrias celulares, y fue concomitante con la fragmentación mitocondrial, que se puede observar directamente en las imágenes de fluorescencia de las mismas células. Fig. 5 muestra gráficas de tiempo que representa el cambio en la relación de aspecto en función del tiempo en las células que sufren apoptosis. Dentro de cada célula, hay una caída en la relación de aspecto a T = 60-100 min en las regiones que se registra en las mitocondrias fluorescentes, pero no en las regiones que se registra en las áreas de fluorescencia de fondo oscuro.
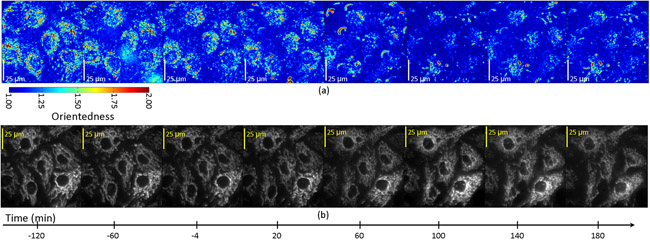
Figura 4: Relación de aspecto (a) y fluorescencia (b) las imágenes de las células endoteliales tratadas con el inductor de la apoptosis, estaurosporina.
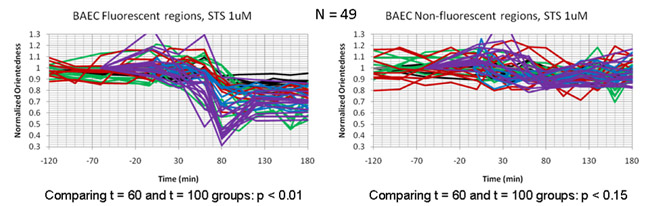
Figura 5: Tiempo parcelas comparar la disminución de la proporción de partículas de aspecto (orientación hacia) en las células endoteliales tratadas con estaurosporina. Las huellas de las parcelas individuales representan el tiempo dentro de las células individuales. La caída de la orientación hacia se limita a las regiones de las células que se registra en las mitocondrias fluorescentes (panel izquierdo) y está ausente de las regiones de fondo restante de fluorescencia (panel derecho).
Ahora que hemos determinado que la caída de la relación de aspecto corresponde a la fragmentación de las mitocondrias, que puede inducir la apoptosis en estas células, medida a la fragmentación usando nuestro método de dispersión óptica sin tener que identificar a las células, y estudiar el efecto de diferentes condiciones genéticas y experimental en este dinámica.
Discusión
El método descrito anteriormente mapas de rendimientos morfométricas del objeto que puede codificar el tamaño de partícula o de orientación, por ejemplo. Esta información estructural se puede utilizar de varias maneras:
- Puede ser utilizado como una pantalla inicial para identificar las regiones de tejido o células que se alteran durante un tratamiento específico y luego seguir analizando estas regiones con particularidades técnicas moleculares y bioquímicos.
- Puede ser usado en conjunció...
Divulgaciones
Agradecimientos
El dispositivo de micro-espejos-en esta investigación fue financiada por beca de la Fundación Whitaker RG-02 a 0,682 a N. Boustany. Trabajo en curso es financiado por la subvención NSF-DBI-0852857 de N. Boustany. Pasternack RM fue parcialmente financiado por una beca de Rutgers Presidencial de Posgrado. También nos gustaría agradecer al Dr. E. White para que las células iBMK utilizados en nuestros estudios y Metaxas Dr. DN útil para el debate sobre las estrategias de filtrado óptico.
Materiales
| Name | Company | Catalog Number | Comments |
| DMEM | Invitrogen | Low glucose DMEM | |
| Liebowitz L15 medium | Invitrogen | Without phenol red | |
| L-glutamine | Invitrogen | ||
| Mitotracker Green | Invitrogen | ||
| Bovine Brain Extract | Clonetics | ||
| Fetal Bovine Serum | Gemini Bio Products | ||
| Heparin | Sigma-Aldrich | ||
| Staurosporine | Sigma-Aldrich | ||
| Dymethylsulfoxide | Sigma-Aldrich | ||
| Inverted microscope | Carl Zeiss, Inc. | Axiovert 200M | |
| DMD | Texas Instruments | TI 0.7 XGA DMD 1100 | |
| CCD | Roper Scientific | Cascase 512B | High (16 bit) dynamic range CCD |
| CCD | Roper Scientific | Coolsnap cf |
Referencias
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., White, E., Boustany, N. N. Measurement of Subcellular Texture by Optical Gabor-Like Filtering with a Digitial Micromirror Device. Optics Letters. 33 (19), 2209-2211 (2008).
- Pasternack, R. M., Qian, Z., Zheng, J. -. Y., Metaxas, D. N., Boustany, N. N. Highly sensitive size discrimination of submicron objects using optical Fourier filtering based on two-dimensional Gabor filters. Optics Express. 17 (14), 12001-12012 (2009).
- Zheng, J. -. Y., Pasternack, R. M., Boustany, N. N. Optical scatter imaging with a digital micromirror device. Optics Express. 17 (22), 20401-20414 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados