Method Article
Primer Libre Selección aptámero utilizar una biblioteca de ADN al azar
En este artículo
Resumen
SELEX protocolos comprenden múltiples rondas de selección, cada uno de los cuales requieren la regeneración de ligandos unidos, que a su vez requieren secuencias fijas de imprimación que flanquean las regiones de la biblioteca al azar. Estas secuencias de los cebadores fija puede interferir con el proceso de selección (falsos positivos y negativos). Aquí presentamos una cartilla sin protocolo.
Resumen
Los aptámeros son oligonucleótidos altamente estructurado (ADN o ARN) que se puede unir a los objetivos con afinidades similares a los anticuerpos 1. Se identifican a través de un proceso de selección in vitro denominado evolución sistemática de ligandos por enriquecimiento exponencial (SELEX) para reconocer una gran variedad de objetivos, a partir de moléculas pequeñas de proteínas y otras macromoléculas 2-4. Aptámeros tienen propiedades que son muy adecuadas para el diagnóstico in vivo y / o aplicaciones terapéuticas: Además de una buena especificidad y afinidad, que son fáciles de sintetizar, sobrevivir las condiciones del proceso más riguroso, son poco inmunogénicas, y su tamaño relativamente pequeño puede dar lugar a la penetración fácil de tejidos.
Aptámeros que se identifican con el proceso SELEX estándar por lo general comprenden ~ 80 nucleótidos (nt), ya que normalmente se selecciona a partir de bibliotecas de ácidos nucleicos con ~ 40 nt de longitud regiones más al azar sitios fijos de imprimación de ~ 20 nt en cada lado. El primer secuencias fijas por lo tanto puede comprender casi el ~ 50% de las secuencias de la biblioteca, y por lo tanto, positiva o negativamente compromiso identificación de los aptámeros en el proceso de selección de tres, aunque los enfoques bioinformáticos sugieren que las secuencias fijas no contribuyen significativamente a la estructura aptamer después de la selección 5 . Para hacer frente a estos problemas potenciales, primer secuencias han sido bloqueados por oligonucleótidos complementarios o cambiar a mitad de camino diferentes secuencias durante las rondas de SELEX 6, o que han sido recortadas a 6-9 nt 7, 8. Wen y Gray 9 diseñó una cartilla sin método SELEX genómica, en la que el primer secuencias fueron completamente eliminadas de la biblioteca antes de la selección y se regenera entonces para permitir la amplificación de los fragmentos del genoma seleccionado. Sin embargo, para emplear la técnica, una biblioteca genómica única tiene que ser construido, el cual posee una limitada diversidad, y la regeneración después de varias rondas de selección se basa en un paso reamplificación lineal. Por otra parte, los esfuerzos para evitar los problemas causados por primer secuencias fijas con el particionado de alta eficiencia se encuentran con problemas en cuanto a amplificación por PCR 10.
Hemos desarrollado un manual gratuito (PF) el método de selección que simplifica los procedimientos de SELEX y efectivamente elimina la interferencia de imprimación problemas 11, 12. Los protocolos de trabajo de una manera directa. La región central al azar de la biblioteca se purifica sin extrañas secuencias de acompañamiento y se une a un objetivo adecuado (por ejemplo, para una proteína purificada o de mezclas complejas, tales como líneas de células). A continuación, las secuencias de atado se obtienen, se reunió con las secuencias de acompañamiento, y re-amplificado para generar seleccionado sub-bibliotecas. Como ejemplo, aquí hemos seleccionado aptámeros para S100B, un marcador de proteína para el melanoma. Los ensayos de unión mostró Kd s en los 10 -7 a 10 -8 M rango después de unas cuantas rondas de selección, y se demuestra que los aptámeros funcionar eficazmente en un formato sándwich vinculante.
Protocolo
1. Breve descripción del Primer protocolos libres de Selección
Una biblioteca de ADN de doble cadena se ha construido utilizando PCR con los oligonucleótidos correspondientes (Figura 1 y 2, el paso a), que contienen un dominio central aleatoria de 30 nt, flanqueado por dos regiones de imprimación. Dos protocolos ligeramente diferente "primer-free" (PF) se han desarrollado. En la biblioteca de ADN de doble cadena, la región 5 'no contiene una endonucleasa "mellar" el sitio para la endonucleasa Nt.BstNBI, esta enzima reconoce dsDNA pero se unirá sólo una hebra del ADN sustrato. La región 3 'de la biblioteca de ADN de doble cadena contiene otra "mellar" el sitio, para la endonucleasa Nt.BbvCI que también reconoce dsDNA pero rompe una sola hebra, dejando un CC en el extremo 3' (PF 1), así como un BspMI endonucleasa sitio de restricción, que divide las dos vertientes sin dejar adicionales 3 'nucleótidos (PF 2). Por lo general, utilizan el protocolo inicialmente PF 1 (porque es más fácil de fragmentos separados producidos durante el proceso de selección, véase más adelante), y luego emplear la PF 2 del protocolo para las siguientes rondas de selección.
El 32 de nt 5'-pN30-CC-3 'fragmento (designado 32 +-fragmento) y el 30 de nt 5'-pN30-3' fragmento (designado 30 +-fragmento) se generaron, respectivamente, por Nt.BbvCI / Nt . BstNBI o BspMI / Nt.BstNBI división de la biblioteca de ADN de doble cadena, y la purificación de gel. Los 32 +-fragmento contiene el dominio de 30 nt al azar, con una secuencia de CC flanqueantes en el extremo 3 '(PF 1). Los 30 +-fragmento (PF 2) consiste sólo en la secuencia de 30 nt de dominio al azar. El "auto-puente" 66 - fragmento (que contiene el azar N30 región con el 5 'y 3' secuencias de acompañamiento) se obtuvo mediante la purificación en gel (Nt.BbvCI o Nt.BstNBI corta sólo la parte superior "+" cadena). Esta auto-puente se obtiene directamente de la PF un protocolo, o se puede generar y aislados después de cortar el ADN de la biblioteca con sólo NtBbv.CI o Nt.BstNBI de la PF 2 del protocolo (Figura 1 y 2, el paso b). Los 32 + - 32 + o-fragmentos se incubaron con las proteínas purificadas o las células de melanoma cultivadas para permitir que los fragmentos se unen a las proteínas o las células, a continuación, los fragmentos no consolidados fueron arrastradas (Figuras 1 y 2, el paso c). Los fragmentos seleccionados obligados o se utiliza para la regeneración de las regiones de imprimación.
En la reacción de hibridación / ligadura, la auto-puente fue producida y purificada en el momento mismo que el 32 + - 30 y + fragmento de purificación; extremo 5 'cartilla fue igual que la construcción de la biblioteca y el extremo 3' primers fueron sustituidos a juego con las cartillas que también contenía una adicional B6 promotor de la transcripción en el extremo 3 '(figura 1 y 2, el paso d). Los productos de la reacción de hibridación / ligadura se utilizaron para la transcripción de ARN in vitro (Figura 1 y 2, el paso e). Después de la transcripción del ARN, el ADN de liga (incluyendo el fragmento de ADN seleccionadas auto-puente) fueron digeridos por DNasa I para eliminar interferencia secuencias de fondo. La transcripción reversa (RT)-PCR datos han demostrado que el primer-regenerado productos se reamplified eficiente, lo que constituye una "ronda" de la selección (Figura 1 y 2, el paso f). La amplificación de fondo se muestra en número de ciclos de alta en la PCR-RT de control no puede ser extirpado completamente por un adicional de digestión con DNasa I, y no es detectable después de los números más bajos del ciclo, que se emplean habitualmente.
Después de siete rondas de selección, aptámeros se caracteriza por las propiedades de unión. Kd es donde están todas en el rango de M 10 -7 10 -8 (Figura 3). En los ensayos de unión, varios pares de aptámeros mostró vinculante aditivo, lo que indica que se dirigen sitios distintos en la proteína S100B. Por lo tanto, a prueba de pares de aptámeros en "sandwich", ensayos de unión, tanto en microarrays de vidrio (diapositivas CodeLink) utilizando fluorescencia etiquetados aptámeros en segundo lugar, y en los nanocables de oro modificado con aptámeros segundo junto a 50 nm nanopartículas de oro (AuNPs; Figura 3). En ambos casos, la especificidad de unión es alta: en microarrays CodeLink, no vinculante sándwich se observó con aptámeros que no mostró aditivo vinculante en las determinaciones de Kd. Con nanocables derivado se observó prácticamente ninguna unión a las proteínas que no son objeto, y los complejos individuales sándwich se puede observar a través de la AuNPs aptamer acoplado (Figura 3).
2. Materiales
2.1. Generación de la Biblioteca de ADN PF y reamplificación de fragmentos enlazados
Los detalles se describen en el Pan y Clawson, 2009; Pan et al, 2008..2.2. Purificada selección a base de proteínas
- 20 mM Tris-HCl, pH 7,4
- 32 +, 30 + y fragmentos-fragmentos
- Selección de Buffer (2,5 mM CaCl2, MgCl2 5 mM en 1X tampón fosfato salino, pH 7,4, Gibco)
- Ni-NTA agarosa (Qiagen)
- Polipropileno columna (Qiagen)
- Tampón de unión (50 mM Na 2 HPO 4 -NaH 2 PO 4, pH 7.2, 150 mM NaCl)
- Expresa, la proteína purificada S100B en tampón de unión (5 mg / l)
- Fenol: CHCl3: IAA (pH 7,9, Ambion)
- 3 M NaAc, pH 5,2
- 100%, y el 70% de etanol
2.3. TOPO Clonación y análisis de secuenciación del ADN de doble cadena seleccionada Bibliotecas
- 7 ª ronda seleccionado productos de la PCR (rondas adicionales se pueden realizar para continuar la optimización de la aptamer vinculante)
- pCR2.1 TOPO-vector (Invitrogen)
- DH5 células componentes (Invitrogen)
- Plásmido Mini Kit (Qiagen)
- M13 cebador
- Informax de Vector NTI (Invitrogen)
2.4. Características de la unión de los aptámeros
- Aptámeros y el control de oligos (N 30 CC y 30 N, IDT)
- Polinucleótido quinasa T4 (New England Biolabs)
- γ-32 P-ATP (3000 Ci / mmol, 10 Ci / ml)
- S100B purificada en tampón de unión (5 mg / l)
2.5. Sandwich ensayos de unión con la proteína S100B purificada y aptámeros seleccionada
- El 1 º de aptámero se acopló a cualquiera de a.) CodeLink diapositivas de microarrays o b.) nanocables de oro.
- Purificado la proteína S100B, diluida ya sea a 0.125 M en 70 l de a) o 1 M en 50 l de b).
- El 2 aptamer fue etiquetado, ya sea con a.) AlexaFluor 546 para las diapositivas serie CodeLink, o b.), junto a 50 nm AuNPs para los estudios con el derivado nanocables de oro.
3. Métodos
3.1. Generación de PF ADN Biblioteca
Métodos y protocolos detallados para la selección de aptamer se puede encontrar en el Pan y Clawson, 2009; Pan et al, 2008..
3.2. Aptámero Selección utilizando proteína purificada S100B
Humanos de enlace de calcio S100 proteína B (S100B. Gene ID # 6285) fue utilizado como un objetivo. El 6-Su etiquetados proteína S100B (98 aminoácidos de longitud) se expresó y se purifica mediante el sistema de QIAexpressionist. (QIAGEN). Si se desea o se indica, el 6-Su etiqueta se puede eliminar por vía enzimática. Durante las rondas de selección, una muestra de l de cada paso se salvó por recuento de centelleo líquido para determinar la eficacia vinculante general.
3.2.1. Preparación de ADN Biblioteca PF-fragmentos
- Vuelva a suspender el + 32 y + 30 fragmentos en 40 l de 20 mM Tris-HCl, pH 7,4.
- Calentar 3 minutos a 85 ° C, y enfriar a 37 ° C durante 3 minutos en una incubadora de 37 ° C.
- Añadir 760 l de buffer de selección (2.5 mM CaCl2, MgCl2 5 mM en 1X tampón fosfato salino, pH 7,4, GIBCO) y se incuba durante 3 min a 37 ° C, a continuación, mantener a temperatura ambiente (RT) durante 10 minutos.
- Pasar a través de una columna de contenido de Ni-NTA agarosa bolas (QIAGEN) pre-lavado con buffer de selección, a continuación, mantener a temperatura ambiente hasta su uso.
3.2.2. Preparación de Ni-NTA agarosa-bolas Límite S100B a temperatura ambiente
- De girar 400 l de Ni-NTA agarosa bolas durante 3 segundos y descartar el sobrenadante.
- Lavar los granos con 400 l de tampón de unión (50 mM Na 2 HPO 4 NaH 2 PO 4, pH 7.2, 150 mM NaCl) pipeteando con suavidad 5 veces, luego de girar y descartar el sobrenadante.
- Repita el paso 2 dos veces.
- Añadir 400 l de agua purificada S100B (5 mg / l, suspendido en tampón de unión) y suavemente pipeta de 5 veces cada 3 minutos para un total de 15 min.
- Centrifugar y descartar el sobrenadante.
- Lavar el grano con destino S100B con 400 l de tampón de unión pipeteando con suavidad tres veces, luego de girar y descartar el sobrenadante.
- Repita el paso 6 dos veces.
3.2.3. La selección de S100B con destino aptámeros
- Transferir el + 32 - y 30 +-fragmentos de la 3.2.1 a la S100B cordón de ruedas.
- Incubar durante 15 minutos y mezclar suavemente cada 3 minutos pipeteando con suavidad.
- Lavar el complejo S100B-ADN con 800 l de tampón de unión pipeteando con suavidad, después de girar y descartar el sobrenadante.
- Repita el paso 3 dos veces.
3.2.4. Recuperación de los aptámeros S100B-seleccionadas
- Añadir 200 l 20 mM Tris-HCl (pH 7,4), el calor 3 min a 85 ° C, 1 min y vórtice de girar, a continuación, transferir el sobrenadante a un tubo nuevo.
- Repita el paso 1 una vez, y combinar los sobrenadantes.
- Purificar el ADN de los fragmentos seleccionados en los pasos por 9.12 de publicaciones anteriores (11, 12).
3.2.5. TOPO Clonación y análisis de secuenciación para la identificación de las secuencias del aptámero Consenso
- Clon de la ronda 10 de determinados productos PCR iNo pCR2.1 TOPO-vector (de Invitrogen). La clonación adicional se puede hacer como más selecciones se llevan a cabo.
- Secuencia de 40 a 50 colonias con un solo cebador M13 (se utiliza el Fondo Genética Molecular de núcleo en el Centro Médico Hershey).
- Alinear las secuencias seleccionadas usando vectores Informax NTI (Invitrogen).
- Determinar las secuencias de consenso basado en las alineaciones.
- Aptámeros consenso fueron adquiridos luego de las tecnologías de ADN integrado.
3.3. Sandwich ensayos de unión con los pares de aptámeros seleccionada
3.3.1. Formato de microarrays con la etiqueta fluorescente 2 ª aptámeros.
- Un consenso aptámeros st fueron sintetizados con un resto 5'-amineC6. Que se suspendieron en tampón de impresión a una concentración final de 15 micras y manchas en CodeLink diapositivas activado (GE Healthcare / Amersham Biosciences) utilizando un Descubrimientos Apogent MicroGrid Arrayer en las instalaciones de ADN Microarray PSU, University Park), siguiendo los protocolos recomendados CodeLink. Cada diapositiva se imprime con 12 matrices.
- La hibridación se llevó a cabo en un 2 x 8 cassettes formato hibridación de microarrays (matriz que, TeleChem Internacional). El cassette de microarrays se selló con nucleasa libre de papel sellado adhesivo (AlumaSeal II, Investigación de Productos Internacional) para evitar la evaporación.
- Proteína S100B se diluyó en PBS a la concentración adecuada en el 70 l, y se aplica a los pozos de la cinta de microarrays. La unión a los aptámeros impreso en la diapositiva CodeLink fue durante 1 hora a temperatura ambiente. Los pozos de la cinta fueron luego individualmente lavado 3X con PBS + 5 mM de MgCl 2 (PBSM), y el exceso de tampón de lavado fue borrado. Los aptámeros marcados con fluorescencia se diluyeron en PBSM a 0.125 M en 70 l, y luego se calienta a 85 ° C durante 3 minutos seguido de 3 minutos en hielo. Aptámeros se aplicaron a los pocillos de la bandeja de microarrays, y se incubó durante una hora a temperatura ambiente. Los pozos fueron lavados 3 veces de forma individual una vez más con PBSM. La cinta se ha llevado además, anuncio la totalidad de la preparación se lavó en PBSM. La corredera era entonces completamente seca por centrifugación, y escaneadas usando un escaneo Packard Biosciences ScanArray 4000XL (Perkin Elmer).
3.3.2. Derivatizado formato de nanocables con 2 º aptámeros, junto a 50 nm AuNPs
Oro del NWS (~ 5 M de largo, 320 nm de diámetro) fueron sintetizados por electrodeposición galvanostático en las membranas de alúmina porosa siguiendo los protocolos previamente publicados (13, 14). Tras la disolución de la membrana porosa, los nanocables se resuspendieron en 1 ml de etanol.
Como se ha señalado, el ADN fue adquirido de las tecnologías de ADN integrado. Las nanopartículas de oro (50 nm) fueron adquiridos de Ted Pella. ADN tiolada se escindió con DTT 100 mM de pH 0,1 M de fosfato de sodio 8,3 durante una hora y luego se purifica en un Centri spin-10 de la columna.
Aptámero apego a NWS oro y PN
Una alícuota de 50 L de nanocables de Au se colocó en un tubo de centrífuga de 0,5 ml que no se pegue y se enjuagó en tampón fosfato 10 mM, NaCl 300 mM, pH 7,4. ADN tiolada en el extremo 5 '(con un espaciador T 10 en el extremo 5') se añadió a una concentración final de 0,4 M a los cables. La muestra se agitó durante 30 minutos y después se aclara por centrifugación (8100 g) tres veces con el tampón fosfato 10 mM, NaCl 300 mM, pH 7,4 y tres veces con tampón fosfato 50 mM, MgCl2 5 mM, pH 7,2. Los cables recubiertos de ADN se resuspendió en buffer de 100 l para diluir a la mitad.
Derivado de ADN AuNPs fueron preparados por la adición de 50 l 100 mM 2 de aptámero (con un espaciador de 10 T en el extremo 5 ') a un AuNPs nm de 50 ml y se calienta a 37 ° C durante una hora. Después de calentar, 10 mM tampón fosfato de sodio, NaCl 1 M, pH 7,4, se añadió (25 l dos veces, seguido de 100 l, l 150 y 128 l) a intervalos de 0,5 horas. Los conjugados fueron dejados en un bloque de 37 º C de calor durante la noche antes de su uso. Las muestras fueron lavadas 3 veces con tampón fosfato 50 mM, MgCl2 5 mM, pH 7,2. Conjugados se resuspendieron en tampón de 120 mL.
Sandwich de hibridación de NWS
Cables recubiertos de ADN, una l diluido, se han añadido al buffer en los tubos de PCR. Proteína S100B o HTRA1 proteína de control (1 mg / l) se añadió a una concentración final de 1 mM en tampón 50 l (~ 10-12 moléculas de proteína añadida por nanómetro cuadrado de superficie de los nanocables Au). Las muestras se agitaron durante ~ 2 hrs. Los cables fueron lavadas por centrifugación (8100 g) tres veces con tampón fosfato 50 mM, MgCl2 5 mM, pH 7,2 y se resuspendió en 20 de cada AUNP l / ADN conjugados. Los cables se agitaron en los conjugados durante 2 horas, y luego se lavaron 5 veces por centrifugación (1300 g) con tampón fosfato 50 mM, MgCl2 5 mM, pH7,2 para eliminar el exceso de las nanopartículas. Las muestras se resuspendieron en 20 l de amortiguación y se secan en las obleas de silicio recubierto de Au FE-SEM análisis.
FE-SEM imágenes de los nanocables se obtuvieron utilizando un Leo 1.530 campo de emisión microscopio electrónico de barrido con un Schottky de campo de fuentes de emisión de electrones a una tensión de 5,00 kV de funcionamiento.
4. Resultados representante
Figura 1. Protocolos del Primer 5'-Libre (PF 1) y Free Primer-(PF 2).
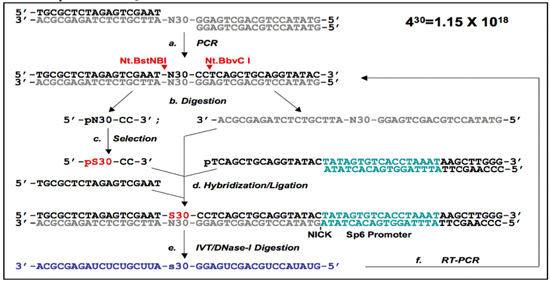
(A) El PF 1 ADN aptamer protocolo de selección. Los oligonucleótidos de la biblioteca son recocidos en conjunto, y sometido a amplificación por PCR para producir una biblioteca de ADN de doble cadena (a). El extremo 5 'cartilla libre (PF 1) Región de azar de las bibliotecas de ADN de cadena sencilla PF se prepara Nt.BstNBI / Nt.BbvCI digestiones (sitios de corte están marcados con flechas rojas) y las purificaciones de gel, y el auto -puente (negro flecha) está aislado por la purificación del gel (b). Después de la selección (c), las secuencias seleccionadas (designado PS30-CC) se hibridan con sus correspondientes oligómeros y el puente de la auto-y se liga para volver a generar las regiones de imprimación que quitó antes. En esta etapa, se introducen los primers que contienen un promotor SP6 en el extremo 3 '(d). Esto se usa para transcribir el ARN que contienen las regiones seleccionadas (e). Finalmente, el ARN es entonces exclusivamente re-amplificado (la plantilla de ADN se digiere) mediante RT-PCR (f), y el seleccionado sub-biblioteca está listo para la próxima ronda de selección.
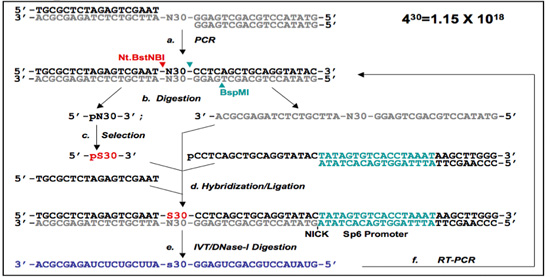
(B) El PF 2 ADN aptamer protocolo de selección. Los oligonucleótidos de la biblioteca son recocidos juntos y sometido a amplificación por PCR para producir una biblioteca de ADN de doble cadena (a). La región de imprimación libre (PF 2) al azar de la biblioteca de ADN de cadena sencilla PF se prepara Nt.BstNBI / BspMI digestiones (sitios de corte se indican con flechas) y purificación en gel. La auto-puente está aislado tanto de la PF un protocolo y / o por una sola digestión de la biblioteca a partir de Nt.BstNBI (b), junto con la purificación en gel. Después de la selección (c), las secuencias seleccionadas (designado PS30) se hibridan con sus correspondientes oligómeros y el puente mismo, y se liga a regenerar las regiones de imprimación que quitó antes. Al igual que en PF 1, primers se introducen que contienen un promotor SP6 en el extremo 3 '(d). Esto se usa para transcribir el ARN que contienen las regiones seleccionadas (e). El ARN es entonces exclusivamente reamplified mediante RT-PCR (f), y el seleccionado sub-biblioteca está lista para la siguiente ronda de la selección.
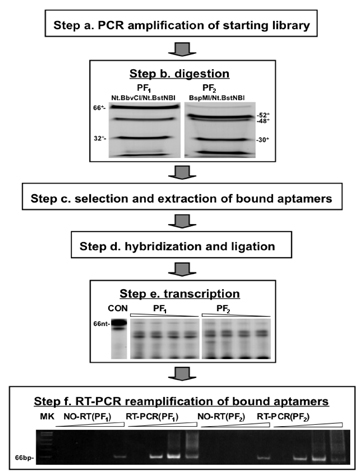
Figura 2. Representación esquemática de los resultados experimentales y / reducción a la práctica del protocolo de selección de PF. Pasos designados corresponden a los que se muestra en la Figura 1. Después de la digestión de la biblioteca PF construido por PCR (paso a) con endonucleasas de restricción (como se indica), los productos digeridos fueron separados por PAGE en un gel al 10% en condiciones desnaturalizantes. Los fragmentos correspondientes obtenidos con el PF 1 y PF 2 protocolos de selección se muestran (paso b). Después de la selección (paso c), los aptámeros obligado se hibridan con sus correspondientes oligómeros y el puente mismo, y se liga a regenerar las regiones de imprimación que quitó antes. En el extremo 3 ', los cebos se introducen que contienen un promotor SP6 en el extremo 3' (paso d). 32 P-etiquetados ARN se transcribe el uso de productos ligados de 1, 0,5, 0,25, 0,125 l de 5 l reacciones, y se separados por PAGE en geles de 6% en condiciones desnaturalizantes (etapa e). Las transcripciones fueron tratados con DNasa para eliminar las regiones seleccionadas al azar de ADN, transcripción inversa en ADNc, y luego reamplified de 7, 14, 21 ó 28 ciclos de PCR. Los productos fueron separados por PAGE en geles de 8% en condiciones no desnaturalizantes. El fragmento de 66 nt representa el producto de larga duración que contiene el seleccionado PS30-CC y secuencias PS30 como re-incorporado dentro de las secuencias 5 'y 3' de acompañamiento. La migración de los marcadores PhiX174/HinfI se muestra a la izquierda (paso f). Los controles incluyen la omisión de la transcripción reversa (RT) paso. Una pequeña cantidad de producto en las muestras se pudo ver en el número de ciclos de alta, lo que probablemente pueda ser eliminado por completo con una segunda (o prolongada) paso de digestión con DNasa.
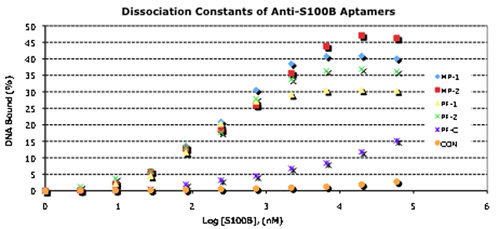
Figura 3. Características de unión de aptámeros seleccionada y su uso emparejados en un formato de Sandwich.
A. dependiente de la concentración P-32 aptámero vinculante para la determinación de Kd. Aptámeros fueron 5'-extremo marcado con g-32 P-ATP (3000Ci /mmol, ~ 10 mCi) y T4 polinucleótido quinasa (New England Biolabs), y la unión se determinó como se describe.
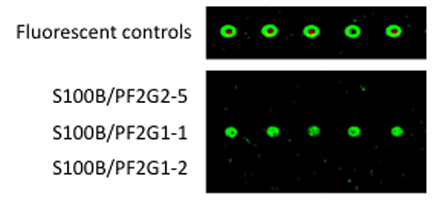
B. 5'-amino-derivado aptámeros 1 º se acoplaron a CodeLink microarrays. Purificado la proteína S100B estaba destinada a la aptamer 1 º, las diapositivas se lavaron a fondo, y AlexaFluor546 marcado 2 de aptamer estaba destinado a la aptamer 1 º: S100B complejos. Después el lavado minucioso, fluorescencia se cuantificó con un escáner ScanArray.

C. 5'-tiol derivado aptámeros 1 º se acoplaron a los nanocables Au utilizando la química tiol estándar. 2 º aptámeros se acoplaron a 50 nm AuNPs de la misma manera. Purificado la proteína S100B (izquierda) o la proteína purificada de control HTRA1 (derecha) se fijó luego la primera a los nanocables derivado. Después de un lavado minucioso, 2 de aptamer-50 AuNPs nm estaban obligados posteriormente a la 1 ª-aptamer nanocables: S100B complejos. Después el lavado minucioso, complejos vinculados sándwich se visualizaron mediante emisión de campo microscopía electrónica de barrido. Las barras de escala = 1 micra.
Divulgaciones
Agradecimientos
Damos las gracias a Craig Pablo en el Fondo para el núcleo de microarrays por su ayuda. Este trabajo fue apoyado por el NIH / NCI subvención # CA118591 del Programa de IMAT. SLD también reconoce un Pennsylvania Space Grant Consortium for beca de apoyo financiero. Esta publicación fue apoyada también por la Universidad Estatal de Pennsylvania Materiales del Instituto de Investigación Nano Fabricación de redes y la Fundación Nacional de Ciencias Acuerdo de Cooperación No. 0335765, la National Nanotechnology Infraestructura de red, con la Universidad de Cornell.
Materiales
| Name | Company | Catalog Number | Comments |
| Tris-HCl | |||
| HotMaster TAQ DNA Polymerase | 5 PRIME | ||
| dNTP Mix | Sigma-Aldrich | ||
| Acrylamide | Fisher Scientific | ||
| UltraPure 10X TBE Buffer | Invitrogen | ||
| Ammonium Persulfate | Bio-Rad | ||
| TEMED | Fisher Scientific | ||
| PhiX174 DNA/Hinf I DNA Marker | Promega Corp. | ||
| Ethidium Bromide | |||
| α-32P-dCTP | |||
| Phenol:ChCl3:IAA | Ambion | ||
| NaCl | |||
| Ethanol | |||
| Restriction Enzymes | New England Biolabs | ||
| Urea | Fisher Scientific | ||
| Photographic Film | ECE Scientific | ||
| PBS | GIBCO, by Life Technologies | ||
| CaCl2 | GIBCO, by Life Technologies | ||
| MgCl2 | GIBCO, by Life Technologies | ||
| Ni-NTA Agarose Beads | Qiagen | ||
| Polypropylene Column | Qiagen | ||
| NaHPO4 | |||
| NaAc | |||
| Detroit-551 cells | |||
| SK-MEL-31 cells | |||
| MEM | |||
| TrypLE Express | GIBCO, by Life Technologies | ||
| T4 DNA Ligase | New England Biolabs | ||
| Dimethyl Sulfoxide | Promega Corp. | ||
| Sp6 RNA Polymerase | Promega Corp. | ||
| RNase-Free DNase I | Promega Corp. | ||
| RNase Inhibitor | Promega Corp. | ||
| SensiScript Reverse Transcriptase | Qiagen | ||
| pCR-2.1-TOPO Vector | Invitrogen | ||
| DH5α Compotent Cells | Invitrogen | ||
| Plasmid Mini Kit | Qiagen | ||
| Vector NTI | Invitrogen | ||
| T4 Polynucleotide Kinase | New England Biolabs | ||
| γ-32P-ATP |
Referencias
- Kulbachinskiy, A. V. Methods for selection of aptamers to protein targets. Biochemistry (Moscow). 72, 1505-1518 (2006).
- Mairal, T. Aptamers: molecular tools for analytical applications. Anal Bioanal Chem. 390, 989-1007 (2008).
- Stoltenburg, R., Reinemann, C., Strehlitz, B. SELEX -- A (r)evolutionary method to generate high-affinity nucleic acid ligands. Biomolec Engineering. 24, 381-403 (2007).
- Que-Gewirth, N. S., Sullenger, B. A. Gene therapy progress and prospects: RNA aptamers. Gene Ther. 14, 283-2891 (2007).
- Cowperthwaite, M. C., Ellington, A. D. Bioinformatic analysis of the contribution of primer sequences to aptamer structures. J Mol Evol. 67, 95-102 (2008).
- Shtatland, T., Gill, S. C., Javornik, B. E. Interactions of Escherichia coli RNA with bacteriophage MS2 coat protein: genomic SELEX. Nucleic Acids Res. 28, E93-E93 (2000).
- Jarosch, F., Buchner, K., Klussmann, S. In vitro selection using a dual RNA library that allows primerless selection. Nucleic Acids Res. 34, E86-E86 (2006).
- Vater, A., Jarosch, F., Buchner, K., Klussmann, S. Short bioactive Spiegelmers to migraine-associated calcitonin gene-related peptide rapidly identified by a novel approach: tailored-SELEX. Nucleic Acids Res. 31, E130-E130 (2003).
- Wen, J. D., Gray, D. M. Selection of genomic sequences that bind tightly to Ff gene 5 protein: primer-free genomic SELEX. Nucleic Acids Res. 32, E182-E182 (2004).
- Musheev, M. U., Krylov, S. N. Selection of aptamers by systematic evolution of ligands by exponential enrichment: addressing the polymerase chain reaction issue. Anal Chim Acta. 564, 91-916 (2006).
- Pan, W. H., Clawson, G. A. Primer-free aptamer selection using a random DNA library. Methods Mol Biol. , (2009).
- Pan, W., Xin, P., Clawson, G. A. Minimal-Primer and Primer-Free SELEX Protocols for Selection of Aptamers from Random DNA Libraries. BioTechniques. 44, 351-360 (2008).
- Nicewarner-Pena, S., Freeman, G., Reiss, B. Submicrometer metallic barcodes. Science. 294, 137-141 (2001).
- JC, H. u. l. t. e. e. n., CR, M. a. r. t. i. n. A general template-based method for the preparation of nanomaterials. J Materials Chem. 7, 1075-1087 (1997).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados