Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Photobleaching ensayos (FRAP y FLIP) para medir la dinámica de la cromatina de proteínas en células vivas madre embrionarias
En este artículo
Resumen
Se describen los métodos de photobleaching incluyendo recuperación de fluorescencia después de photobleaching (FRAP) y la pérdida de fluorescencia en photobleaching (FLIP) para controlar la dinámica de la cromatina de proteínas en células madre embrionarias (ES) las células. Dinámica de la cromatina proteína, que es considerado como uno de los medios para estudiar la plasticidad de la cromatina, es mayor en las células pluripotentes.
Resumen
La recuperación de fluorescencia después de photobleaching (FRAP) y la pérdida de fluorescencia en photobleaching (FLIP) permiten el estudio de la dinámica de proteínas en las células vivas con una buena resolución espacial y temporal. Aquí se describe cómo llevar a cabo ensayos FRAP y FLIP de proteínas de la cromatina, como H1 y HP1, en madre embrionarias de ratón (ES) las células. En un experimento FRAP, las células son transfectadas, ya sea transitoria o estable, con una proteína de interés fusionada con la proteína verde fluorescente (GFP) o sus derivados (YFP, PPC, cerezo, etc.) En las células transfectadas, fluorescentes, un intenso haz de láser enfocado blanqueadores una región relativamente pequeña de interés (ROI). La longitud de onda se selecciona de acuerdo a la proteína fluorescente utilizado para la fusión. La luz del láser irreversiblemente blanquea la señal fluorescente de moléculas en el retorno de la inversión, e inmediatamente después de blanqueo, la recuperación de la señal fluorescente en la zona blanqueada - mediado por la sustitución de las moléculas de blanqueado con las moléculas de crudo - se vigila por medio de imágenes de lapso de tiempo. Las curvas de fluorescencia generada recuperación de proporcionar información sobre la movilidad de la proteína. Si las moléculas fluorescentes son inmóviles, no hay recuperación de fluorescencia se observó. En un enfoque complementario, la pérdida de fluorescencia photobleaching (FLIP), el rayo láser blanquea el mismo lugar varias veces y la intensidad de la señal se mide en otras partes de la célula de fluorescencia. Experimentos FLIP por lo tanto, medir la descomposición de la señal en lugar de la recuperación de la fluorescencia y son útiles para determinar la movilidad de proteínas, así como proteínas y venir entre los compartimentos celulares. Unión transitoria es una característica común de las proteínas asociadas a la cromatina. Aunque la mayor fracción de cada proteína de la cromatina se une a la cromatina en un momento dado en el estado estacionario, la unión es temporal y la mayoría de las proteínas de la cromatina tienen una alta rotación en la cromatina, con un tiempo de residencia en el orden de segundos. Estas propiedades son cruciales para la generación de alta plasticidad en la expresión del genoma 1. Photobleaching experimentos son particularmente útiles para determinar la plasticidad de la cromatina mediante la fusión GFP-versiones de la cromatina proteínas estructurales, especialmente en las células madre embrionarias, donde el intercambio dinámico de las proteínas de la cromatina (incluyendo la proteína de heterocromatina 1 (HP1), la histona H1 y enlazador histonas) es mayor que en las células diferenciadas 2,3.
Protocolo
1. Revestimiento de las células madre embrionarias
T = 0 horas
MEF placas
- Escudo de la imagen en vivo de 8 y μ-Slides (ibidi, Munich, Alemania) con gelatina o en cámaras cubiertas de vidrio (Lab-Tek, Rochester, NY) o en placas de cultivo con fondo de cristal (MatTek, Ashland, MA). Deja de 5-30 min y aspirar lejos de la gelatina sin.
- Semillas de 22.000 MEFs / pocillo en 250 l de volumen total de DMEM [complementado con fetal al 10% de suero bovino (FBS)]. Permiten a las células crecer en un cultivo de tejidos incubadora (37 ° C, 5% CO2).
T = 6 horas
ES recubrimiento celular
- Aspirar DMEM.
- Semillas de cada MEF pozo cubierto con 15.000 células R1 / pocillo en 250 l de volumen total de los medios de comunicación de las células ES [complementado con ESC de grado 10% de suero fetal bovino (FBS), 1 mM piruvato de sodio, 0,1 mM de aminoácidos no esenciales, 0,1 mM de β- mercaptoetanol, y 1000 U / ml de factor inhibidor de leucemia (LIF)], para obtener un 30% a 50% de confluencia al día siguiente.
2. Transfección de las células madre embrionarias
T = 24 horas
Transfección transitoria
- Vuelva a colocar los medios de comunicación de las células ES, con 250 l / pocillo de nuevo los medios de comunicación de células ES.
- En un tubo de ensayo 1,5 ml estéril, añadir 100 ml de suero libre de los medios de comunicación [Opti-MEM (Gibco)], a continuación, añadir 10 TransIT-LT1 l reactivo de transfección (Mirus) directamente en los medios de comunicación libres de suero. Mezclar por pipeteo suave e incubar a temperatura ambiente durante 50-20 min.
- Añadir 1,5 g GFP ADN plásmido de fusión (H1E, H1o o HP1) de los gases de tránsito LT1 reactivo. Mezclar por pipeteo suave e incubar a temperatura ambiente durante 15-30 min.
- Añadir 13,5 l / pocillo de la mezcla de transfección. Agitar el 8 y μ-Slides para asegurar una dispersión. Después de 24 horas reemplazar los medios de comunicación ES celulares viejos, con 250 l de nuevo los medios de comunicación de células ES.
3. Realización de FRAP y FLIP
T = 48 a 72 hrs
- El experimento se puede realizar en cualquier microscopio láser confocal de barrido (CLSM) pero como en una normal FRAP / FLIP experimento, muchas imágenes consecutivas son adquiridos, se recomienda el uso de un microscopio confocal de disco giratorio, que permite que la velocidad de adquisición y asegura que no muestra no deseada decoloración se produce después de la inicial de eventos intencionales de blanqueo. En este caso, recomendamos el uso de la Revolución giro de disco del sistema confocal (www.Andor.com), con la Yokogawa CSU-X cabeza del disco giratorio. Este sistema tiene la doble capacidad de photobleach con un módulo especializado FRAPPA con un sistema de puntos de exploración, y cambiar rápidamente la luz de fondo para recoger las imágenes con el disco giratorio. Los tres proteínas fluorescentes más comunes utilizados para los experimentos se photobleaching GFP, YFP y Cherry. Si GFP o YFP se utilizan, a ~ 488 nm láser es necesario. Para Cherry, el uso de un láser ~ 560 nm. En todos los casos, recomendamos el uso de láseres de estado sólido. Tener una etapa automatizado es útil pero no necesario. Dado que las células vivas son imágenes, es fundamental utilizar una cámara de medio ambiente (se puede utilizar un LIS, Suiza), el control de oxígeno, humedad, CO 2 y la temperatura. FRAP se realiza con la intensidad del láser, mientras que el máximo de imágenes se hace con la potencia del láser mínimo requerido (por lo general en el área del 10%, cuando el nivel de fluorescencia es adecuada).
Observar las células con luz fluorescente de la longitud de onda apropiada y seleccione una célula que expresa GFP usando un lente de 60x de inmersión en aceite. Asegurar una distribución subcelular correcta. En ocasiones, cuando los niveles de expresión son muy altos, la localización de la proteína puede "derrame" de otros compartimentos como el nucléolo. Estas células no se debe elegir. - Ahora establezca un protocolo de imagen: recolectar 5.3 cuadros antes que el photobleaching, entonces photobleach en eucromatina o heterocromatina (vistos como focos condensada GFP) y recoger 90 a 120 fotogramas después de la photobleach, con intervalos de 250 a 1.000 ms: H1E-GFP, 1000 ms, H1o y HP1-GFP, 250 ms (intervalo de tiempo los cambios de acuerdo a la dinámica de las proteínas, donde las proteínas altamente dinámicos requieren menor tiempo de intervalo). Normalmente se usan 80 a 100% la intensidad del láser para photobleaching con un pulso láser de μseconds 20-40 (1-2 repeticiones), pero estos números pueden cambiar de acuerdo a la proteína analizados y los niveles de expresión. Cuando photobleaching es apropiado, se debería observar un "agujero negro" en la fluorescencia de GFP. El agujero negro se va re-lleno de recuperación después de la fluorescencia. Aunque el disco giratorio se puede obtener un máximo de alrededor de 60 imágenes por segundo (con buena intensidad de la fluorescencia y el zoom en una sola célula), no se recomienda utilizar el sistema a una velocidad tan alta debido a la baja calidad de imagen y mayor potencial de fototoxicidad.
- Para un experimento de la FLIP, establecer un protocolo de imágenes diferentes: recolectar 5.3 cuadros antes del blanqueo, y comienza a repetir blanqueo en el mismo lugar durante la percepción de imágenes. Para H1E-GFP, lejía cada 5 segundos, para H1o-GFP, cloro cada 2 segundos, y para HP1-GFP cloro cada 1 seg. Bleach und recoger imágenes en repetidas ocasiones a lo largo de todo el experimento.
- Para cualquiera de estas técnicas, repita el proceso en 20-30 células. A efectos estadísticos, repita el experimento 3 veces o más, preferentemente en días diferentes. En poblaciones homogéneas y configuración correcta, la desviación estándar es generalmente baja (<5%).
Por tanto FRAP y FLIP, el tamaño y la forma de la zona blanqueada influye en la dinámica de recuperación y debe permanecer constante dentro de un experimento. Además, cuando se comparan dos células, los protocolos debe ser idéntico y las células deben ser analizadas de forma secuencial en el mismo día que la potencia del láser y otras condiciones pueden variar y pueden afectar el resultado del experimento.
4. FRAP y Análisis de Datos FLIP
- En todos los cuadros recogidos FRAP, medir la intensidad de fluorescencia en el retorno de la inversión (ROI b = área blanqueada), el área de fondo (ROI bg), y el área de no proliferación nuclear, blanqueadas (ROI nb) como una función de tiempo antes y después del blanqueamiento. Cuando la región blanqueada es insignificante todo el núcleo puede ser seleccionado para fines de normalización.
- Por cada punto del tiempo, normalizar los datos de acuerdo con la fórmula: (b ROI - ROI bg) / (ROI nb - ROI bg) / (b pbROI - pbROI bg) / (pbROI nb - pbROI bg), pb indica pre-blanqueado. Para pre-cloro imágenes que debería obtener un valor aproximado de 1. La primera imagen después de que el cloro se indica la profundidad de Bleach. Restar el valor de 1 para el valor de Bleach profundidad real. Repita la operación para todas las células y el promedio de 20 a 30 células de cada experimento.
- En todos los cuadros recogidos FLIP, medir la intensidad de fluorescencia en la zona de no proliferación nuclear, blanqueados, y el área de fondo (ROI nb = no blanquea la zona, el ROI de fondo bg =). Cálculo de los datos de la FLIP es similar a una curva de FRAP, sólo el retorno de la inversión analizada (ROI nb) debe ser diferente de la propia región blanqueada, que no se utiliza para el cálculo: (ROI nb - ROI bg) / (pbROI nb - pbROI bg) . También es posible utilizar una celda contigua (ROI n celular vecino =) para fines de normalización: (ROI nb - ROI bg) / (ROI n - ROI bg) / (pbROI nb - pbROI bg) / (pbROI n - pbROI bg ).
Tras la recogida de datos, es posible ajustar los datos experimentales a la simulación por ordenador. Esto permite calcular, con una buena proximidad, la fracción móvil, la fracción de inmovilidad y la mitad del máximo. No vamos a discutir los aspectos matemáticos y estadísticos de análisis FRAP aquí y remitimos al lector a otras publicaciones excelentes 4-9. La profundidad de cloro se refiere a la distancia (en el eje y) entre el pre-cloro (100%) de la señal y la primera imagen después de cloro, la fracción móvil se refiere a la distancia (en el eje y) entre la profundidad de cloro y la señal recuperada cuando la cinética alcanza una meseta, y la fracción inmóvil refiere a la distancia (en el eje y) entre la señal recuperada y la señal de pre-cloro (100%) (ver Figuras 1B y 2B). Más allá de este análisis, hay buenos modelos matemáticos para ajustar los datos. Para un exponente único, la ecuación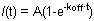
donde T es el tiempo, A es la fracción móvil, 1-A es la fracción inmóvil y fuera de k es la constante de disociación, se puede utilizar para ajustar los datos, y una estimación directa de la tasa de descuento de la unión (k off) se puede obtenidos, así como para el parámetro A, que se puede utilizar para calcular el tipo de asociación.
5. Los resultados representativos:
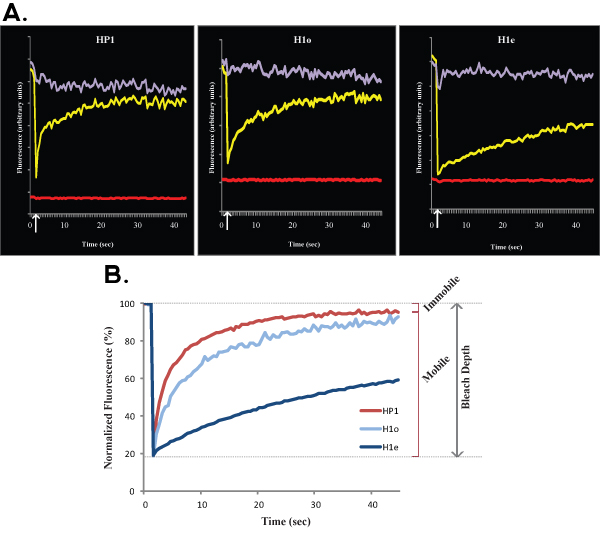
Figura 1. A y B muestran las curvas de FRAP representante de HP1 (izquierda), H1o (centro) y H1E (derecha) en las células ES R1. La figura de la simplicidad y claridad 1A muestra los datos en bruto de una sola célula antes de que la normalización y el cálculo. La curva amarilla corresponde a la región blanqueada, la curva de color púrpura se corresponde con el área de no proliferación nuclear, blanqueadas (cuando la región blanqueada es insignificante todo el núcleo puede ser seleccionado para fines de normalización), y la línea roja corresponde a la fluorescencia de fondo, que es mínima en este caso. Flecha vertical representa el tiempo de blanqueo. Los datos normalizados y un promedio se muestra en la Figura 1B. Tenga en cuenta la recuperación más lenta de H1 (azul) en comparación con HP1 (rojo). También la variante H1E (azul oscuro) es más lenta que la variante H1o (azul claro). Fracciones móviles e inmóviles, y la profundidad de Bleach están indicados para HP1.
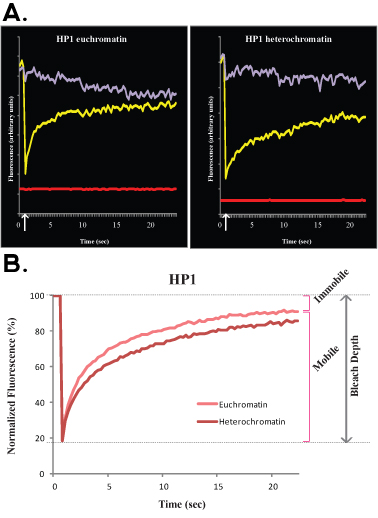
Figura 2. A y B muestran las curvas de FRAP representante comparar eucromatina (izquierda) con la heterocromatina (derecha) de HP1 en las células ES R1. Al igual que en la Figura 1, Figura 2A muestra los datos en bruto de una sola célula, la curva amarilla corresponde a la región blanqueada, la curva de color púrpura se corresponde con el área de no proliferación nuclear, blanqueados, y la línea roja corresponde a la fluorescencia de fondo. Flecha vertical representa el tiempo de blanqueo. Los datos normalizados y un promedio se muestra en la Figura 2B. Tenga en cuenta la recuperación más lenta de la heterocromatina (rojo oscuro) en comparación con la eucromatina (luz roja). Fracciones móviles e inmóviles, y la profundidad de Bleach están indicados para la eucromatina.
Figura 3.. Un experimento típico de la FLIP H1o R1 células madre embrionarias se muestra en la Figura 3 (en bruto, sin normalizar los datos) y B (datos normalizados y medio). En este experimento, la curva de color púrpura se corresponde con el área nuclear no blanqueado, la línea verde corresponde a un núcleo de la célula vecina y la línea roja corresponde a la fluorescencia de fondo.
Discusión
A diferencia de la mayoría de las técnicas disponibles, que incluyen la cromatina purificada a partir de poblaciones de células o células fijadas, los experimentos FRAP seguir los cambios en la dinámica de las proteínas de la cromatina en las células vivas. Encontramos dinámica de la cromatina proteínas para ser un buen indicador de la plasticidad de la cromatina. Sin embargo, debido a que requiere la fusión del gen de interés con las buenas prácticas agrarias, la adición de la etiqueta fluorescente puede i...
Divulgaciones
Agradecimientos
Agradecemos a los miembros del laboratorio de Meshorer, especialmente Shai Melcer, Alajem Adi, Edupuganti Raghu Ram, Badi Sri Sailaja, Mattout Anna y Biran Alva, por comentarios críticos y ensayos de solución de problemas photobleaching sobre una base diaria. EM es una H. Joseph y Belle R. Braun Profesor titular de Ciencias de la Vida y con el apoyo de la Fundación Ciencias de Israel (ISF 943/09), el Ministerio de Salud de Israel (6007) de la Unión Europea (IRG-206872 y 238176), la Fundación para la Investigación del Cáncer de Israel, las subvenciones internas Aplicativo de Medicina de la Universidad Hebrea y del Instituto de Psicobiología Israel.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
|---|---|---|---|
| DMEM | Sigma | D5671 | |
| Gelatina | Merck | 1,04078 | |
| Opti-MEM | Gibco | 31985 | |
| TransIT-LT1 | Mirus | MIR2300 | |
| 8 y μ-Diapositivas | ibidi | 80826 |
Referencias
- Phair, R. D. Global nature of dynamic protein-chromatin interactions in vivo: three-dimensional genome scanning and dynamic interaction networks of chromatin proteins. Mol Cell Biol. 24, 6393-6402 (2004).
- Meshorer, E., Girard, L. . Imaging chromatin in embyonic stem cells in StemBook. , (2008).
- Meshorer, E. Hyperdynamic plasticity of chromatin proteins in pluripotent embryonic stem cells. Dev Cell. 10, 105-116 (2006).
- Bancaud, A., Huet, S., Rabut, G., Ellenberg, J. . Fluorescence perturbation techniques to study mobility and molecular dynamics of proteins in live cells: FRAP, photoactivation, photo conversion, and FLIP. , (2009).
- Dundr, M., Misteli, T. Measuring dynamics of nuclear proteins by photobleaching. Curr Protoc Cell Biol. Chapter 13, Unit 13-Unit 13 (2003).
- Ellenberg, J. Nuclear membrane dynamics and reassembly in living cells: targeting of an inner nuclear membrane protein in interphase and mitosis. J Cell Biol. 138, 1193-1206 (1997).
- Lenser, T., Weisshart, K., Ulbricht, T., Klement, K., Hemmerich, P. Fluorescence fluctuation microscopy to reveal 3D architecture and function in the cell nucleus. Methods Cell Biol. 98, 2-33 (2010).
- Mueller, F., Mazza, D., Stasevich, T. J., McNally, J. G. FRAP and kinetic modeling in the analysis of nuclear protein dynamics: what do we really know. Curr Opin Cell Biol. 22, 403-411 (2010).
- Phair, R. D., Misteli, T. Kinetic modelling approaches to in vivo imaging. Nat Rev Mol Cell Biol. 2, 898-907 (2001).
- Poser, I. BAC TransgeneOmics: a high-throughput method for exploration of protein function in mammals. Nat Methods. 5, 409-415 (2008).
- Sigal, A. Generation of a fluorescently labeled endogenous protein library in living human cells. Nat Protoc. 2, 1515-1527 (2007).
- Cohen, A. A. Dynamic proteomics of individual cancer cells in response to a drug. Science. 322, 1511-1516 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados