Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La extracción de ADN de alto peso molecular a partir de tapetes microbianos
En este artículo
Resumen
Ofrecemos un protocolo mejorado para la extracción de ADN de alto peso molecular de tapetes microbianos hipersalinos. Las células microbianas son separados de la matriz de alfombra antes de la extracción y purificación del ADN. Esto mejora la concentración, calidad y tamaño de la DNA. El protocolo puede ser utilizado para otras muestras de refractario.
Resumen
Análisis de éxito y preciso y la interpretación de los datos de metagenómica depende de la extracción eficiente de alta calidad, la comunidad de ADN de alto peso molecular (HMW). Sin embargo, las muestras ambientales colchoneta a menudo plantean dificultades para la obtención de grandes concentraciones de alta calidad, de alto peso molecular del ADN. Tapetes microbianos hipersalinos contienen altas cantidades de sustancias poliméricas extracelulares (EPS) 1 y las sales que pueden inhibir aplicaciones posteriores del ADN extraído. Los métodos directos y las duras a menudo se utilizan en la extracción de ADN de muestras de refractario. Estos métodos se utilizan normalmente debido a que la EPS en esteras, una matriz adhesiva, une el ADN 2,3 durante la lisis directa. Como resultado de los métodos de extracción más dura, el ADN se fragmenta en pequeños tamaños 4,5,6.
El ADN se convierte en inadecuado para la reproducción de vectores de gran inserción. Con el fin de sortear estas limitaciones, se presenta una metodología mejorada para extraer el ADN de alto peso molecular de buena calidad y cantidad de tapetes microbianos hipersalinos. Se empleó un método indirecto, que supone la separación de las células microbianas de la matriz de alfombra de fondo a través de la mezcla y centrifugación diferencial. Una combinación de procedimientos mecánicos y químicos se utilizó para extraer y purificar el ADN de las células microbianas extraídos. El protocolo de los rendimientos de aproximadamente 2 g de ADN de alto peso molecular (35-50 kb) por gramo de muestra de alfombra, con un ratio de 1,6 260/280. Por otra parte, la amplificación de genes del 16S rRNA 7 sugiere que el protocolo es capaz de minimizar o eliminar los efectos inhibitorios de los contaminantes. Nuestros resultados proporcionan una metodología adecuada para la extracción de ADN de alto peso molecular de tapetes microbianos de los estudios de metagenómica funcional y puede ser aplicable a otras muestras ambientales de la extracción de ADN, que es un reto.
Protocolo
1. La extracción de células microbianas:
- Homogeneizar las alfombras microbianas con un mortero estéril molienda mezclando a fondo. Lugar aproximadamente a los 30 g (peso húmedo) de material de alfombra homogeneizada en un recipiente estéril de Waring batidora, añadir aproximadamente 100 ml de 1 M de NaCl (o una concentración específica de la muestra en uso), y la mezcla de tres veces a velocidad media durante 1 minuto con enfriamiento intermitente en un congelador a -20 ° C durante 1 min. La transferencia de la mezcla en una botella de 250 ml de centrífuga y llenar el volumen vacío restante con 1 M NaCl. Prepare una solución de NaCl en autoclave de agua DI y el filtro de esterilización de la misma.
- Además desprender las células microbianas de la matriz por agitación (150 rpm) usando un vórtice (Vortex-Genie R 2) a temperatura ambiente durante 30 min.
- Centrifugar a baja velocidad (500x g) durante 15 min a 4 ° C. Suavemente transferir el sobrenadante a un matraz con el menor trastorno posible a los sedimentos.
- Utilizando el sedimento pellets, repita los pasos de fusión y 1.2 y 1.3 cuatro veces más con la adición de NaCl fresco a cada uno de los sedimentos de pellets en la final de la etapa 1.3. El sobrenadante de cada extracción de NaCl se transfiere a un frasco de dulce.
- Combine los sobrenadantes de la extracción de células y centrifugar 5 en alícuotas de 200 ml a alta velocidad (25.000 xg) durante 15 min a 4 ° C. Descartar el sobrenadante, resuspender cada pellet de células en 10 ml de hexametafosfato de sodio al 2%, se combinan pastillas y completar el volumen a 200 ml con hexametafosfato de sodio al 2%. Lavar las células de agitación (150 rpm) a temperatura ambiente durante 30 min. Al final de este paso, debe tener un solo tubo de células para proceder al siguiente paso.
- Se centrifuga a alta velocidad (25.000 xg) durante 15 min a 4 ° C. Descartar el sobrenadante, resuspender sedimento celular en 200 ml de TE (50 mM EDTA), y lavar las células de agitación (150 rpm) a temperatura ambiente durante 10 min.
- Se centrifuga a alta velocidad (25.000 xg) durante 15 min a 4 ° C, descartar el sobrenadante, resuspender en 15 ml de TE (10 mM EDTA), y la tienda de 200 l alícuotas a -80 ° C hasta que sea necesario para la extracción de ADN.
2. Extracción de ADN y purificación:
- Añadir 200 l de NaCl 5 M, 200 l 10% de SDS, y 100 l 14,3 M β-mercaptoetanol a la alícuota microbiana de 200 l de células. Mezclar suavemente por inversión de 4.6 veces.
- Someter la mezcla a 3 rondas de congelación-descongelación sumergiendo en nitrógeno líquido durante 2 min seguido de un descongelamiento en un baño de agua de 65 ° C durante 5 min. Ampliar última descongelación a 10 min.
- Añadir 200 l de acetato de potasio 5 M (pH 5,5) y el lugar en hielo durante 10 min. Centrifugar a 10.000 xg durante 10 min a 4 ° C y la transferencia del sobrenadante a un nuevo tubo con una punta de la pipeta de gran calibre. Añadir 3 l de RNasa A y se incuba a 37 ° C durante 1 hora.
- Añadir un volumen igual de cloroformo, agitar brevemente por inversión y centrifugar a 15.000 xg durante 10 min a temperatura ambiente.
- Transferir cuidadosamente la capa superior del sobrenadante a un tubo nuevo con una punta de la pipeta de gran calibre. Evite el pipeteo de la capa de interfaz de color blanco situado entre las capas superior e inferior. Repita el procedimiento de extracción de cloroformo.
- Añadir un volumen igual de frío (a -20 ° C) isopropanol al sobrenadante e incubar en hielo durante 30 minutos para precipitar el ADN.
- Centrifugar a máxima velocidad (20.800 xg) durante 10 min a 4 ° C para precipitar el ADN.
- Descartar el sobrenadante y lavar el ADN precipitado con 1 ml de frío (a -20 ° C) de etanol al 70%.
- Centrifugar a máxima velocidad (20.800 xg) durante 10 min a 4 ° C, descartar el sobrenadante, repetir el 70% de etanol de lavado y secado al aire pellet de ADN por 10 min. Repita el 70% de etanol de lavado.
- Resuspender el ADN en 25 l TE (10 mM EDTA), caliente a 65 ° C durante 5 minutos, se combinan en un tubo si varios tubos se han utilizado, y el maquillaje volumen a 500 ml con TE.
- Añadir 500 l helado (o menos 4 ° C) 20% de glicol polietileno (PEG) (preparado en 1,2 M NaCl). Mezclar suavemente por inversión e incubar en hielo durante 10 min y centrifugar a máxima velocidad durante 10 minutos a 4 ° C para precipitar el ADN. Retire y deseche el sobrenadante, lavar el ADN granulado con 1 ml de etanol frío al 70%, de secado al aire durante 10 minutos, y resuspender en 30-50 l TE o agua de grado molecular. Facilitar la resuspensión de ADN por calentamiento a 65 º C durante 5 minutos y el ADN de la tienda a -80 ° C.
3. La pureza del ADN, la concentración y determinación del tamaño:
- Determinar la calidad del ADN mediante la medición del A y A 260/280 260/230 ratios usando un espectrofotómetro Nanodrop 1000 o cualquier instrumentación apropiada.
- Determinar la concentración de ADN utilizando Quant-iT kit de ADN de doble cadena de ensayo de acuerdo a las instrucciones del fabricante.
- Determinar el tamaño de ADN utilizando el campo de pulsos electroforesis en gel. Prepare un 1% en gel de agarosa con TBE 0.5X y utilizar los siguientes parámetros en el CHEF Mapper XA sistema, el tiempo de cambio inicial de 0,35 s, un tiempo de cambio de final de 7,67 s, y 120 ° ángulo incluido de un gradiente de 6,0 V / cm en una r linealamplificación de los factores. Mancha de gel con 1 x SYBR Green I durante 30 minutos. Carga entre 700-1000 ng de ADN total en gel. Ver fig. 3 de una forma condensada de todo el protocolo.
Los resultados representativos:
Celda de extracción:
Secuencial de los extractos de células microbianas (sobrenadantes) han demostrado que la turbidez de los extractos disminuye a medida que aumenta el número de extracciones. Esto sugiere una reducción en el número de células después de cada extracción de células sucesivas. Es importante señalar aquí es que cada paso célula de extracción adicional proporciona la oportunidad para la introducción de contaminantes, el número de extracciones de células debe ser minimizado para que el final precipitado de células es representativa de la comunidad microbiana en general. Otras fuentes de contaminación se controla mediante la adopción de técnicas adecuadas de laboratorio estéril. Por ejemplo, las soluciones se prepararon en autoclave agua desionizada y esterilizada filtro. Los contenedores fueron esterilizados con alcohol, autoclave, y se trata a la luz ultravioleta y la radiación ultravioleta reticulante.
Concentración de ADN y la determinación de la calidad:
El protocolo produjo aproximadamente 2 g de ADN de alto peso molecular (35-50 kb) (Fig. 4) por gramo de muestra de tapete con un A 260/280 ratio de 1,6, y un A 260/230 ratio de 0,7 (Tabla 1). Aunque la relación A 260 / 230 pareció ser baja, no hay inhibición de aguas abajo de base molecular de aplicaciones como PCR-amplificación de los genes del 16S rRNA se observó en un estudio separado 7. Es importante tener en cuenta que el ADN de las esteras hipersalinas tiene dos fuentes principales de contaminación, EPS y las sales. Por tanto, es posible que los restos de estos contaminantes pueden estar influyendo en la A 260/230 relaciones a pesar de los enormes esfuerzos para reducir sus efectos sobre las aplicaciones de aguas abajo.
La determinación del tamaño del ADN:
Electroforesis en gel de campo de pulsos utiliza un flujo de corriente pulsante con interruptores intermitentes de dirección que resulta en una prueba de ADN, como se muestra en la figura 3. Nuestro protocolo produjo un ADN de alto peso molecular de aproximadamente 35 a 50 kb. Mientras que algunos de tamaño de ADN puede ser menor de 30 kb, es importante que una parte de la mancha por encima de 35 kb, ya que la clonación fosmid requiere ~ 40 kb inserciones de ADN y grandes fragmentos de ADN proporcionan un mayor acceso a las vías de biosíntesis intacto. En nuestros estudios, la prueba de ADN por encima de 35 kb se cortó y se purifica para la clonación de vectores de gran inserción y otras aplicaciones molecular.

Figura 1. Microbianas hipersalinas alfombra sitio de muestreo (Laguna Grande) se encuentra en Eleuthera, Bahamas.

Figura 2. Una sección de la alfombra microbiana hipersalina utilizados en este estudio. El tapete microbiano se obtuvo de una laguna hipersalina situada en Eleuthera, Bahamas.
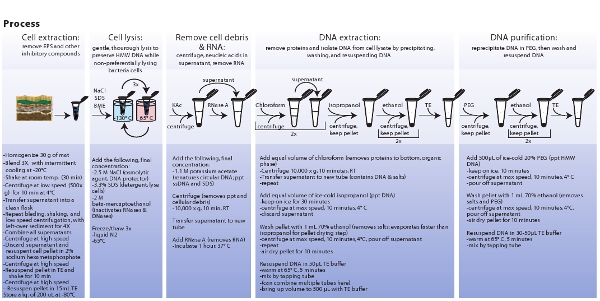
Figura 3. Representación esquemática de los procedimientos involucrados en la extracción de células microbianas, la lisis celular, la extracción y purificación del ADN metagenómica. Haga clic aquí para ver una imagen más grande.

Figura 4. Caracterización peso molecular del ADN extraído utilizando el campo de pulsos electroforesis en gel. Carril M es un marcador de alto peso molecular, y las calles 1 y 2 son repeticiones de ADN extraído de la metagenómica alfombra Eleuthera hipersalinos utilizando el protocolo anterior.
| Método de extracción | Concentración ng / g | A 260 / 280 | A 260 / 230 | |
| PEG | Representante de una | 1806.1 | 1.64 | 0.76 |
| Rep 2 | 2010.8 | 1.61 | 0.75 |
Tabla 1. Medición de la concentración y la calidad del ADN extraído de alfombra microbiana hipersalinos.
Discusión
Teniendo en cuenta que la eliminación total de las células de las muestras de alfombra complejos y muy variados microbiana no es práctico, la principal preocupación es qué tan bien las células extraídas representar a la comunidad microbiana alfombra en general. En un estudio previo, PCR-DGGE análisis de microorganismos genes del 16S rRNA mostró que la eliminación de cinco pasos de células utilizadas en este protocolo de extractos de células que son representativos de la comunidad microbiana alfombra en gener...
Divulgaciones
Agradecimientos
Este trabajo fue financiado por el Programa Nacional Science Foundation Ambiental Genómica (Grant No. EF-0723707).
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
|---|---|---|---|
| β-mercaptoetanol | Sigma-Aldrich | M3148 | |
| Polietilenglicol 8000 | Promega | V3011 | 20% en 1,2 M NaCl |
| Acetato de potasio | Fisher Scientific | Fisher Scientific | |
| Quant-iT ensayo dsDNA kit | Invitrogen | Q33130 | |
| RNasa | Epicentro | MRNA092 | |
| Cloruro de sodio | BDH productos químicos | BDH8014 | Conc apropiado. |
| Dodecilsulfato de sodio | Fisher Scientific | 03-500-509 | 10% en agua |
| hexametafosfato de sodio | EMD Chemicals | SX0583-3 | 2% en agua |
| TBE | Fisher Scientific | BP1333-1 | |
| CHEF Mapper XA Sistema | Bio-Rad Laboratories | 170-3670 | |
| NanoDrop 1000 espectrofotómetro | Thermo Scientific | ND-1000 | |
| Vórtice | Scientific Industries Inc. | ||
| Crosslinker ultravioleta | UVP | ||
| Mezclador Waring | Waring laboratorio | LB10S |
Referencias
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados