Method Article
La visualización de ADN recombinante y complejos de proteínas utilizando microscopía de fuerza atómica
En este artículo
Resumen
A modo de aprovechar microscopio de fuerza atómica (AFM) el método para la visualización de ADN plásmido, proteínas citoplasmáticas, y los complejos ADN-proteína se describe. El método incluye los enfoques alternativos para la preparación de muestras para el AFM imágenes después de la manipulación bioquímica. ADN que contiene regiones específicas que interactúan las proteínas se observan en las condiciones de amortiguación casi fisiológico.
Resumen
Microscopía de fuerza atómica (AFM) permite la visualización de proteínas individuales, las moléculas de ADN, los complejos de proteína-proteína, y los complejos ADN-proteína. En el extremo del voladizo del microscopio es una sonda de nano-escala, que atraviesa zonas de la imagen que van desde los nanómetros a micrómetros, que mide la elevación de las macromoléculas que descansa sobre la superficie del sustrato en un momento dado. Las fuerzas electrostáticas hacen que las proteínas, lípidos y ácidos nucleicos para fijar libremente sobre el sustrato en orientaciones aleatorias y permitir imágenes. Los datos generados se asemejan a un mapa topográfico, donde las macromoléculas resolver en tres dimensiones las partículas de tamaño discreto (Figura 1) 1,2. Al tocar el modo de AFM implica la oscilación repetida del voladizo, que permite imágenes de los biomateriales relativamente suave, como el ADN y las proteínas. Uno de los notables beneficios de la AFM respecto a otras técnicas de microscopía nanométrica es su adaptabilidad con respecto a visualizar las proteínas individuales y complejos macromoleculares en tampones acuosos, incluyendo casi fisiológica condiciones de buffer, en tiempo real y sin manchas o revestimiento de la muestra que se va a examinar.
El método que aquí se presenta describe la imagen de ADN y un factor de transcripción immunoadsorbed (es decir, el receptor de glucocorticoides, GR) en la solución tampón (Figura 2). Immunoadsorbed proteínas y complejos de proteínas se puede separar de la immunoadsorbing anticuerpos cordón de pellets por la competencia con el epítopo de anticuerpos y luego imágenes (Figura 2). Esto permite una manipulación bioquímica de las biomoléculas de interés antes de la imagen. Una vez purificada, el ADN y las proteínas pueden ser mezclados y el complejo de la interacción resultante puede formar una imagen así. La unión del ADN a la mica requiere un catión divalente 3, tales como Ni 2 + o Mg 2 +, que se pueden añadir a las reservas de la muestra sin embargo, mantener la actividad de la proteína. Utilizando un enfoque similar, AFM se ha utilizado para visualizar las enzimas individuales, como la ARN polimerasa 4 y una enzima de reparación de cinco, con destino a las hebras individuales de ADN. Estos experimentos proporcionar información significativa en la proteína-proteína y proteína-ADN interacciones biofísicas que tienen lugar a nivel molecular. Partículas de imágenes individuales macromoleculares con AFM puede ser útil para determinar la homogeneidad de las partículas y para la identificación de la disposición física de los elementos constitutivos de las partículas de imágenes. Mientras que el presente método fue desarrollado para la visualización de los complejos chaperona GR-proteína y ADN 1,2 líneas de actuación a la que el GR se unen, se puede aplicar ampliamente el ADN de imágenes y muestras de proteínas de una variedad de fuentes.
Protocolo
1. Preparación de muestras de ADN y las proteínas para obtener imágenes libres de contaminantes
- Aislar a las biomoléculas para formar una imagen y colocarla en un tampón acuoso adecuado. Proteínas para formar una imagen puede ser purificado mediante cromatografía líquida de 6, inmunoadsorción seguido de la eliminación de los anticuerpos y los pellets de 1,2, o purificación Profinity exacta y eliminación de etiquetas 7 (Figura 2A). Aislar las moléculas de ADN para ser captado por la purificación minipreparación (Figura 2B).
- Confirmar la composición y la pureza de las muestras de proteínas para formar una imagen utilizando SDS-PAGE y Western Blot. Para el ADN, la secuencia y confirmar la pureza de completar un análisis de restricción y electroforesis en gel de agarosa.
- Incubar las muestras en un buffer de AFM de adsorción para promover la adhesión de la muestra a la mica. Para las muestras de proteínas, 10 mM HEPES buffer puede ser utilizado. Adicionales con baja fuerza iónica sales fuerza o cofactores se pueden añadir otros también. Para las muestras de ADN, incluyen un catión divalente (por ejemplo, Mg 2 + o Ni 2 +) para promover la adsorción sobre el sustrato de mica. Una adecuada Mg 2 +-tampón que contiene el ADN es de 10 mM Tris, pH 7,5, 10 mM NaCl, 2 mM de MgCl 2 3. Una alternativa Ni 2 + buffer es de 10 mM HEPES, pH 6,8, 10 mM NiCl2.
- Diluir la muestra a una concentración de 5 mg / ml en tampón de adsorción. Mezclar con cuidado. Guárdelo en hielo, mientras que el microscopio se está preparando.
2. Montaje de Sonda de AFM
- Coloque el soporte de celda de la sonda de líquido en la estación de acoplamiento en voladizo de instalación correspondientes.
- Localice el voladizo largo y grueso (denominado en voladizo de la 'B') de la palanca de fuerte nitruro (SNL) de la sonda que se utiliza para obtener imágenes. Cuidadosamente traslado al titular de celda de la sonda de líquido, asegurando que la punta permanece en posición vertical. Nota técnica: Deberá ser pagado mientras que la sonda se encuentra en tránsito, dejándolo caer desde cualquier altura puede dañar o destruir el voladizo o la punta. A micropalanca SNL (MSNL) de la sonda se puede utilizar como una alternativa a la sonda de SNL.
- Retire el soporte de la sonda de la estación de acoplamiento y examinar el voladizo bajo un microscopio de luz para asegurarse de que está intacta y bien asentado en el soporte de la celda de la sonda de líquido.
- Retire con cuidado la cabeza AFM del conjunto de cola de milano instrumento apretando el tornillo de cabeza moleteada pinza. Nota técnica: Deberá ser pagado durante la manipulación de la cabeza del AFM, dejándolo caer incluso de una distancia corta (por ejemplo, si no está sentado correctamente en el conjunto de cola de milano) puede causar daños considerables.
- Invertir la cabeza AFM y presione firmemente el soporte de la celda de la sonda de líquido en los cuatro pernos en la base de la cabeza del AFM. Con cuidado, volver la cabeza AFM en el conjunto de cola de milano instrumento. Suelte el tornillo de cabeza moleteada pinza para sujetar la cabeza del AFM en el instrumento.
3. Localización de la punta en voladizo, la alineación de láser, y el ajuste de fotodetector
- Utilizando el software del instrumento AFM, localice en su punta, moviendo los mandos de la microscopía óptica a bordo. Ajustar la iluminación del microscopio, si es necesario para mejorar la identificación de la punta. Lleve el extremo de la punta del voladizo en la proximidad de la cruz que aparece en el software del instrumento.
- Acerque la punta del voladizo en el foco mediante el ajuste de las flechas arriba y abajo del controlador óptica en el software. Use lenta (S) o la velocidad media (M), mientras que el ajuste de la óptica.
- Alinear el láser en su punta con los mandos de ajuste de láser. Mover los botones de ajuste hasta que el punto rojo de láser se encuentra dentro de la mancha iluminación blanca. Recorrido a lo largo del brazo en voladizo con un patrón de zig-zag hasta que el láser se coloca en el extremo en voladizo. Cuando está bien alineado, un punto fuerte reflejo aparecerá en la ventana frontal de la cabeza AFM.
- El centro de la célula fotoeléctrica ajustando las perillas fotodetector en la cabeza AFM. Esto alineará el punto láser en el punto de mira de la pantalla fotodetector del software del instrumento. La suma de la señal observada debe ser de aproximadamente 4.6.
4. Posicionamiento de la cabeza y el voladizo AFM tuning
- Coloque una hoja recién cortado de mica unido a una platina de metal AFM en el soporte de la muestra magnetizada encima de la placa porta-AFM. Esta configuración se utiliza para colocar la cabeza antes de la AFM imágenes de la muestra de ADN o de proteína preparada en el paso 1.
- Nota técnica: El titular de la muestra magnetizada y titular de líquido de las células de la sonda son más gruesas que otra muestra de fijación de mecanismos y de los titulares de la sonda. Si no existe actualmente depuración insuficiente entre el conjunto titular de la mica de la muestra y la cabeza AFM, mover la cabeza AFM por seleccionar el icono de retirar en el software del instrumento en varias ocasiones.
- Gire la placa de sujeción AFM para la platina se coloca para la imagen inicial. Asegúrese de que el sustrato de mica se centra en la cabeza AFM.
- Mueve la cabeza hacia el AFMsuperficie del disco mica-muestra utilizando el control de la superficie objeto de los instrumentos de software. Seleccione el medio (M) la velocidad en el motor Z, y continuar hacia la superficie hasta que la cabeza AFM es de aproximadamente 2 mm por encima de la mica. En este momento, pasar a utilizar el lento (S) Z velocidad del motor con el fin de evitar el contacto brusco entre la sonda y la superficie (en adelante, rompiendo la punta).
- Proceder a mover la sonda más cerca de la superficie del sustrato de mica hasta rasgos distintivos en la superficie de la mica o el reflejo extremo están en foco. Alternar entre la superficie y los planos de reflexión punta focal a través del "se centran en:" desde el software del instrumento.
- Seleccione el icono de la canción del software del instrumento y ajustar el cantilever. La frecuencia de resonancia del SNL recomendado y sondas MSNL cantilever es 20-60 kHz. Seleccione un máximo del 5% de compensación. Nota técnica: El ajuste voladizo adecuada es esencial para obtener imágenes de muestra adecuado.
5. Mica superficie de la muestra de imágenes
- Involucrar a la sonda sobre la superficie de la mica. Definición del tamaño de exploración inicial de 10 micras y velocidad de barrido de 1 Hz. Ganancia integral y el aumento proporcional puede ser inicialmente a 0,2 y 0,4, respectivamente. Ajustar las ganancias, según sea necesario a fin de que las exploraciones de seguimiento y volver más o menos se superponen. La disminución de la amplitud de consigna se incrementará la interacción entre la sonda y la mica y garantizar la participación.
- Capturar imágenes exploración completa del campo m 10. Disminución de tamaño de digitalización a 5, 1 y 0,5 micras y la captura de imágenes completas de cada campo. Ajustar las ganancias y velocidad de barrido es necesario. La captura de imágenes de la superficie de mica ofrece una línea de base para la comparación con el ADN y las proteínas que contienen las muestras.
- Desconectar la sonda con un solo clic en el icono de desactivar el software del instrumento.
6. Biomoléculas imágenes de interés
- Mezclar suavemente el ADN o las proteínas de la muestra preparada en el paso 1 y se preparan para cargarla en la superficie de mica imagen. Mezclar 5 l de la muestra preparada con 45 l de tampón fresco de adsorción (concentración de la muestra final = 0,5 mg / ml).
- Añadir con cuidado 50 mg l del 0,5 / ml de solución de biomoléculas que contienen directamente sobre la superficie de la mica. Hacer esto sin mover la cabeza de la sonda o AFM, y asegurar la solución de muestra (pero no punta de la pipeta) hace contacto con la sonda.
- Pausa de 5 minutos para permitir que las biomoléculas en la muestra que se adhieren a la superficie de la mica.
- Al término de 5 minutos, pipeta de un adicional de 50 a 100 l de tampón de adsorción en el soporte de celda de la sonda de líquido, teniendo cuidado de no tocar la sonda, la cabeza AFM, o mica con la punta de la pipeta. Esto forma un menisco y permiten aprovechar imagen AFM modo en el líquido. Figura 3 muestra la disposición final de la celda de fluidos, en voladizo, muestra, titular de la muestra, y el menisco.
- Reajustar el fotodetector y realinear el láser para el voladizo según sea necesario. Nota técnica: ver la punta de la sonda y la alineación láser es complicada debido a la difracción causada por la adición de tampón de muestra en el soporte de la sonda. Sintonizar manualmente el voladizo, si es necesario.
- Vuelva a instalar la sonda usando el software del instrumento. Ajustar la desviación vertical, ganancia integral, y el aumento proporcional según sea necesario. Esto comenzará el proceso de re-imagen de la sección de mica escaneados inmediatamente antes de la adición de la muestra y capturar imágenes.
- Aumentar el tamaño del escaneado (500 nm-10 micras) para identificar regiones de interés. Zoom-in y reducir la velocidad de barrido a 0,5 Hz para mejorar la resolución de la imagen
- Una vez se haya completado imágenes, desconecte la sonda mediante la selección de retirar icono en el software del instrumento en varias ocasiones. De suficiente espacio libre entre la cabeza del AFM y el disco muestra antes del desmontaje de la celda de líquidos o platina.
- Enjuague bien el titular de la celda de la sonda de líquido y platina con agua destilada para evitar que los cristales de sal que se formen como residual se evapora tampón de adsorción. Seca con aire comprimido.
7. Los resultados representativos:
Ejemplos de imágenes de AFM se presentan en la Figura 4. Sustrato de mica (A) proporciona una superficie plana sobre la cual molecular del ADN y las proteínas, pueden absorber. Mica de imágenes antes de que las muestras de biomoléculas proporciona un control negativo y la evaluación del ruido de imagen. También proporciona un nivel de seguridad de que el voladizo se adapta de forma correcta y muestra imágenes posteriores será un éxito. De doble cadena de ADN plásmido (B, D), presuntamente superenrollado, se identifica fácilmente por su apariencia asimétrica y uniforme de depositar sobre el sustrato de mica previamente poco notable. Complejos de proteínas de tamaños de partículas discretas (C, E) también se distingue de forma exclusiva el sustrato de mica. Las diferencias de tamaño de partículas indican heterogeneidad de la muestra, y puede ser útil para la aproximación de estequiometría complejo de proteínas o la actividad bioquímica. La forma general en diagonal y la orientación constante de la prolas partículas de proteína observado en el Panel de C es un artefacto de imagen, como las proteínas era de esperar que se orientarán sobre el sustrato de mica en forma aleatoria. Las posibles causas de que el artefacto se observa es una anormalidad física o punta de AFM de imágenes en demasiado rápido de una velocidad de barrido. Mientras que la convolución punta (ilustrado en la Figura 1B) evita los cálculos de medición de la longitud absoluta en los ejes X e Y, medición de la altura (eje z) y x relativos y las mediciones y puede ser sin embargo útil para estimar las propiedades biofísicas de las biomoléculas imágenes.
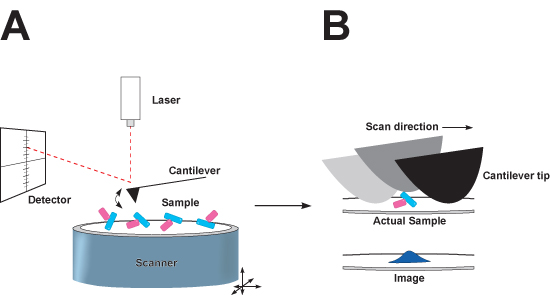
Figura 1: Representación esquemática de aprovechar el modo de microscopia de fuerza atómica (AFM). A, el microscopio. Al final del cantilever es una punta afilada que oscila arriba y abajo, ya que las exploraciones sobre la superficie de un sustrato de mica que absorben biomoleculares complejos. A medida que el escáner se mueve en las direcciones x e y, un rayo láser se refleja en la parte posterior del cantilever en una posición sensible detector de fotodiodos para trazar la vertical (z) de distancia de la punta se mueve a su paso por los complejos biomoleculares sentado en el mica. B. La distorsión de la imagen en las direcciones x e y causada por convolución punta. El radio nominal de una punta de nitruro de silicio convencionales, es más grande que las partículas sean imágenes, y el borde de los contactos de la punta de la muestra, ya que atraviesa la superficie. Resultados de la punta de convolución en el que aparecen imágenes de partículas más grande en la direcciones x e y, pero no la dirección z. Este efecto puede ser minimizado por el uso de las puntas con un radio de giro nominal menor.

Figura 2: Ilustración de la proteína immunoadsorbed y preparación de muestras de ADN. Un lanzamiento de GR immunoadsorbed • complejo de la proteína Hsp70 de los anticuerpos monoclonales (mAb)-proteína A-Sepharosa (PAS) de pellets. Incubando la immunopellet con un péptido que contiene el epítopo MAB facilitar la liberación de la GR complejos • proteína HSP70 de la pastilla, lo que permite que el complejo se recogerán a partir del sobrenadante y se visualizan mediante AFM. B, Preparación de ADN para el AFM imagen requiere el uso de cationes divalentes de adsorción de amortiguamiento. El catión divalente aumenta la afinidad de ADN para el sustrato de mica.

Figura 3: Disposición Final de la cabeza AFM, titular de líquido de las células de la sonda, muestra, soporte de la muestra, y el menisco de amortiguamiento. Un cuidado, debe tener cuidado al depositar la muestra y cobertura adicional en el sustrato de mica para evitar el contacto físico entre la punta de la pipeta y la cabeza AFM, llame al líquido, y el sustrato de mica. B, de ampliación de la A. El menisco tampón se extiende desde la platina al titular de la celda de la sonda de líquido.
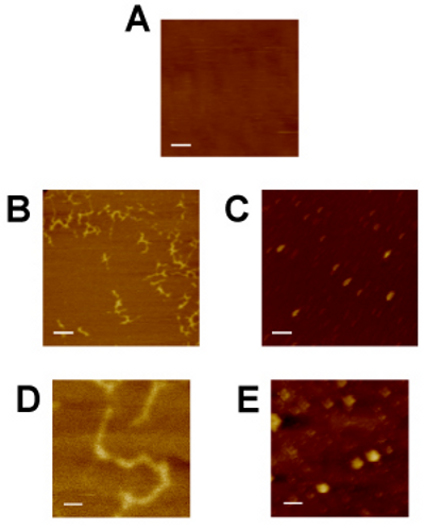
Figura 4: Micrografías de fuerza atómica de sustrato de mica (A), 2xGal4-2xGRE-luciferease ADN plásmido 8 (B, D), y GR • complejos de la proteína HSP70 (C, E) en la solución tampón. Mica sirve como una imagen de control para comparar con la visualización de ADN y proteínas. GR complejos de proteína Hsp70 • fueron preparados por inmunoadsorción de GR preparado con Hsp70 y luego se libera de la pastilla de anticuerpos de cuentas utilizando un anticuerpo mAb de la competencia (como se ilustra en la Figura 2A). ADN fue preparado por minipreparación plásmido convencional. Los grupos A, B y C son de un aumento equivalente (barras de escala = 200 nm), al igual que los paneles D y E (las barras de escala = 40 nm).
Discusión
AFM ofrece una técnica única capaz de obtener imágenes microscópicas individuales biomoléculas sin recubrir en soluciones tampón acuoso y cerca fisiológicos en tiempo real. Esto permite la visualización de proteínas y moléculas individuales de ADN, así como complejos multiproteicos y los complejos ADN-proteína. Partículas macromoleculares con imágenes AFM puede ser útil para evaluar la homogeneidad de la muestra y para la identificación de la disposición física de los elementos constitutivos de las partículas observadas. Este enfoque de la observación de las partículas individuales macromoleculares pueden ser un complemento útil a las técnicas convencionales de bioquímicos, tales como ensayos de inmunoprecipitación, electroforesis en gel de poliacrilamida, y la exclusión tamaño de cromatografía en columna, que proporcionan datos significativos sumativa en representación de la 10 3 -10 11 complejos de biomoléculas individuales de interés actual en una muestra.
La investigación sobre estequiometría multiproteico complejos, las interacciones biomoleculares, y los requisitos de cofactor todos pueden ser investigados mediante AFM. El método que aquí se presenta puede ser adaptado para dar cabida a preguntas específicas biofísicos de interés. Por ejemplo, utilizando un soporte de la celda de la sonda de líquido, capaz de intercambiar buffer, es posible que los requisitos de cofactor de ensayo para la formación de proteínas complejas. Proteína-ADN se pueden formar asambleas y disociado casi en tiempo real e incluso mientras se toman las radiografías. ADN se pueden generar con secuencias específicas de interés (por ejemplo, los elementos de respuesta o presunta nueva secuencia de unión a proteínas), mezclado con la hipótesis de la proteína de unión, y la imagen que proporcionan una evidencia directa de las interacciones intermoleculares.
Al tocar el modo de AFM de imágenes es mucho menos complicada si las muestras secas se utilizan en lugar de las muestras en soluciones acuosas, y se encuentra de ADN lineal y cerrado círculo de plásmidos de ADN han sido dos imágenes de esta manera 3,9. El método que aquí se presenta utiliza muestras en soluciones tamponadas con el fin de proporcionar un entorno de imagen más fisiológica. Otros métodos de imágenes notables de proteínas también deben ser considerados, incluyendo cerca de microscopía de campo infrarrojo 10. El uso complementario de inmunoadsorción en consorcio con AFM ofrece la oportunidad de preparar una gran variedad de complejos de proteínas, utilizando técnicas bien establecidas bioquímicos, y luego a visualizar después de la liberación de la immunoadsorbing anticuerpos cordón de pellets. Por ejemplo, GR immunoadsorbed ha sido ensayada por su asociación con la proteína chaperona molecular Hsp70 1, así como la proteína dineína motor 2 a través de este enfoque con el fin de estimar el tamaño del complejo y estequiometría. Tamaño de las partículas y las características biofísicas (rigidez por ejemplo) han sido útiles para determinar la identidad de las biomoléculas en las muestras AFM imágenes 11,12. También es posible confirmar la identidad de biomoléculas por la adición de una biomolécula que interactúan (por ejemplo, un ligando o anticuerpo monoclonal), que se unen a su objetivo y provocar un aumento del tamaño de las partículas si la biomolécula objetivo está presente.
Divulgaciones
Agradecimientos
Este trabajo fue financiado por los Institutos Nacionales de Salud Grant GM086822. Los autores desean agradecer a los Dres. Alec Pakhomov Wallace y Pablo (Univ. de Washington Fondo Usuario Nanotecnología (NTUF)) y Andrea Slade (Bruker AXS) por su asistencia técnica de expertos. ADN AFM de imágenes se llevó a cabo en la Universidad. de Washington NTUF, un miembro de la Red de Nanotecnología Nacional de Infraestructura. El plásmido 2xGal4-2xGRE-luciferease fue proporcionado amablemente por el laboratorio del Dr. Keith Yamamoto (Universidad de California en San Francisco).
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
|---|---|---|---|
| Dimension 3100 | Bruker | Dimension 3100 | |
| Dimensión de la celda de fluidos | Bruker | DTFML-DD-HE | |
| Palanca de nitruro de Sharp (SNL) de nitruro de silicio Sonda de AFM | Bruker | SNL-10 | |
| Micropalanca fuerte nitruro de palanca (MSNL) de nitruro de silicio Sonda de AFM | Bruker | MSNL-10 | |
| NanoScope AFM de instrumentos de software | Bruker | 004-132-000 | |
| De metal AFM platinas portamuestras | Ted Pella | 16208 | |
| Grado V1 Mica discos de 12 mm | Ted Pella | 50-12 |
Referencias
- Murphy, P. J. M. Visualization and mechanism of assembly of a glucocorticoid receptorhsp70 complex that is primed for subsequent Hsp90-dependent opening of the steroid binding cleft. J Biol Chem. 278, 34764-34773 (2003).
- Harrell, J. M. Evidence for glucocorticoid receptor transport on microtubules by dynein. J Biol Chem. 279, 54647-54654 (2004).
- Pastre, D. Adsorption of DNA to mica mediated by divalent counterions: a theoretical and experimental study. Biophys J. 85, 2507-2518 (2003).
- Guthold, M. Direct observation of one-dimensional diffusion and transcription by Escherichia coli RNA polymerase. Biophys J. 77, 2284-2294 (1999).
- Petrucco, S., Volpi, G., Bolchi, A., Rivetti, C., Ottonello, S. A nick-sensing DNA 3'-repair enzyme from Arabidopsis. J Biol Chem. 277, 23675-23683 (2002).
- Murphy, P. J. M., Morishima, Y., Kovacs, J. J., Yao, T. P., Pratt, W. B. Regulation of the dynamics of hsp90 action on the glucocorticoid receptor by acetylation/deacetylation of the chaperone. J Biol Chem. 280, 33792-33799 (2005).
- Ruan, B., Fisher, K. E., Alexander, P. A., Doroshko, V., Bryan, P. N. Engineering subtilisin into a fluoride-triggered processing protease useful for one-step protein purification. Biochemistry. 43, 14539-14546 (2004).
- Meijsing, S. H., Elbi, C., Luecke, H. F., Hager, G. L., Yamamoto, K. R. The ligand binding domain controls glucocorticoid receptor dynamics independent of ligand release. Mol Cell Biol. 27, 2442-2451 (2007).
- Shen, X. C. A simple and effective sample preparation method for atomic force microscopy visualization of individual DNA molecules in situ. Mol Biol Rep. 38, 965-969 (2010).
- Paulite, M., Fakhraai, Z., Akhremitchev, B. B., Mueller, K., Walker, G. C. Assembly, tuning and use of an apertureless near field infrared microscope for protein imaging. J Vis Exp. , (2009).
- Cretu, A., Castagnino, P., Assoian, R. Studying the effects of matrix stiffness on cellular function using acrylamide-based hydrogels. J Vis Exp. , (2010).
- Brunger, A. T., Weninger, K., Bowen, M., Chu, S. Single-molecule studies of the neuronal SNARE fusion machinery. Annu Rev Biochem. 78, 903-928 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados