Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Los ensayos biofísicos investigará las propiedades mecánicas del núcleo celular Interfase: Aplicación del sustrato deformación y manipulación de Microneedle
En este artículo
Resumen
Se presentan dos independientes, basados en microscopio de herramientas para medir las deformaciones inducidas nuclear y del citoesqueleto en habitaciones individuales, que viven las células adherentes en respuesta a la aplicación de la tensión global o localizada. Estas técnicas se utilizan para determinar la rigidez nuclear (es decir, la deformabilidad) y para investigar la transmisión de fuerza intracelular entre el núcleo y el citoesqueleto.
Resumen
En la mayoría de las células eucariotas, el núcleo es el más grande del orgánulo y es de 2 a 10 veces más rígido que el citoesqueleto de los alrededores y, en consecuencia, las propiedades físicas del núcleo de contribuir de manera significativa al comportamiento general de biomecánica de las células en condiciones fisiológicas y patológicas. Por ejemplo, en la migración de neutrófilos y la invasión de las células cancerosas, rigidez nuclear puede suponer un gran obstáculo en la extravasación o paso a través de espacios estrechos dentro de los tejidos. 1 Por otro lado, el núcleo de las células en el tejido mecánicamente activos como el músculo requiere un soporte estructural suficiente para soportar la tensión mecánica repetitiva. Es importante destacar que el núcleo está estrechamente integrado en la arquitectura celular, que está conectado físicamente con el citoesqueleto de los alrededores, que es un requisito crítico para el movimiento intracelular y la posición del núcleo, por ejemplo, en células polarizadas, los núcleos sinápticas en las uniones neuromusculares o en la migración de las células. 2 No es sorprendente que las mutaciones en proteínas de la envoltura nuclear, tales como laminas y nesprins, que juegan un papel crítico en la determinación de la rigidez nuclear y el acoplamiento núcleo-citoesqueleto, se ha demostrado recientemente para dar lugar a una serie de enfermedades humanas, incluyendo Emery- La distrofia muscular Dreifuss, la integridad física, distrofia muscular, y la miocardiopatía dilatada. 3 Para investigar la función biofísica de diversas proteínas de la envoltura nuclear y el efecto de mutaciones específicas, hemos desarrollado métodos experimentales para estudiar las propiedades físicas del núcleo de las células individuales, salón sometido a la perturbación mecánica global o localizada. La medición de deformaciones inducidas nuclear en respuesta a la solicitud aplicadas precisamente la tensión del sustrato proporciona información importante sobre la deformabilidad del núcleo y permite la comparación cuantitativa entre las diferentes mutaciones o líneas celulares deficientes de proteínas específicas de membrana nuclear. Aplicación localizada cepa del citoesqueleto con una microaguja se utiliza para complementar este ensayo, y puede proporcionar información adicional sobre la transmisión de la fuerza intracelular entre el núcleo y el citoesqueleto. El estudio de la mecánica nuclear en las células vivas intactas conserva la arquitectura intracelular normal y evita posibles artefactos que pueden surgir cuando se trabaja con núcleos aislados. Además, la aplicación del sustrato cepa presenta un buen modelo para el estrés fisiológico que experimentan las células en los músculos u otros tejidos (por ejemplo, células del músculo liso vascular expuesto a la cepa del barco). Por último, si bien estas herramientas han sido desarrolladas principalmente para estudiar mecánica nuclear, también se puede aplicar a investigar la función de las proteínas del citoesqueleto y la señalización mecanotransducción.
Protocolo
1. Substrato cepa
La medición de la tensión nuclear normalizado incluye la preparación de platos de presión con membranas transparentes, elástico de silicona como la superficie de cultivo de células, las células de revestimiento sobre la vajilla, y la adquisición de imágenes de las células antes, durante y después de la aplicación cepa (uniaxial o biaxial).
Preparación de platos membrana de silicona y la adhesión de las células
- Cada plato se compone de una cepa a medida plato de fondo de plástico con un diámetro de 3 "y un anillo de plástico para mantener una membrana de silicona, que sirve como sustrato de cultivo celular. Para la preparación de los platos de la tensión, la abrazadera de 4" x 4 "pedazo de membrana de silicona entre el anillo y el plato. corte con cuidado el exceso de la membrana, enjuague con agua destilada y autoclave los platos cepa.
- Marca un punto de referencia en la parte inferior central de la membrana (por fuera) antes de aplicar la membrana de silicona con las moléculas de la matriz extracelular (por ejemplo, la fibronectina). Este punto de referencia le ayudará a identificar las células del mismo durante los experimentos de tensión. (Opcional: Para la aplicación tensión uniaxial, dos franjas paralelas de cinta adhesiva se aplican alrededor del punto de referencia para limitar la deformación de la membrana en una dimensión.)
- Para proporcionar fijación óptima de la célula, la capa de las membranas de silicona con 3 mg / ml fibronectina diluido en 10 ml de PBS o proteínas adecuadas de la matriz extracelular. Cubra el plato con la cepa de un plato de 10 cm de poliestireno invertida, e incubar las placas durante la noche a 4 ° C.
- Al día siguiente, enjuague de las membranas una vez con tampón fosfato salino (PBS) para eliminar el exceso de proteínas. Rellene plato con 10 ml de medio de crecimiento (Dulbecco modificado Eagles Medium (glucosa) suplementado con suero fetal bovino al 10% y el 1% penicilina-estreptomicina /) y dejar de lado.
- Fibroblastos de ratón embrionario se trypsinized con tripsina 0,05% y se sembraron en medio de crecimiento en la confluencia de aproximadamente 30% sobre el plato de silicona recubierto de membrana e incubar durante 24 - 48 horas bajo condiciones de cultivo normales.
Experimentos sustrato cepa
- Puesta a punto del microscopio para realizar experimentos. Los experimentos se llevan a cabo en un microscopio invertido con una cámara digital ideal para microscopía de fluorescencia, contraste de fase o de coagulación intravascular diseminada, con un objetivo de 60x y el software apropiado de adquisición de imágenes (por ejemplo, o IPLab Metamorph). Un microscopio vertical no es adecuado para esta aplicación. El dispositivo de presión consiste en una placa base que encaja en la platina del microscopio y tiene una placa central cilíndrico que sirve para aplicar presión a la sección central de la membrana de silicona, una placa móvil que sostiene el plato y la tensión que se puede deslizar hacia arriba y hacia abajo cuatro pasadores de guía, así como una placa de peso de 5 libras para aplicar una carga.
- Con el fin de visualizar los núcleos, se incuban las células en el plato de presión con 1 mg / ml de Hoechst 33342 durante 15 min a 37 ° C. Aspirar fuera del medio y reemplazarlo con 15 ml de fenol-rojo medio de crecimiento libre (Modificado rojo fenol libre Dulbecco medio Eagles (glucosa alta) con 25 mM Hepes suplementado con suero fetal bovino al 10% y 1% de penicilina / estreptomicina). Tornillo del plato tensión en la placa de soporte de plato. Aplique con cuidado la grasa (grasa de vacío Braycote 804) en el perímetro de la parte inferior de la membrana de silicona para asegurar el deslizamiento de la membrana a lo largo del centro de la platina. Asegúrese de mantener la sección central de la membrana transparente.
- Colocar la placa base sobre la platina del microscopio. Plato de montaje en soporte de la placa cuidadosamente sobre la placa base. Asegurar que en la posición de reposo inicial, la membrana de silicona de la placa de tensión libremente se basa en la placa central.
- En primer lugar, se centran en la parte inferior de la membrana de silicona y encontrar el punto de referencia central negro. El punto será el punto de partida para todas las adquisiciones de imágenes y ayuda en la localización de las mismas células durante y después del estiramiento. Nosotros usamos un programa personalizado escrito de imágenes automatizado para almacenar las posiciones de las células y para reubicar estas células durante el experimento, pero también se puede lograr de forma manual.
- A partir del punto, ajustar el enfoque para visualizar las células y la parte superior de la membrana de silicona. Ubicar bien extendidas las células con núcleo situado en el centro y adquirir un contraste de fase y una imagen de fluorescencia de la nuclear Hoechst mancha. La imagen de contraste de fase debe centrarse en el contorno celular y la membrana de silicona, mientras que las imágenes de fluorescencia debe centrarse en el plano central del núcleo.
- Después de la adquisición de imágenes de 5 a 15 células, volver al punto central. Aplique lentamente el peso hasta el plato cepa, resulte de la aplicación uniforme de la tensión en el centro del plato. La máxima tensión aplicada sustrato está limitada por espaciadores de nylon colocada en los pasadores de alineación vertical (pines guía).
- Se centran en la parte inferior de la membrana de silicona y localizar el punto de referencia una vez más. A partir de lapunto, la posición de la misma celda y otra vez adquirir un contraste de fase y una imagen de fluorescencia de las células y los núcleos bajo presión completa, tratando de asemejarse a los planos focales de la imagen inicial. Este proceso no debe exceder de 10 minutos para evitar la remodelación y adaptación activa de la célula al sustrato tensas.
- Después de todas las imágenes correspondientes se han adquirido, mover la platina del microscopio de nuevo al punto de partida. Retire con cuidado el peso de la placa de soporte de antena y permitir que la membrana de silicona para relajarse. Si es necesario, empuje suavemente el plato hasta que la tensión está en la posición inicial. Luego adquirir las imágenes de contraste de fases y fluorescencia de las células después de la tensión como se describió anteriormente para las imágenes de la tensión.
Análisis
- Imágenes de las células y los núcleos marcados con fluorescencia, antes, durante y después de la aplicación cepa se analizan para calcular la tensión nuclear normalizado. En nuestro laboratorio, usamos un script escrito MATLAB para el análisis, sino varias opciones alternativas están disponibles. El análisis se realiza en tres pasos.
- En primer lugar, para calcular la tensión del sustrato aplicado, las posiciones de 3 a 6 puntos de control situados en la membrana de forma manual compensada entre las correspondientes pre-completo, y la tensión post-imágenes. El programa MATLAB a continuación, calcula la tensión de membrana aplicada mediante la comparación de las posiciones de juego los puntos de control entre las imágenes pre-tensión y la tensión total, y también la tensión residual entre la tensión pre-y las imágenes de la tensión post-. Al mismo tiempo, los puntos de control se utilizan para registrar los pares de imágenes, lo que ayudará a detectar células dañadas o separar (ver Figura 1).
- En un segundo paso, los núcleos se seleccionan manualmente utilizando un programa separado de MATLAB que calcula la tensión nuclear para cada núcleo individual, haciendo coincidir el tamaño ya sea nuclear o marcadores intranucleares entre correspondiente previo, completo, y la tensión post-imágenes fluorescentes. Para tener en cuenta las pequeñas variaciones en la tensión aplicada entre la membrana experimentos diferentes, expresar los resultados como cepa normalizada nuclear, que se define como el cociente de la tensión inducida nuclear a la tensión aplicada la membrana que se calcula para cada núcleo. Las secuencias de comandos de MATLAB están disponibles en el laboratorio Lammerding bajo petición.
- Finalmente, cada núcleo se valida, con exclusión de las mediciones de las células que se desprenden o se dañan durante la aplicación de la cepa (Figura 1).
2. Microagujas ensayo de la manipulación
Preparación de los platos, las células adherentes, y microagujas
- Incubar 35 mm con fondo de cristal placas de cultivo celular con una baja concentración de fibronectina (0.5ug/ml) en solución salina de Hank salina tamponada (HBSS) o proteínas adecuadas de la matriz extracelular durante 2 horas a 37 º C. Lave los platos con HBSS dos veces y se añaden 2 ml de medio de cultivo en el plato antes de proceder al siguiente paso.
- Fibroblastos de ratón embrionario se trypsinized con tripsina 0,05% y se sembró en 2 ml de medio de cultivo sembrados a 7,5 x 10 4 células / ml en los platos de fondo de vidrio recubiertos de fibronectina. Vuelva a colocar las células en la incubadora durante la noche. Vuelva a colocar las células en la incubadora durante la noche. Se debe optimizar el número de células para obtener una sola, adherente, no confluente células de otros tipos de células.
- Tire de las microagujas, hechos de borosilicato capilares, a punta de un diámetro de aproximadamente 1 a 3 micras, con un extractor de pipeta comerciales (por ejemplo, la compañía de Sutter Instrument).
Microagujas experimento de manipulación
- Al día siguiente, se incuban las células con tinción MitoTracker mitocondrial (600 M; Invitrogen) y Hoechst 33342 tinción nuclear (1 mg / mL) añadido al medio de cultivo durante 30 minutos en una incubadora a 37 ° C.
- Lave las células una vez en HBSS durante 5 minutos a temperatura ambiente y luego agregar rojo fenol medio de cultivo libre de las células para obtener imágenes.
- Adquirir una imagen de la célula sin la microaguja se inserta en el citoesqueleto de contraste de fases, una imagen fluorescente de la imagen de Hoechst 33342 tinción fluorescente y una de las mitocondrias mancha, con un objetivo de 60x (NA 0.70, Plan-acromático) en una invertida microscopio con una cámara digital, dispositivo de carga acoplada.
- El uso de un micromanipulador (por ejemplo, InjectMan NI 2, Eppendorf), inserte con cuidado el microagujas en el citoplasma de una célula a una distancia fija (normalmente 5 m) de distancia de la periferia nuclear y tomar una imagen de contraste de fase, una imagen fluorescente de la Hoechst 33342 mancha y una imagen de fluorescencia de la mancha mitocondrial. Para esta aplicación, ayuda a controlar el micromanipulador a través de un ordenador, por ejemplo, Windows Hyperterminal, para lograr procedimientos uniformes de micromanipulación.
- Mueva la microaguja, a una distancia específica (normalmente 10 o 20 micras) hacia la periferia de la célula en un m / seg y al mismo tiempo la recopilación de imágenes de fluorescencia y contraste de fase cada 10segundos. A medida que la microaguja se está moviendo a una distancia específica hacia la periferia de la célula. Con los parámetros elegidos, como la microaguja se está moviendo a una distancia específica hacia la periferia de la célula, esto corresponde a 3.2 cuadros durante el proceso de manipulación.
- Finalmente adquirir imágenes adicionales después de la microaguja se retira del citoesqueleto.
Análisis
- Mapas de desplazamiento se calculan usando un script escrito MATLAB basado en funciones de seguimiento de la etiqueta fluorescente del núcleo y el citoplasma. (La secuencia de comandos de MATLAB está disponible en el laboratorio Lammerding bajo petición). El programa utiliza un normalizado de correlación cruzada entre las regiones algoritmo de imagen pequeña (aproximadamente 10 m x 10 m de tamaño y espacio 5 micras de diferencia) en los marcos de imagen posterior. Para cada centro de la región, el desplazamiento en el x-e y direcciones, se calcula como el cambio entre la ubicación original y la posición recientemente identificados y se muestra como un vector de desplazamiento y también se almacenan como valores numéricos. De los mapas de desplazamiento, el desplazamiento promedio dentro de las regiones predefinidas se pueden calcular. Tenga en cuenta que los desplazamientos del citoesqueleto se basan en el canal de fluorescencia de los marcadores del citoesqueleto (por ejemplo, MitoTracker mitocondrial mancha), mientras que los desplazamientos nuclear se calculan a partir del canal de fluorescencia correspondiente a la señal de Hoechst 33342. Para nuestra aplicación, de manera rutinaria examinar las siguientes regiones: (i) la tensión del citoesqueleto en el sitio de aplicación cepa, es decir, el sitio de inserción de microagujas, (ii) la tensión nuclear en una región dentro del núcleo hacia el lugar de aplicación cepa, (iii) la energía nuclear la tensión en una región nuclear fuera de la zona de aplicación, y (iv) la tensión del citoesqueleto en una región citoplásmica a través del núcleo. Además, también se puede medir directamente la elongación nuclear de contraste de fase o Hoechst 33342 secuencias de imágenes de fluorescencia. En este caso, aplicar la tensión nuclear se calcula dividiendo la elongación nuclear (ΔL = L - L 0) por la longitud inicial, L 0, donde L es la longitud final del núcleo al final de la aplicación de la tensión y L 0 es la longitud inicial del núcleo. De células con un núcleo-intacta citoesqueleto de acoplamiento, el núcleo se alarga hacia el lugar de aplicación cepa. Por el contrario, en las células en las que se interrumpe núcleo-citoesqueleto de acoplamiento, es decir, las fuerzas se transmiten menos eficiente entre el citoesqueleto y el núcleo, el núcleo se espera que se alargan mucho menos en la dirección del sitio de aplicación cepa. Por lo tanto, la disminución de las deformaciones nucleares en respuesta a la solicitud cepa del citoesqueleto implica un desacoplamiento (parcial) entre el núcleo y el citoesqueleto.
3. Los resultados representativos:
Substrato cepa
Hemos adquirido las imágenes antes, durante y después de la aplicación de la tensión fibroblastos embrionarios de ratón a partir de heterocigotos y homocigotos lamina A / C-deficiente (LMNA + / - y LMNA - / -), y de tipo salvaje (LMNA + / +) y, posteriormente, los ratones calcula la tensión normalizada nuclear para cada celda. Tras el análisis, los núcleos son validados y las células que se dañan o se retraen durante la aplicación de la tensión son excluidos del análisis. La figura 1A muestra los núcleos de las tres células que son válidos, mientras que la Figura 1B muestra las células que deben ser excluidos del análisis. Normalizado de datos tensión nuclear se reúnen por lo menos tres experimentos independientes (cada uno con las mediciones de los núcleos de ~ 5-10) y en comparación con otras células o grupos de tratamiento mediante el análisis estadístico. Aumento de la tensión nuclear normalizado indica menor rigidez nuclear, como se ve en las células con una menor expresión de las proteínas de envoltura nuclear lamina A / C (Figura 2).
Microagujas ensayo de la manipulación
Para la prueba de la manipulación de microagujas, que imágenes desplazamientos nucleares y del citoesqueleto durante la aplicación localizada cepa del citoesqueleto. Las células que se dañan o separados son excluidos del análisis. Para el análisis, podemos medir la magnitud de los movimientos nucleares y del citoesqueleto hacia el lugar de aplicación de la fuerza en las células individuales, adherente. Por ejemplo, en la figura 3, hacemos un seguimiento de las mitocondrias (marcador para el citoesqueleto) desplazamientos antes y después de un esfuerzo del citoesqueleto y luego trazar los desplazamientos como vectores. Cada vector representa el desplazamiento calcula como el cambio entre la ubicación original y la posición recientemente identificados. Las regiones con baja intensidad de la imagen o textura insuficiente (por ejemplo, las regiones fuera de la célula) se excluyeron del análisis. Los desplazamientos del citoesqueleto y nucleares son entonces cuantificado en áreas selectas a distancias cada vez mayores de la zona de aplicación cepa (Figura 4, las áreas correspondientes a los colores boequis en el recuadro). En fibroblastos de embriones de ratón con el citoesqueleto intacto núcleo-acoplamiento, las fuerzas se transmiten a través de las células de todo, lo que indujo deformaciones nucleares y del citoesqueleto que poco a poco se disipan fuera de la zona de aplicación cepa (Figura 4). Por el contrario, los fibroblastos con trastornos núcleo-citoesqueleto de acoplamiento (o la organización del citoesqueleto alterado) muestran desplazamientos localizados cerca del lugar de aplicación, como se muestra en la Figura 4 y sólo deformaciones inducidas por poco más lejos. Aplicación comparables cepa del citoesqueleto en el sitio de inserción de microagujas (recuadro naranja) se observa tanto para los fibroblastos control (mCherry solos) y los fibroblastos con un núcleo-interrumpió el citoesqueleto de acoplamiento (DN KASH). Sin embargo, los desplazamientos inducidos nuclear y del citoesqueleto (azul, amarillo, rojo y cajas) en otras regiones fueron significativamente menores en los fibroblastos con núcleo-interrumpió citoesqueleto de acoplamiento (DN KASH) que en las células control (mCherry solo) (Figura 4). Por lo tanto, la disminución de los desplazamientos del citoesqueleto y nucleares lejos de la zona de aplicación cepa, indica que la transmisión de fuerza entre el citoesqueleto y el núcleo estaba perturbado.
Es importante destacar que también hemos validado que las mitocondrias son marcadores citoesqueleto adecuada, mediante la realización de la manipulación de microagujas en fibroblastos embrionarios de ratón transfectadas con GFP-actina o mCherry y las buenas prácticas agrarias vimentina y marcados con fluorescencia con Mitotracker verde o rojo. Mapas del citoesqueleto de desplazamiento se calculan de forma independiente de la señal fluorescente de la mitocondria y el citoesqueleto de actina y vimentina. El desplazamiento absoluto promedio se calculó para cuatro regiones distintas del citoesqueleto a distancias cada vez mayores fuera de la zona de aplicación cepa. Los valores de pendiente y R-cuadrado se calcula a partir de la regresión lineal entre las medidas obtenidas de las mitocondrias y de la actina y vimentina, respectivamente. Para la actina, la pendiente fue de 0,99 y el valor de R 2 fue 0.986, para la vimentina, la pendiente fue de 1,04 y el valor de R2 fue 0,971, lo que confirma que los desplazamientos de las mitocondrias sirven como indicadores fiables de las deformaciones del citoesqueleto.
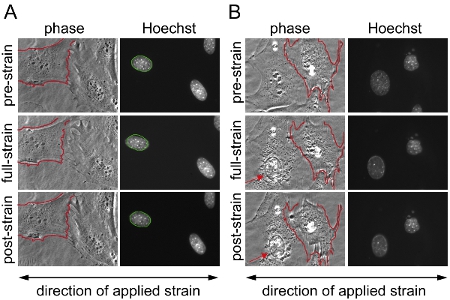
Figura 1. Aplicación sustrato tensión en fibroblastos embrionarios de ratón (MEFs). Fibroblastos embrionarios de ratón en dos ámbitos distintos en la membrana de silicona son imágenes con contraste de fases y microscopía de fluorescencia antes, durante y después de la aplicación del 20% de deformación uniaxial. (A) Ejemplo de un experimento exitoso con núcleos válidos a partir de células que sobrevivieron a la aplicación de la tensión sin ningún tipo de daño o desprendimiento y (B) ejemplo de las células que se retraen / parcial de la retina durante la aplicación de la tensión, los resultados de las células se muestra en (B) están excluidos del análisis. En (B), la célula en el lado izquierdo muestra signos de daño del citoesqueleto y el colapso nuclear (flecha), mientras que la célula en el lado derecho se desprende en parte y se retrae durante la aplicación de la tensión. Esto puede ser una indicación de la aplicación excesiva tensión. Para una mejor comparación, en (A) y (B) de la frontera de una de las membranas celulares sin estirar se resume en rojo y se superponen en la misma celda durante y después de la aplicación cepa. En (A) de la frontera del núcleo sin estirar se describe en verde y se superponen en el mismo núcleo durante y después de la aplicación cepa.
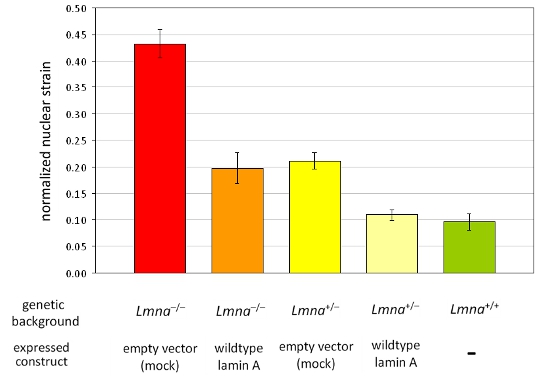
Figura 2. Análisis de la tensión normalizada nuclear en un panel de diferentes líneas de células MEF MEFs del LMNA -. / - Y LMNA + / - los antecedentes genéticos ectópica manifieste un vector vacío o lamina de tipo salvaje A se analizaron. En comparación con MEFs de tipo salvaje littermates (LMNA + / +), la pérdida de la lamina A los resultados / C expresión en la rigidez diminuyen nuclear que puede ser totalmente restaurado por la reintroducción de tipo salvaje lamina A. En particular, la reducción de la rigidez nuclear se refleja en aumento de los valores de la tensión nuclear normalizado. Las barras de error representan los errores típicos.

Figura 3. Ensayo de la manipulación de microagujas para medir la transmisión de la fuerza intracelular. Contraste de fases (A, B) y fluorescencia (C, D) las imágenes de un fibroblasto etiquetados con tinción nuclear (azul) y MitoTracker mitocondrial mancha (verde). A microagujas se insertó en el citoesqueleto a una determinada distancia del núcleo (A y C) y, posteriormente, se dirigió hacia la periferia de la célula (B, D). Desplazamientos del citoesqueleto y nucleares fueron cuantificados por el núcleo de seguimiento de la etiqueta fluorescente y las mitocondrias con una custom-written algoritmo de correlación cruzada. (E) del mapa de desplazamiento del citoesqueleto final (verde) deformaciones calculadas a partir de series de imágenes de fluorescencia, longitud de la flecha se multiplica por 2 para una mejor visibidad. Las barras de escala, 10 micras.
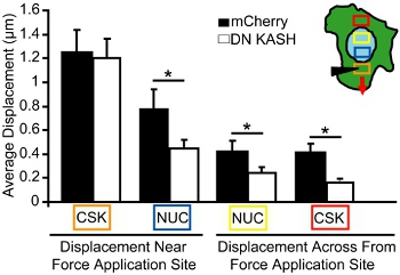
Figura 4. Análisis de la transmisión de la fuerza intracelular durante la manipulación de microagujas. Inducida por desplazamientos del citoesqueleto y nucleares durante la manipulación de microagujas, medida en las áreas correspondientes a las casillas de color (el recuadro en la A). El cuadro naranja es el lugar de aplicación cepa. A pesar de la aplicación cepa similar en el citoesqueleto (recuadro naranja), inducida por el desplazamiento nuclear y del citoesqueleto (azul, amarillo, rojo y cajas) fueron significativamente menores en los fibroblastos de embriones de ratón que con un núcleo-interrumpió el citoesqueleto de acoplamiento (DN KASH) en comparación con el control ( mCherry solo) las células.
Access restricted. Please log in or start a trial to view this content.
Discusión
Sustrato de ensayo de tensión
Aplicación de la tensión se ha utilizado con éxito por nosotros y otros grupos para estudiar las deformaciones inducidas nuclear en las células sometidas a esfuerzos mecánicos y para investigar la contribución de determinadas proteínas de la envoltura nuclear a la rigidez nuclear. 4.8 La ventaja de esta técnica es que las sondas de las propiedades mecánicas de vida de los núcleos de su entorno celular normal y el citoesqueleto y que la aplicac...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue financiado por los Institutos Nacionales de Salud (R01 HL082792 y NS059348 R01) y el Hospital Brigham and Cardiovascular Premio de Liderazgo Femenino Grupo.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| Fibronectina | Millipore | FC010 | |
| MitoTracker Red FM y FM Verde | Invitrogen | M22425 y M-7514 | |
| Hoechst 33342 | Invitrogen | H3570 | |
| Buffered Saline sal de Hank | Invitrogen | 14185 | |
| Fenol libre, DMEM | Invitrogen | 21063 | |
| De suero fetal bovino | Aleken Biológicos | FBSS500 | |
| Penicilina / estreptomicina | Sigma | P0781-100ML | |
| Vidrio borosilicato con filamento | Sutter Instrumento | BF100-78-10 | |
| Brillo / brillo no reforzado láminas de silicona, 0.005 " | Especialidad Manufacturing Inc. | ||
| Fosfato de Dulbecco solución salina tamponada | Invitrogen | 14200 | |
| 35 mm de vidrio platos fondo la cultura (FluoroDish) | World Precision Instruments, INC | FD35-100 | |
| Braycote 804 Grasa de vacío | SPI Supplies | 05133A-AB |
Referencias
- Friedl, P., Wolf, K., Lammerding, J. Nuclear mechanics during cell migration. Curr Opin Cell Biol. 23, 55-64 (2011).
- Mejat, A., Misteli, T. LINC complexes in health and disease. Nucleus. 1, 40-52 (2010).
- Worman, H. J., Fong, L. G., Muchir, A., Young, S. G. Laminopathies and the long strange trip from basic cell biology to therapy. J Clin Invest. 119, 1825-1836 (2009).
- Caille, N., Tardy, Y., Meister, J. J. Assessment of strain field in endothelial cells subjected to uniaxial deformation of their substrate. Ann Biomed Eng. 26, 409-416 (1998).
- Lammerding, J. Lamins A and C but not lamin B1 regulate nuclear mechanics. J Biol Chem. 281, 25768-25780 (2006).
- Lammerding, J. Abnormal nuclear shape and impaired mechanotransduction in emerin-deficient cells. J Cell Biol. 170, 781-791 (2005).
- Lammerding, J. Lamin A/C deficiency causes defective nuclear mechanics and mechanotransduction. J Clin Invest. 113, 370-378 (2004).
- Verstraeten, V. L., Ji, J. Y., Cummings, K. S., Lee, R. T., Lammerding, J. Increased mechanosensitivity and nuclear stiffness in Hutchinson-Gilford progeria cells: effects of farnesyltransferase inhibitors. Aging Cell. 7, 383-393 (2008).
- Brooks, S. V., Zerba, E., Faulkner, J. A. Injury to muscle fibres after single stretches of passive and maximally stimulated muscles in mice. J Physiol. 488, 459-469 (1995).
- Maniotis, A. J., Chen, C. S., Ingber, D. E. Demonstration of mechanical connections between integrins, cytoskeletal filaments, and nucleoplasm that stabilize nuclear structure. Proc Natl Acad Sci. 94, 849-854 (1997).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados