Method Article
Un día de flujo de trabajo Método de detección de patógenos bacterianos y pruebas de resistencia a los antimicrobianos de cultivos de sangre
En este artículo
Resumen
El diseño de un esquema de flujo de trabajo sencillo de un día para el diagnóstico de patógenos bacterianos permite el reconocimiento rápido de infecciones del torrente sanguíneo. La inclusión de los ocho objetivos bacterianos clínicamente relevantes y sus perfiles de resistencia a los antibióticos ofrece al clínico una visión inicial en el mismo día, que puede conducir a una terapia más adecuada.
Resumen
Infecciones del torrente sanguíneo se asocia con altas tasas de mortalidad a causa de la manifestación probable de sepsis, sepsis grave y shock séptico 1. Por lo tanto, la administración rápida de la terapia antibiótica adecuada es de importancia principal en el tratamiento de infecciones del torrente sanguíneo. El elemento crítico en este proceso es el tiempo, depende en gran medida los resultados de la identificación de bacterias y pruebas de susceptibilidad a los antibióticos. Ambos de estos parámetros son rutinariamente obtenida por cultivo basado en pruebas, que consume tiempo y necesita una media de 24-48 horas 2, 4. El objetivo del estudio fue desarrollar ensayos basados en el ADN para la identificación rápida de infecciones del torrente sanguíneo, así como pruebas rápidas de susceptibilidad antimicrobiana. El primer ensayo es un 16S rDNA eubacterial real basado en PCR en tiempo complementadas con especies del género-o sondas específicas 5. El uso de estas sondas, bacterias Gram-negativas, incluyendo Pseudomonas spp., Pseudomonas aeruginosay Escherichia coli, así como las bacterias Gram-positivas como Staphylococcus spp., Staphylococcus aureus, Enterococcus spp., Streptococcus spp. y Streptococcus pneumoniae pueden ser distinguidos. El uso de este ensayo de Multiprobe, una primera identificación del microorganismo causal se da después de 2 horas.
En segundo lugar, hemos desarrollado un ensayo molecular para el semi-pruebas de sensibilidad antibiótica de S. aureus, Enterococcus spp. y (facultativo) aerobios bacilos Gram-negativos 6. Este ensayo se basa en un estudio en el que la PCR se utilizó para medir el crecimiento de bacterias 7. Las bacterias recolectadas directamente de los cultivos de sangre se incubaron durante 6 horas con una selección de los antibióticos, y después de un SYBR Green-tiempo real basado en el ensayo de PCR determina la inhibición del crecimiento. La combinación de estos dos métodos podría dirigir la elección de una terapia antibiótica apropiada, en el mismo día (Figura 1). En conclusión, El análisis molecular de ambos identificación y susceptibilidad a los antibióticos ofrece una alternativa más rápida para la detección de patógenos, y podría mejorar el diagnóstico de infecciones del torrente sanguíneo.
Protocolo
PARTE I: la identificación de patógenos
1. Preparación de la muestra
Nota: El flujo de trabajo molecular que se describe en el siguiente protocolo se debe realizar de acuerdo a las recomendaciones para garantizar la calidad en el diagnóstico molecular 3.
- Añadir una alícuota de 0,1 ml de cultivo de sangre a 0,9 ml de NaCl al 0,9% en un tubo de 1,5 ml de reacción para convertirse en una muestra diluida 1:10. (Diluir con el fin de prevenir la inhibición qPCR).
- Centrifugar la muestra a 13400 xg durante 5 minutos para sedimentar el ADN bacteriano.
- Resuspender el sedimento bacteriano en 100 l estéril desmineralizada H 2 O.
- Almacenar la muestra de ADN a 4 ° C hasta su uso posterior.
2. Ensayo de identificación: en tiempo real PCR 16S rDNA
- Preparar las mezclas de reacción de la siguiente manera. El ensayo consiste en cuatro reacciones separadas por muestra. Cada mezcla incluye 12,50 maestro de lmezcla, 0,9 mM cebador (5-TCCTACGGGAGGCAGCAGT-3) 7, cebador 0,6 mM inversa (5-GGACTACCAGGGTATCTAATCCTGTT-3) 8 y un panel de sondas. La cantidad de sondas se da para cada una de las cuatro reacciones separadas en adelante.
- La primera reacción incluye:
- 0,2 mM universal de la sonda (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1) 8
- 0,2 mM p aeruginosa sonda (5-JOE-CCAAAACTACTGAGCTAGAGTACG-3-BHQ1)
- La segunda reacción incluye:
- 0,2 mM E. la sonda coli (5-JOE-GGAGTAAAGTTAATACCTTTGCTCATT-3-BHQ1)
- 0,2 mM de Pseudomonas spp. de la sonda (5-NED-CCTTCCTCCCAACTTAAAGTGCTT-3-MGBNFQ)
- La tercera reacción incluye:
- 0,2 mM Staphylococcus spp. de la sonda (5-NED-AATCTTCCGCAATGGGCGAAAGC-3-MGBNFQ)
- 0,2 mM de S. la sonda aureus (5-FAM-AGATGTGCACAGTTACTTACACATAT-3-BHQ1)
- 0,2 mM de Enterococcus spp. (5-JOE-TCCTTGTTCTTCTCTAACAACAGAG-3-BHQ1)
- La reacción cuarto incluye:
- 0,2 mM universal de la sonda (5-FAM-CGTATTACCGCGGCTGCTGGCAC-3-BHQ1)
- 0,3 mM Streptococcus spp. de la sonda (5-NED-CCAGAAAGGGACSGCTAACT-3-MGBNFQ)
- 0,2 mM de S. pneumoniae de la sonda (5-JOE-CCAAAGCCTACTATGGTTAAGCCA-3-BHQ1)
- Añadir desmineralizada estéril, H 2 O para llegar a un volumen total de 20 l. Añadir 20 l de cada mezcla de reacción a los pocillos de una placa de 96 pocillos de PCR.
- Añadir 5 l de muestra a cada pocillo.
- Utilizar una película adhesiva para sellar la placa 96-así PCR.
- Ejecutar la placa en el ABI PRISM 7900HT Sistema de Tiempo Real PCR utilizando los siguientes ciclos térmicos condiciones óptimas:
- Pre-calentamiento a 50 ° C durante 10 min
- Desnaturalización a 95 ° C durante 15 minutos
- 42 ciclos de
- Desnaturalización a 95 ° C durante 15 s
- El recocido a 60 ° C durante 1 minuto
3. Análisis de los resultados
Ajuste el umbral del análisis de Ct a 0,1 en la pestaña Configuración de análisis. Limitar las configuraciones de referencia para empezar (Ciclo): 6 y el fin (del ciclo): 15.
- Registre el ciclo umbral (Ct), el valor de todas las muestras. El valor de corte para considerar un resultado positivo por PCR como se puede ajustar a un valor de CT-35. La cantidad de bacterias presentes en los cultivos de sangre osciló entre 07 al 10 10 11 UFC / ml, la generación de Ct valores por debajo de 35.
PARTE II: pruebas de susceptibilidad antimicrobiana
4. El aislamiento de bacterias de cultivos de sangre positivos 9
- Aspirar 5 ml de caldo de una botella de hemocultivo positivo y la transfiere en un tubo separador de suero.
- Centrifugar el tubo separador de suero a 2000 xg durante 10 min.
- Se desecha el sobrenadante del tubo separador de suero.
- Transferencia de bacbacterias de la capa de gel del tubo con una torunda de algodón estéril en solución salina al 0,9% hasta un 0,5 McFarland suspensión estándar se obtiene.
5. La inoculación de las placas Micro Titre
- Diluir la suspensión 0,5 McFarland en doble concentrado caldo Mueller Hinton II para formar una suspensión de 5 x 10 5 UFC / ml.
- Añadir esta suspensión a los pocillos de una placa de micro título que contiene una selección de antibióticos (Tabla 1).
- Se incuba la placa micro título a 37 ° C durante 6 h.
- Almacenar una alícuota de la suspensión a 4 ° C (como testigo de crecimiento negativo).
- Después de 6 horas de incubación, transferir el contenido de cada pocillo en un tubo estéril, así como la muestra de control de crecimiento negativo que se almacenó a 4 ° C.
- Centrifugar los tubos a 16000 xg durante 5 min.
- Retire con cuidado el sobrenadante, sin perturbar el sedimento bacteriano.
- Resuspender el precipitado en H desmineralizada estéril 2 O.
- Diluir las muestras de 10 veces en estériles desmineralizada H 2 O.
6. En tiempo real de PCR del rDNA 16S 10
- Preparar la mezcla de PCR como sigue:
- 12,50 l iQ SYBR Green Supermix
- 0,5 mM cebador 16S-1 (5-TGGAGAGTTTGATCCTGGCTCAG-3) 11
- 0.25 16S-2 micras de imprimación inversa (5-TACCGCGGCTGCTGGCAC-3) 11
- estéril desmineralizada H 2 O hasta un volumen total de 20 l
- Añadir 20 l de mezcla de PCR a los pocillos de una placa de 96 pocillos de PCR.
- Añadir 5 l de muestra a cada pocillo.
- Utilizar una película adhesiva para sellar la placa 96-así PCR.
- Ejecutar la placa en el MyiQ un solo color en tiempo real del sistema de detección por PCR, utilizando las siguientes condiciones óptimas ciclos térmicos:
- Desnaturalización a 95 ° C durante 4 minutos
- Inicial recocido a 65 ° C durante 30 s
- 35 ciclos de
- Desnaturalización a 95 ° C durante 15 s
- El recocido a 60 ° C durante 1 minuto
- Fundir análisis de la curva (60 a 95 ° C en 20 minutos con incrementos de 0,57 ° C)
7. Análisis de los resultados
- Calcular el valor de corte de Ct utilizando una de las fórmulas siguientes (dependiendo del tipo de antibiótico).
- En general:
De corte el valor Ct = Ct de control de valor de un crecimiento positivo + 0,5 x (control de Ct valor de crecimiento negativo - el control de Ct valor de crecimiento positivo) - Piperacilina, piperacilina / tazobactam y ceftazidima en bacilos Gram-negativos, amoxicilina, oxacilina y trimetoprim / sulfametoxazol en el S. aureus, Enterococcus spp amoxicilina en:. De corte Ct = valor de control de valor de Ct crecimiento positivo + 0,25 x (crecimiento Ct valor negativo - Control Ct valor de crecimiento positivo)
- En general:
- Use la muestra se incuba con agua estéril desmineralizada H 2 O, como testigo de crecimiento positivo.
- Dependiendo del microorganismo, utiliza el control apropiado de crecimiento negativo:
- Gram-negativos Muestra varillas se incubaron con la mezcla de antibióticos
- Enterococcus spp. Muestra almacenada a 4 ° C
- Ejemplo de S. aureus se almacena a 4 ° C
- Determinar la susceptibilidad (S) o resistencia (R) de la cepa para el antibiótico ensayó como sigue:
- Un valor Ct superior al valor de corte de Ct indica la susceptibilidad
- Un valor de Ct inferior al valor de corte de Ct indica que la resistencia
8. Los resultados representativos
Dos organismos modelo, es decir, una E. Gram-negativos coli y S. una Gram-positivas aureus, se eligen para visualizar el procedimiento combinado para la detección e identificación de bacterias patógenas y la determinación de su perfil antimicrobiano. La primera parte del protocolo comprende la identificación de patógenos. Spesondas específicas están diseñadas para la detección de ocho microorganismos clínicamente relevantes. En presencia de un objetivo incluido en el panel bacteriana, curvas de amplificación se generan y Ct valores se calculan (Figura 2). El valor de corte para considerar un resultado positivo por PCR como se establece en un valor de Ct de 35 años. En la Figura 2A, el perfil de identificación de un cultivo de sangre infectada con E. coli se muestra. La sonda 16S universal está incluida en dos mezclas de reacción separadas y, en consecuencia genera dos curvas de amplificación (Ct de 25,20 y 25,95). La tercera señal se deriva de la sonda específica para E. E. (Ct de 27.04). La identificación de una S. aureus infectada cultivo de sangre se muestra en la Figura 2B. La sonda 16S universal tiene señales de amplificación de 33,35 y 33,71. Las dos señales restantes se derivan de las sondas específicas para Staphylococcus spp. y S. aureus (Ct de 32,48 y 30,59).
Después de la primera parte del protocolo, el microorganismo causal es conocido y el perfil de los antimicrobianos se puede determinar. La figura 3 es un ejemplo de un gráfico de amplificación pruebas de susceptibilidad a los antibióticos, en representación de la cepa de E. coli que se mostró también en la Figura 2A. Cada línea representa un antibiótico que la muestra bacteriana se incubó con. Una muestra con un bajo valor de CT es una muestra en la que se ha producido crecimiento en presencia de una resistencia a los antibióticos, lo que indica que el antibiótico probada. Por el contrario, un alto valor Ct representa una muestra en la que no hay crecimiento se ha producido por el funcionamiento efectivo de la susceptibilidad a los antibióticos, lo que indica que el antibiótico probada. La Tabla 1 ilustra la determinación del perfil de los antimicrobianos de la E. coli y S. aureus aislados. Todos los valores de Ct se informan y, utilizando las fórmulas mencionadas en el texto del protocolo (7,1), dos de corte los valores de Ct se calcularoncionado a distinguir entre la resistencia y susceptibilidad. La cepa es resistente a los antibióticos, si el valor de Ct informado es más bajo que el calculado valor de corte de Ct (y viceversa).
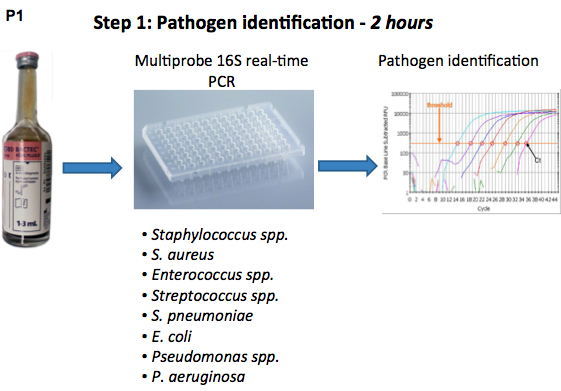
Figura 1. Diagrama de flujo del procedimiento de identificación de patógenos y la susceptibilidad a los antibióticos mediante pruebas en tiempo real PCR del rDNA 16S.
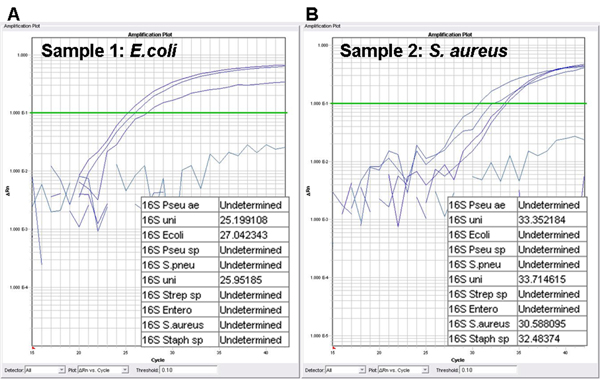
Figura 2 Ensayo de identificación:. Parcelas de amplificación y los valores de ciclo umbral (Ct valores) Un cultivo de sangre positivo se detecta por lo universal de la sonda de ADNr 16S, mientras que las sondas específicas se utilizan para la identificación del agente causal.. Parcela A. amplificación de cultivo de sangre que contiene E. coli; parcela B. amplificación de la cultura de la sangre que contiene S. aureus, Pseudomonas ae, Pseudomoncomo P. aeruginosa, uni, 16S universal de la sonda; Ecoli, Escherichia coli de la sonda, Pseudomonas sp, Pseudomonas spp. la sonda, S. pneumoniae, Streptococcus pneumoniae sonda; Strep sp, Streptococcus spp. la sonda, Entero, Enterococcus spp. la sonda, S. aureus, la sonda de Staphylococcus aureus, estafilococo sp, Staphylococcus spp. sonda.
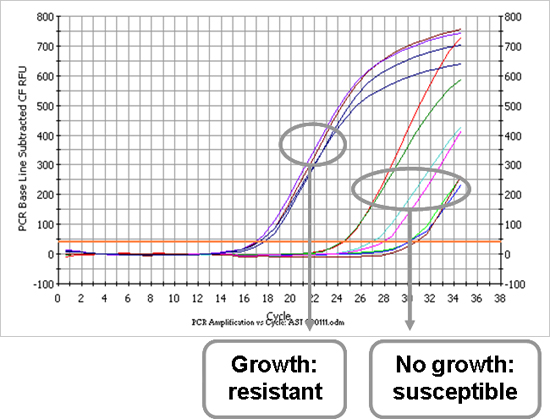
Figura 3. Amplificación parcela de las pruebas de sensibilidad antibiótica de una E. E. aislar (muestra 1). Cada curva representa un antibiótico que la cepa se incubó con. Una señal temprana es causada por una alta carga bacteriana, lo que significa que la cepa ha crecido en presencia del antibiótico probado y es por lo tanto resistentes al antibiótico. Señales tardías indican que la cepa no ha crecido en presencia del antibiótico, en otras palabras, es susceptible.
| Ejemplo 1: E. E. | Ejemplo 2: S. Staphylococcus | ||||
| AST | Ct | R / S | AST | Ct | R / S |
| La amoxicilina 8 mg / l | 16,83 | R | La amoxicilina 0,25 mg / l | 21,03 | R |
| La amoxicilina-ácido clavulánico 4.8 mg / L | 17,36 | R | Oxacilina 2 mg / l | 25,80 | S |
| Piperacilina 16 mg / l | 16,67 | R | La vancomicina 2 mg / l | 25,20 | S |
| Piperacilina-tazobactam 16 /4 mg / l | 24,15 | S | La gentamicina 4 mg / l | 25,86 | S |
| La ciprofloxacina 1 mg / l | 29,72 | S | Trimetoprim-sulfametoxazol 2/38 mg / L | 24,62 | S |
| La ceftazidima 1 mg / l | 24,03 | S | |||
| La ceftazidima 8 mg / l | 26,58 | S | |||
| La gentamicina 4 mg / l | 29,83 | S | |||
| Trimetoprim-sulfametoxazol 2/38 mg / L | 27,60 | S | |||
| Control de crecimiento negativo (mezcla de antibióticos) | 30,41 | Testigo de crecimiento negativo (muestra almacenada a 4 ° C) | 27,42 | ||
| Control de crecimiento positivo | 16,90 | Control de crecimiento positivo | 20,22 | ||
| De corte CT-valor de 1 * | 21,76 | De corte CT-valor 1 *** | 23,82 | ||
| De corte CT-valor 2 ** | 18,75 | De corte CT-valor 2 **** | 22,02 | ||
| * Para la amoxicilina, amoxicilina-clavulánico, ciprofloxacino, gentamicen, trimetoprim-sulfametoxazol ** Para piperacilina, piperacilina-tazobactam, ceftazidime | *** Para vancomicina y gentamicina **** Para amoxicilina, oxacilina y trimetoprim-sulfametoxazol |
Tabla 1. Determinación de las pruebas de sensibilidad antibiótica de las dos muestras (E. coli y S. aureus). Ct valores de la PCR-ensayo fueron copiados a este archivo de Excel, ya que automáticamente se calcula las dos línea de corte alores Ct del control de crecimiento positivo y negativo, utilizando las fórmulas que aparecen en el texto del protocolo. Si un antibiótico muestra un valor de Ct menor que el valor de corte Ct, la cepa es resistente a los antibióticos, si el valor de CT fue mayor que la de corte, la cepa es susceptible.
Discusión
El protocolo aquí descrito permite la identificación rápida de patógenos y ofrece un perfil funcional a los antimicrobianos que puedan conducir a la administración precoz de antibióticos adecuados, mejorando así el pronóstico de los pacientes con infecciones del torrente sanguíneo. Dependiendo de las condiciones exigidas de una prueba, es decir, costo bajo, alto rendimiento, un mínimo de tiempo de vuelta, las condiciones de prueba se puede ajustar. Todo el procedimiento se puede realizar en un día de trabajo. Además, las dos partes del protocolo puede realizarse simultáneamente, lo cual reduce el tiempo de giro alrededor de manera significativa. Por lo aquí expuesto, el panel de identificación es una selección de las bacterias con mayor relevancia clínica en nuestro hospital. Desde el principio principal se dirige a la región del gen 16S, las sondas específicas para otros microorganismos pueden ser diseñados y se añade a la del ensayo. El ensayo completo fue originalmente pensado para el análisis rápido de los cultivos de sangre, pero también pueden ser utilizados para el procesamiento de Otros materiales de la muestra. Este es también el caso de los antibióticos que se utilizaron para las pruebas de susceptibilidad a los antibióticos: los antibióticos más o se pueden agregar otros, con base en los patrones de resistencia locales y directrices.
Divulgaciones
No tenemos nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el azm Profileringsfonds (PF245).
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| De cloruro de sodio (NaCl) | Merck Chemicals | 106404 | 0,9% en agua |
| Vacutainer SST suero tubo separador 5 ml | BD Diagnostic Systems | 367986 | |
| Mueller Hinton II caldo de | BD Diagnostic Dystems | 212322 | 44 g / l en agua |
| Centrifugar Rotixa 50 rs | Andreas Hettich GmbH & Co. KG | 4910 | |
| Centrífuga 5415 D | Eppendorf | Suspendido | |
| Primers | Sigma-Aldrich | na | |
| Sondas | Sigma-Aldrich/Applied Biosystems | na | |
| TaqManEnvironmental maestro de la mezcla 2,0 | Applied Biosystems | 4396838 | |
| iQ SYBRGreen Supermix | Bio-Rad Laboratories BV | 170-8880 | |
| MicroAmp óptica de 96 pocillos de reacción | Applied Biosystems | N8010560 | |
| Película MicroAmp adhesivo óptico | Applied Biosystems | 4311971 | |
| iQ PCR de 96 pocillos placas | Bio-Rad Laboratories BV | 223-9441 | |
| Microseal B burletes adhesivos en | Bio-Rad Trabajoatories BV | MSB-1001 | |
| PCR en tiempo real del sistema de detección | Applied Biosystems | ABI PRISM 7900HT | |
| PCR en tiempo real del sistema de detección | Bio-Rad Laboratories BV | MyiQ un solo color |
Referencias
- Wallet, F. Preliminary clinical study using a multiplex real-time PCR test for the detection of bacterial and fungal DNA directly in blood. Clin. Microbiol. Infect. 16, 774 (2010).
- Beekmann, S. E., Diekema, D. J., Chapin, K. C., Doern, G. V. Effects of rapid detection of bloodstream infections on length of hospitalization and hospital charges. J. Clin. Microbiol. 41, 3119 (2003).
- Raymaekers, M., Bakkus, M., Boone, E., de Rijke, B., Housni, H. E. l., Descheemaeker, P., De Schouwer, P., Franke, S., Hillen, F., Nollet, F., Soetens, O., Vankeerberghen, A. Molecular Diagnostics working group. Reflections and proposals to assure quality in molecular diagnostics. Acta. Clin. Belg. 66, 33 (2011).
- Peters, R. P. New developments in the diagnosis of bloodstream infections. Lancet Infect. Dis. 4, 751 (2004).
- Hansen, W. L., Beuving, J., Bruggeman, C. A., Wolffs, P. F. Molecular probes for the diagnosis of clinically relevant bacterial infections in blood cultures. J. Clin. Microbiol. 48, 4432-4432 (2010).
- Beuving, J. Antibiotic susceptibility testing of grown blood cultures by combining culture and real-time polymerase chain reaction is rapid and effective. PLoS ONE. 6, (2011).
- Rolain, J. M., Mallet, M. N., Fournier, P. E., Raoult, D. Real-time PCR for universal antibiotic susceptibility testing. J. Antimicrob. Chemother. 54, 538 (2004).
- Nadkarni, M. A., Martin, F. E., Jacques, N. A., Hunter, N. Determination of bacterial load by real-time PCR using a broad-range (universal) probe and primers set. Microbiol. 148, 257 (2002).
- Waites, K. B., Brookings, E. S., Moser, S. A., Zimmer, B. L. Direct susceptibility testing with positive BacT/Alert blood cultures by using MicroScan overnight and rapid panels. J. Clin. Microbiol. 36, 2052 ( ).
- Vliegen, I. Rapid identification of bacteria by real-time amplification and sequencing of the 16S rRNA gene. J. Microbiol. Meth. 66, 156 (2006).
- Hall, L., Doerr, K. A., Wohlfiel, S. L., Roberts, G. D. Evaluation of the MicroSeq system for identification of mycobacteria by 16S ribosomal DNA sequencing and its integration into a routine clinical mycobacteriology laboratory. J. Clin. Microbiol. 41, 1447 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados