Method Article
Aislamiento de la cromatina mediante la purificación de ARN (chirp)
En este artículo
Resumen
Chirp es una técnica novedosa y rápida a los sitios de unión del Mapa del Genoma de los ARNs no codificantes de largo (lncRNAs). El método tiene la ventaja de la especificidad de los oligonucleótidos antisentido baldosas para permitir la enumeración de los sitios genómicos lncRNA enlazados.
Resumen
RNAs no codificantes largos son los principales reguladores de los estados de la cromatina de importantes procesos biológicos como la compensación de la dosis, la impresión y la expresión de genes en el desarrollo 1,2,3,4,5,6,7. El reciente descubrimiento de miles de lncRNAs en asociación con determinados complejos de la cromatina modificación, tales como el complejo Polycomb represiva 2 (PRC2) que media la histona H3 lisina 27 trimethylation (H3K27me3), sugiere que las funciones generales de lncRNAs numerosos estados en la gestión de la cromatina en un gen específico 8,9 de la moda. Mientras que algunos lncRNAs se cree que actúa en cis de los genes vecinos, lncRNAs otros trabajan en trans para regular los genes yacimiento lejano. Por ejemplo, Drosophila lncRNAs roX1 roX2 y regiones se unen numerosos en el cromosoma X de las células masculinas, y son fundamentales para la compensación de la dosis 10,11. Sin embargo, la ubicación exacta de sus sitios de unión no se conocen en alta resolución. Del mismo modo, humana lncRNA AIRE CALIENTE puede afectar a la ocupación de PRC2 hientos de los genes en todo el genoma 3,12,13, pero ¿cómo se logra la especificidad no está claro. LncRNAs también puede servir como andamios modulares para contratar el montaje de complejos de proteínas múltiples. El clásico de acción trans ARN andamio es el TERC ARN que sirve como la plantilla y andamio para la telomerasa complejo 14; AIRE CALIENTE también puede servir como un andamio para PRC2 y un desmetilasa H3K4 complejo 13.
Los estudios previos de cartografía de ocupación del ARN en la cromatina han revelado una sólida perspectiva de 15,16, pero sólo en un lugar único gen a la vez. Los sitios de ocupación de la mayoría de lncRNAs no se conocen, y las funciones de regulación de la cromatina en lncRNAs han sido en su mayoría infiere de los efectos indirectos de la perturbación lncRNA. Al igual que la cromatina immunoprecipitation seguida de microarrays o la secuenciación profunda (chip-chip o chip siguientes, respectivamente) ha mejorado enormemente nuestra comprensión de las interacciones proteína-ADN en una escala genómica, aquí se ilustra una recentemente publicada estrategia para asignar tiempo de ocupación en todo el genoma ARN en alta resolución 17. Este método, Aislamiento cromatina por purificación del RNA (CHIRP) (Figura 1), se basa en la captura de afinidad de destino lncRNA: complejo cromatina por embaldosado antisentido-oligos, que luego se genera un mapa de sitios de unión genómicas a una resolución de varios cientos de bases con sensibilidad alta y baja de fondo. Chirp es aplicable a muchos lncRNAs porque el diseño de sondas de afinidad es sencillo dada la secuencia de ARN y no requiere conocimiento de la estructura del ARN o dominios funcionales.
Protocolo
1. Sonda de Diseño
Diseño anti-sentido ADN embaldosado sondas para la recuperación selectiva del ARN diana por Chirp.
- Diseño anti-sentido sondas de oligonucleótidos mediante el diseñador de la sonda en línea en singlemoleculefish.com 18.
- Utilice los siguientes parámetros: número de sondas = 1 sonda / 100 pb de longitud ARN, 2) el objetivo% GC = 45, 3) longitud del oligonucleótido = 20, 4) de longitud Espacio = 60-80. Romper el ARN en segmentos, si es demasiado larga para el diseñador. Omitir las regiones de repeticiones o extensa homología.
- Orden anti-sentido sondas de ADN con BiotinTEG a las 3 de alto riesgo extremo.
- Sondas de etiqueta de acuerdo con sus posiciones a lo largo del ARN. Separarlos en dos grupos para que el "par" de piscina contiene todas las sondas de numeración 2, 4, 6, etc, y el "extraño" de piscina contiene sondas de numeración 1, 3, 5, etc Diluir grupo de sondas a la concentración mM 100 y almacenar a -20 ° C.
- Todos los experimentos se realizaron utilizandoambos grupos, que sirven como controles internos para los demás. Real RNA-dependiente de la señal estará presente en las dos piscinas, mientras que la sonda específicos de ruidos será único para cada piscina. Esto se aplica tanto para CHIRP-qPCR y CHIRP ss.
2. Las células de la cosecha
Recoger las células que serán usadas en el experimento CHIRP.
- Se cultivan las células en placas de cultivo de tejidos o frascos a la confluencia. Enjuague con tampón fosfato salino (PBS) de una vez trypsinize. Apaga la tripsina con un volumen de> 2 veces los medios de comunicación, la pipeta hacia arriba y abajo para separar las células y resuspender en suspensión de células individuales. Transfiera todos los medios de comunicación y las células resuspendidas en 50 ml tubos Falcon. 20 millones de células son normalmente suficientes para una muestra Chirp.
- Derivado células en 800RCF durante 4 min. Aspirar y medios Resuspender las células en 40 millones de 40 ml de PBS, se combinan los tubos si es necesario. Derivado células en 800RCF durante 4 min. Decantar PBS, cuidadosamente aspirar en un ángulo del líquido restante.
3. Enlace de la Cruz-Las células y recoger sedimento celular
Crosslink recogen las células con glutaraldehído para preservar la cromatina-ARN interacciones y preparar sedimento celular.
- Siga todos los pasos a temperatura ambiente.
- Preparar 1% de glutaraldehído en PBS temperatura ambiente. Preparar 10 ml por 10 millones de células (0,4 ml de 25% de valores glutaraldehído + 9,6 ml de PBS). El glutaraldehído debe ser utilizado fresco.
- Toque en la parte inferior de los tubos Falcon para desalojar gránulos. Resuspender pellet de células en 1% de glutaraldehído, comenzando con un pequeño volumen para evitar trozos, luego arriba hasta el volumen completo. Invertir para mezclar. Crosslink durante 10 minutos a temperatura ambiente en un agitador de extremo a extremo o rotador.
- Sofocar la reacción de reticulación con el volumen 1/10o de 1,25 M de glicina a temperatura ambiente durante 5 min.
- Girar a 2000RCF durante 5 min. Aspirar el sobrenadante y el precipitado lavado con 20 ml de PBS enfriado una vez, girando a 2000RCF durante 5 min.
- Aspirar y resuspender el wincinera, reticulado precipitado con 1 ml de PBS enfriado por 20 millones de células. Transfiera cada ml a un tubo Eppendorf y giran a 2000RCF durante 3 min a 4 ° C. Quite la mayor cantidad posible con PBS punta de la pipeta con cuidado.
- Flash congelar los gránulos de las células en nitrógeno líquido y se almacena a -80 ° C indefinidamente.
4. Lisis de la célula
Lisar células reticuladas para preparar lisado celular.
- Descongele gránulos congelados de células a temperatura ambiente. Toque duro para desalojar y mezclar el sedimento celular. Girar por la pastilla en el 2000RCF durante 3 min a 4 ° C. Utiliza la esquina 10 l punta de la pipeta para eliminar cualquier resto de PBS.
- En una balanza electrónica (precisión de 1 mg) de tara la masa de un vacío tubo Eppendorf (los tubos pesan 1.060 gramos forma muy consistente). Pese cada pastilla y registrar su peso. Un total de 15 cm plato de células HeLa reticulados normalmente pesa 100 mg.
- Suplemento tampón de lisis (10 veces la masa de sedimento, por ejemplo, 1 ml de 100 mg) con Fresh inhibidor de la proteasa, PMSF y SUPERase en (véase la lista adjunta de búfer). Mezclar bien.
- Añadir un volumen 10 veces complementado tampón de lisis a cada tubo y resuspender el precipitado. Para pequeños gránulos <25 mg, resuspender en 250 l de amortiguación complementado lisis. La suspensión debe ser lisa. Si no es así, dividir la suspensión en 500 ml de alícuotas y utilizar un motor mezclador de pellets para disgregar los grumos. Proceder de inmediato a tratamiento con ultrasonidos.
5. Sonicación
ADN cizallamiento por sonicación reticuladas lisados celulares.
- Someter a ultrasonidos lisado celular en Bioruptor en 15 ml tubos Falcon. Utilice <1,5 ml de lisado en cada tubo, y para acelerar la sonicación, sonicar no más de dos tubos a la vez.
- Someter a ultrasonidos en un baño de agua a 4 ° C en la posición más alta con 30 segundos en ON, OFF 45 segundos intervalos de pulso. Compruebe lisado cada 30 minutos. Continuar sonicando hasta que el lisado de células ya no se turbia. Esto puede tomar tan poco como 30 minutos y horas hasta 4. El númerode los tubos, el volumen de la muestra, la temperatura del baño, y el período de tiempo de sonicación afectará cuánto tiempo toma el proceso. Tubos probablemente sonicar a un ritmo diferente, así que la piscina juntos cada 30 minutos y redistribuir en los tubos originales para asegurar la homogeneidad. Nota: glutaraldehído reticulado células toman mucho más tiempo para sonicar que los equivalentes de formaldehído.
- Al lisado se vuelve clara, transferir 5 l lisado a un tubo Eppendorf. Añadir 90 l de ADN de la proteasa K (PK) Buffer (véase la lista de amortiguación) y 5 PK l. Vortex para mezclar y centrifugar brevemente. Incubar durante 45 min a 50 ° C.
- Extraer el ADN con el kit de Qiagen purificación de PCR. ADN se eluye en 30 l del buffer de elución Qiagen (EB) y comprobar el tamaño del ADN, el 1% en gel de agarosa. Si el frotis grueso de ADN es 100-500 pb, sonicación es completa. Si no es así, continúe con ultrasonidos.
- Centrifugar las muestras de ultrasonidos a 16100RCF durante 10 min a 4 ° C. Combine sobrenadantes, alícuotas en muestras de 1 ml y flash de congelación en nitroge líquidon. Conservar a -80 º C.
6. CHIRP
Hibridar sondas biotiniladas de ADN a ARN y aislar la cromatina enlazado.
- Descongele los tubos de la cromatina a temperatura ambiente.
- Prepare el tampón de hibridación (véase la lista de buffers, preparar 2 ml por cada ml de la cromatina). Vortex para mezclar.
- Para una muestra típica CHIRP utilizando 1 ml de lisado, quitar 10 l para INPUT ARN y 10 l de entrada de ADN y el lugar en tubos Eppendorf. Mantener en hielo hasta su uso posterior.
- Transferir 1 ml de la cromatina a 15 ml tubo Falcon. Añadir 2 ml tampón de hibridación a cada tubo. Por volumen total <1,5 ml, utilice tubos Eppendorf.
- Descongele sondas a temperatura ambiente. NanoDrop sondas para comprobar la cantidad si no lo ha usado en mucho tiempo (100 uM sondas deberían especificaciones ~ 500-600 ng / l usando el ajuste de una sola cadena de ADN). Añadir un volumen adecuado de sondas específicas para tubos (100 pmol por sonda 1 ml de la cromatina, 1 l de 100 pmol / l sonda por 1 ml de la cromatina).Mezclar bien. Incubar a 37 ° C durante 4 horas con agitación.
- Con 20 minutos restantes para la hibridación, preparar las perlas magnéticas C-1 (almacenado a 4 ° C). Utilizar 100 l por 100 pmol de las sondas. Lavar con 1 ml de tampón de lisis sin suplementos tres veces, usando la cinta magnética Dynamag-2 a cuentas separadas de búfer.
- Resuspender las perlas en el volumen original de tampón de lisis, y Suplemento con fresco PMSF, PI y SUPERase en-. Después de 4 h reacción de hibridación es completa, añadir 100 l perlas a cada tubo. Mezclar bien. Incubar a 37 ° C durante 30 min con agitación.
- Preparar el tampón de lavado (5 ml por muestra). Vortex para mezclar. Pre-caliente a 37 ° C. Añadir PMSF antes de su uso.
- Lávese las cuentas con 1 ml de solución de lavado cinco veces. En el primer lavado, utilizar Dynamag-15 banda magnética a perlas separadas, decantación y se resuspenden en 1 ml de tampón de lavado. Transferir el volumen a 1,5 ml tubo Eppendorf. Incubar a 37 ° C con agitación durante 5 min.
- En lavados posteriores, la desaceleración de cada tubo en un minicentrifuge, Ajuste de la muestra en Dynamag-2 banda magnética durante 1 min. Se decanta la muestra, limpie cualquier derrame con un Kimwipe, resuspender en 1 ml de solución de lavado. Incubar a 37 ° C con agitación durante 5 min. Repetir durante cinco lavados totales.
- En el último lavado, resuspender las cuentas bien. Sacar 100 l, y reservado para el aislamiento de ARN. Reserva de 900 l para la fracción de ADN. Coloque todos los tubos de Dynamag-2 la banda magnética y quitar el tampón de lavado. Girar todos los tubos durante un breve instante, colocarlos en cinta magnética. Eliminar el último bit de buffer de lavado completamente con una fuerte punta de la pipeta 10 l.
7. Aislamiento del ARN
Extracto de la fracción de ARN de las muestras de un pitido para cuantificar de QRT-PCR.
- Tomar muestras de 100 l de talón y 10 l de muestra ENTRADA ARN. Añadir 85 l de ARN PK buffer de pH 7,0 a ARN de entrada. Volver a suspender las bolas en 95 l de pH 7,0 ARN PK búfer. Añadir 5 K Proteinease l e incubar a 50 ° C durante 45 min con agitación de extremo a extremo.
- En pocas palabras de girar todos los tubos yhervir las muestras durante 10 minutos en el bloque de calor a 95 ° C.
- Enfriar en hielo, añadir 500 l TRIzol vórtice, vigorosamente durante 10 segundos. Incubar a temperatura ambiente durante 10 min. Conservar a -80 ° C o vaya al paso 4.
- Añadir 100 l de cloroformo TRIzol muestras tratadas. Vortex vigorosamente durante 10 segundos. Girar a 16100RCF en una centrífuga de mesa durante 15 min a 4 ° C.
- Retire ~ 400 l sobrenadante acuoso, evitando orgánica y la interfaz.
- Añadir 600 l (1,5 volúmenes) 100% de etanol y mezclar bien. Haga girar la muestra a través MIRNeasy mini columnas. Lavar 1x con TAR (MIRNeasy mini kit), 2x con RPE de acuerdo al protocolo del fabricante. Eluir con 30 l de nucleasa libre de H 2 O (HNF 2 O).
- Tratar el eluato de ARN con ADN libre por el protocolo del fabricante. Después de la reacción es completa, calentar la muestra durante 15 minutos a 65 ° C para inactivar completamente cualquier resto de DNasa.
- Utilice 1 l de ARN aislado por pocillo de QRT-PCR análisis para confirmar lncRNArecuperación. GAPDH se utiliza a menudo como un control negativo.
8. El aislamiento del ADN
Extracto de la fracción de ADN de muestras de un pitido para identificar o cuantificar mediante secuenciación por PCR cuantitativa.
- Prepare el tampón de elución del ADN (ver lista de buffers), 150 l por ejemplo, incluyendo la entrada de ADN.
- Añadir 1 0μL RNasa A (10 mg / ml) y 10 l RNasa H (10 U / l) por ml de tampón de elución del ADN, y vórtice para mezclar.
- Resuspender cada muestra de las perlas en 150 l de tampón de elución con ADN RNasas. (ENTRADA Resuspender el ADN en 140 l) Se incuba a 37 ° C durante 30 min con agitación.
- Cuentas separadas y el sobrenadante de Dynamag-2 la banda magnética. Eliminar el sobrenadante y añadir a los tubos etiquetados.
- Preparar una segunda alícuota de tampón de elución de ADN con 10 l de ribonucleasa A (10 mg / ml) y RNasaH (10 U / l) exactamente como se hace en 8,2). Añadir 150 l de cada muestra (incluida la entrada de ADN), se incuba, y eliminar el sobrenadante. Recoge todas sobrenadante (hombrod es ~ 300 l).
- Añadir 15 PK l de cada muestra. Incubar a 50 ° C durante 45 min con agitación.
- Pre-spin hacia abajo de color amarillo fase de bloqueo de tubos de gel (5PRIME). Transferir las muestras de ADN a los tubos de gel de enganche de fase, y añadir 300 l PhOH: cloroformo: isoamílico por muestra. Agitar enérgicamente durante 10 minutos, y de girar en una centrífuga de mesa a 16100RCF durante 5 min a 4 ° C. Tome acuoso de la parte superior (~ 300 l). Añadir 3 GlycoBlue l, 30 l de NaOAc y 900 l de EtOH 100%. Mezclar bien y guardar a -20 ° C durante la noche.
- Haga girar las muestras a 16100RCF durante 30 min a 4 ° C.
- Decantar el sobrenadante con cuidado. Añadir 1 ml de EtOH 70% y agitar para mezclar. Girar a 16100RCF durante 5 min. Eliminar sobrenadante con una pipeta. Deje secar al aire durante 1 min. Resuspender en 30 l EB.
- Las muestras de ADN están listos para el análisis de qPCR o la preparación de las bibliotecas de secuenciación de alto rendimiento por el protocolo de Illumina.
10. Los resultados representativos
Figura 1 representa el flujo de trabajo CHIRP. Un experimento CHIRP éxito típicamente enriquece ARN diana significativamente más ARNs no específicos. Figura 2 muestra el enriquecimiento de la telomerasa humana ARN (TERC) a partir de células HeLa por GAPDH, un ARN abundantes celulares que sirve como un control negativo. La mayoría de los ARNs TERC (~ 88%) presentes en la célula se empujado hacia abajo por la realización de chirrido, mientras que sólo el 0,46% de GAPDH RNA fue recuperada, demostrando un factor de enriquecimiento de ~ 200 veces. Las sondas no específicas, tales como sondas dirigidas LacZ ARN, que no se expresa en células de mamíferos (Figura 2), se puede utilizar como controles negativos adicionales.
Regiones de ADN esperados de obligar a la lncRNA objetivo son típicamente enriquecida sobre regiones negativos cuando se mide por qPCR. Figura 3 muestra la validación qPCR de cuatro AIRE CALIENTE enlazados a sitios en fibroblastos primarios de prepucio humano que determina realizando CHIRP-ss en la misma línea celular, mientras que TERC y GAPDH ADN sitios sírve como regiones de control negativo. Tanto el "par" y la sonda "raro", establece el enriquecimiento producido comparable de plazos previstos AIRE CALIENTE-sitios a través de las regiones negativas, un sello distintivo de los verdaderos lncRNA sitios de unión.
De alto rendimiento de secuenciación de ADN CHIRP enriquecido se obtiene un mapa global de los sitios de unión lncRNA. La Drosophila lncRNA roX2 se sabe que interactúan con el cromosoma X en una forma que se requiere para la compensación de la dosis. La Figura 4 muestra roX2 perfil de unión sobre una sección del cromosoma X. Tanto "incluso" y "extraño" las muestras han sido secuenciados y sus únicos ruidos han sido eliminados para producir un registro de las señales superpuestas. Cada "pico" aquí indica un sitio fuerte de roX2 vinculante. La pista completa y la lista de roX2 genes diana se han descrito en Chu et al. 2011 17.
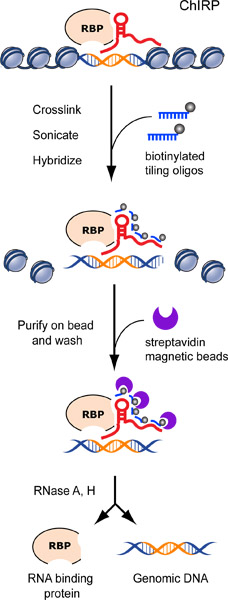
Figura 1. Diagrama de flujo del procedimiento CHIRPDuré. La cromatina se entrecruza con lncRNA: aductos de proteínas in vivo Biotinylated sondas suelo de baldosas se hibridan para apuntar lncRNA, y los complejos de la cromatina se purifica mediante perlas magnéticas estreptavidina, seguido de lavados rigurosos.. Nos eluir el ADN o las proteínas lncRNA unido con un cóctel de RNasa A y H. se esquematiza una supuesta secuencia de lncRNA vinculante en color naranja. Anteriormente publicado en Chu et al. 2011. 17
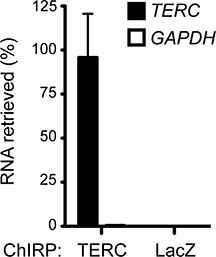
Figura 2. CHIRP enriquece de ARN humano TERC. TERC-asDNA sondas recuperar ~ 88% de los celulares TERC RNA no detectable y GAPDH. LacZ asDNA sondas se utilizaron como controles negativos y recuperar ni ARN. La media ± desviación estándar se muestran. Anteriormente publicado en Chu et al. 2011. 17
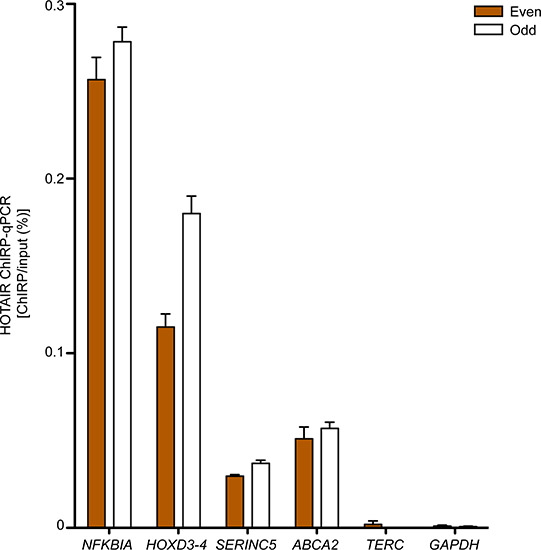
Figura 3. AIRE CALIENTE CHIRP-qPCR en humanos primariafibroblastos Eskin. NFKBIA, HOXD3 4, SERINC5 y ABCA2 son las regiones que interactúan con AIRE CALIENTE. TERC y GAPDH sirvieron como controles negativos. La media ± desviación estándar se muestran. Anteriormente publicado en Chu et al. 2011. 17
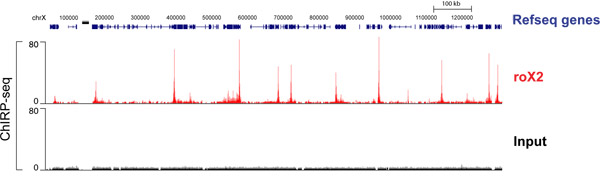
Figura 4. CHIRP-ss datos de roX2 ARN en células de Drosophila SL2. "Aún" y "extraño" fueron secuenciados por separado, sus datos se unen para reflejar sólo los picos comunes en ambos. La pista fusionada se muestra. Anteriormente publicado en Chu et al. 2011. 17
Discusión
Aquí se describe CHIRP-ss, un método de asignación en vivo sitios lncRNA vinculantes en todo el genoma. Los parámetros clave para el éxito son las piscinas abiertas de mosaico sondas de oligonucleótidos y el entrecruzamiento glutaraldehído. El diseño de las sondas de afinidad es sencillo dada la secuencia de ARN y no requiere conocimiento previo de la estructura del ARN o dominios funcionales. Nuestro éxito con roX2, TERC, y AIRE CALIENTE - tres ARN en lugar de dos especies diferentes - sugiere que CHIRP-ss es probable generalizable a muchos lncRNAs. Al igual que con todos los experimentos, los controles de asistencia y adecuada están obligados a interpretar los resultados. LncRNA diferentes pueden requerir un ajuste de las condiciones, y el cambio razonable de condiciones, como la selección de sondas de afinidad o reticulantes diferentes, puede poner de relieve diferentes aspectos de las interacciones ARN-cromatina. Al igual que el chip-ss, no todos los eventos son necesariamente vinculantes funcional, y se requieren estudios adicionales para determinar las consecuencias biológicas de ARN occupancy en la cromatina. No obstante, prevemos muchas aplicaciones interesantes de esta tecnología para los investigadores de otras asociadas a la cromatina lncRNAs, que ahora se cuentan por miles 8,9. Así como chip-ss ha abierto la puerta a todo el genoma de la exploración de las interacciones proteína-ADN, chirrido-ss estudios de la "interactoma ARN" puede revelar muchas nuevas vías de la biología.
Divulgaciones
C. Chu y Chang HY se nombran como inventores en una solicitud de patente basada en este método.
Agradecimientos
Damos las gracias a T. Hung, MC. Tsai, O. Manor, E. Segal, M. Kuroda, Swigut T., y yo Shestopalov para los debates. Con el apoyo de la Agencia de Ciencia, Tecnología e Investigación de Singapur (CC), el NIH R01-R01 CA118750 y HG004361-(HYC), y California Instituto de Medicina Regenerativa (HYC). HYC es un científico de Carrera Temprana del Instituto Médico Howard Hughes.
Materiales
| Name | Company | Catalog Number | Comments |
| Buffer List: | |||
| Dissolve a pellet of complete protease inhibitor in 1 ml water as 50x stock. Make 100 mM PMSF in isopropanol (100x stock). Superase-in is used as 200x stock. Store all at -20 °C. | |||
| Lysis Buffer: | |||
| 50 mM Tris-Cl pH 7.0 10 mM EDTA 1% SDS Always add PMSF, P.I. and Superase-in fresh before use except when washing beads | |||
| Proteinase K Buffer (for DNA) | |||
| 100 mM NaCl 10 mM TrisCl pH 8.0 (For RNA use pH 7.0) 1 mM EDTA 0.5% SDS Add 5% by volume Proteainse K (Ambion AM2546 20 mg/ml) fresh before use | |||
| Hybridization Buffer | |||
| 750 mM NaCl 1% SDS 50 mM Tris-Cl pH 7.0 1 mM EDTA 15% formamide (store in the dark at 4 °C) Always add PMSF, P.I. and Superase-in fresh before use | |||
| Wash Buffer | |||
| 2x NaCl and Sodium citrate (SSC) (diluted from 20x SSC Invitrogen stock) 0.5% SDS Always add PMSF fresh before use | |||
| DNA elution Buffer | |||
| 50 mM NaHCO3 1% SDS | |||
| Table of specific reagents and equipment: | |||
| Glutaraldehyde (EM grade) | Sigma-Aldrich | G5882-10x10ml | |
| Motorized pellet mixer | VWR international | V8185-904 | |
| Protease inhibitor | Roche Group | 11873580001 | |
| PMSF | Sigma-Aldrich | 78830 | |
| Superase-in | Ambion | AM2696 | |
| Bioruptor | Diagenode | UCD-200 | |
| Falcon tubes (for sonication) | Corning | 430790 | |
| Proteinase K | Ambion | AM2546 | |
| PCR purification kit | Qiagen | 28106 | |
| C-1 magnetic beads | Invitrogen | 65002 | |
| PMSF | Sigma-Aldrich | P7626-25G | |
| DynaMag-15 magnet | Invitrogen | 123-01D | |
| DynaMag-2 magnet | Invitrogen | 123-21D | |
| MIRNeasy mini kit | Qiagen | 217004 | |
| Rnase H | Epicentre Biotechnologies | R0601K | |
| Rnase A | Sigma-Aldrich | R4875-100MG | |
| Phase Lock Gel Heavy | 5 PRIME | 2302810 | |
| Trizol | Invitrogen | 15596-018 | |
| Phenol:chloroform:Isoamyl | Invitrogen | 15593-031 | |
| Chloroform | Ricca | RSOC0020-1C | |
| GlycoBlue | Ambion | AM9515 | |
| Glycine | JT Baker | 4057-06 | |
| PBS, pH 7.4 | Invitrogen | 10010-049 | |
| Elution Buffer (EB) | Qiagen | 19086 | |
| 20x SSC | Invitrogen | 15557-036 | |
| 10% SDS | Invitrogen | 15553-027 | |
| DNA-free | Ambion | AM1906 | |
| Buffer kit | Ambion | AM9010 | |
| Formamide | Invitrogen | 15515-026 | |
Referencias
- Koziol, M. J., Rinn, J. L. RNA traffic control of chromatin complexes. Curr. Opin. Genet. Dev. 20, 142-148 (2010).
- Mercer, T. R., Dinger, M. E., Mattick, J. S. Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10, 155-159 (2009).
- Rinn, J. L. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs. Cell. 129, 1311-1323 (2007).
- Zhao, J., Sun, B. K., Erwin, J. A., Song, J. J., Lee, J. T. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome. Science. 322, 750-756 (2008).
- Kelley, R. L. Epigenetic spreading of the Drosophila dosage compensation complex from roX RNA genes into flanking chromatin. Cell. 98, 513-522 (1999).
- Pandey, R. R. Kcnq1ot1 antisense noncoding RNA mediates lineage-specific transcriptional silencing through chromatin-level regulation. Mol. Cell. 32, 232-246 (2008).
- Wang, K. C. A long noncoding RNA maintains active chromatin to coordinate homeotic gene expression. Nature. 472, 120-124 (2011).
- Khalil, A. M. Many human large intergenic noncoding RNAs associate with chromatin-modifying complexes and affect gene expression. Proc. Natl. Acad. Sci. U.S.A. 106, 11667-11672 (2009).
- Zhao, J. Genome-wide identification of polycomb-associated RNAs by RIP-seq. Mol. Cell. 40, 939-953 (2010).
- Meller, V. H., Wu, K. H., Roman, G., Kuroda, M. I., Davis, R. L. roX1 RNA paints the X chromosome of male Drosophila and is regulated by the dosage compensation system. Cell. 88, 445-457 (1997).
- Franke, A., Baker, B. S. The rox1 and rox2 RNAs are essential components of the compensasome, which mediates dosage compensation in Drosophila. Mol. Cell. 4, 117-122 (1999).
- Gupta, R. A. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature. 464, 1071-1076 (2010).
- Tsai, M. C. Long noncoding RNA as modular scaffold of histone modification complexes. Science. 329, 689-693 (2010).
- Zappulla, D. C., Cech, T. R. RNA as a flexible scaffold for proteins: yeast telomerase and beyond. Cold Spring Harb. Symp. Quant. Biol.. 71, 217-224 (2006).
- Nagano, T. The Air noncoding RNA epigenetically silences transcription by targeting G9a to chromatin. Science. 322, 1717-1720 (2008).
- Carter, D., Chakalova, L., Osborne, C. S., Dai, Y. F., Fraser, P. Long-range chromatin regulatory interactions in vivo. Nature. 32, 623-626 (2002).
- Chu, C., Qu, K., Zhong, F. L., Artandi, S. E., Chang, H. Y. Genomic Maps of Long Noncoding RNA Occupancy Reveal Principles of RNA-Chromatin Interactions. Mol. Cell. , (2011).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nat. Methods. 5, 877-879 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados