Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Único ovocito Mutagénesis bisulfito
En este artículo
Resumen
Bisulfito de mutagénesis es el estándar de oro para el análisis de la metilación del ADN. Nuestro protocolo modificado permite el análisis de metilación del ADN en el ámbito de una sola célula y fue diseñado específicamente para los ovocitos individuales. También se puede utilizar para los embriones etapa de escisión.
Resumen
La epigenética abarca todos hereditarias y las modificaciones reversibles a la cromatina que la accesibilidad alter gen, y por lo tanto son los principales mecanismos para la regulación de la transcripción de genes 1. Metilación del ADN es una modificación epigenética que actúa sobre todo como una marca represivo. A través de la adición covalente de un grupo metilo a las citosinas en dinucleótidos CpG, se puede contratar a otras proteínas de represión y modificaciones de las histonas para iniciar procesos involucrados en la condensación de la cromatina y el silenciamiento de genes 2. Metilación del ADN es esencial para el desarrollo normal, ya que desempeña un papel fundamental en la programación del desarrollo, la diferenciación celular, la represión de elementos retrovirales, la inactivación del cromosoma X y la impronta genómica.
Uno de los métodos más poderosos para el análisis de la metilación del ADN es la mutagénesis bisulfito. Bisulfito de sodio es un mutágeno ADN que desamina citosinas en uracilos. Tras la amplificación por PCR y Seque ncing, estos eventos de conversión se detectan como timinas. Citosinas metiladas están protegidos de la desaminación y por lo tanto permanecen como citosinas, que permita la identificación de la metilación del ADN en el nivel individual de nucleótidos 3. Desarrollo del ensayo mutagénesis bisulfito ha avanzado de las originalmente presentadas 4-6 hacia los que son más sensibles y reproducibles 7. Un avance clave fue incrustar pequeñas cantidades de ADN en un cordón de agarosa, protegiendo así el ADN del tratamiento bisulfito dura 8. Este análisis de metilación habilitado para llevar a cabo en las piscinas de los ovocitos y blastocitos, embriones de 9. El protocolo de mutagénesis bisulfito de los más sofisticados hasta la fecha es de cada etapa de blastocisto, embriones de 10. Sin embargo, puesto que los blastocistos tienen un promedio de 64 células (que contiene 120-720 pg de ADN genómico), este método no es eficaz para los estudios de metilación en oocitos individuales o embriones etapa de escisión. tienda "> Tomando pistas de la incrustación de agarosa de las cantidades de ADN minutos incluyendo 11 ovocitos, aquí se presenta un método por el cual los ovocitos están directamente incrustado en una solución de agarosa y lisis cordón de recuperación inmediatamente después y la eliminación de la zona pelúcida del ovocito. Esto nos permite pasar por alto los dos principales retos de la mutagénesis sola bisulfito de ovocitos:. la protección de una pequeña cantidad de ADN de la degradación y la pérdida subsecuente durante los pasos del protocolo numerosos importante, ya que los datos se obtienen a partir de ovocitos individuales, el problema del sesgo de PCR en las piscinas se elimina otra. , contaminación inadvertida célula cúmulo es detectable por este método, puesto que cualquier muestra con patrón de metilación más de un pueden ser excluidos de análisis 12. Este protocolo proporciona un método mejorado para el análisis de éxito y reproducible de la metilación del ADN en el nivel de una sola célula y se adapta idealmente de oocitos individuales, así como embriones en estado de división.
Protocolo
DIA 1
Prepare las siguientes soluciones frescas en el día de la recogida de ovocitos con agua destilada estéril, como el agua GIBCO. Para reducir el riesgo de contaminación de ADN, cambiar los guantes con frecuencia y utilizar puntas con filtro. Mantener tubos ángulo lejos cuando está abierto, y recapitular todos los tubos cuando no esté en uso. Se recomienda que las soluciones se hacen como n +1.
3% de agarosa LMP
Punto 30 mg de fusión bajo (LMP) de agarosa
hasta 1 ml GIBCO H 2 O
disolver @ 70 ° C
Solución de lisis
8 l de buffer de lisis
1 l de proteinasa K
1 l IGEPAL 10%
lugar en hielo hasta que esté listo para su uso
02:01 agarosa: solución de lisis (10 l por persona de ovocitos, la cantidad es de 3 ovocitos)
20 l 3% de agarosa LMP
10 Solución de Lisis l
mezclar @ 70 ° C
SDS Lysis de búfer (501 l por persona ovocito)
| 1x TE pH 7,5 | 450 l |
| 10% de SDS | 50 l |
| Proteinasa K | 1 l |
| 501 l |
1. Recolección de los ovocitos
- Coloque los oviductos de ratones disecados en los medios de comunicación M2, y romper la ampolla para extraer el complejo de células del cumulus.
- Separar los oocitos a partir del complejo de células cúmulos utilizando 0,3 mg / ml de solución de hialuronidasa en una caída de 30 l de medios M2. Mantenga los ovocitos en solución sólo durante el tiempo que sea necesario para eliminar las células del cumulus, ya que la exposición prolongada puede dañar. Lave los ovocitos 3x en 30 l de los medios de comunicación caída de M2, la eliminación de las células del cumulus periódicamente.
- Retire la zona pelúcida con solución ácida de Tyrode. Coloque los ovocitos en un 30 l de solución de gota, y luego transferir a otros 30gota l, como cualquier medio llevadas a lo largo va a diluir el ácido y reducir su eficacia. Mantenga los ovocitos en solución sólo durante el tiempo que sea necesario para eliminar la zona, como la exposición prolongada puede dañar. Nota: un aumento de la concentración de la solución ácida de Tyrode o pronasa puede ser utilizado para muestras humanas, como la humana zona pelúcida es más resistente al tratamiento con una solución ácida de Tyrode que el ratón.
- Lavar los oocitos una vez más en una caída de 30 l de medios M2.
2. Incorporación de agarosa y Lysis
- Para realizar la incrustación de agarosa, poner la solución de lisis en un 70 heatblock ° C. Añadir el precalentado agarosa LMP a la solución de lisis, produciendo una agarosa 2:1: solución de lisis.
- Coloque un único ovocito en un portaobjetos de vidrio limpio en medio mínimo M2. Tomar hasta 10 l de la agarosa: solución de lisis en una punta de la pipeta, y (bajo un microscopio) suavemente expulsar una pequeña cantidad (~ 1 l o menos) sobre el portaobjetos de vidrio, permitiendo que se mezcle con la mínimaAl medios de comunicación. Suavemente levante el ovocito en la punta de la pipeta y poner todo l 10 en un tubo Eppendorf con 300 l de aceite mineral por lo que el cordón forma una esfera.
Nota: este proceso debe hacerse con bastante rapidez como la agarosa se endurecerá si la temperatura desciende por lo menos 5 ° C por debajo de 70 ° C. - Incubar el tubo en hielo durante 10 minutos. Para llevar a cabo la lisis, quitar el aceite mineral de 300 l, y añadir 500 l de la solución amortiguadora de lisis SDS. Incubar toda la noche en un baño de agua a 50 ° C.
Nota: La solución de lisis (Tabla 1) también se pueden utilizar para este propósito.
DÍA 2
Preparar las siguientes soluciones frescas en el día de mutagénesis bisulfito. Para reducir la posibilidad de contaminación de ADN, cambiar los guantes con frecuencia y utilizar puntas con filtro. Mantener tubos ángulo lejos cuando está abierto, y recapitular todos los tubos cuando no esté en uso. Se recomienda que las soluciones se hacen como n +1.
| 3 M de NaOH | 2,4 g de NaOH en 20 ml autoclave ddH2O |
| 0,1 M de NaOH | 0,5 ml de 3M en 14,5 ml autoclave ddH2O |
| 0,3 M de NaOH | 1,5 ml de 3M en 13,5 ml autoclave ddH2O |
2,5 M solución de bisulfito
- 3,8 g de bisulfito de sodio
5,5 ml GIBCO H 2 O destilada
1 ml de 3 M de NaOH
disolver @ temperatura ambiente - 110 mg de hidroquinona
1 ml GIBCO H 2 O destilada
disolver @ 90 ° C (sólo durante el tiempo que sea necesario para disolver, mezclar con regularidad)
Cuando está completamente disuelto, mezclar la solución (a) y (b)
* Manténgase alejado de la luz *
3. Bisulfito de Mutagénesis
- Totalmente eliminar el tampón 500 l lisis SDS y añadir 300 l de aceite mineral (~ 20 horas). Cualquier tampón de lisis restante se diluya ªe agarosa cuando se calienta y el cordón será más susceptible a la disolución en los pasos subsiguientes. Proceda con bisulfito de mutagénesis de inmediato, o tienda a -20 ° C durante un máximo de 5 días.
- Si procede, extraiga los ovocitos del congelador y dejar descongelar (sólo hasta agarosa al grano es relativamente transparente). Incubar durante 2,5 minutos en un bloque de 90 ° C de calor, después de lo cual Incubar en hielo durante 10 minutos.
Nota: No mezclar o revolver, se extienden más de 2,5 minutos, o variar la temperatura. - Para llevar a cabo la desnaturalización, eliminar el aceite mineral y se añade 1 ml de NaOH 0,1 M a cada tubo, película e invierta 5-6 veces.
- Incubar durante 15 minutos en un baño de agua a 37 ° C, invirtiendo cada 3-4 minutos. El talón debe flotar en el NaOH.
- Para realizar el tratamiento de bisulfito, hacer girar el tubo suavemente, luego retire el NaOH y añadir 300 l de aceite mineral y 500 solución de bisulfito de l. Incubar el tubo durante 3,5 horas en un baño de agua a 50 ° C. * Mantenga alejado de la luz *
Nota: Longitud de la incubación puede necesitar ser determinado empíricamente para el gen de interés. - Para llevar a cabo desulfonation, incubar en hielo durante 3 minutos, luego retire el aceite mineral y la solución de bisulfito, girar suavemente, y se añade 1 ml de 0,3 M de NaOH. Flick e invertir 5-6 veces.
- Incubar durante 15 minutos en un baño de agua a 37 ° C, invirtiendo cada 3-4 minutos. El talón debe flotar en el NaOH.
- Lávese las muestras, en primer lugar, girando con suavidad, luego retire el NaOH y añadir 1 ml de TE 1x pH 7,5. Agitar durante 5-10 minutos a temperatura ambiente (en un agitador). Haga girar suavemente de nuevo, a continuación, quitar el TE 1x. Repita este proceso de lavado dos veces.
- Añadir 1 ml de autoclave ddH 2 O. Agitar durante 5-10 minutos a temperatura ambiente (en un agitador). Haga girar suavemente, a continuación, retire el H 2 O. Repita el lavado ddH 2 O dos veces.
- Comprobar el pH del sobrenadante, sino que debe ser 5,0 pH. Si todavía muy básica, lavar otra vez con H 2 O. Retire todo el sobrenadante, dejando sólo la agarosa esanuncio.
4. 1 ª y 2 ª ronda de amplificación por PCR
- Preparar 1 ª ronda de PCR ** mezcla mientras se lava **
| 10 mM adelante externo Primer | 0,5 l |
| 10 mM Inversa cebador externo | 0,5 l |
| 240 ng / ml de ARNt | 1 l |
| H 2 O | 13 l |
Añadir a Illustra cuentas Ready-to-Go Hot Start PCR
Deslice con cuidado el sólido cordón de agarosa en el tubo de PCR (~ 10 l)
Calentar a 70 ° C y la mezcla
Añadir 25 l de aceite mineral
Total: 50 l
- Ampliar
Nota: Un ejemplo de ciclismo condiciones para ratón SNRPN es desnaturalización durante 2 minutos a 94 ° C, seguido por 40 ciclos de 30 segundos a 94 ° C, 1 minuto a 50 ° C, y 1minutos a 68 ° C, y un final de 10 minutos paso de elongación a 68 ° C. Recocido temperatura durante 1 ª ronda de PCR para el ratón H19 y Peg3 es de 50 ° C. - Prepare 2 ª ronda de PCR mezcla
| 10 mM Adelante Interior Primer | 0,5 l |
| 10 mM cebador inverso interior | 0,5 l |
| H 2 O | 19 l |
Añadir a Illustra cuentas Ready-to-Go Hot Start PCR
Añadir 5 l 1 producto redondo pt como una plantilla. Calentar el producto 1 ª ronda a 70 ° C durante 1 minuto para ablandar la agarosa. Asegúrese de que la pipeta por debajo de la capa de aceite mineral.
Añadir 25 l de aceite mineral
Total: 50 l
Nota: las secuencias anidadas de imprimación para SNRPN, H19, y Peg3 se pueden encontrar en el mercado Velker et al 10,12.
- Ampliar
Nota: Ciclismo condiciones para el ratón SNRPN es desnaturalización durante 2 minutos a 94 ° C, seguido por 40 ciclos de 30 segundos a 94 ° C, 1 minuto a 50 ° C, y 1 minuto a 68 ° C, y una elongación final 10 minutos paso a 68 ° C 10. Ratón H19 y Peg3 requieren un 50 ° C la temperatura de recocido para 2 ª ronda de PCR. - Como una prueba de diagnóstico, la segunda ronda muestras puede ser cortado con una enzima de restricción que es la metilación o cepa específica.
| 2 ª Ronda del producto | 4 l |
| Enzima de restricción | 1 l |
| Buffer | 1 l |
| H 2 O | 4 l |
- Electroforesis los productos de digestión en un gel de acrilamida al 8%. Las bandas heterogéneas representan más de un Sequena vez.
5. La clonación y la asistencia técnica de PCR colonia
- Para clonar 2 ª producto redondo, calor primero a 70 ° C durante 1 minuto para ablandar la agarosa, a continuación, ligante en el vector usando la Promega vector pGEM-T del sistema (Fisher Scientific Cat. # A1360).
| 2 ª ronda de PCR | 1 l |
| pGEMT-fácil vector | 1 l |
| Ligasa | 1 l |
| H 2 O | 2 l |
| Tampón de ligación 2x | 5 l |
Incubar toda la noche @ 4 ° C en máquina de PCR.
- Descongele competentes células de E. coli en hielo durante 15 minutos (Zymo Investigación Cat Corp # T3009). Añadir 3 l de reacción de ligación a 8 l E. coli y la ligadura se incuba en hielo durante 15 minutos.
- De choque térmico durante 40 segundos en un baño de agua a 42 ° C,y se incuba en hielo durante 2 minutos. Añadir 60 l medio SOC y se incuba a 37 ° C durante 1 hora (en coctelera).
- Coloque toda la mezcla de reacción sobre una placa LB / agar / IPTG / Xgal / Amp y la placa se incuba a 37 ° C durante la noche.
- Prepare la mezcla de PCR de colonias
| 20 mM M13 cebador | 0,7 l |
| 20 mM M13 cebador inverso | 0,7 l |
| Go Green 5X buffer Taq | 7,0 l |
| 10 mM dNTP | 0,7 l |
| Taq ADN polimerasa | 0,28 l |
| H 2 O | 25,62 l |
| 35 Total l |
Añadir 35 l colonia PCR Master Mix en un tubo de PCR. Elija una colonia de bacterias de la placa blanca con una punta de la pipeta, y se arremolinan en la reacción de PCR.
- Amplificar con desnaturalización durante 10 minutos a 94 ° C, seguido por 30 ciclos de 45 segundos a 94 ° C, 30 segundos a 57 ° C, y 1 minuto a 72 ° C, y un paso final 10 minutos de elongación a 72 ° C. 4 l electroforesis en un gel de agarosa al 1,5%. Enviar ~ 30 l del producto de PCR para la secuenciación.
Nota: Para obtener los ovocitos, 5 colonias de los productos de PCR se secuenciaron. - Una vez que los resultados de la secuenciación se obtienen, los patrones de metilación puede ser leído. Cualquier CG original que se mantuvo como un centro de gravedad fue metilado, y cualquier CG original que ahora es un TG se unmethylated.
6. Los resultados representativos
En nuestro trabajo, prueba de metilación impresa en ovocitos y embriones individuales (Figura 1). Tras anidada amplificación por PCR utilizando cebadores bisulfito convertidos, es posible confirmar una conversión con éxito mediante la visualización de un tamaño de fragmento correcto en un gel de agarosa (Figura 2). Un oocito individuorepresenta uno de los alelos de los padres, y, en teoría, tiene un patrón de metilación impresa. Como tal, la segunda ronda los productos de PCR puede hacerse la prueba de la contaminación accidental. Una enzima de restricción sensible a la metilación del ADN (tales como HinfI o DpnII) se puede utilizar para digerir el producto segunda ronda de PCR para evaluar si contiene un alelo metilado o no metilado (Figura 3). A metilado dentro de la secuencia de reconocimiento de la enzima se escinde mientras un C unmethylated que se convierte a T ya no es reconocido por la enzima y es sin cortar. Cualquier muestra que contenga tanto oocitos MII metilado y alelos metilados debe ser desechado, ya que es indicativa de contaminación cúmulos de células (Figura 3). Después de la ligación y transformación, colonia éxito amplificación por PCR puede ser visualizada en un gel de agarosa para asegurar las muestras con el tamaño correcto del producto se envían para la secuenciación (Figura 4). Finalmente, la secuencia de cinco cl individuolos de un ovocito MII debe producir cinco patrones de metilación idénticos y las mismas tasas de conversión nonCpG (Figura 5A). Todas las muestras que contienen más de un patrón debe desecharse (Figura 5b). Desde ovuladas oocitos MII tienen dos copias del cromosoma o un cuerpo polar adjunto, hay una posibilidad para la obtención de dos patrones de secuencia similar (Figura 5c). Se recomienda descartar los datos a partir de ovocitos que tienen los patrones de metilación muy disímiles ya que la contaminación de cúmulos de células que no se puede descartar.

Figura 1. Esquema del ensayo de mutagénesis único ovocito bisulfito.
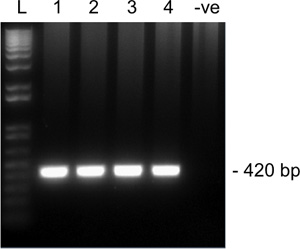
Figura 2. Los resultados representativos de amplificación de 2 ª ronda para SNRPN de un cantarLe MII ovocitos en un 1,5% en gel de agarosa. Los carriles 1-4 son cuatro individuales ovocitos MII y el carril 5 es un control negativo (sin oocito). Tamaño esperado del amplicón de 420 pb es SNRPN. L, escalera.
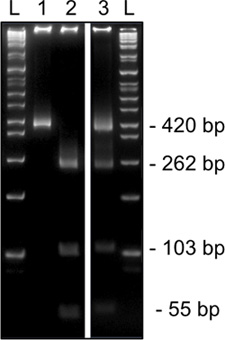
Figura 3. Los resultados representativos de 2 ª ronda digestión de restricción metilación específica para SNRPN de una sola oocitos MII en un gel de acrilamida al 8%. Digestión HinfI restricción diagnóstico muestra de ADN metilado que alberga una T que suprime el sitio de restricción (420bp, calle 1) o ADN metilado que contiene un sitio de reconocimiento dentro de C (corte, 262, 103, y 54 pb, calle 2). La digestión que muestra los sitios de restricción tanto metilado y unmethylated enzima bandas cortadas y sin cortar, carril 3) son indicativos de contaminación cúmulos de células. L, escalera.
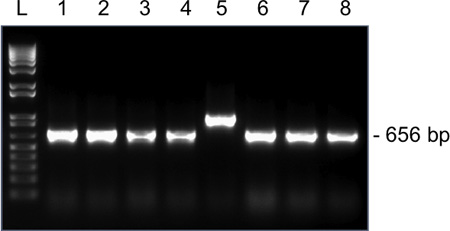
Figura 4. Los resultados representativos para la colonia de la amplificación por PCR de SNRPN de un solo ovocito MII en un 1,5% en gel de agarosa. Tamaño esperado del amplicón después de la ligadura de SNRPN en el vector pGEM-T Easy y el uso de M13 adelante y atrás primers es 656 pb. Carril 1-8, 1-8 amplicones de los clones. Clon 5 tiene un tamaño de amplificación incorrecta y no deben ser enviados para la secuenciación.

Figura 5. Representante de secuenciación resultados para SNRPN de un solo ovocito MII. SNRPN es metilado en los ovocitos. Los círculos negros indican metilado CpGs. Los círculos blancos indican unmethylated CpGs. Número de CpG y la colocación es representativo de un ratón B6 cepa femenino. a) Se espera los resultados de secuenciación de SNRPN de un solo ovocito MII. Sólo una sola hebra de ADN se amplifica en los cinco clones. Los ovocitos con un patrón de metilación sola y misma no CpG golpeteo de conversiónn deben ser incluidos en los análisis (porcentaje de conversión de los no CpGs indica a la derecha se calculó como el número de no-CpG citosinas convertidos a timina como un porcentaje del total de no-CpG citosinas). b) Secuenciación de los resultados de SNRPN de un solo ovocito MII con la contaminación de células del cumulus. Tenga en cuenta la disimilitud entre los estados y los patrones de metilación de conversión que indica la amplificación cadena múltiple. c) La secuenciación resultados para SNRPN de un solo ovocito MII con las copias de cromosomas o la inclusión tanto de cuerpo polar.
Discusión
Este ensayo contiene un solo ovocito muchos pasos con un número que son críticas y requieren un cuidado especial. El primero es el lavado de los ovocitos. Es particularmente importante para lavar cada oocito varias veces en medio fresco cae después del tratamiento hialuronidasa para eliminar las células cumulus como sea posible. Por otra parte, cuando la transferencia de ovocitos a la solución ácida de Tyrode para la zona pelúcida asegurarse de la eliminación medio circundante está libre de células del cumulus...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por la Universidad de Western Ontario, el Departamento de Obstetricia y Ginecología, y una donación ER06-02-188 de la Investigación y la Innovación Ministryof, Premio al Investigador temprana. MMD fue apoyada por un programa de capacitación CIHR en la reproducción, el desarrollo temprano y el Impacto en la Salud de Becas de Posgrado (REDIH).
Materiales
Tabla de los reactivos y equipos específicos.
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | Comentarios |
| Recolección de los ovocitos | |||
| Hialuronidasa | Sigma | H4272 | |
| Ácida de Tyrode | Sigma | T1788 | |
| Proteinasa K | Sigma | P5568 | |
| 10% IGEPAL | Bioshop | NON999.500 | |
| Solución de lisis | |||
| Tris pH 7,5 | Bioshop | TRS001.5 | |
| LiCl | Sigma | L9650 | & Nbsp; |
| EDTA pH 8,0 | Sigma | E5134 | |
| LiDS | Bioshop | LDS701.10 | |
| TDT | Invitrogen | P2325 | |
| Tampón de lisis SDS | |||
| TE pH 7,5 | Bioshop (Tris) Sigma (EDTA) | TRS001.5 E5134 | |
| 10% de SDS | Bioshop | SDS001.500 | |
| Bisulfito de conversión | |||
| Hidróxido de sodio | Sigma | S8045 | |
| De sodio bisulfito (bisulfito de sodio) | Sigma | 243973 | ; |
| La hidroquinona | Sigma | H9003 | |
| Bajo punto de fusión (LMP) de agarosa | Sigma | A9414 | |
| Aceite mineral | Sigma | M8410 | |
| Medio M2 | Sigma | M7167 | |
| GIBCO Agua destilada | Invitrogen | 15230-196 | |
| Autoclave de doble destilada (DD) | |||
| PCR | |||
| Illustra Hot Start Mix RTG | GE Healthcare | 28-9006-54 | |
| 240 ng / ml de tRNA de levadura | Invitrogen | 15401-011 | |
| 5x verde GoTaq tampón de reacción | ProMega | M7911 | |
| Cebadores internos y externos | Sigma | ||
| Ligadura | |||
| Promega pGEM-T Easy Vector | Fisher Scientific | A1360 | |
| Cloning TA | |||
| Células de E. coli competentes | Zymo Research Corp. | T3009 | |
| Equipo | |||
| Microscopio de disección | |||
| 70 ° C y 90 ° C Bloques de calor | |||
| 37 ° C y 50 ° C, tanque de agua (42 ° C para las transformaciones) | |||
| Mecedora | |||
| Máquina de PCR | |||
Referencias
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. 33, 245-254 (2003).
- Rodenhiser, D., Mann, M. Epigenetics and human disease: translating basic biology into clinical applications. CMAJ. 174, 341-348 (2006).
- Frommer, M. A genomic sequencing protocol that yields a positive display of 5-methylcytosine residues in individual DNA strands. Proc. Natl. Acad. Sci. U.S.A. 89, 1827-1831 (1992).
- Clark, S. J., Harrison, J., Paul, C. L., Frommer, M. High sensitivity mapping of methylated cytosines. Nucleic Acids Res. 22, 2990-2997 (1994).
- Feil, R., Charlton, J., Bird, A. P., Walter, J., Reik, W. Methylation analysis on individual chromosomes: improved protocol for bisulphite genomic sequencing. Nucleic Acids Res. 22, 695-696 (1994).
- Raizis, A. M., Schmitt, F., Jost, J. P. A bisulfite method of 5-methylcytosine mapping that minimizes template degradation. Anal. Biochem. 226, 161-166 (1995).
- Patterson, K., Molloy, L., Qu, W., Clark, S. DNA Methylation: Bisulphite Modification and Analysis. J. Vis. Exp. (56), e3170 (2011).
- Olek, A., Oswald, J., Walter, J. A modified and improved method for bisulphite based cytosine methylation analysis. Nucleic Acids Res. 24, 5064-5066 (1996).
- Mann, M. R. Selective loss of imprinting in the placenta following preimplantation development in culture. Development. 131, 3727-3735 (2004).
- Market-Velker, B. A., Zhang, L., Magri, L. S., Bonvissuto, A. C., Mann, M. R. Dual effects of superovulation: loss of maternal and paternal imprinted methylation in a dose-dependent manner. Hum. Mol. Genet. 19, 36-51 (2010).
- Meng, L. H., Xiao, S. Q., Huang, X. F., Zhou, Y., Xu, B. S. A study on bisulfite sequencing method for methylation status of imprinted genes in single human oocytes. Zhonghua Yi Xue Yi Chuan Xue Za Zhi. 25, 289-292 (2008).
- Denomme, M. M., Zhang, L., Mann, M. R. Embryonic imprinting perturbations do not originate from superovulation-induced defects in DNA methylation acquisition. Fertil. Steril. 96, 734-738 (2011).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Hajj, N. E. l. Limiting dilution bisulfite (pyro)sequencing reveals parent-specific methylation patterns in single early mouse embryos and bovine oocytes. Epigenetics. 6, 1176-1188 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados