Method Article
Multi-objetivo enfoque de procesamiento paralelo para Gene a la estructura de determinación de la polimerasa subunidad PB2 Influenza
* Estos autores han contribuido por igual
En este artículo
Resumen
El diseño de fármacos basado en la estructura juega un papel importante en el desarrollo de fármacos. La búsqueda de objetivos múltiples en paralelo aumenta considerablemente las posibilidades de éxito para la detección de plomo. El siguiente artículo pone de relieve cómo el Centro de Genómica Estructural Seattle de Enfermedades Infecciosas utiliza un enfoque multi-objetivo para la determinación genética de la estructura de la influenza A subunidad PB2.
Resumen
Brotes pandémicos de las cepas de influenza altamente virulentas pueden causar morbilidad y mortalidad generalizada en las poblaciones humanas en todo el mundo. Sólo en los Estados Unidos, un promedio de 41.400 muertes y 1.860.000 hospitalizaciones se deben a infección por virus de la gripe cada año 1. Las mutaciones puntuales en la proteína básica de la polimerasa subunidad 2 (PB2) se han relacionado con la adaptación de la infección viral en seres humanos 2. Los resultados de tales estudios han puesto de manifiesto la importancia biológica de PB2 como un factor de virulencia, destacando así su potencial como una diana de fármaco antiviral.
El programa de genómica estructural presentado por el Instituto Nacional de Alergias y Enfermedades Infecciosas (NIAID) provee fondos para Emerald Bio y otras tres instituciones del noroeste del Pacífico que componen el Centro de Genómica Estructural Seattle de Enfermedades Infecciosas (SSGCID). El SSGCID se dedica a proporcionar a la comunidad científica threestructuras de proteínas e-dimensionales de NIAID Categoría AC patógenos. Hacer tal información estructural disponible para la comunidad científica sirve para acelerar la estructura basada en el diseño de fármacos.
El diseño de fármacos basado en la estructura juega un papel importante en el desarrollo de fármacos. La búsqueda de objetivos múltiples en paralelo aumenta considerablemente las posibilidades de éxito para el nuevo descubrimiento de cabezas apuntando a un camino o una familia de proteínas entero. Esmeralda Bio ha desarrollado una canalización de procesamiento paralelo de alto rendimiento, multi-objetivo (MTPP) para la determinación de genes a la estructura de apoyo del consorcio. Aquí se describen los protocolos utilizados para determinar la estructura de la subunidad PB2 de cuatro diferentes cepas de la gripe.
Protocolo
Una visión general del protocolo se presenta en la Figura 1.
Biología Molecular
1. Construir Diseño
Utilice el software Compositor Gene para diseñar construcción de la proteína y el codón de ingeniería secuencias de genes sintéticos. El uso de software Compositor Gene ha sido ofrecido en detalle en otra parte 3.
- Utilice el módulo Visor de Alineación y Construcción de Módulo de Diseño para comparar secuencias de proteínas alineaciones y definir construcción de la proteína. Alinear la secuencia de aminoácidos diana a ambos los elementos estructurales primarios y 3D de homólogos en el Banco de Datos de Proteínas (PDB), si está disponible (Figura 2).
- Utilizar la información de alineación para hacer diseños constructo estructura-guiadas por la elección de nuevos terminales basados en la conservación de la estructura primaria y estructuras 3D de homólogos.
- Diseño de PCR inserto (iPCR) y el vector PCR (vPCR) amplímeros (terminal de cebadores).
- Usando Gene Calgoritmo de proteína-ADN de omposer, de vuelta-traducir la secuencia de aminoácidos constructo en secuencia de ácido nucleico de ingeniería codón.
- Utilice la tabla de uso de codones adecuada (CUT) para optimizar la secuencia para la expresión en E. coli.
- Prácticamente clonar inserción en pET28 vector modificado para incorporar una etiqueta N-terminal histidina 6x y proteína de fusión Smt3/SUMO que permite una fácil purificación.
- Poner fin gen sintético con DNA 2.0 y cebadores orden de Integrated DNA Technologies.
2. Polimerasa incompleta Extensión Clonación Primer (PIPE)
- Prepare Primers y Genes
- Centrifugar las placas suministrados por el proveedor que contienen cebadores a 1.000 rpm durante 1 min.
- Traiga primer concentración de 100 mM y añadir tampón TE 50 l.
- Diluir cebadores a 10 mM con agua desionizada (DI) en una placa de fondo en v de 96 pocillos.
- Centrifugar el gen suministrado por el proveedor en un tubo de 1,5 ml a 1300 rpm durante 1 min.
- Uso de tampón TE, llevar la concentración de ADN de cada tubo a 50 ng / l.
- En tubos de 1,5 ml, hacer diluciones de cada cebador a 10 ng / l.
- Primers Tienda y genes a -20 ° C cuando no esté en uso.
- Prepare Insertar PCR (iPCR)
- Descongelar un vial de Pfu Master Mix en hielo; mantener los genes y los cebadores a temperatura ambiente.
- Crear un mapa de asignación de los pozos de la placa a un conjunto de cebadores y construir.
- Añadir 13 l de agua DI en cada pocillo de una placa de PCR de 96 pocillos.
- Añadir 5 l de hacia adelante y 5 l de cebador inverso a cada reacción en la placa de 96 pocillos de acuerdo con el mapa placa, asegurando a cambiar las puntas entre cada pocillo.
- Añadir 2 l de cada gen de longitud completa para su adecuada bien según el mapa de la placa.
- Añadir 25 l de Pfu maestro de la mezcla a cada pocillo, asegurando a cambiar las puntas entre cada pocillo.
- Ciclo de las reacciones con las siguientes condiciones de PCR:
- 95 ° C 2 min
- 95 ° C 30 seg
- 50 ° C 45 seg
- 68 ° C 3 min
- 4 ° C ∞
- Repita los pasos bd durante 25 ciclos.
- Transferir 10 l de cada reacción iPCR a una nueva placa de PCR de 96 pocillos.
- Añadir 3 l de colorante de carga 6X a cada muestra.
- Separar cada muestra en un gel de 1% de agarosa TAE EtBr a 110 V al lado de una escalera de 100-500 pb de ADN para confirmar la amplificación de fragmentos.
- Tienda producto iPCR a -20 ° C cuando no está en uso (evitar la congelación descongelación tanto como sea posible).
3. Preparar vectorial PCR (vPCR)
- Iniciar cultivo nocturno de E. transformado coli con el plásmido vector pET28.
- Inocular dos tubos de 5 ml de caldo de 2-YT con 50 mg / ml de kanamicina.
- Los cultivos durante la noche a 37 ° C en el agitador a 220 rpm.
- Centrifugar las culturas después de una noche de crecimiento por centrifugación a 3000 rpm durante 15 min.
- Utilice un Qiagen QIApreparación Spin Miniprep Kit para extraer vector pET28 de sedimentos bacterianos de acuerdo con las instrucciones del fabricante.
- Configuración de digestiones con enzimas de restricción del plásmido pET28 extrajeron.
- Añadir 2,2 l de tampón BamHI 10X y 1 l de BamHI y HindIII para 20 l de vector pET28.
- Se incuba la reacción durante 1 hora a 37 ° C.
- Producto de digestión separada en un gel.
- Consulte el paso 2.2.10.
- Cortar banda del vector a partir de gel y se purifica utilizando el kit de extracción de gel QIAquick según las instrucciones del fabricante.
- El uso de un NanoDrop, cuantificar la concentración de ADN.
- Diluir corte vector de 10 ng / l. Almacenar a -20 ° C cuando no esté en uso.
- Prepare primers vPCR.
- Centrifugar IDT oligonucliotides suministrados durante 1 minuto a 1300 rpm.
- Traiga la concentración de 100 mM con agua DI.
- Preparar una dilución 10 mM de ambos adelante y atrás en un tubo de 1,5 ml.
- Tienda cebadores y las diluciones de imprimación a -20 ° C.
- Descongelar Pfu Master Mix en molde y los cebadores de hielo y la descongelación a temperatura ambiente.
- Reacciones vPCR instalación de una placa de PCR de 96 pocillos:
- En la primera fila de una placa de 96 pocillos se combinan 60 l de ambos cebadores directo e inverso vPCR y 24 l de pET28 digerido plantilla (10 ng / l).
- Usando una pipeta multicanal 12 de punta, la transferencia de 12 l de la imprimación y la mezcla maestra a cada plantilla restante pocillo de la placa. Esto debe resultar en 12 l de cebador y la plantilla de mezcla maestra en cada pocillo de la placa.
- Añadir 13 l de agua DI a cada pocillo.
- Añadir 25 l de Pfu Master Mix en cada pocillo.
- Ciclo de las reacciones a través de las condiciones de PCR utilizadas en el paso 2.2.7.
- Piscina todas las reacciones vPCR en un tubo de 15 ml Falcon.
- Verifique amplificación de fragmentos separando 10 l de producto de PCR agrupado en un gel (duración prevista de vector digerido pET28es de aproximadamente 6 kb).
- Consulte el paso 2.2.10.
- Prepare combinar platos.
- Alícuota 3 l de producto vPCR en cada pocillo de una placa de fondo en v de 96 pocillos.
- Placas Almacenar a -20 ° C hasta merge con el producto iPCR.
4. Combinar iPCR y vPCR Productos
- Productos iPCR Descongelar y pre-vPCR alícuota de 96 pocillos se unen la placa a temperatura ambiente.
- Añadir 3 l de cada producto iPCR a su respectivo pocillo de la placa de mezcla.
- Transformar fusionar placa en Top Ten células químicamente competentes.
- Añadir 2 l de cada reacción se funden en un solo tubo de 50 l de células químicamente competentes suministrado por el proveedor y proceda con el protocolo suministrado por el fabricante.
- Preparar cultivos de una noche para cada construcción de la placa de la transformación.
- Alícuota de 5 ml de caldo de TB (con 50 g / ml de kanamicina) a partir de un depósito de 25 ml estéril en cada pocillo de un bloque de pozo profundo.
- Usando sterile técnica, elige una colonia aislada de cada placa de transformación e inocular el pozo correspondiente del bloque de pozo profundo.
- Cubra el bloque con una cubierta Airpore.
- Agitar bloque a 220 rpm a 37 ° C durante la noche.
- Precipitar las células por centrifugación del bloque durante 30 min a 4000 rpm.
- Retirar el sobrenadante y acariciar la parte superior del bloque de secar con una toalla de papel.
- Mini-Prep utilizando un Qiagen de 96 pocillos aparato de vacío de acuerdo con las instrucciones del fabricante.
5. Preparación de Acciones de glicerol de construcciones con éxito clonados
- Transform clonado con éxito secuencia de ADN validado en BL21 (DE3) células químicamente competentes de acuerdo con las instrucciones del fabricante.
- Para cada constructo, recoger una sola colonia aislada de la (DE3) transformación e inocular BL21 en 1 ml de caldo de 2-YT (con 50 g / ml de kanamicina).
- Agite culturas a 220 rpm durante 3-4 horas a 37 ° C.
- Etiquetar un 1,5 mlenroscar la tapa del tubo con el número único de identificación de constructo, cepa de células, y la fecha. Añadir 500 l de glicerol al 50% y 500 l de cultivo celular y se invierte varias veces. Inmediatamente almacenar de glicerol en hielo seco o en un congelador a -80 ° C.
6. Pruebas de Expresión
| Lisis Buffe r | Tampón de lavado | Tampón de elución |
| 50 mM NaH 2 PO 4, pH 8,0 300 mM de NaCl 10 mM de imidazol 1% de Tween 20 2 mM MgCl 2 0,1 l / ml de Benzonase 1 mg / ml de lisozima | 50 mM NaH 2 PO 4, pH 8,0 300 mM de NaCl 20 mM de imidazol 0,05% de Tween 20 | 25 mM de Tris, pH 8,0 300 mM de NaCl 250 mM de imidazol 0,05% de Tween 20 |
* Añadir Benzonase, lisozima,y el inhibidor de la proteasa inmediatamente antes de la lisis.
- Racha de una muestra de glicerol en agar selectivo kanamicina y se incuba durante la noche a 37 ° C.
- Iniciar un pre-cultivo no induce en un bloque de 96 pocillos de fondo redondo; inocular 1,2 ml de caldo de TB (con 50 mg / ml de kanamicina) suplementado con 0,5% de glucosa con una E. recién crecido aislar coli. Crecer durante la noche con agitación a 220 rpm a 37 ° C.
- Después de un crecimiento durante la noche, iniciar cultivos de inducción mediante la inoculación de 1,2 ml de caldo de TB (con 50 mg / ml de kanamicina) suplementado con Novagen noche a la mañana Sistema Express 1 (de acuerdo con el protocolo del fabricante) con 40 l de la pre-cultivo.
- Cultivar las culturas de inducción pequeña escala a 20 ° C durante 48 horas, agitando a 220 rpm.
- Células de la cosecha por centrifugación a 4000 rpm durante 15 min, se vierte el sobrenadante y se almacena a -20 ° C durante al menos 1 hora antes de la elaboración.
- En el bloque de 96 pocillos, volver a suspender los sedimentos de células en 300 l de tampón de lisis.
- Se incuban las células en tampón de lisis a temperatura ambiente durante 30 min seguido por lisis mecánica agitando vigorosamente durante 30 min a temperatura ambiente.
- Aclarar el lisado crudo por centrifugación durante 30 min a 4000 rpm a 4 ° C.
- Utilizar una pipeta de múltiples canales para transferir 200 l del lisado clarificado (fracción soluble) a una bandeja de 96 pocillos de fondo plano (Qiagen). Para cada pocillo que contiene una muestra, añadir 40 perlas de Ni-NTA magnéticos l (Qiagen).
- Agitar suavemente la placa en un agitador durante 1 hora a 16 ° C.
- Colocar la placa en una placa posterior magnética (Qiagen) y retirar la fracción soluble no unido. Tenga cuidado de no pipeta a cabo cualquiera de las perlas de Ni-NTA.
- Retirar la placa de la placa posterior y se resuspendieron suavemente las perlas en 200 l de tampón de lavado. Pipetear hacia arriba y abajo durante 30 segundos y luego colocar la placa posterior de la placa posterior.
- Retirar el tampón de lavado y repita el paso 6.12.
- Retire la placa de la placa posterior y eluir el Ni-NTA obligado taproteína rget por lavado con 50 l de tampón de elución durante 5 min.
- Volver placa de fondo plano a la placa de poste magnético y transferir la elución a una placa de fondo en v de 96 pocillos fresca.
- Transferir 20 l de la elución a una placa de fondo en v de 96 pocillos fresca y reaccionar con 1 l Ulp1 proteasa.
- De acuerdo con el protocolo del fabricante, analizar la fracción eluida y se eluyó + Ulp1 por electroforesis capilar utilizando un LabChip 90.
- Alternativamente, todas las fracciones de la prueba de expresión pueden ser analizados a través de SDS-PAGE.
7. Grande fermentación a escala
- Utilizar una punta de pipeta estéril para obtener una raspadura de un stock de glicerol, inocular 100 ml de caldo de TB (con 50 mg / ml de kanamicina) y crecer durante la noche. Agitar a 220 rpm y 37 º C.
- Después de un crecimiento durante la noche, ampliar de pre-cultivo mediante la inoculación de 1 L de caldo TB con soluciones autoinducción EMD (véase el protocolo del fabricante) (con 50 mg / ml de kanamicina) en un matraz de 2 L desconcertado con 10 ml de lapre-cultivo (dilución 1:100).
- Agitar los cultivos expandidos 1 L a 37 ° C; cambiar la temperatura de la incubadora de agitación a 20 ° C cuando se alcanza una densidad óptica de 0,6 (OD 600).
- Después del crecimiento durante la noche, tener un representante alícuota de 10 ml de cada construcción para las pruebas de expresión.
- Cosecha de células pegar por centrifugación a 5000 rpm durante 15 min y se descartó el sobrenadante.
- Celular Freeze pega a -80 ° C.
Purificación de proteínas
Buffers:
| Tampón de Lisis | Buffer A (Equilibrio) | Tampón B (elución) | Acerca de tampón de columna |
| 25 mM de Tris, pH 8,0 200 mM de NaCl 0,5% de glicerol 0,02% de CHAPS 10 mM de imidazol 1 mM TCEP 50 mM de arginina 5 l Benzonase 100 mg lisozima 3 tabletas inhibidor de la proteasa (EDTA libre) | 25 mM de Tris, pH 8,0 200 mM de NaCl 10 mM de imidazol 1 mM TCEP 50 mM de arginina 0,25% Glicerol | 25 mM de Tris, pH 8,0 200 mM de NaCl 200 mM de imidazol 1 mM TCEP | 25 mM de Tris, pH 8,0 200 mM de NaCl 1% Glicerol 1 mM TCEP |
* Añadir Benzonase, lisozima, y las tabletas de inhibidores de la proteasa a cada muestra de 150 ml inmediatamente antes de la lisis.
8. Lisis celular
- Hacer 2 L de tampón de lisis; no añadir lisozima, tabletas inhibidores de la proteasa o benzonasa (cada muestra se lisaron por separado en 150 ml de tampón de lisis).
- Descongelar y volver a suspender pasta de células en tampón de lisis a una masa 1:05: relación de volumen agitando vigorosamente durante 30 min a 4 ° C. Romper trozos sueltos de los lados del vaso de precipitados con una espátula limpia. Durante este período de tiempo preparar Ni y DialyBuffers sis
- El hielo, la lisis de las células utilizando un aparato de ultrasonidos Misonix (70% de potencia, 2 s encendido / 1 segundo apagado pulsos durante 3 minutos) y con cuidado envase agitar para evitar el sobrecalentamiento. Guardar una pequeña (200 l) alícuota de lisado bruto para el análisis futuro.
- Aclarar el lisado crudo por centrifugación a 18.000 xg durante 35 min a 4 ° C, recoger el sobrenadante y guardar una pequeña alícuota (200 l) para el análisis futuro. La tienda de pellets a 4 ° C hasta que se confirmó la proteína ha sido lisadas en la fracción soluble.
9. Fabricante de configuración Protein Pre-run
- Con el fabricante de la proteína activada y el software abierto, inicializar el instrumento.
- Una vez inicializado, conectar una columna 5,0 ml GE Healthcare HisTrap FF níquel-quelato (Ni columna) en una línea separada del pórtico para cada una de las muestras.
- Ejecutar 3-4 volúmenes de columna (CV) de tampón de equilibrado a través de cada columna.
- Cebe el equilibrio y las líneas de tampón de elución.
- Equilibrcomió las columnas aspirando tampón A través de la columna una vez.
10. Nickel 1 (Ni1) Columna
- Lave cada columna con 20 ml de agua Milli-Q para eliminar tampón de almacenamiento. Ejecutar 5 ml de tampón B y 25 ml de tampón A para el equilibrado.
- Cargar el lisado clarificado que contiene proteína solubilizada en las columnas a una velocidad de 2 ml / min, a continuación, siga por un lavado de 15 ml con tampón A.
- Se eluye la proteína unida en un gradiente por etapas con tampones A y B por las siguientes relaciones respetuosamente: 5 ml de 95:5, 5 ml de 60:40, 10 ml de 0:100. Recoger cada fracción de elución por separado.
- Analizar: las fracciones eluidas, lisado crudo, lisado aclarado, y el flujo a través de SDS-PAGE. Fracciones piscina que contiene la proteína y el uso de un Nanodrop para medir Un 280 para determinar aproximadamente la cantidad de proteína presente.
11. Ulp1 escisión
- Mantener una pequeña alícuota (250 l) de la piscina columna Ni1 para posterior análisis en gel. Llevar el restode la piscina Ni1 de 10 ml y añadir ubiquitina proteasa 1 (Ulp1) a 1 l / 5 mg de proteína total para quitar la etiqueta de afinidad Su-SMT.
- Dializar la piscina Ni1 + Ulp1 frente a 2 l de tampón A durante 4 horas a 4 ° C en unos 10 kDa de corte de peso molecular (MWCO) sobre una placa de agitación a 4 ° C.
- Después de la diálisis, ejecute SDS-PAGE de Ni1 piscina y Ni1 pool + Ulp1 para determinar si Ulp1 división se ha realizado correctamente.
12. Nickel 2 (Ni2) Columna
- Carga de proteína escindida sobre la misma columna de Ni y repita el paso 9,3 a un caudal reducido de 1 ml / min. La etiqueta escindido se unirá a la columna y la proteína diana tagless ahora fluirá a través. Recoger el flujo a través en un contenedor fresco.
- Lavar la columna de Ni con 3 ml de tampón A seguido por 5 ml de tampón B para eluir todas proteína marcada con His y unida no específicamente. Recoger cada fracción por separado.
- Ejecutar SDS-PAGE de Ni2 flujo a través, lavar y Ni2 fracciones de elución para verificar Ulp1 escote y que prOtein está presente en el flujo a través. Utilice un Nanodrop para medir Un 280 para determinar aproximadamente la presencia de la proteína.
13. Concentrándose
- Se concentra el flujo a través de Ni2 (y Ni2 elución si la proteína está presente) a 5 ml con un Ultra 10 kDa MWCO tubo de centrífuga Amicon. Haga girar en intervalos de 10 min a 4000 rpm a 4 ° C. Mezclar con una pipeta entre cada giro para evitar que la proteína de un exceso de concentración a lo largo de la membrana.
14. Cromatografía de exclusión por tamaño (SEC)
- Configurar una S-100 10/30 GL columna de Sephacryl (GE Healthcare) equilibrando con tampón SEC 200 ml a una velocidad de flujo de 0,5 ml / min en un sistema AKTApurifier (GE Healthcare).
- Preparar 10 ml superloops para su uso en la columna de la SEC de acuerdo con las instrucciones del fabricante.
- Usando una jeringa de 5 ml, cargar muestras en superloops y comience la carrera SEC.
- Supervisar el seguimiento de absorbancia UV a 280 nm durante la percepción de pequeño volumen fracciones.
- Ejecutar fracciones SEC a través de SDS-PAGE.
- Piscina para las fracciones de la SEC que muestran las bandas de mayor intensidad.
- Concéntrese fracciones SEC agrupados. Consulte el paso 13.1.
- Proteína alícuotas en 100 muestras l, congelación ultrarrápida en nitrógeno líquido y se almacenan a -80 ° C.
CRISTALIZACIÓN
15. La cristalización de proteínas
- Pre-llenar cada depósito de una placa de 96 pocillos compacto cristalización Jr (Emerald Bio) con 80 l de pantalla de cristalización (Esmeralda Bio) de su elección.
- Diluir proteína con tamaño de búfer de 2-20 mg / ml y almacenar en hielo.
- Vierta 0,4 l de proteína y 0,4 l de la pantalla de la cristalización en cada uno de los 96 pozos y cubrir con cinta de sellado de cristal claro (Manco).
- Guarde la placa a 16 ° C, mientras que la comprobación de cristalización de proteínas periódicamente en las próximas semanas bajo un microscopio de disección.
16. Crystal cosecha
- Crear un crioprotector a partir del licor madre y el glicol de etileno. Corte la cinta transparente que cubre el pozo con el cristal de proteína diana. Para un pozo vacío, añadir 1,6 l de la condición de cristalización correspondiente y se combinan con 0,4 l de etilenglicol produciendo una concentración final de 20% de glicol de etileno y 80% de cristalización condiciones. Nota: para optimizar difracción de cristal de probar diferentes crioprotectores tales como: glicerol, aceites, polietilenglicoles de bajo PM, y / o en porcentajes variables de la crioprotector.
- Antes de la cosecha se enfríe un disco de estilo ALS en un Dewar llena con nitrógeno líquido y cubierta con la tapa.
- Se recoge el cristal mediante la colocación de un CryoLoop con el diámetro interior que corresponde al tamaño del cristal en una varita de cristal magnético (Hampton Research) y recoger directamente de la solución así.
- Sumergir inmediatamente el CryoLoop con el cristal cosechado en el crioprotector luego sumergirse en el disco de estilo ALS a parpadear congelar la crystal. Repita este procedimiento para un número deseado de cristales.
17. Crystal Screening / Data Collection
- Una vez que la cosecha se ha completado utilizar una varita puck colocar la tapa magnética crio puck puck en la ALS. Con pinzas dobladas, mueva el disco al revés.
- Transferir el puck a un Rigaku ACTOR dewar, atornillar un Pusher Puck en el disco, y perforar la tapa dejándola en el dewar con alfileres hacia arriba.
- El uso de software JDirector, pantalla de cada cristal bajo los siguientes parámetros: hendidura haz ajustado a 0,5 grados, la distancia detector ajustado a 50 mm, paso de la imagen a 70 grados, y la duración de la exposición establece en 30 segundos.
- Ejecutar MOSFLM en las imágenes de prueba te disparó con JDirector para determinar cuál es el mejor cristal y la estrategia es para la recolección de datos.
- Recoge un conjunto completo de datos sobre la base de los resultados de MOSFLM.
18. Data Processing / Determinación de la estructura
- Ejecutar XDS / XSCALE 4 de procesar el conjunto de datos.
- Abra el paquete de software CCP4.
- Ejecutar Phaser 5 para calcular una solución de reemplazo molecular utilizando un modelo de búsqueda de alta homología, cuando esté disponible. En este caso se utilizó el 3CW4 PDBID como un modelo de búsqueda 6.
- Ejecutar Refmac 7 para refinar el modelo molecular en contra de la reflexión que se recogen en el conjunto de datos. Resolución final debe basarse fuera de la más alta shell y determinado por los siguientes parámetros: factor R> 50%, I / sigma> 2, y la exhaustividad> 90%.
- Construir un modelo de densidad de electrones de 3 dimensiones con el software de gráficos moleculares FOCHA 8.
- Antes de depositar la estructura en el AP validarlo con el software MolProbity 9 para verificar que la calidad de la estructura es adecuada para la deposición.
Resultados
Los siguientes resultados ilustran los resultados esperados del protocolo descrito, y en el caso de PB2, los resultados observados.
Usando Compositor Gene, cinco secuencias de aminoácidos diana de longitud completa de la influenza virus polimerasa subunidad PB2 fueron diseñadas (Figura 2). Las secuencias fueron PB2 volver traducidos y sometidos a muchas medidas de ingeniería 3, dando lugar a secuencias de codones armonizadas optimizados para la expresión en E. coli. Desde los productos iPCR (Figura 3b), un total de treinta y cuatro construcciones se clonaron con éxito en un sistema de vector pET28 modificado 10 con un N-terminal de 6x His-Smt marcador de fusión Uso del tubo de clonación 3 como se muestra en la Figura 3a. Un resumen de la clonación de flujo de trabajo se presenta en la Figura 4.
Después de la clonación con éxito, expresión de la proteína de micro-escala de cada construcción se ensayó en células BL21 (DE3) de E.células de E. coli. Las células se cultivaron en medio TB suplementado con Novagen noche a la mañana Express 1 medio (de acuerdo con el protocolo del fabricante) durante 48 horas a 20 ° C en un conjunto incubador con agitación a 220 rpm. Después del crecimiento, las células se recogieron y se ensayaron para la expresión de proteína soluble utilizando electroforesis capilar con una pinza de LabChip 90. Catorce de los treinta y cuatro PB2 constructos dirigidos a la proteína diana soluble y entró en fermentación a gran escala. Cultivos a gran escala de cada construcción se cultivaron en medio TB suplementado con 1 noche a la mañana medio de Novagen urgente según el protocolo del fabricante. Después del crecimiento, las células se recogieron por centrifugación y se almacenaron a -80 ° C. La expresión de proteínas a gran escala de cada cultivo se confirmó a través de análisis de SDS-PAGE (Figura 5) antes de proceder con la purificación a gran escala.
El fabricante de proteína se utiliza para llevar a cabo la purificación paralela de los catorce constructos PB2. Los lisados clarificados de all catorce construcciones se llevaron a cabo a través de una columna de níquel-quelato. Después de determinar que fracciones contenía proteína diana por SDS-PAGE, las fracciones correspondientes se combinaron para cada muestra y la concentración de cada uno se determinó por una A 280 lectura. La eliminación de la etiqueta 6x His-Smt se llevó a cabo mediante la adición de Ulp1 seguido por diálisis durante la noche y una segunda columna de níquel. La confirmación de la eliminación de etiquetas de His-Smt se llevó a cabo por SDS-PAGE (Figura 6), y cada muestra se concentró con un tubo de centrífuga kDa Amicon Ultra 10. Después de la concentración usando los tubos de centrífuga Amicon Ultra, cada muestra se llevó a cabo sobre una columna de dimensionamiento para lograr la pureza cristalográfica. Una segunda concentración se llevó a cabo para aumentar la concentración de proteína a un nivel necesario para la cristalización. Los catorce construcciones se purificaron con éxito y entró en ensayos de cristalización.
La cristalización se inició por la descongelación previamente frproteína ozen. La cristalización se lleva a cabo en una habitación de clima controlado a 16 º C con placas especialmente diseñadas (Esmeralda Bio) para la difusión de vapor de gota sentado (Figura 7). La selección inicial se realizó con cuatro pantallas de matriz escasa; JCSG +, Pacto, Wizard completo y CryoFull (Emerald Bio), siguiendo una estrategia de Newman extendida. 0,4 l de solución de proteína se mezcló entonces con 0,4 l de crystallant (o solución de reserva) desde el depósito correspondiente usando placas de cristalización Jr de 96 pocillos compactos (Esmeralda Bio). De los catorce muestras purificadas nueve de ellos produjo cristales adecuados para estudios de difracción (Figura 8). Un conjunto de datos de difracción en-casa se recogió en cinco de las nueve construcciones cristalizadas en Cu radiación K alfa de longitud de onda usando un generador de rayos X Rigaku ultrabrillante FR-E + rotativo-ánodo equipado con Osmic VariMAX HF óptica y un detector de Saturno 944 + CCD (Figura 9 ). Cada conjunto de datos se procesa con XDS / XSCALE 4 < / Sup> y ampliarse a una resolución final. Los intentos para resolver las estructuras por reemplazo molecular se llevaron a cabo con Phaser 5 de la CCP4 suite de 7. Los modelos finales se obtuvieron después de refinamiento en REFMAC 7 y reconstrucción manual con focha 11. Las estructuras fueron evaluados y corregidos por la geometría y de la aptitud con MolProbity 9. Se determinó un total de cuatro estructuras de la subunidad PB2 (Figura 10) y se deposita en el AP. Figura 11 ilustra el resultado general en cada etapa de la tubería MTPP.

Figura 1. Descripción general de la gen-a-estructura SSGCID vía para el procesamiento paralelo de múltiples objetivo en el Emerald Bio.
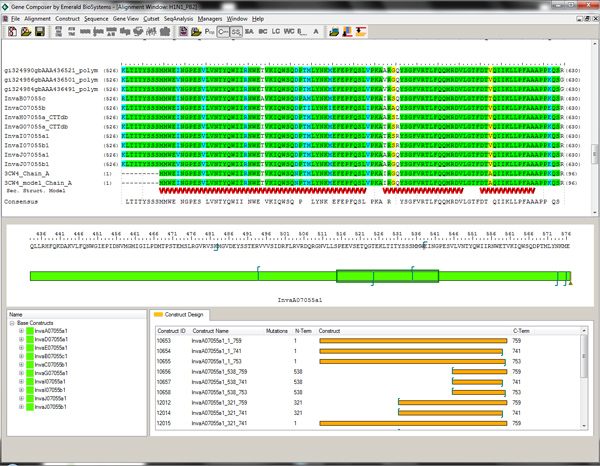
Figura 2. Visor de Alineación y Protein Design Construct módulo de software Compositor Gene. Base de amino-ácido constructo de destino se muestra en verde (ventana del medio) y la estructura de los truncamientos guiadas de construcciones alternativas se muestran en oro (ventana inferior). Un alineamiento de múltiples secuencias de la gripe virales PB2 se muestra en comparación con la secuencia y elementos de estructura secundaria del dominio C-terminal de 3CW4 PDBID. El conocimiento de la estructura de dominios y elementos de estructura secundaria permite truncamientos N-terminal a elección dentro del módulo Diseño Compositor Gene, haga clic en el residuo de aminoácido deseado. Haz clic aquí para ver más grande la figura .
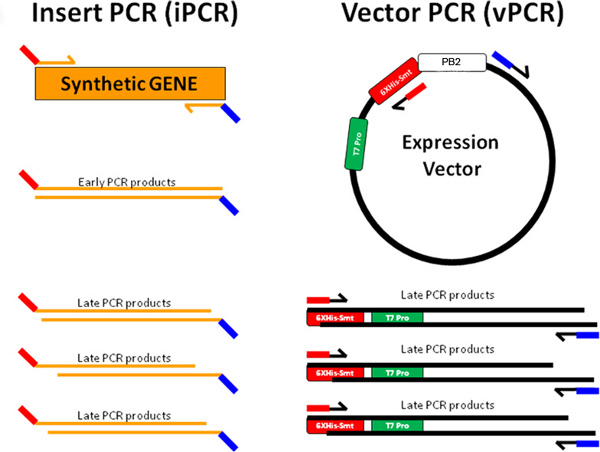
La Figura 3a. TUBO clonación se ilustra en la que la inserción de genes sintéticos (naranja) se amplifica por diseñadas forward (líneas de color rojo-naranja) y atrás primers (líneas naranja y azul) para generar insertar material de PCR. El vector de expresión se amplifica con líneas inversa (rojo-negro ) y forward (líneas de color negro azulado) primers para generar material de vectores PCR. Las secuencias de los productos de iPCR terminales son complementarias a las secuencias terminales de productos vPCR (rojo de iPCR complementa rojo de vPCR y azul de iPCR complementos de azul de vPCR). Esto permite que los productos iPCR y vPCR a hibridan para formar plásmidos que se replican después de la transformación en el huésped BL21 (DE3) de E. químicamente competente células de E. coli.

La Figura 3b. Análisis en gel de agarosa de iPCR producción ts de la subunidad PB2. iPCR fallos pueden ser vistos como bandas débiles o graso, mientras que los productos iPCR exitosos están representados por bandas robustas. la calidad del producto iPCR generalmente se puede correlacionar con el éxito de clonación. Marcadores de peso molecular están en kilodaltons. La figura se reproduce de Raymond et al., 2011 12.

La Figura 4. Pasos de ingeniería de genes de proteínas PB2 objetivo se realizaron utilizando el software Compositor Gene. Después de que se estableció la secuencia de ácido nucleico de ingeniería para cada objetivo, 6-7 construcciones alternativas de proteínas se han diseñado para cada uno. El procesamiento en paralelo de múltiples objetivo en los pasos iniciales de diseño de genes y clonación resultó en 34 construcciones, 14 de los cuales eran objetivos viables que producen proteínas solubles en E. coli.
re 5 "src =" / files/ftp_upload/4225/4225fig5.jpg "/>
Figura 5. Representante análisis de SDS-PAGE de fermentación a gran escala que muestra la expresión de proteína sólida (tamaño esperado de 25,76 kDa), más o menos 50% soluble (carril 4) y alrededor de 50% de escisión de la etiqueta 6x His-Smt de proteína eluida (carril 7).
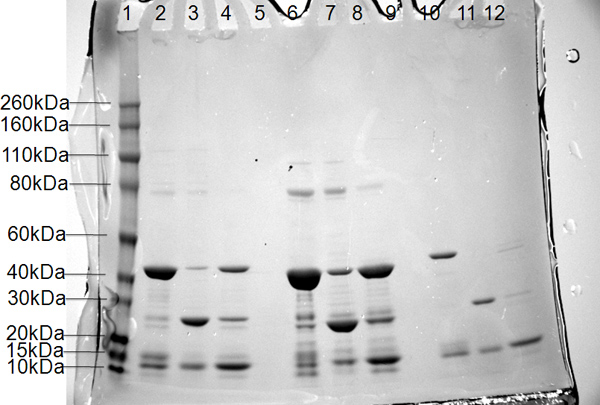
La Figura 6. Resultados de SDS-PAGE para tres constructos de la subunidad PB2 de polimerasa carril 1, marcadores de peso molecular (marcados a la izquierda en kDa);. Carriles 2, 6, y 10, proteína agrupada procedente de la columna de níquel 1; carriles 3, 7, y 11, flujo a través de la proteína escindida en tampón A de níquel 2, carriles 4, 8, y 12, la eliminación de la etiqueta 6x His-Smt en tampón B a partir de níquel 2.
d/4225/4225fig7.jpg "/>
Figura 7. Un esquema de difusión de vapor por el método de la gota que se sienta. El método de la gota que se sienta para la cristalización de proteínas cae dentro de la categoría de difusión de vapor. Este método se basa en una muestra purificada de la proteína y precipitante que se equilibre con un depósito más grande que contiene condiciones similares en una concentración más alta. Cuando el agua se evapora de la muestra de proteína y traslados al depósito, la concentración de precipitación se incrementa a un nivel óptimo para la cristalización de proteínas.

Figura 8. Proteína de cristal de la polimerasa subunidad PB2 de una cepa de virus de la gripe.

La Figura 9. Imagen de difracción de rayos X de la polimerasa subunidad PB2 de uncepa del virus de la influenza.

La Figura 10. Diagramas de cinta de las moléculas en la unidad asimétrica cristalográfica de 4 PB2 estructuras. Estructuras secundarias en el patrón de colores del arco iris con los códigos PDB correspondientes. (A) 3K2V (A/Yokohama/2017/2003/H3N2) (b) 3KHW (A / Mexico / InDRE4487/2009/H1N1) (c) 3KC6 (A/Vietnam/1203/2004/H5N1) (d) 3L56 (A/Vietnam/1203/2004/H5N1).

La Figura 11. El análisis de resultados de la influenza PB2 objetivos de los métodos descritos. La estructuracióntubería de determinación de correo se ilustra en cinco pasos: determinación Clonación, solubilidad, purificación, cristalización y la estructura.
Discusión
Multi-Target procesamiento paralelo
El diseño de fármacos basado en la estructura juega un papel importante en el descubrimiento de fármacos. El SSGCID se dedica a proporcionar a la comunidad científica con estructuras de proteínas en tres dimensiones del NIAID Categoría AC patógenos. Hacer tal información estructural ampliamente disponible en última instancia servirá para acelerar la estructura basada en el diseño de fármacos.
El primer paso crítico del enfoque MTPP es el diseño de constructo. Varias construcciones de cada proteína diana aumenta la probabilidad de éxito de la determinación de la estructura y aumenta la respuesta. Es inevitable que algunas construcciones de proteína se producirá un error en las etapas de la tubería. Aplicación del método de clonación TUBO soporta el método de MTPP por lo que permite la generación de muchas construcciones en formato de 96 pocillos sin pasos de purificación intensivos en mano de obra. Emparejamiento de clonación tubo con la capacidad de analizar la expresión de proteínas en el mismo formato de 96 pocillos (Pinza de laboratorioViruta 90) agiliza aún más el flujo global. El emparejamiento de estos métodos permite la rápida identificación de los constructos que producen proteína soluble que asegura el éxito de la producción de proteínas a gran escala y purificación.
Un aspecto esencial para el éxito de la MTPP de alto rendimiento es el fabricante de la proteína (patente de EE.UU. N º 6.818.060, Esmeralda Bio) instrumento. El fabricante de proteína es un sistema de cromatografía líquida-paralelo de 24 canales diseñado específicamente para aumentar la eficiencia de la producción de proteínas de alto rendimiento y aplicaciones de la investigación genómica estructural de tuberías relacionadas. Utilizando el protocolo descrito anteriormente para el fabricante de la proteína, las ventajas son evidentes en comparación con un único sistema de FPLC de línea. Una sola persona puede purificar hasta 48 objetivos en paralelo dentro de un período de ocho horas. Por el contrario, una sola persona que utiliza un único sistema FPLC línea sólo puede purificar un máximo de cuatro objetivos dentro de ese mismo plazo. Los altos niveles de pureza para cada objetivoconsigue con el Hacedor de proteínas son un factor crítico en el éxito posterior de crecimiento de cristales de proteínas para el análisis de estructuras.
Limitaciones y problemas
Resolución de estructuras tridimensionales mediante cristalografía de rayos X es un esfuerzo multi-etapas con muchos retos, uno de los cuales es la incapacidad para obtener grandes cantidades de proteína diana soluble. Una estrategia que puede ser implementado para superar el problema de solubilidad es el uso de un huésped de expresión E. como alternativa células de E. coli son incapaces de realizar varias importantes modificaciones postraduccionales eucariotas. La expresión en diversos levadura, líneas celulares de insecto y de mamífero que son capaces de llevar a cabo estas modificaciones post-traduccionales son a menudo una alternativa adecuada. Las proteínas diana se expresan a veces, pero completamente insoluble en las condiciones de lisis estándar. El fabricante de proteína puede ser un recurso valioso para la prueba rápida de las condiciones de lisis celular alternativoscomo se describe en Smith et al. 2011 13. Esta estrategia es a menudo necesario para mantener objetivos moviéndose a través de la tubería. En cualquier línea de la genómica estructural, protocolos estandarizados pueden no ser adecuadas para todos los objetivos que viene a través de la tubería y los objetivos pueden necesitar optimización individual. Por ejemplo, se ha optado por utilizar el 20% de etilenglicol por cada crioprotector. En los casos en que esta condición no es adecuado, pueden necesitar ser probado crioprotectores o concentraciones alternativas.
Debido a la naturaleza única de cada objetivo proteína individual, la limitante de la velocidad y el paso impredecible en la determinación de una estructura es la cristalización. Los desplazamientos de tuberías MTPP la frecuencia baja tasa de éxito de cristalización de proteínas con la optimización de las pantallas iniciales matrices dispersas. Cada cristal inicial golpear las pantallas de matriz dispersa disponibles en el mercado se ha optimizado aún más con un generador de E-Screen (Emerald Bio). La pantalla de optimización está diseñado arONIDO la condición de la exitosa cristal inicial, la alteración de las concentraciones de los tampones, sales, y aditivos. Pantallas de optimización exitosos producen cristales adecuados para estudios de difracción y la determinación de la estructura.
El programa de genómica estructural presentado por el Instituto Nacional de Alergias y Enfermedades Infecciosas (NIAID) provee fondos para Emerald Bio y otras tres instituciones del noroeste del Pacífico, que en conjunto son la SSGCID (Emerald Bio, SeattleBiomed, la Universidad de Washington y el Laboratorio Nacional del Pacífico Noroeste) . Cada miembro del consorcio fue seleccionado por su experiencia en la aplicación de tecnologías de última generación necesarios para el cumplimiento de los objetivos del programa de genómica estructural NIAID. Hasta la fecha, SSGCID ha depositado 461 estructuras en el ranking de AP como el séptimo mayor contribuyente en el mundo, y en 2011, la más productiva. Los protocolos y metodologías de la SSGCID se proporcionan con la intención de beneficiar a lacomunidad científica y la perpetuación de la investigación de las enfermedades infecciosas.
Divulgaciones
Los autores son empleados de Emerald Bio, Inc.
Agradecimientos
Los autores desean agradecer a todos los miembros del consorcio SSGCID. El logro de los objetivos de la SSGCID es posible gracias a los enormes esfuerzos de todos los miembros del equipo en Emerald Bio. Esta investigación fue financiada bajo el contrato No. HHSN272200700057C Federal del Instituto Nacional de Alergias y Enfermedades Infecciosas de los Institutos Nacionales de Salud y el Departamento de Salud y Servicios Humanos.
Materiales
| Name | Company | Catalog Number | Comments |
| Primers | IDT | ||
| Genes | DNA 2.0 | ||
| TE buffer | Qiagen | provided in kit | |
| 96-well half skirt PCR plates | VWR | 10011-248 | |
| PFU Master Mix | |||
| 6X Orange Loading Dye | Fermentas | R0631 | |
| 10X TAE | Teknova | T0280 | |
| Agarose | Sigma-Aldrich | A9414-10G | |
| pET28 vector | |||
| 2-YT Broth | VWR | 101446-848 | |
| Kanamycin | Teknova | K2151 | |
| Restriction Enzymes | Fermentas | ||
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Top 10 chemically comp cells | Invitrogen | C4040-06 | |
| Disposable Troughs (Sterile, 25 ml) | VWR | 89094-662 | |
| Airpore covers (Rayon films for bio cultures) | VWR | 60941-086 | |
| 24-well blocks | VWR | 13503-188 | |
| QIAvac 96 | Qiagen | 19504 | |
| BL21(DE3) cells chemcomp (phageR) | NEB | C2527H | |
| 50% Glycerol | VWR | 100217-622 | |
| TB Media | Teknova | T7060 | |
| IPTG | Sigma-Aldrich | ||
| 1 M Tris pH 8.0 | Mediatech | 46-031-CM | |
| 5 M NaCl | Teknova | S0251 | |
| Glycerol | Aldrich | G7893-4L | |
| CHAPS | JT Baker | 4145-01 | |
| Imidazole | Sigma | 56749-1KG | |
| TCEP | Amresco | K831-10G | |
| L-arginine | Amresco | 0877-500G | |
| Benzonase | EMD | 70746-3 | |
| Lysozyme | USB | 1864525GM | |
| 10 kDa MWCO dialysis tubing | Thermo | 68100 | |
| Amicon Ultra 10 ka MWCO concentrators | Millipore | UFC901024 | |
| HisTrap FF columns | GE | 17-5255-01 | |
| HiTrap Chelating columns | GE | 17/0408-01 | |
| Compact Jr crystallization plates | Emerald Bio | EBS-XJR | |
| Crystalization screens | Emerald Bio | ||
| Ethylene Glycol 100% | Emerald Bio | EBS-250-EGLY | |
| Crystal Wand Magnetic Straight | Hampton Research | HR4-729 | |
| Mounted CryoLoop 0.1-0.2 mm | Hampton Research | HR4-955 | |
| ALS style puck | |||
| Puck Wand | |||
| Bent Tongs | |||
| Puck Pusher |
Referencias
- Lowen, A. C., Mubareka, S., Steel, J., Palese, P. Influenza virus transmission is dependent on relative humidity and temperature. PLoS Pathogens. 3 (10), 1470-1476 (2007).
- Yamada, S., et al. Biological and structural characterization of a host-adapting amino acid in influenza virus. PLoS Pathog. 6, e1001034 (2010).
- Lorimer, D., Raymond, A., Walchli, J., Mixon, M., Barrow, A., Wallace, E., Grice, R., Burgin, A., Gene Stewart, L. Composer: database software for protein construct design, codon engineering, and gene synthesis. BMC Biotechnol. 9, 36 (2009).
- Kabsch, W. Integration, scaling, space-group assignment and post-refinement. Acta Cryst. D. 66, 125-132 (2010).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. J. Phaser crystallographic software. Appl. Cryst. 40, 658-674 (2007).
- Kuzuhara, T., Kise, D., et al. Structural basis of the influenza A virus RNA polymerase PB2 RNA-binding domain containing the pathogenicity-determinant lysine 627 residue. J. Biol. Chem. 284, 6855-6860 (2009).
- Murshudov, G. N., Skubàk, P., Lebedev, A. A., Pannu, N. S., Steiner, R. A., Nicholls, R. A., Winn, M. D., Long, F., Vagin, A. A. REFMAC5 for the refinement of macromolecular crystal structures. Acta Cryst. D. 67, 355-367 (2011).
- Cowtan, K. Recent developments in classical density modification. Acta Cryst. D. 66, 470-478 (2010).
- Chen, V. B., Arendall, W. B., Headd, J. J., Keedy, D. A., Immormino, R. M., Kapral, G. J., Murray, L. W., Richardson, J. S., Richardson, D. C. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Cryst. D. 66, 12-21 (2010).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Mol. Cell. 5, 865-876 (2000).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Cryst. D. 66, 486-501 (2010).
- Raymond, A. C., Haffner, T. E., Ng, N., Lorimer, D., Staker, B. L., Stewart, L. J. Gene design, cloning and protein-expression methods for high-value targets at the Seattle Structural Genomics Center for Infectious Disease. Acta Cryst. F. 67, 992-997 (2011).
- Smith, E. R., Begley, D. W., Anderson, V., Raymond, A. C., Haffner, T. E., Robinson, J. I., Edwards, T. E., Duncan, N., Gerdts, C. J., Mixon, M. B., Nollert, P., Staker, B. L., Stewart, L. J. The Protein Maker: an automated system for high-throughput parallel purification. Acta Cryst. F. 67, 1015-1021 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados