Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Captura selectiva de 5-hidroximetilcitosina a partir de ADN genómico
En este artículo
Resumen
Se describe un proceso de etiquetado en dos etapas utilizando β-glucosiltransferasa (β-GT) para transferir una azida-glucosa al 5-HMC, seguido de química para transferir un enlazador de biotina para el enriquecimiento fácil y densidad independiente. Este método de marcaje eficaz y específico permite el enriquecimiento de 5-HMC con fondo extremadamente bajo y de alto rendimiento de mapeo epigenómico a través de secuenciación de próxima generación.
Resumen
5-metilcitosina (5-mC) constituye ~ 2-8% de las citosinas en el total de ADN genómico humano y afecta a una amplia gama de funciones biológicas, incluyendo la expresión génica, el mantenimiento de la integridad del genoma, impronta parental, inactivación del cromosoma X, la regulación de desarrollo, el envejecimiento y el cáncer 1. Recientemente, la presencia de un oxidó 5-MC, 5-hidroximetilcitosina (HMC-5), fue descubierto en células de mamífero, en particular en madre embrionarias (ES) de células y células neuronales 2-4. 5-HMC es generada por la oxidación de 5-mC catalizada por TET familia del hierro (II) / α-cetoglutarato dependiente de dioxigenasas 2, 3. 5-HMC es propuesto para que participen en el mantenimiento de la madre embrionarias (ES) de células, la hematopoyesis normal y tumores malignos, y cigoto desarrollo 2, 5-10. Para comprender mejor la función de 5-HMC, un sistema de secuenciación fiable y sencilla es esencial. Secuenciación de bisulfito tradicionales no pueden distinguir 5 HMC de 5-MC 11 12.
Aquí se describe un sencillo procedimiento en dos etapas para el etiquetado de productos químicos selectivos de 5-HMC. En la primera etapa de etiquetado, 5-HMC en el ADN genómico se marca con un catalizada 6-azida-glucosa por β-GT, una glucosiltransferasa a partir del bacteriófago T4, de una manera que transfiere el 6-azida-glucosa al 5-HMC de la modificado cofactor, UDP-6-N3-Glc (6-N3UDPG). En la segunda etapa de biotinilación,, un enlazador disulfuro de biotina se une al grupo azida por la química clic. Ambas etapas son altamente específicos y eficiente, llevando a completar el etiquetado independientemente de la abundancia de la 5-HMC en regiones genómicas y dando fondo extremadamente bajo. Después de biotinilación de 5-HMC, los fragmentos de ADN de 5 HMC que contienen son entonces selectivamente capturadoutilizando perlas de estreptavidina de una manera independiente de la densidad. El resultante 5-HMC enriquecidos con fragmentos de ADN se podría utilizar para los análisis posteriores, incluyendo secuenciación de próxima generación.
Nuestra etiquetado selectivo y protocolo de captura confiere una alta sensibilidad, aplicable a cualquier fuente de ADN genómico con variables / diversa 5-HMC abundancias. Aunque el propósito principal de este protocolo es su aplicación aguas abajo (es decir., Secuenciación de próxima generación para mapear la distribución de 5-HMC en el genoma), es compatible con una sola molécula, en tiempo real SMRT (ADN) secuenciación, que es capaz de ofrecer una sola base de secuenciación resolución de 5-HMC.
Protocolo
1. Fragmentación de ADN genómico
El fragmento de ADN genómico utilizando sonicación a un intervalo de tamaño deseado adecuado para la plataforma de secuenciación de todo el genoma. (Por lo general, sonicar a ~ 300 pb.) Verificar la distribución del tamaño del ADN genómico fragmentado en 1% de gel de agarosa (Figura 1).
2. Preparación de ADN
Determinar las cantidades de ADN de partida basado en la abundancia de la 5-HMC en el ADN genómico. Dado que los niveles de 5-HMC varían significativamente en diferentes tipos de tejidos, a partir cantidades de ADN dependen de los niveles de 5-HMC de las muestras. Por favor, consulte la Tabla 1 para ver ejemplos.
3. β-GT reacción catalizada (reacción de glucosa Transfer)
- Mezclar pipeteando la mezcla como se detalla en la Tabla 2 y se incuba en un baño a 37 ° C durante 1 hr.
- Después de la incubación, limpiar la reacción con el kit QIAquick eliminación de nucleótidos, usando 10 g de ADN porcolumna. Se eluye con 30 l de agua por columna y combinar.
4. Biotinilación Reacción (Instrucciones Química)
- Añadir DBCO-SS-PEG3-biotina conjugado solución de trabajo (1 mM) en la solución de ADN eluido (de la etapa 3) a una concentración final de 150 mM (es decir, 5 l de solución de trabajo por solución de ADN 30 l).
- Mezclar mediante pipeteo y se incuba en un baño a 37 ° C durante 2 horas.
- Limpie la reacción con el kit QIAquick eliminación de nucleótidos. El volumen ideal de elución total es de 100 l.
- Cuantificar la cantidad de ADN recuperado mediante espectrofotómetro microlitro escala (por ejemplo., NanoDrop).
5. Captura de la 5-HMC que contienen ADN
- Lavar 50 l de Dynabeads MyOne estreptavidina C1 3 veces con 1 ml de 1X tampón B & W de acuerdo con las instrucciones del fabricante. Se separan las perlas con un soporte magnético.
- Añadir un volumen igual de 2X tampón B & W al recuperado D biotiniladoNA (100 ul) a las perlas lavadas.
- Incubar durante 15 min a temperatura ambiente con rotación suave, en un rotador.
- Se separan las perlas con un soporte magnético y lavar las perlas 3 veces con 1 ml de 1X tampón B & W.
- Eluir el ADN incubando las perlas en 100 l de DTT 50 mM recién preparado durante 2 horas a temperatura ambiente con rotación suave, en un rotador.
- Se separan las perlas con un soporte magnético. Aspirar el eluyente y se carga en un Micro Bio-Spin 6 columna de acuerdo con la instrucción de fabricación para eliminar el DTT. El ADN diana se encuentra en la solución ahora.
- Se purifica el ADN eluido de la etapa anterior por Qiagen kit de purificación de PCR MinElute y el ADN se eluye en 10 l de tampón EB. Cuantificar el ADN utilizando fluorómetro Qubit, o NanoDrop si la concentración es superior a 20 ng / ul. El ADN está listo para aguas abajo en todo el genoma preparación de bibliotecas de secuenciación.
6. Los resultados representativos
Si la calidad of ADN genómico es alto, los rendimientos típicos de recuperación después de la β-GT y reacciones biotinilación son ~ 60-70%. Sin embargo, la eficiencia de la captura variar significativamente con tipos de tejidos diferentes en función de los niveles de 5-MCS de las muestras. Típicamente, la eficiencia de la captura de ADN genómico cerebro es de ~ 4.9%, y en algunos casos extremos, la eficiencia puede llegar hasta 12%. Para células madre embrionarias, la eficiencia de la captura promedio es de ~ 2-4%, en contraste con ~ 0,5% para las células madre neurales. La menor eficiencia visto hasta ahora era para el ADN genómico de las células cancerosas. Todo el ADN enriquecido está listo para el estándar de la próxima generación de protocolos de preparación de la biblioteca. Además, el ADN capturado también puede ser utilizado como molde para la PCR en tiempo real para detectar el enriquecimiento de algunos fragmentos en comparación con el ADN de entrada, si los cebadores relacionados están disponibles.
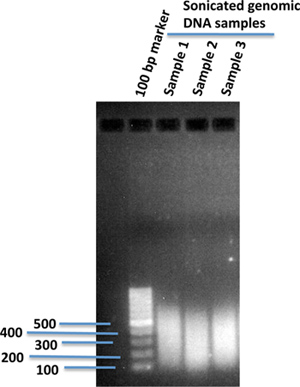
Figura 1. Sonicadas humanos fragmentos de ADN genómico en1% en gel de agarosa. 10 g de ADN genómico aislado de células iPS humanas en 120 l de tampón TE 1X se sometió a ultrasonidos usando un dispositivo de sonicación (Covaris). Después de la sonicación, 2 l de la ADN sonicado se cargó en agarosa al 1% en gel utilizando 100 pb del marcador de ADN para comparar los tamaños de los fragmentos de ADN sometidos a ultrasonidos.
| Componente | Volumen | Concentración final |
| Agua | _ L | |
| 10 X β-GT Reaction Buffer | 2 l | 1 X |
| Hasta 10 g de ADN genómico | _ L | Hasta 500 ng / l |
| UDP-6-N-3 Glc (3 mM) | 0,67 l | 100 mM |
| β-GT (40 mM) | 1 l | 2 uM |
| Volumen total | 20 l |
i) Para el ADN genómico de tejido (mayor de 5 HMC contenido> 0,1%)
| Componente | Volumen | Concentración final |
| Agua | _ L | |
| 10 X β-GT Reaction Buffer | 10 l | 1 X |
| Hasta 20 g de ADN genómico | _ L | Hasta 500 ng / l |
| UDP-6-N3-Glc (3 mM) | 1,33 l | 100 mM |
| β-GT (40 mM) | 2 l | 2 uM |
| Volumen total | 40 l |
ii) Para el ADN genómico de células madre (mediana 5 HMC contenido ~ 0,05%)
| Componente | Volumen | Concentración final |
| Agua | _ L | |
| 10 X β-GT Reaction Buffer | 10 l | 1 X |
| Hasta 50 g de ADN genómico | _ L | Hasta 500 ng / l |
| UDP-6-N3-Glc (3 mM) | 3,33 l | 100 mM |
| β-GT (40 mM) | 5 l | 2 uM |
| Volumen total | 100 l |
iii) En el caso del ADN genómico de las células cancerosas (de menos de 5 HMC contenido de ~ 0,01%)
Tabla 1. Ejemplos de cantidades de ADN de entrada y etiquetado reacciones utilizando las muestras con diferentes niveles de 5-HMC por el método de etiquetado químico selectivo.
| Muestra | 5 HMC nivel | ADN de partida (g) | La recuperación después de etiquetado (entrada a las perlas) (g) | Recuperación de rendimiento | Tire hacia abajo del ADN (ng) | Pull-down producir |
| Cerebelo de ratón adulto | 0,4% | 10 | 7,5 | 75% | 236 | 3,1% |
| Día 7 postnatal ratón cerebelo | 0,1% | 11 | 9 | 82% | 140 | 1,6% |
| Ratón de células ES E14 | 0,05% | 60 | 42 | 70% | 350 | 0,8% |
Tabla 2. Los resultados representativos de los tejidos del cerebro de ratón y células ES.
Access restricted. Please log in or start a trial to view this content.
Discusión
5-hidroximetilcitosina (HMC-5) es un presente identificado recientemente modificación epigenética en cantidades sustanciales en ciertos tipos de células de mamíferos. El método presentado aquí es para determinar la distribución en todo el genoma de la 5-HMC. Usamos bacteriófago T4 β-glucosiltransferasa para transferir un resto de glucosa de ingeniería que contiene un grupo azida en el grupo hidroxilo de la 5-HMC. El grupo azida se pueden modificar químicamente con biotina para la detección, enriquecimiento d...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este estudio fue apoyado en parte por los Institutos Nacionales de la Salud (GM071440 a CH y NS051630/MH076090/MH078972 a PJ).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre | Empresa | Catálogo | Comentario |
| Reactivos | |||
| El cloruro de sodio 5 M (NaCl) | Promega | V4221 | |
| 0,5 M pH 8,0 ácido etilendiaminotetraacético (EDTA) | Promega | V4231 | |
| Trizma base 1 M (Tris), pH 7,5 | Invitrogen | 15567-027) | |
| HEPES 1 M, pH 7,4 | Invitrogen | 15630 | |
| Cloruro de magnesio (MgCl 2) 1M | Ambion | AM9530g | |
| Sulfóxido de dimetilo (DMSO) | Sigma | D8418 | |
| Tween 20 | Fisher BioReagents | BP337-100 | |
| DBCO-SS-biotina-conjugado PEG3 | Haga clic en Herramientas Química | A112P3 | |
| 1,4-ditiotreitol, ultrapura (TDT) superpuro | Invitrogen | 15508-013 | |
| QIAquick Kit de eliminación de nucleótidos | Qiagen | 28304 | |
| Micro Bio-Spin 6 Columna | Bio-Rad | 732-6222 | |
| Dynabeads MyOne | Invitrogen | 650-01 | |
| Estreptavidina C1 | |||
| Qiagen MinElute PCR Purification Kit | Qiagen | 28004 | |
| UltraPure agarosa | Invitrogen | 16500500 | |
| UDP-6-N 3-glucosa | Active Motif | 55013 | |
| Enzima | |||
| β-glucosiltransferasa (β-GT) | New England Biolab | M0357 | |
| Equipo | |||
| La sonicación dispositivo | Covaris | ||
| Escritorio centrífuga | |||
| Baño de agua | Fisher Scientific | ||
| Gel corriendo aparato | Bio-Rad | ||
| NanoDrop1000 | Thermo Scientific | ||
| Labquake Tube Shaker | Barnstead | ||
| Labquake Tube Shaker | Thermolyne | ||
| Soporte Separación Magnética | Promega | Z5342 | |
| Qubit 2,0 Fluorómetro | Invitrogen | ||
| Reactivo de configuración 10 X β-GT de tampón de reacción (500 mM de HEPES pH 7,9, 250 mM de MgCl 2) 2 X Encuadernación y lavado (B & W) tampón (10 mM Tris pH 7,5, 1 mM EDTA, 2 M de NaCl, 0,02% Tween 20) . |
Referencias
- Jaenisch, R., Bird, A. Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nat. Genet. , Suppl 33. 245-254 (2003).
- Ito, S. Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification. Nature. 466, 1129-1133 (2010).
- Tahiliani, M. Conversion of 5-methylcytosine to 5-hydroxymethylcytosine in mammalian DNA by MLL partner TET1. Science. 324, 930-935 (2009).
- Kriaucionis, S., Heintz, N. The nuclear DNA base 5-hydroxymethylcytosine is present in Purkinje neurons and the brain. Science. 324, 929-930 (2009).
- Ko, M. Impaired hydroxylation of 5-methylcytosine in myeloid cancers with mutant TET2. Nature. 468, 839-843 (2010).
- Koh, K. P. Tet1 and tet2 regulate 5-hydroxymethylcytosine production and cell lineage specification in mouse embryonic stem cells. Cell Stem Cell. 8, 200-213 (2011).
- Iqbal, K., Jin, S. G., Pfeifer, G. P., Szabo, P. E. Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine. Proceedings of the National Academy of Sciences of the United States of America. 108, 3642-3647 (2011).
- Wossidlo, M. 5-Hydroxymethylcytosine in the mammalian zygote is linked with epigenetic reprogramming. Nat. Commun. 2, 241(2011).
- Gu, T. P. The role of Tet3 DNA dioxygenase in epigenetic reprogramming by oocytes. Nature. 477, 606-610 (2011).
- Dawlaty, M. M. Tet1 is dispensable for maintaining pluripotency and its loss is compatible with embryonic and postnatal development. Cell Stem Cell. 9, 166-175 (2011).
- Huang, Y. The behaviour of 5-hydroxymethylcytosine in bisulfite sequencing. PLoS One. 5, e8888(2010).
- Song, C. X. Selective chemical labeling reveals the genome-wide distribution of 5-hydroxymethylcytosine. Nat. Biotechnol. 29, 68-72 (2011).
- Pastor, W. A. Genome-wide mapping of 5-hydroxymethylcytosine in embryonic stem cells. Nature. 473, 394-397 (2011).
- Matarese, F., Pau, C. arrillo-deS. anta, E,, Stunnenberg, H. G. 5-Hydroxymethylcytosine: a new kid on the epigenetic block. Mol. Syst. Biol. 7, 562(2011).
- Szwagierczak, A., Bultmann, S., Schmidt, C. S., Spada, F., Leonhardt, H. Sensitive enzymatic quantification of 5-hydroxymethylcytosine in genomic DNA. Nucleic Acids Res. 38, 181(2010).
- Terragni, J., Bitinaite, J., Zheng, Y., Pradhan, S. Biochemical characterization of recombinant β-glucosyltransferase and analysis of global 5-hydroxymethylcytosine in unique genomes. Biochemistry. , (2012).
- Rusmintratip, V., Sowers, L. C. An unexpectedly high excision capacity for mispaired 5-hydroxymethyluracil in human cell extracts. Proc. Natl. Acad. Sci. U.S.A. 97, 14183-14187 (2000).
- Globisch, D. Tissue distribution of 5-hydroxymethylcytosine and search for active demethylation intermediates. PLoS One. 5, e15367(2010).
- Yildirim, O. Mbd3/NURD Complex Regulates Expression of 5-Hydroxymethylcytosine Marked Genes in Embryonic Stem Cells. Cell. 147, 1498-1510 (2011).
- Szulwach, K. E. Integrating 5-hydroxymethylcytosine into the epigenomic landscape of human embryonic stem cells. PLoS Genet. 7, e1002154(2011).
- Szulwach, K. E. 5-hmC-mediated epigenetic dynamics during postnatal neurodevelopment and aging. Nat. Neurosci. 14, 1607-1616 (2011).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados