Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
ARN de predicción de estructura secundaria Utilizando SHAPE de alto rendimiento
En este artículo
Resumen
De alto rendimiento selectiva 2 'acilación hidroxilo analizados por extensión del cebador (FORMA) utiliza un software de predicción de estructura secundaria de producto químico nuevo sondeo de la tecnología, la transcripción inversa, electroforesis capilar y para determinar las estructuras de ARN de varios cientos a varios miles de nucleótidos en una sola resolución de nucleótidos.
Resumen
Comprender la función del ARN que participan en los procesos biológicos exige un conocimiento profundo de la estructura del ARN. Con este fin, la metodología denominada "acilación hidroxilo de alto rendimiento 2 selectivo 'analizado mediante extensión de cebador", o forma, permite la predicción de la estructura secundaria de ARN con una sola resolución de nucleótidos. Este enfoque utiliza agentes químicos de sondeo que preferentemente acilato de regiones de cadena sencilla o flexible de ARN en solución acuosa. Sitios de modificación química se detectan mediante transcripción inversa del ARN modificado, y los productos de esta reacción son fraccionados por electroforesis capilar automatizado (CE). Dado que la transcriptasa inversa se detiene en esos nucleótidos de ARN modificados por los reactivos de la forma, la biblioteca de cDNA resultante asigna indirectamente estos ribonucleótidos que son de cadena sencilla en el contexto de los ARN plegadas. El uso de software ShapeFinder, los producidos por electroferogramas automatizado CE son procesados y convertidos en nutablas de reactividad cleotide que se convierten ellos mismos en las limitaciones del pseudo-energía utilizadas en el RNAStructure (v5.3) algoritmo de predicción. Las estructuras de ARN de dos dimensiones obtenidos por combinación de una forma sondeo con ARN in silico de predicción de estructura secundaria se han encontrado para ser mucho más precisa que la estructuras obtenidas usando cualquier método solo.
Introducción
Para comprender las funciones de los ARN catalíticos y no codificantes implicadas en la regulación del corte y empalme, traducción, replicación del virus y el cáncer, se requiere un conocimiento detallado de la estructura del ARN 1,2. Desafortunadamente, la predicción exacta de ARN plegado presenta un reto formidable. Agentes de sondeo clásicas sufren de muchas desventajas tales como la toxicidad, la cobertura incompleta de nucleótidos y / o un caudal limitado a 100-150 nucleótidos por experimento. Algoritmos de predicción de estructura secundaria sin ayuda son igualmente desventajoso, debido a las inexactitudes que resultan de su incapacidad para distinguir efectivamente entre estructuras energéticamente similares. Grandes de ARN, en particular, también son a menudo refractario a los métodos de determinación de la estructura 3D, tales como la cristalografía de rayos X y resonancia magnética nuclear (RMN), debido a su flexibilidad conformacional y grandes cantidades de muestras muy puras requeridas para estas técnicas.
HFORMA igh rendimiento resuelve muchos de estos problemas al proporcionar un enfoque eficaz, sencilla de sondeo de las grandes estructuras de ARN a una resolución de un solo nucleótido. Por otra parte, los reactivos utilizados para la forma son seguros, fáciles de manejar y, en contraste con la mayoría de otros productos químicos de sondeo reactivos, reaccionan con los cuatro ribonucleótidos. Estos reactivos también pueden penetrar en las membranas celulares, por lo que es posible para sondear RNAs en su contexto in vivo (s) 3. Originalmente desarrollado en las Semanas de laboratorio 4, la forma ha sido utilizado para analizar una amplia variedad de ARN, el ejemplo más notable es la determinación de la estructura secundaria completa del ~ 9 kb VIH-1 ARN del genoma 5. Otros logros notables incluyen SHAPE utilizando elucidación de las estructuras de los viroides infecciosas 6, ARNs no codificantes largas humanos 7, ribosomas de levadura 8 y 9 riboswitches así como para identificar los sitios de unión de proteínas de virión asociada a VIH-1 RNA 3. While las variaciones originales y de alto rendimiento del protocolo SHAPE se han publicado en otros lugares 10 a 12, el presente trabajo ofrece una descripción detallada de la determinación de la estructura secundaria del ARN por la forma de alto rendimiento utilizando oligonucleótidos fluorescentes, la Beckman Coulter CEQ 8000 Genetic Analyzer y software SHAPEfinder y RNAStructure (v5.3). Detalles técnicos inéditas y consejos de solución de problemas también se incluyen.
Las variaciones de la forma
La esencia de la forma y sus variaciones es la exposición de ARN en solución acuosa a anhídridos electrófilos que acilar selectivamente grupos ribosa 2'-hidroxilo (2'-OH), produciendo aductos voluminosos en los sitios de modificación. Esta reacción química sirve como un medio de interrogar a la dinámica estructural de ARN locales, como los nucleótidos de cadena simple son más propensas a adoptar conformaciones favorables para ataque electrofílico por estos reactivos, mientras que la base emparejado o arquitectónicamente Constrnucleótidos objetivó son menos o no reactivo 10. Los sitios de formación de aductos se detectan mediante transcripción inversa a partir de la iniciación fluorescentemente o radiomarcado cebadores hibridados a un sitio específico en el ARN modificado (el "(+)" reacción de extensión del cebador). Cuando la transcriptasa inversa (RT) no atravesar los ribonucleótidos acilados, un grupo de productos de ADNc se produce cuyas longitudes coinciden con los sitios de modificación. Un control, "(-)" cebador de extensión reacción utilizando ARN que no ha sido expuestos a reactivo también se realiza de modo que la terminación prematura de la síntesis de ADN (es decir, "se detiene") debido a la estructura del ARN, no específica de ARN de hebra rotura, etc, mayo. distinguirse de deteniéndose producido por modificación química. Por último, dos reacciones de secuenciación didesoxi-inician a partir de los mismos cebadores se utilizan como marcadores para correlacionar nucleótidos reactivos con la secuencia primaria de ARN después de la electroforesis.
En la solicitud original de SHAPE, el mismo 32-P marcado en el extremo cebador se utiliza para los (+), -, y dos reacciones de secuenciación (). Los productos de estas reacciones se cargan en pocillos adyacentes en una placa de gel de poliacrilamida al 5-8%, y se fraccionó por electroforesis en gel desnaturalizante de poliacrilamida (PAGE; Figura 1). El análisis cuantitativo de las imágenes de gel producidas por la forma convencional se puede realizar utilizando SAFA, un software de análisis de la huella de semi-automatizado 13.
Por el contrario, la forma de alto rendimiento emplea cebadores marcados con fluorescencia y electroforesis capilar automatizado. Específicamente, para cada región de ARN bajo investigación, un conjunto de cuatro cebadores de ADN que tienen una secuencia común pero diferentes 5 'etiquetas fluorescentes deben ser sintetizados o comprados. Estos oligonucleótidos marcados de manera diferente-sirven para dos reacciones principales forma y dos reacciones de secuenciación, los productos de los cuales se agrupan y se fraccionó / detectaron por electroforesis capilar automatizado (CE). WherEAS el perfil de reactividad de los nt 100-150 de ARN se puede obtener a partir de un conjunto de cuatro reacciones utilizando el enfoque original, la forma de alto rendimiento permite la resolución de 300-600 nt a partir de una sola muestra agrupada 3. Hasta 8 conjuntos de reacciones puede ser fraccionado simultáneamente, mientras que tanto como 96 muestras pueden preparado para el fraccionamiento en el transcurso de 12 ejecuciones consecutivas de la CE (Figura 2). Por otra parte, el software SHAPEfinder, desarrollado para procesar y analizar los datos que se desprenden del CEQ y otros analizadores genéticos, es más automatizado y requiere mucho menos intervención del usuario de SAFA 13 u otros paquetes de gel de análisis.
Más avanzadas metodologías de alto rendimiento han surgido recientemente tales como PARS (análisis paralelo de la estructura del ARN) 14 y Frag-Seq (fragmento de secuenciación) 15, que utilizan enzimas específicos de la estructura en lugar de reactivos de alquilación en conjunción con técnicas de secuenciación de próxima generación para obtener information sobre la estructura del ARN. A pesar del atractivo de estas técnicas, las muchas limitaciones inherentes a la nucleasa de sondeo siguen siendo 16. Estos problemas pueden ser evitados en la secuenciación FORMA (SHAPE-Seq) 17 protocolo, donde la secuenciación de próxima generación está precedida por modificación química y la transcripción inversa del ARN de una manera similar a la realizada en la forma convencional. Si bien estos métodos pueden representar el futuro de la determinación de la estructura de ARN, es importante recordar que la secuenciación de próxima generación es muy caro, y sigue sin estar disponible para muchos laboratorios.
Análisis de datos SHAPE
Los datos producidos en el analizador genético se presentan en la forma de un electroferograma, en el que la intensidad de fluorescencia de la muestra (s) que fluye a través del detector capilar se representa en función de un índice de tiempo de migración. Esta parcela tiene la forma de las huellas superpuestas, según el canal cuatro de fluorescencias utiliza para detectar los diferentes fluoróforos, y donde cada traza se compone de los picos correspondientes a los productos de ADNc o secuenciación individuales. Datos electroferograma se exporta desde el analizador genético como un archivo de texto delimitado por tabuladores y se importan en la transformación ShapeFinder y software de análisis 18.
ShapeFinder se utiliza inicialmente para llevar a cabo una serie de transformaciones matemáticas en los datos para asegurar que los tiempos de migración y volúmenes pico reflejan con precisión las identidades y cantidades de los productos de reacción, respectivamente. Los picos son entonces alineados e integrados, y los resultados se tabulan junto con la secuencia de ARN primario. Un "perfil de reactividad" para el segmento pertinente de ARN se obtiene restando los valores de control a partir de la (+) valores asociados con cada ARN de nucleótidos, y la normalización de los datos tal como se describe a continuación. Este perfil se importa en RNAstructure (v5.3) Software de 19,20, lo que convierte la val reactividad normalizadaUES en limitaciones pseudo-energía que se incorporan en el algoritmo de plegado estructura secundaria del ARN. La combinación química de sondeo y plegado algoritmos de esta manera mejora significativamente la exactitud de la predicción de la estructura en comparación con cualquiera de los métodos solos 12,21. La salida de RNAstructure (v5.3) incluye imágenes de la energía más baja ARN estructuras secundarias con código de colores con el perfil FORMA reactividad (s), así como las mismas estructuras en Pruebas notación de punto-soporte. Este último puede posteriormente ser exportado al software dedicado a la representación gráfica de la estructura secundaria del ARN tales como Varna PseudoViewer 22 y 23.

Figura 1. Organigrama de la estructura de la determinación de ARN a través de SHAPE 4,10. (A) RNA may se obtiene a partir de muestras biológicas o por transcripción in vitro. (B) Dependiendo de la fuente, el ARN se pliega o se procesa y se modificó con el reactivo FORMA lo contrario. (C) La transcripción inversa usando cebadores marcados radiactivamente o fluorescentemente. (D) son productos de ADNc fraccionado ya sea a través de electroforesis capilar a base de gel o de poca altura. (E) Análisis de fragmentos. (F) la predicción de estructura de ARN. Haz clic aquí para ver más grande la figura.
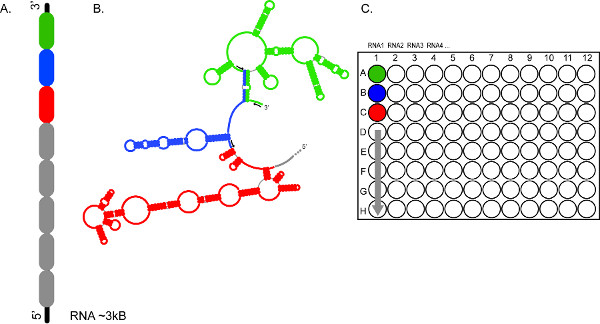
Figura 2. El carácter de alto rendimiento de FORMA basados en CE permite el análisis rápido de múltiples ARN, y / o múltiples segmentos de los mismos ARN. (A) Representa cómo una RNA puede ser dividido en secciones 300-600 nt (código de color en verde, azul y rojo) (B) Las secciones del ARN se probaron de forma independiente utilizando diferentes conjuntos de cebadores fluorescentes (flechas negras) (C) juegos de reacciones se combinaron y se cargaron en los pocillos A1, B1, C1, etc, respectivamente, proporcionando una cobertura completa para el ~ 3 kb ARN1. Productos de reacción de RNAs 2, 3, 4, etc pueden prepararse de forma similar para el fraccionamiento de carreras electroforéticas consecutivos. Haz clic aquí para ver más grande la figura.
Protocolo
Diseño de cebadores y extensión de la terminal del ARN 3 '
Para el análisis de ARN de largo por FORMA de alto rendimiento, una serie de sitios de hibridación de cebadores debe ser seleccionado de tal manera que (i) están separados por ~ 300 nt, (ii) son 20-30 nt de longitud, y (iii) que el ARN / híbridos de ADN producidos por el recocido de ADN a estos sitios tienen una temperatura de fusión esperada de> 50 ° C. Además, los segmentos de ARN que se predicen para ser altamente estructurado se deben evitar, a pesar de hacer tal determinación requiere algo de conocimiento previo de la estructura de ARN, que es a menudo no está disponible. Cebadores de ADN que se hibridan a estos sitios a continuación, deben ser diseñados, teniendo cuidado de asegurarse de que no se espera que para formar dímeros estables o estructuras secundarias intrastrand.
Una vez diseñado, conjuntos de cebadores deben ser comprados (por ejemplo, de Integrated DNA Technologies, Ames, Iowa) o sintetizados 24,25. Los cebadores 5'-etiquetados con Cy5, Cy5.5,WellRedD2 (Beckman Coulter) y IRDye800 (Lycor) / WellRedD1 (Beckman Coulter) son los más adecuados para el Beckman Coulter CEQ 8000, proporcionando una buena intensidad de la señal y reducir al mínimo la interferencia. Oligonucleótidos marcados se pueden almacenar indefinidamente en pequeñas partes alícuotas de 10 micras, con -20 ° C, evitar los ciclos de congelación / descongelación repetidos.
Mediante el uso de cebadores diseñados de esta manera, es posible obtener datos de la forma para prácticamente todo un ARN de cualquier longitud. Sin embargo, la secuencia en o cerca del extremo 3 'de un ARN es siempre inaccesibles a la forma, a menos que el ARN está diseñado para contener una extensión 3' terminal (por ejemplo un "casete de estructura") a la que un cebador puede hibridar 4.
Preparación de ARN a través de Electroforesis Capilar
Aunque los ARN de las muestras biológicas pueden ser utilizados para la forma de alto rendimiento, el protocolo dado aquí está optimizado para el ARN producido por la transcripción in vitro. Comercial trakits de nscription tales como MEGAshortscript (Ambion) se utiliza junto con MegaClear columnas de purificación de ARN (Ambion) se adaptan bien a la generación de grandes cantidades de ARN puros. ARN se deben almacenar en tampón TE entre -20 ° C y -80 ° C. Para obtener los mejores resultados, los ARN deberían aparecer homogénea tanto por electroforesis en gel de poliacrilamida desnaturalizante y no desnaturalizante.
1. ARN plegable
- En un tubo de microcentrífuga de 0,5 ml, diluir 12 pmol de ARN de 18 l con agua y añadir 2 l de tampón de renaturalización 10 veces. Mezclar bien.
- Calentar a 85 ° C durante 1 min, después se enfría a 4 ° C a una velocidad de 0,1 ° C / seg.
- Añadir 100 l de agua y 30 l de tampón de plegamiento 5X.
- Incubar a 37 ° C durante 30-60 minutos, dependiendo de los ARN de ser doblado. En general, Mg 2 + dependientes de plegamiento de ARN más largas, y más estructurado requiere tiempos de incubación más largos.
- Transferir una alícuota de 72 l a cada uno de dos tubos de microcentrífuga de 0,5 ml: Modified (+) y de control (-).
2. Modificación química del ARN
Bueno caracteriza, reactivos electrófilos SHAPE incluyen anhídrido isatoico (IA), el anhídrido N-metilisatoico (NMIA), anhídrido 1-metil-7-nitro-isatoico (1M7) 26, y cianuro de benzoílo (BzCN) 27. De ellos, los más utilizados para la forma de alto rendimiento son 1M7 y NMIA, y sólo este último se encuentra disponible comercialmente (Life Technologies). La concentración final de la modificación de reactivo debe ser optimizada para cada ARN para obtener "un solo ataque" cinética de modificación, es decir, la condición en la que la mayoría de los ARN en solución se modifican una vez en la región de ARN que se analiza 11. Esta concentración óptima se puede determinar mediante la realización de múltiples reacciones en las que la concentración de reactivo es variada en toda la gama (s) indicados en la tabla en la Sección 2.1 a continuación. Usar la concentración de reactivo que produce una señal fácilmente detectable mientras minimizing la diferencia en la intensidad de la señal entre los productos de síntesis de ADN largas y cortas (por ejemplo, Figura 3).
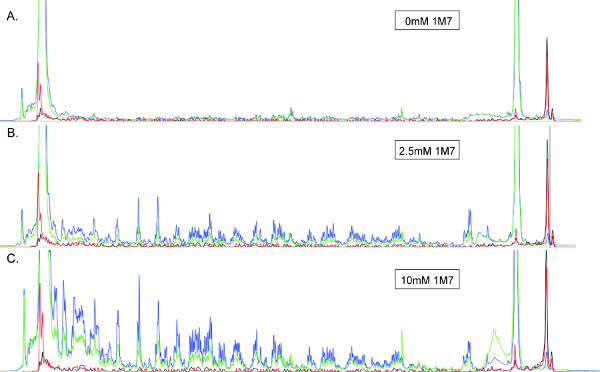
Figura 3. Electroferogramas SHAPE producidos a partir de un ~ 360 nt ARN tratado con (A) 0 (B) 2,5 mM o (C) 10 mM 1M7. Todos los electroferogramas se muestran en la misma escala. Azul, huellas verde, rojo y negro corresponden a (+) productos de reacción (Cy5), (-) productos de reacción (Cy5.5), y las dos escaleras de secuenciación (D2 WellRed y IRDye800), respectivamente. El ARN se utilizan para producir la imagen (B) se ha tratado con la cantidad óptima de 1M7, lo que demuestra una buena resolución máxima y la intensidad, con una mínima caída de la señal a lo largo de la traza (izquierda). Leer longitud es máxima en estas condiciones. En contraste, la ausencia de int medioensity, picos bien resueltos en (A) sugiere una concentración subóptima de 1M7. Por el contrario, la caída de la señal evidente en (C) indica que solo la cinética de ataque no se observa, y el ARN es más de-modificado. En tales casos, sobre todo cuando no se espera RT para encontrarse con el extremo 5 'de la plantilla de ARN, leer longitud será subóptima.
- Prepare valores 10 veces de reactivo SHAPE (NMIA o 1M7). Esto se consigue mejor mediante la adición de una pequeña cantidad de reactivo a un tubo de microcentrífuga de 1,5 ml, a continuación, la adición de DMSO para alcanzar la concentración deseada Atención:. Soluciones de reactivo forma debe permanecer anhidro hasta que se mezcle con el ARN. Tienda DMSO en un desecador a temperatura ambiente, y preparar soluciones madre inmediatamente antes de su uso con el fin de minimizar la exposición a vapor de agua a temperatura ambiente.
Reactivo La concentración óptima de 10 veces (en DMSO) Tiempo parala degradación completa de reactivo 27 NMIA 10-100 mM ~ 20 min 1M7 10-50 mM 70 seg
Tabla 1. Reactivos electrófilos utilizados para la modificación del ARN. - Añadir 8 l de DMSO 10 veces NMIA/1M7 o anhidro para (+) modificado y el control (-) se mezcla, respectivamente Nota:. 2,5 mM ha demostrado ser una concentración de partida eficaz para tanto NMIA y 1M7, independientemente de la ARN que se analiza.
- Incubar a 37 ° C durante 50 min (NMIA) o 5 minutos (1M7), según sea apropiado.
- Precipitar el ARN mediante la adición de 8 l (0,1 vol) de NaOAc 3 M (pH 5,2), 8 l 100 mM de EDTA, 240 l (3 vol) de etanol frío y 1 l 10 mg / ml de glucógeno. Refrigerar durante 2 horas y después centrifugar a 14.000 xg durante 30 min a 4 ° C. Lavar la pella dos veces con etanol frío al 70% Atención:. Es importanteminimizar el tiempo de refrigeración, el tiempo y la velocidad de centrifugación con el fin de minimizar la co-precipitación de la sal, ya que esto puede afectar negativamente a pico de resolución durante la electroforesis.
- Aspirar el sobrenadante con una micropipeta, y el aire de gránulo seco durante 5 min a temperatura ambiente.
- Disolver ARN precipitados en tampón TE 10 l e incubar 5 min a temperatura ambiente. Esto es lo suficientemente disuelto RNA por dos reacciones de transcripción inversa. Guarde la parte no utilizada a -20 ° C. Atención: resuspensión Mecánica de la pastilla no suele ser necesario, y puede dañar el ARN.
3. La transcripción inversa
Este paso genera los productos de ADNc marcadas con fluorescencia que se utilizan para identificar indirectamente el grado en que los nucleótidos de ARN han sido modificados por un reactivo FORMA. Para la forma, el rendimiento de Superíndice III (Invitrogen) RT fue superior a todas las demás probada RTS, y es la enzima elegido para su uso con esteprotocolo. Los oligonucleótidos marcados con Cy5 y Cy5.5 se utilizan para cebar el (+) y (-) reacciones, respectivamente. Para los ARN más cortas, los cebadores se hibridan con una extensión 3 'terminal del ARN nativo (por ejemplo, un "casete de estructura") con el fin de obtener información sobre el 3' terminal 4 Atención:. Desde este punto a través del CE, las muestras deben ser protegidos de luz.
- Preparar (+) y (-) para la transcripción reversa muestras en tubos de microfuga de 0,5 ml. Para la reacción de RT (+), mezcle 5 l de ARN modificado (+), 6 l de agua y 1 l cebador marcado con Cy5 (10 M), porque el (-) la reacción RT, mezcle 5 Control de l de ARN (-), . 6 l de agua, y 1 l cebador marcado con Cy5 (10 mM) Atención: Sarstedt tubos PCR (REF 72.735.002) se recomienda para esta aplicación.
- Colocar los tubos en un termociclador y Anneal cebador de ARN y prepararse para la transcripción inversa mediante la aplicación del siguiente programa: 85 ° C, 1 min, 60 ° C, 5 min;35 ° C, 5 min, 50 ° C, sostiene.
- Durante la etapa de recocido, preparar 2,5 X RT mezcla suficiente para el número de reacciones que se deben realizar, más el 50% (por ejemplo, para dos (+) y dos (-) reacciones, escala de 4,5 veces). Una reacción requiere 8 l, de la siguiente manera: 4 l 5x tampón de RT, 1 l 100 mM DTT, 1,5 l de agua, 1 l dNTP 10 mM, 0,5 l superíndice III RT. Mantenga en hielo Atención. Tampón RT 5X y DTT 100 mM se proporcionan con el III RT superíndice.
- Una vez que la temperatura de las mezclas de recocido alcanza 50 ° C, añadir 8 l de 2,5 veces el RT de la mezcla de los (+) y (-) reacciones Recomendación:. Caliente la mezcla de RT a 37 ° C durante 5 min antes de añadir a las reacciones .
- Incubar durante 50 min a 50 ° C, después se enfría a 4 ° C y / o el lugar en el hielo Nota:. La incubación de las reacciones de RT durante más de 50 minutos puede dar lugar a productos de ADNc aberrantes.
- Hidrolizar ARN mediante la adición de 1 l de NaOH 4 M y calentando a 95 ° C durante 3 min. Reacciones frescos en hielo y después se neutralizan mediante la adición de 2 l de HCl 2 M Atención:. La omisión de este paso resulta en la separación mala calidad de los productos de ADNc.
- Combinar (+) y (-) reacciones y precipitar el ADNc mediante la adición de 0,1 volúmenes de NaOAc 3 M, 0,1 vol de 100 mM de EDTA, 1,5 vol de etanol frío y 1 l de 10 mg / ml de glucógeno. Refrigerar durante 2 horas y después centrifugar a 14.000 xg durante 30 min a 4 ° C. Lavar el pellet dos veces con etanol frío al 70% Atención:. Centrifugación a tasas más altas o durante un período más largo resulta en dificultades para resuspender el pellet (s).
- Resuspender ADNc sedimentaron en 40 l de formamida desionizada por calentamiento a 65 ° C durante 10 min, seguido de agitación vigorosa durante más de 30 min Atención:. Pellets puede ser invisible. La falta de señal o señal débil después de la electroforesis puede ser el resultado de la insuficiencia adecuadamente para disolver el precipitado en esta etapa.
Escaleras de secuenciación sirven como marcadores para la determinación de la posición de nucleótido durante el procesamiento de datos. Estos se generan utilizando un kit USB Ciclo de Secuenciación (# 78500), ADN que tiene la misma secuencia que el ARN que se estudian, y los cebadores etiquetados con D2 WellRed o D1/Lycor 800. Típicamente, el ADN utilizado en esta reacción será que utiliza como una plantilla para la transcripción del ARN en cuestión. Aunque el protocolo de reacción que aquí se presenta se parece mucho a la recomendada por el fabricante del kit, la reacción se escala hasta varias veces. Mientras ddA y el DDT se utilizan como terminadores de cadena en las reacciones descritas a continuación, cualquier par de terminadores puede ser utilizado para generar las escaleras de secuenciación.
- Mezclar 40 l de la mezcla de terminación ddA, 5 pmol de molde de ADN, 4,6 l de tampón Sequenase 10 veces, 10 l de WellRed cebador marcado D2, 4,6 l de Sequenase y agua para llevar el volumen total a 82 l. Agregue la Sequenase pasado. Prepavolver a una segunda reacción de secuenciación de la misma manera, la utilización de DDT y Licor IR800 cebador marcado en su lugar.
- Proceder a la amplificación de PCR usando las condiciones recomendadas USB Atención:. No se requiere la adición de aceite mineral ni se recomienda para los protocolos / termocicladores que utilizan una tapa térmica.
- Combine el PDD y ddT reacciones de secuenciación en un tubo de microcentrífuga de 1,5 ml (~ 164 l en total).
- Precipitar el ADN de la siguiente manera: Añadir 16 l de NaOAc 3 M (pH 5,2), 16 mM de EDTA 100 l, 1 l 10 mg / ml de glucógeno, y 480 l de etanol al 95%. Mezclar bien, se incuba a 4 ° C durante 30 min y se centrifuga a 14.000 xg durante 30 min a 4 ° C.
- Resuspender ADNc sedimentaron en 100 l de formamida desionizada por calentamiento a 65 ° C durante 10 min, seguido de agitación vigorosa durante al menos 30 min.
5. Fraccionamiento de los productos de reacción por electroforesis capilar
La electroforesis capilar permite simultáneaseparación de los productos de síntesis de cDNA de cuatro reacciones agrupados en una sola muestra. Ocho muestras pueden ser fraccionado simultáneamente, mientras que tanto como 96 muestras pueden ser fraccionada durante una sola pasada (Figura 2).
- Mezclar 40 l de muestras SHAPE agrupados con 10 l de las escaleras de secuenciación agrupados, y la transferencia de muestra a las placas de 96 pocillos Atención:. Es imperativo que Beckman reactivos y las placas (incluyendo LPA-I de gel, tampón, aceite mineral, muestra Coulter solución de carga y de la muestra y las placas de tampón) pueden utilizar con el Beckman-Coulter CEQ 8000 Genetic Analyzer.
- Programa y preparar instrumento de electroforesis capilar e iniciar ejecutar según las instrucciones del fabricante Nota:. Para una mejor resolución de las muestras, utilice los parámetros del método CAFA previamente publicados 28.
Lo ideal sería que fuera de la cartilla y los picos fuertes de parada, señales para cada pico en los cuatro electroferograma trazas deben estar en el rango lineal, una bajada gradual de la señal es aceptable. A veces, sin embargo, grandes picos (paradas) son evidentes incluso en la reacción de control, y éstos pueden interferir con el procesamiento posterior de los datos. ADNc truncados que dan lugar a estos picos pueden ser el resultado de un obstáculo natural durante la transcripción inversa (por ejemplo, estructura secundaria del ARN), o la degradación del ARN. En el primer caso, aditivos, tales como betaína pueden mejorar la capacidad de procesamiento y reducir RT RT pausa de terminación / prematura.
Proceso de datos
ShapeFinder software permite al usuario visualizar y transformar las huellas de la CE y convertirlos en perfiles de reactividad forma 18. Una vez que los valores de reactividad se tabulan, se normalizan y se importan en RNAStructure (v5.3) para generar y refinar los modelos estructurales secundarios.
6. ShapeFinder Software
Una extensión de la BaseFinder rastro procesamiento platformulario 29, la versión publicada de ShapeFinder está disponible gratuitamente para uso no comercial 18. Las instrucciones detalladas para el manejo de datos en ShapeFinder cuentan con la documentación del software.
- Electroferogramas son importados de la CEQ en ShapeFinder, donde se ajustan para corregir (i) de fondo fluorescente, (ii) la superposición espectral entre canales fluorescentes, (iii) la movilidad turnos impartida por primers etiquetados de manera diferente, (iv) las diferencias en la intensidad de fluorescencia de productos comunes etiquetados con diferentes fluoróforos, y (v) la caída de la señal resultante de la terminación prematura de la transcripción inversa.
- La función de "Configuración" de la función "Alinear e integrar" en ShapeFinder asigna automáticamente las identidades de los picos individuales y se relaciona esto con la secuencia de ARN según la definición de la entrada del usuario y las dos escaleras de secuenciación. Aunque las asignaciones iniciales son generalmente imperfecto, los errores se pueden corregir manualmente utilizando la función "Modificar" dela misma herramienta. Por último, la función de "Fit" calcula las áreas bajo el alineado (+) y (-) picos de reacción, y tabula los valores de reactividad junto con el número de nucleótidos correspondiente en un archivo de texto delimitado por tabuladores.
Nota: El análisis de los datos es fundamental para la precisión de la forma, y algunas consideraciones son muy importantes en este análisis, incluyendo:
- De señal-a-ruido: La relación señal-a-ruido tiene que ser tal que los picos individuales deben ser fácilmente identificable incluso para posiciones con baja reactividad. Aunque ShapeFinder proporciona una opción de suavización de datos; esta alternativa se debe utilizar mucha cautela, ya que puede sesgar el análisis posterior.
- Región de análisis: Normalmente, los datos fiables se pueden obtener a partir de ADNc 300-600 nt de longitud, a partir de una región 40-80 nt eliminado a partir del cebador 3 'terminal y terminando como las señal decae a niveles difíciles de distinguir del ruido de fondo. El uso de múltiplesconjuntos de primer ple deberán analizar tramos más largos de ARN. En este caso, se recomienda que la coincidencia de señal fiable entre los conjuntos de cebadores está en el intervalo de 30-50 nt. En los ARN más cortas, donde la transcriptasa inversa alcanza con frecuencia al final de la plantilla de ARN, se debe tener cuidado para excluir esos picos cuya relación señal a ruido se ve afectada por la síntesis de ADN fuerte parada.
- Decaimiento de la señal: La caída de la señal está relacionada con el grado de modificación del ARN durante el experimento, así como la capacidad de procesamiento imperfecta de la RT. Idealmente, la cinética de un solo golpe con respecto a la región de ARN que se analiza debe lograrse con el fin de maximizar la lectura longitud. Shapefinder contiene una herramienta que es efectiva en la corrección de caída de la señal, sin embargo, porque esto tiende a introducir errores en el análisis - sobre todo cuando no se observan cinéticas de un solo golpe, se utiliza mejor cuando degradación de la señal es mínima (es decir, cuando la distribución de los picos es consistente con un solo golpe kinetics). Recientemente, mejora de los algoritmos de transformación de señal de caída de la señal se han publicado 30 y deben ser investigado si caída de la señal es de especial preocupación en un experimento particular.
- Señal de escala. Podría decirse que el paso más arbitraria en el procesamiento de datos de la forma, el perfil de control debe ser ajustado de modo que las intensidades de los picos entre mínimamente reactivo (+) y (-) restos son iguales. La ampliación de la traza de control para una extensión demasiado grande dará lugar a una gran cantidad de valores de reactividad negativos en el primer cuartil (ver Normalización de los datos a continuación). En este caso, el factor de escala se debe reducir en consecuencia y los datos reintegrado.
- Asignación Peaks. En general, la versión automatizada de la atribución de los picos funciona bien. Cuando falla el proceso, sin embargo, es imperativo que el usuario se asegure de que todos los picos han sido reconocidos por el software, en particular cuando la relación señal-a-ruido es bajo. Picos de hombro, por ejemplo, no siempre se detectan, y el G-ricos sícuencias a menudo se comprimen.
7. Normalización de datos
Para incorporar perfiles de reactividad de nucleótidos en el algoritmo de estructura secundaria que utiliza el software RNAStructure (v5.3), y / o para comparar los perfiles de ARN estrechamente relacionados, los datos de forma se deben normalizar de una manera estandarizada 12. Esto implica (i) con exclusión de los valores atípicos de los cálculos posteriores, (ii) determinar la reactividad "máxima efectiva" (es decir, la media de la más alta 8% del valor de la reactividad, con exclusión de los valores atípicos), y (iii) normalización dividiendo todos los valores de reactividad por la "máxima efectiva", de la siguiente manera:
- Abra el archivo de texto delimitado por tabuladores generado después de la alineación y la integración y copiar su contenido en una hoja de cálculo Excel. La columna más a la derecha de este archivo (RX.area-BG.area) contiene los valores de reactividad SHAPE absolutos calculados para cada uno de nucleótidos del ARN. Las columnas de la izquierda se refieren esta reactividad al ARN SEQUcia.
- Calcular y almacenar el primer cuartil y terceras partes (por ejemplo, el percentil 25 y 75) los valores de (RX.area-BG.area) utilizando la función de Excel "= cuartil (array, cuarto)"
- Calcular y almacenar la diferencia intercuartil "= cuartil (array, 3)-cuartil (array, 1)"
- Calcular y almacenar el "valor de corte atípico" por medio de la fórmula "= (cuartil, serie, 3) 1.5 * ((cuartil (array, 3)-cuartil (array, 1))." Todo reactividad de los valores superiores a este valor se ser excluido de los cálculos posteriores.
- Copie los valores de reactividad de (RX.area-BG.area) y pegarlos en una columna vacía adyacente, a continuación, ordenar estos valores tales que el mayor se encuentran en la parte superior de la columna.
- En "columna ordenada los valores de" la nueva creación, elimine los valores mayores que el valor de corte atípico.
- Calcular y almacenar el promedio del 8% mayor de valores de reactividad restantes en el "ordenados valores column ". Este valor es la" máxima reactividad eficaz ".
- Divida la no clasificados (RX.area-BG.area) de cada nucleótido (incluyendo los valores extremos) por el valor de reactividad "efectiva máxima" para obtener los valores de reactividad "normalizados". Almacenar estos en una columna vacía, dejando una columna vacía a la izquierda. A continuación, copie el número de nucleótidos en la parte izquierda de la tabla y pegarlos en la columna vacía a la izquierda de los "valores de reactividad normalizados".
- Copia y pega los pares de valores de reactividad posición normalizados nucleótidos en un editor de texto.
- Eliminar los valores por debajo de -0,09 (es decir, dejar el espacio en blanco espacios), ya que estos son probablemente el resultado de la RT pausa durante la síntesis de ADNc por razones distintas de la modificación química de la plantilla. Por otra parte, los valores de reactividad de nucleótidos a la que se observa una fuerte pausa en la plantilla sin modificar (según lo determinado por la inspección visual de la "Alinear e integrar" perfil ShapeFinder), deben ser excluidos.
- SaVe el archivo con una extensión. forma para su uso en el análisis estructural con el software RNAstructure (v5.3).
8. Modelado de datos
Software de RNAstructure (v5.3) se utiliza para predecir la estructura secundaria del ARN experimentalmente-compatible (s) con las limitaciones de la energía seudo-libres derivados del análisis de la forma 19. El software proporciona representaciones gráficas de las estructuras más bajas de energía 2D ARN así como la representación textual de estas estructuras en la notación de punto-soporte. Este último puede ser importado en una estructura de ARN espectador de la preferencia del usuario, por ejemplo, Pseudoviewer 23 o 22 Varna, para producir imágenes con calidad de publicación.
Nota: Se debe tener cuidado al considerar las estructuras producidas por el software RNAstructure (v5.3). Por ejemplo, el software no puede resolver interacciones terciarias tales como pseudonudos y bucles que se besan, ni puede distinguir si la falta dereactividad en una cierta región es debido a la protección estérica por emparejamiento de bases o proteínas unidas. Como consecuencia, estos factores, junto con las energías reportados para las estructuras individuales, deben tenerse en cuenta en la presentación de un modelo estructural definitiva.
Resultados
ARN que contiene el VIH-1 elemento de respuesta rev (RRE) y un 3 'casete estructura de la terminal 4 se preparó a partir de un plásmido linealizado por la transcripción in vitro, después de lo cual se pliega por calentamiento, enfriamiento, y la incubación a 37 ° C en la presencia de MgCl 2. ARN fue expuesto a NMIA y luego a transcripción inversa a partir de un cebador de ADN 5'-marcado en el extremo hibridó con el casete de estructura de la terminal 3 '. La fo...
Discusión
Se presenta aquí un protocolo detallado para la forma de alto rendimiento, una técnica que permite la determinación de la estructura secundaria de la resolución de un solo nucleótido de ARN de cualquier tamaño. Por otra parte, el acoplamiento de datos experimentales forma con algoritmos de predicción de estructura secundaria facilita la generación de modelos de ARN 2D con un mayor grado de precisión que es posible con cualquiera de los métodos solos. La combinación de cebadores marcados con fluorescencia y au...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch y SFJ Le Grice son apoyados por el Programa de Investigación Intramural del Instituto Nacional del Cáncer, de los Institutos Nacionales de Salud, EE.UU..
Materiales
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5' labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied - including any 3'terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies | ||
Referencias
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 '-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 '-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues - A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados