Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un Ensayo Fenotípico Microscópico Para La Cuantificación De Micobacterias Intracelulares Adaptadas Para La Detección De Alto Rendimiento / Alto Contenido
En este artículo
Resumen
Aquí, se describe un ensayo fenotípico aplicable a las pantallas de alto rendimiento / alto contenido de ARN sintético de interferencia pequeña (siRNA), compuesto químico, y bibliotecas de mutantes de Mycobacterium tuberculosis. Este método se basa en la detección de tuberculosis de la micobacteria fluorescentemente etiquetada dentro de la célula huésped etiquetada fluorescentemente usando microscopia confocal automatizada.
Resumen
A pesar de la disponibilidad de terapia y vacuna, la tuberculosis (TB) sigue siendo una de las infecciones bacterianas más mortales y extendidas del mundo. Desde hace varias décadas, la explosión repentina de cepas multirresistentes y ampliamente resistentes a los medicamentos es una grave amenaza para el control de la tuberculosis. Por lo tanto, es esencial identificar nuevas dianas y vías críticas para el agente causal de la tuberculosis, Mycobacterium tuberculosis (Mtb)y buscar nuevos productos químicos que podrían convertirse en fármacos contra la tuberculosis. Un enfoque es establecer métodos adecuados para las pantallas genéticas y químicas de bibliotecas a gran escala que permitan la búsqueda de una aguja en un pajar. Con este fin, desarrollamos un ensayo fenotípico que se basa en la detección de Mtb marcado fluorescentemente dentro de las células huésped etiquetadas fluorescentemente utilizando microscopía confocal automatizada. Este ensayo in vitro permite una cuantificación basada en imágenes del proceso de colonización de Mtb en el huésped y se optimizó para el formato de microplaca de 384 pozos, que es adecuado para pantallas de bibliotecas de mutantes de siRNA, compuestos químicos o Mtb. Las imágenes se procesan entonces para el análisis multiparamétrico, que proporciona la lectura hacia fuera que infiere en la patogenesia del Mtb dentro de las células huesped.
Introducción
Entre los patógenos infecciosos emergentes y reemergentes reportados durante los últimos años, Mycobacterium tuberculosis (Mtb)ocupa un lugar destacado siendo responsable de 1,4 millones de muertes y 8,7 millones de nuevas infecciones en 2011 (Informe Mundial de Tuberculosis 2012, www.who.int/topics/tuberculosis/en/). A pesar de la disponibilidad de terapias multidrogas, el número de personas infectadas sigue en aumento y el Mtb multirresistente (MDR) y ampliamente farmacorresistente (XDR) se está extendiendo rápidamente por todo el mundo1. Por otra parte, si se tiene en cuenta la presencia de antígenos mtb, es evidente que un tercio de la población mundial se considera que está infectada latentemente por mtb. Estadísticamente, en un caso de cada diez, hay evolución hacia la forma activa de la enfermedad con síntomas clínicos posteriores2. Por lo tanto, se necesitan con urgencia nuevos medios para luchar contra el Mtb. En este contexto, desarrollamos un ensayo fenotípico visual in vitro basado en el seguimiento de la invasión de Mtb y la multiplicación en las células huésped mediante microscopía de fluorescencia confocal automatizada3. La adaptación del ensayo en placas de microtíter de 384 pozos en combinación con la adquisición y el análisis automatizados de imágenes, permitió la detección de alto contenido / alto rendimiento (HC / HTS) de bibliotecas de escala media de compuestos, siRNAs y mutantes bacterianos. La investigación de una biblioteca amplia del genoma RNAi en este análisis fenotípico permitió así la identificación de los anfitrión-factores dominantes implicados en tráfico de Mtb y la réplica intracelular pero también la aclaración de los anfitrión-caminos explotados por el bacilo del tubérculo. Otra adaptación de este análisis fenotípico particular estaba para la identificación de los factores bacterianos esenciales a la persistencia intraphagosomal del Mtb. Por ejemplo, la detención de la maduración del fagosoma se considera como uno de los principales mecanismos que facilita la supervivencia y replicación de Mtb en los macrófagos. El monitoreo de la localización subcelular de mutantes knock-out mtb en compartimentos fluorescentemente etiquetados como ácidos permitió la identificación de genes bacterianos involucrados en el proceso de supervivencia4. Finalmente, la imagen de alto contenido de Mtb también ofrece un excelente método para cuantificar la eficiencia de los fármacos para inhibir diversos fenómenos como el crecimiento bacteriano intracelular3. En conjunto, este tipo de ensayo fenotípico de alto rendimiento permite acelerar el descubrimiento de fármacos contra la TB y los datos recogidos por estos diferentes enfoques contribuyen a una mejor comprensión de la manipulación del huésped ejercida por Mtb.
Protocolo
1. Detección de siRNA en todo el genoma de alto rendimiento
La investigación realizada en un tipo humano de los pneumocytes del tipo II modelo A549 la línea de células sobre la infección con mtb H37Rv que expresa la proteína fluorescente verde (GFP). Este procedimiento se describe en la Figura 1A.
- Resuspend la biblioteca de siRNA seca que se almacena en placas madre (placas de 96 pozos) con 1x tampón de siRNA para alcanzar una concentración de 4 μM, luego transfiera 10 μl de la mezcla en una placa hija de 384 pozos (placa hija 1).
- Añadir 10 μl de 1x tampón de siRNA en la placa hija 1 para diluir el siRNA por 2 veces. Después de la resuspensión de siRNA, las placas se sellan con sello de aluminio pelable y se pueden almacenar a -20 ° C al menos 6 meses y hasta 2-3 años, pero el tiempo de almacenamiento puede variar dependiendo de las recomendaciones del fabricante de la biblioteca de siRNA.
- Diluir los siRNAs en la placa hija 1 en la placa hija 2 para alcanzar una concentración de 500 nM. Después de la resuspensión de siRNA, las placas se sellan con sello de aluminio pelable y se pueden almacenar a -20 ° C al menos 6 meses y hasta 2-3 años, pero el tiempo de almacenamiento puede variar dependiendo de las recomendaciones del fabricante de la biblioteca de siRNA.
- Antes de su uso, descongelar la placa hija 2 a temperatura ambiente.
- Tome 2,5 μl de siRNA de la placa hija 2 y colóquelos en una placa de ensayo de 384 pozos.
- En la misma placa de ensayo de 384 pozos en el paso 1.5, agregue 2.5 μl de siRNA de control negativo y positivo a sus respectivos pozos.
- Diluya el reactivo de transfección en 1x D-PBS para producir suficiente solución para proporcionar un reactivo de transfección de 0,1 μl en cada pozo y preincuba la solución de transfección diluida a temperatura ambiente durante 5 min.
- Añadir 7,5 μl de la mezcla de reactivo de transfección/solución de PBS a cada pozo de la placa de ensayo e incubar durante 30 min a temperatura ambiente.
- Añadir 40 μl de células A549 (1.500 células/pozo) suspendidas en medio RPMI 1640 suplementado con suero fetal bovino al 10% (FBS). Mantener las células durante un período de incubación de 3 días a 37 °C en una atmósfera que contenga un 5% deCO2. Estas células se dividen cada 24 horas, por lo que unas 12.000 células están en los pozos tres días después de la transfección.
- Lave un cultivo de Mtb H37Rv que exprese GFP de dos semanas de edad con D-PBS (libre de MgCl2 y CaCl2)por centrifugación a 4,000 x g durante 5 min y deseche los lavados. Repita este paso 3x. (Para las condiciones de cultivoGFP-Mtb H37Rv, consulte el Protocolo 3).
- Suspender el pellet bacteriano en 10 ml de RPMI 1640 medio suplementados con 10% de FBS y decantar durante 1 hora a temperatura ambiente para permitir que los agregados bacterianos se sedimenten.
- Recoger el sobrenadante bacteriano y medir OD600 (OD600 debe estar entre 0.6-0.8) y GFP-fluorescencia (valor de RFU) utilizando un lector de microplacas para determinar la concentración bacteriana. Calcule el título de la suspensión utilizando una línea de regresión de referencia que muestre el valor de RFU = f (valor de CFU) que se había generado antes del experimento en otro cultivo que se había preparado en las mismas condiciones. Preparar suspensión bacteriana que contenga 2,4 x 106 bacterias/ml, que corresponde a una multiplicidad de infección (MOI) de 5.
- Retire el medio en la placa de ensayo de 384 pozos y agregue 25 μl de suspensión bacteriana recién preparada.
- Incubar la placa de ensayo de 384 pozos a 37 °C durante 5 horas en una atmósfera que contenga un 5% de CO2.
- Retire el medio y lave suavemente las células con el medio RPMI complementado con 10% FBS 3x.
- Para matar las bacterias extracelulares restantes, trate las células con 50 μl de medio RPMI-FBS fresco que contenga 50 μg/ml de amikacina a 37 °C durante 1 hora en una atmósfera que contenga 5% de CO2.
- Retire la amikacina que contiene el medio y agregue 50 μl de medio RPMI fresco complementado con 10% de FBS. Incubar la placa de ensayo de 384 pozos a 37 °C durante 5 días en una atmósfera que contenga un 5% deCO2.
- Antes de la adquisición de la imagen, añadir 10 μl de 30 μg/ml de DAPI recién preparados en PBS (concentración final de 5 μg/ml) e incubar durante 10 min a 37 °C.
- Cargue la placa en un microscopio confocal automatizado.

Haga clic aquí para ver la imagen más grande. - Establezca los parámetros de exposición. Registre la fluorescencia DAPI utilizando láser de excitación de 405 nm con filtro de emisión de 450 nm y fluorescencia GFP utilizando láser de excitación de 488 nm con filtro de emisión de 520 nm.
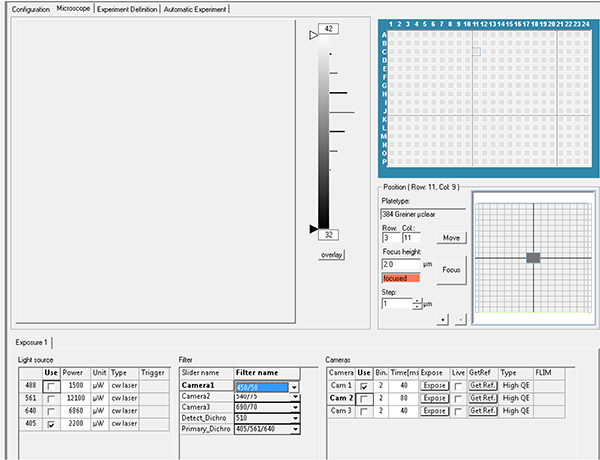
Haga clic aquí para ver la imagen más grande.
Haga clic aquí para ver la imagen más grande. - Seleccione los pozos y los campos en cada pozo que se va a adquirir, que luego se conoce como parámetros de diseño y subdiseño.
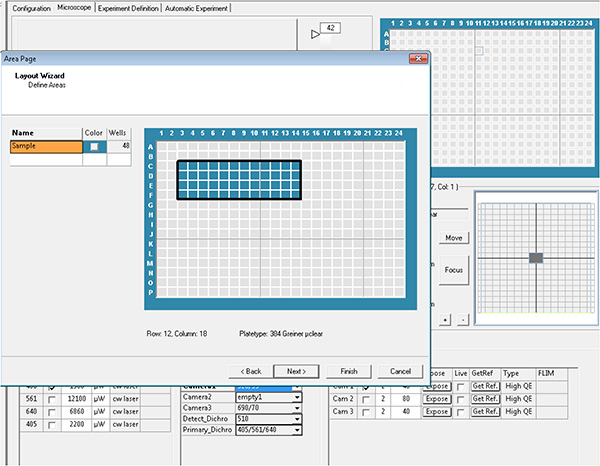
Haga clic aquí para ver la imagen más grande.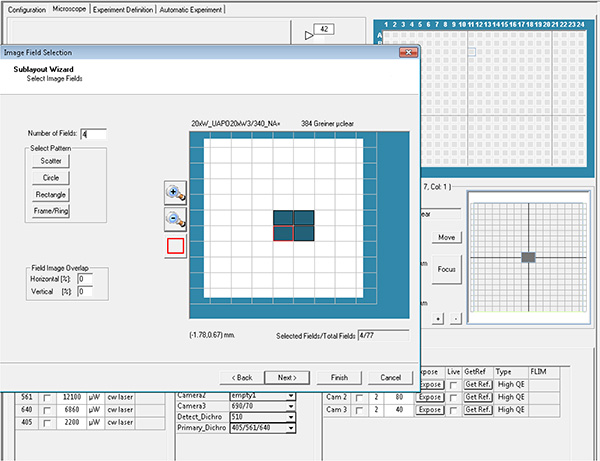
Haga clic aquí para ver la imagen más grande. - Genere el archivo de experimento utilizando los parámetros de los pasos 1.20 y 1.21 y ejecute la adquisición automática.
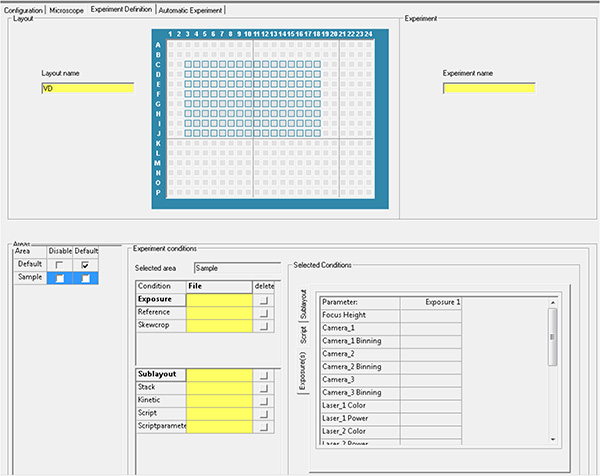
Haga clic aquí para ver la imagen más grande.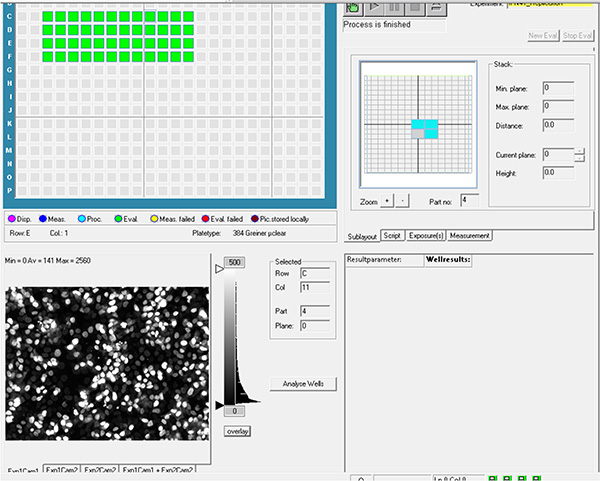
Haga clic aquí para ver la imagen más grande. - Transferir imágenes al servidor remoto.
Haga clic aquí para ver la imagen más grande. - Evalúe las imágenes utilizando el software de análisis de imágenes. Detectar núcleos celulares del canal DAPI utilizando el algoritmo de detección de núcleos y el área bacteriana del canal GFP utilizando el algoritmo de propiedades de intensidad de píxeles (Figura 4A).
Nota: Este protocolo está optimizado para estudiar el efecto del silenciamiento génico sobre el crecimiento intracelular de Mtb. Mtb es una bacteria de crecimiento lento que se divide cada 20 horas en condiciones óptimas. Después de 5 días después de la infección, la cantidad de Mtb extracelular sigue siendo baja en ausencia de lisis celular y no afectó la calidad del análisis. Este protocolo debe optimizarse en términos de duración del tratamiento con antibióticos y tiempo de incubación para adaptarse a las pantallas de siRNA utilizando bacterias de crecimiento rápido como Mycobacteria smegmatis y Escherichia coli que se liberan ampliamente y pueden infectar nuevas células.
2. Cribado de compuestos de alto rendimiento
La investigación realizada en las células huésped infectadas mtb H37Rv. Este procedimiento se describe en la Figura 1B.
- Descongelar las placas madre de 384 pozos que contienen la biblioteca compuesta solubilizada en DMSO al 100%. Transferir 0,5 μl de los compuestos en placas hijas de 384 pozos que contienen 10 μl de medio RPMI 1640 complementado con FBS al 10%.
- Lave un cultivo de Mtb H37Rv que exprese GFP de dos semanas de edad con D-PBS (libre de MgCl2 y CaCl2)por centrifugación a 4,000 x g durante 5 min y deseche los lavados. Repita este paso 3x (para las condiciones de cultivoGFP-Mtb H37Rv, consulte el Protocolo 3).
- Suspender el pellet bacteriano en 10 ml de RPMI 1640 medio suplementados con 10% de FBS y decantar durante 1 hora a temperatura ambiente para permitir que los agregados bacterianos se sedimenten.
- Recoger el sobrenadante bacteriano y medir OD600 (OD600 debe estar entre 0.6-0.8) y GFP-fluorescencia (valor de RFU) utilizando un lector de microplacas. Calcule el título de la suspensión utilizando una línea de regresión de referencia que muestre el valor de RFU = f (valor de CFU) que se había generado antes del experimento. La concentración típica es de 1 x 108 bacterias/ml.
- Cosechar 6 días de edad de macrófagos humanos primarios a 4 x 105 células/ml en rpmi 1640 medio complementado con 10% de suero bovino fetal (FBS) y 50 ng/ml M-CSF humano recombinante (Para la purificación de células de monocitos de sangre periférica humana y la diferenciación de macrófagos ver Protocolo 4).
- Incubar las células primarias diluidas con bacilos a diferentes MOI, variando de 1-5, en suspensión con temblor leve a 90 rpm durante 2 horas a 37 °C.
- Lave las células infectadas por centrifugación a 350 x g para eliminar las bacterias extracelulares. Después de cada paso de centrifugación, resuspend el pellet en rpmi 1640 medio complementado con 10% FBS. Repita este paso 2x.
- Suspender las células infectadas en medio RPMI 1640 suplementado con FBS al 10% y amikacina a 50 μg/ml e incubar la suspensión con temblores leves durante 1 hora a 37 °C.
- Retire el medio de cultivo celular que contiene amikacina por centrifugación a 350 x g y lave las células infectadas con un medio RPMI 1640 completo complementado con 10% de FBS y 50 ng/ml de M-CSF humano recombinante. Repita una vez.
- Añadir 40 μl de la suspensión de macrófagos infectados en la misma placa de ensayo en el paso 2.1, que ya contiene 10 μl de las diluciones compuestas. La concentración final de DMSO en cada pozo ha alcanzado ahora el 1%.
- Incubar las placas de ensayo durante 5 días a 37 °C en una atmósfera que contenga un 5% deCO2.
- Manche las células vivas con el tinte fluorescente lejano-rojo célula-permeante.
- Cargue la placa en un microscopio confocal automatizado.

Haga clic aquí para ver la imagen más grande. - Establezca los parámetros de exposición. Registre la fluorescencia de color rojo lejano utilizando láser de excitación de 640 nm con filtro de emisión de 690 nm y fluorescencia GFP utilizando láser de excitación de 488 nm con filtro de emisión de 520 nm.

Haga clic aquí para ver la imagen más grande.
Haga clic aquí para ver la imagen más grande. - Seleccione los pozos y los campos en cada pozo que se va a adquirir, que luego se conoce como parámetros de diseño y subdiseño.

Haga clic aquí para ver la imagen más grande.
Haga clic aquí para ver la imagen más grande. - Genere el archivo de experimento utilizando los parámetros de los pasos 2.14 y 2.15 y ejecute la adquisición automática.

Haga clic aquí para ver la imagen más grande.
Haga clic aquí para ver la imagen más grande. - Transferir imágenes al servidor remoto.

Haga clic aquí para ver la imagen más grande. - Evalúe las imágenes utilizando el software de análisis de imágenes. Detecte el área celular del canal rojo lejano utilizando el algoritmo de propiedades de intensidad de píxeles y el área bacteriana del canal GFP utilizando el algoritmo de propiedades de intensidad de píxeles (Figura 4B).
Nota: Este protocolo se puede adaptar para la detección de la biblioteca de mutantes Mtb mediante la sustitución de compuestos por mutantes que expresan una proteína fluorescente (un pozo / un mutante) (Figura 1C, véase también Brodin et al. 4). Los mutantes fluorescentes se siembran por primera vez en pozos (20 μl de suspensión bacteriana por pozo). Las bacterias se recuperan entonces por 30 μl de suspensión celular. Después de la centrifugación a 350 x g durante 1 min, la placa se incuba a 37 °C en una atmósfera que contiene un 5% deCO2. El tiempo de incubación y el MOI dependen del ensayo. Como ejemplo, para la visualización de eventos celulares tempranos como la acidificación del fagosoma, las células pueden infectarse durante 2 horas con MOI que van desde 1-20. Los lisosomas se tiñen con tinte Lysotracker a 2 μM durante 1,5 horas a 37 °C en una atmósfera que contiene 5% de CO2 y luego se fijan con formol al 10% o al 4% de paraformadehído (PFA). Las imágenes confocales se adquieren y finalmente se analizan utilizando scripts de análisis de imágenes con algoritmos apropiados para la detección de lisosomas y la localización subcelular4.
3. Proteína fluorescente verde que expresa mycobacterium tuberculosis H37Rv (GFP-H37Rv) Condiciones de cultivo
Para el almacenamiento a largo plazo, GFP-H37Rv se congelaron en D-PBS (alrededor de 1 x 108 micobacterias por vial).
- Resuspend un vial de GFP-H37Rv congelado en un matraz Erlenmeyer que contenga 50 ml de medio caldo 7H9 complementado con enriquecimiento de Middlebrook OADC al 10%, glicerol al 0,5%, tween-80 al 0,05% e higromicina B (50 μg/ml).
- Incubar 8 días a 37 °C.
- Mida OD600 de la cultura GFP-H37Rv.
- Diluir el cultivo de GFP-H37Rv para obtener OD600= 0,1 en medio caldo fresco 7H9 suplementado con enriquecimiento de Middlebrook OADC al 10%, Glicerol al 0,5%, Tween-80 al 0,05% e Higromicina B (50 μg/ml).
- Incubar el GFP-H37Rv a 37 °C durante 8 días más antes de usarlo para el ensayo.
4. Purificación de células de monocitos de sangre periférica humana de sangre entera o preparación de capa de Buffy
- Diluir la bolsa de sangre 2x en 1x D-PBS (libre de MgCl2 y CaCl2)que contiene 1% de FBS.
- Aislar los monocitos por Centrifugación de gradiente de densidad de Ficoll a 400 x g durante 20 min.
- Recoger los monocitos aislados.
- Lavar los monocitos 3x con 1x D-PBS (libre de MgCl2 y CaCl2)que contiene 1% de FBS por centrifugación a 400 x g durante 10 min a temperatura ambiente.
- Concentrar las células hasta 1 x 107 células/ml.
- Purificar monocitos usando perlas magnéticas CD14 de acuerdo con el protocolo del fabricante (ver Materiales).
- Después de la purificación de los monocitos CD14, sembraron las células a 1,5 x10 6 células/ml en RPMI 1640 complementadas con 10% de FBS y 40 ng/ml de factor estimulante de colonias de macrófagos humanos (hM-CSF) e incubadas durante 4 días a 37 °C en una atmósfera que contiene 5% de CO2.
- Después de 4 días sustituir el medio por RPMI 1640 fresco complementado con 10% de FBS y 40 ng/ml de hM-CSF e incubado durante 2 días a 37 °C en una atmósfera que contiene un 5% deCO2.
- Después de 6 días, las células se pueden utilizar para el ensayo.
Resultados
Detección de siRNA en todo el genoma de alto rendimiento
Mtb es capaz de colonizar las células inmunes in vitro, así como varias otras células epiteliales pulmonares. Por ejemplo, Mtbes capaz de infectar y dañar las células epiteliales A549 que se utilizan comúnmente como modelo para los neumocitos humanos tipo II5-7. La dectina-1 fue reportada como un receptor de células huésped implicado en la captación de Mtb, la respuesta proinflamatori...
Discusión
Se describen aquí los métodos necesarios para un ensayo fenotípico utilizando una cepa de Mtb H37Rv que expresa GFP para infectar las células huésped etiquetadas fluorescentemente, lo que lo hace apropiado para pantallas de alto contenido / alto rendimiento. Este protocolo podría aplicarse a una amplia gama de compuestos, sondas fluorescentes y mutantes mtb. Para cada protocolo descrito anteriormente, se podrían realizar pasos de fijación e inmunoetiquetado antes de la adquisición de imágenes....
Divulgaciones
No se declaran conflictos de intereses.
Agradecimientos
El apoyo financiero para este trabajo fue proporcionado por la Comunidad Europea (ERC-STG INTRACELLTB Grant n° 260901, MM4TB Grant n° 260872), la Agence Nationale de Recherche, el Feder (12001407 (D-AL) Equipex Imaginex BioMed) y la Region Nord Pas de Calais. Agradecemos la asistencia técnica de Gaspard Deloison, Elizabeth Werkmeister, Antonino Bongiovanni y Frank Lafont de la plataforma BICeL.
Materiales
| Name | Company | Catalog Number | Comments |
| µclear-plate black, 384-well | Greiner Bio-One | 781091 | 127.8/86/15 MM with Lid, TC treated |
| CellCarrier 384-well plate | PerkinElmer | 6007550 | Black, Clear Bottom, with Lid, TC treated |
| V-bottom white, 384-well plate | Greiner Bio-One | 781280 | |
| sealing tape, breathable, sterile | Corning | 3345 | |
| Lipofectamine RNAiMax | Life Technologies | 13778150 | Transfection reagent |
| Dimethyl sulfoxide | Sigma-Aldrich | 34943 | |
| RPMI 1640 + GlutaMAX-I | Life Technologies | 61870-010 | Cell culture medium |
| D-PBS 1x [-]MgCl2/[-]CaCl2 | Life Technologies | 14190-094 | Dulbecco's Phosphate Salin Buffer |
| D-PBS 1x [+]MgCl2/[+]CaCl2 | Life Technologies | 14190-091 | Dulbecco's Phosphate Salin Buffer |
| Fetal bovine serum | Life Technologies | 2610040-79 | |
| Ficoll Paque PLUS | Dutscher | 17-1440-03 | Ficoll for Peripherical Blood Monocyte Cells purification |
| CD14 MicroBeads, human | Miltenyi | 130-050-201 | Purification of CD14+ Monocytes |
| Human M-CSF, premium gr. (1000 μg) | Miltenyi | 130-096-493 | Macrophage Colony Stimulating Factor |
| LS Columns | Miltenyi | 130-042-401 | Columns for CD14+ Monocytes isolation |
| Tween 80 | Euromedex | 2002-A | Mycobacteria culture |
| Glycerol high purity | Euromedex | 50405-EX | Mycobacteria culture |
| Middlebrook OADC enrichment | Becton-Dickinson | 211886 | Mycobacteria culture |
| 7H9 | Becton-Dickinson | W1701P | Mycobacteria culture |
| Versene 1x | Life Technologies | 15040033 | Nonenzymatic cell dissociation solution |
| DAPI | Life Technologies | D1306 | Nuclei dye |
| Hoechst 33342 | Life Technologies | H3570 | Nuclei dye |
| Syto60 | Life Technologies | S11342 | Nuclei/cytoplasm dye |
| Formalin | Sigma-Aldrich | HT5014 | Cell fixation solution |
| siRNA targeting Dectin-1 | Santa-Cruz | sc-63276 | |
| *siGenome* Nontargeted siRNA pool | Dharmacon | D-001206-14 | |
| Rifampicin | Sigma-Aldrich | R3501 | antibiotic |
| Isoniazid (INH) | Sigma-Aldrich | I3377-50G | antibiotic |
| Hygromycin B | Life Technologies | 10687-010 | antibiotic |
| Amikacin | Sigma-Aldrich | A1774 | antibiotic |
| Automated Confocal Microscope OPERA | PerkinElmer | Image acquisition | |
| Columbus 2.3.1 Server Database | PerkinElmer | Data transfer, storage, and analysis | |
| Acapella 2.6 software | PerkinElmer | Image-based analysis | |
| GraphPad Prism5 software | GraphPad | Statistical analysis | |
| Excel 2010 | Microsoft | Statistical analysis |
Referencias
- Gandhi, N. R., et al. Multidrug-resistant and extensively drug-resistant tuberculosis: a threat to global control of tuberculosis. Lancet. 375, 1830-1843 (2010).
- Barry, C. E., et al. The spectrum of latent tuberculosis: rethinking the biology and intervention strategies. Nat. Rev. Microbiol. 7, 845-855 (2009).
- Christophe, T., Ewann, F., Jeon, H. K., Cechetto, J., Brodin, P. High-content imaging of Mycobacterium tuberculosis-infected macrophages: an in vitro model for tuberculosis drug discovery. Future Med. Chem. 2, 1283-1293 (2010).
- Brodin, P., et al. High content phenotypic cell-based visual screen identifies Mycobacterium tuberculosis acyltrehalose-containing glycolipids involved in phagosome remodeling. PLoS Pathog. 6, (2010).
- Bermudez, L. E., Goodman, J. Mycobacterium tuberculosis invades and replicates within type II alveolar cells. Infect. Immun. 64, 1400-1406 (1996).
- Dobos, K. M., Spotts, E. A., Quinn, F. D., King, C. H. Necrosis of lung epithelial cells during infection with Mycobacterium tuberculosis is preceded by cell permeation. Infect. Immun. 68, 6300-6310 (2000).
- McDonough, K. A., Kress, Y. Cytotoxicity for lung epithelial cells is a virulence-associated phenotype of Mycobacterium tuberculosis. Infect. Immun. 63, 4802-4811 (1995).
- Lee, H. M., Yuk, J. M., Shin, D. M., Jo, E. K. Dectin-1 is inducible and plays an essential role for mycobacteria-induced innate immune responses in airway epithelial cells. J. Clin. Immunol. 29, 795-805 (2009).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J. Biomol. Screen. 4, 67-73 (1999).
- Birmingham, A., et al. Statistical methods for analysis of high-throughput RNA interference screens. Nat. Methods. 6, 569-575 (2009).
- Carralot, J. P., et al. Automated high-throughput siRNA transfection in raw 264.7 macrophages: a case study for optimization procedure. J. Biomol. Screen. 14, 151-160 (2009).
- Zhang, X. D. Illustration of SSMD, z score, SSMD*, z* score, and t statistic for hit selection in RNAi high-throughput screens. J. Biomol. Screen. 16, 775-785 (2011).
- Song, O. R., et al. Confocal-based method for quantification of diffusion kinetics in microwell plates and its application for identifying a rapid mixing method for high-content/throughput screening. J. Biomol. Screen. 15, 138-147 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados