Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
De alta resolución espacio-temporal Análisis de la Dinámica del receptor por Casa molécula de microscopía de fluorescencia
En este artículo
Resumen
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
Resumen
Microscopía sola molécula está surgiendo como un enfoque poderoso para analizar el comportamiento de las moléculas de señalización, en particular en lo referente a los aspectos (por ejemplo., La cinética, la coexistencia de los diferentes estados y poblaciones, interacciones transitorias), que normalmente se ocultan en las mediciones del conjunto, tales como los obtenidos con métodos bioquímicos o de microscopía estándar. Por lo tanto, los eventos dinámicos, como las interacciones receptor-receptor, se pueden seguir en tiempo real en una célula viva con una alta resolución espacial y temporal. Este protocolo describe un método basado en el etiquetado con fluoróforos orgánicos pequeños y brillantes y el total de fluorescencia de reflexión interna (TIRF) microscopía para visualizar directamente los receptores individuales en la superficie de las células vivas. Este enfoque permite a localizar con precisión los receptores, medir el tamaño de los complejos receptores, y la captura de eventos dinámicos, como las interacciones receptor-receptor transitorio. El protocolo proporciona una descripción detallada de la forma de performa un experimento de una sola molécula, incluyendo la preparación de muestras, adquisición de imágenes y análisis de imágenes. Como un ejemplo, la aplicación de este método para analizar dos receptores G acoplados a la proteína,, β 2-adrenérgicos y γ-aminobutírico de tipo ácido B (GABA B), se informó es decir.. El protocolo se puede adaptar a otras proteínas de la membrana y diferentes modelos de células, métodos de transfección y estrategias de etiquetado.
Introducción
Receptores localizados en la superficie celular detectan el medio ambiente extracelular y responden a una variedad de estímulos, tales como odorantes, iones, pequeñas neurotransmisores y hormonas proteicas grandes. La naturaleza fluida de las membranas celulares permite movimientos de los receptores y otras proteínas de membrana. Esto es esencial para la formación de complejos de proteínas y la aparición de interacciones proteína-proteína transitorios, tales como los utilizados por los receptores para ensamblar en unidades funcionales y transducir señales al interior de la célula. Por ejemplo, los receptores de proteína G acoplados (GPCR), que constituyen la mayor familia de receptores de superficie celular 1, se han sugerido para formar di-/oligomers, que parece estar implicada en la regulación afinada de la transducción de señales y podría tener importantes consecuencias fisiológicas y farmacológicas 2-5.
Microscopía sola molécula tiene el gran potencial de visualizar directamente con alta spatiotempresolución oral, el comportamiento dinámico de los receptores individuales situados en la superficie de las células vivas, incluyendo su asociación para formar dímeros y de orden superior complejos moleculares 6-10. Esto ofrece varias ventajas en comparación con los métodos bioquímicos y de microscopía estándar, que por lo general reportan el comportamiento medio de miles o millones de moléculas.
El etiquetado de proteínas con un fluoróforo suficientemente brillante y fotoestable es esencial para la microscopía de una sola molécula. Este protocolo se aprovecha de la etiqueta SNAP recientemente introducido 11 para unir covalentemente fluoróforos orgánicos pequeños y brillantes a los receptores de la superficie celular. SNAP es un 20 kD etiqueta de proteína derivada de la enzima O-ADN alquiltransferasa 6-alquilguanina de reparación del ADN humano, que puede ser irreversible etiquetado con fluoróforo conjugado-bencilguanina derivados (fluoróforo-BG). CLIP, una etiqueta de ingeniería adicional derivado de SNAP, puede en cambio marcado con fluoróforo-cderivados bencilcitosina onjugated 12.
El protocolo en este manuscrito se explica cómo transfectar y la etiqueta de SNAP-11 marcado con receptores pequeños fluoróforos orgánicos y el uso total de fluorescencia de reflexión interna (TIRF) microscopía para visualizar los receptores individuales o complejos de receptores en la superficie de las células vivas 10. Los resultados del protocolo reportados en> 90% de eficiencia en el etiquetado de una proteína de la superficie celular de etiquetado SNAP extracelular 10. Más información sobre cómo utilizar los datos de una sola molécula para analizar el tamaño y la movilidad de los complejos receptores, así como para capturar las interacciones del receptor-receptor transitorio, se proporciona. Un flujo de trabajo esquemática de todo el protocolo se da en la Figura 1. Como un ejemplo, la transfección de células de ovario de hámster chino (CHO) con los receptores etiquetados SNAP-G-acoplados a proteínas (GPCRs) seguida por el etiquetado con un derivado de fluoróforo-BG como así como su aplicación a quantifse describen y y di-/oligomerization receptor de monitor. Este protocolo se puede extender a otras proteínas de la superficie celular y etiquetas fluorescentes (por ejemplo., CLIP), así como a otros métodos de transfección y etiquetado.
Protocolo
1. Preparación de la muestra
- Limpieza Cubreobjetos
NOTA: Trabaje bajo una campana de humos.- Utilice pinzas limpias para colocar cubreobjetos de vidrio (24 mm de diámetro) en un soporte de portaobjetos que separa cubreobjetos individuales.
- Coloque el soporte con cubreobjetos en un vaso de precipitados y agregar cloroformo hasta que los cubreobjetos están cubiertos. Cubrir el vaso con papel de aluminio para reducir la evaporación y se somete a ultrasonidos en un baño de ultrasonido durante 1 hora a TA. Tome el titular cubreobjetos fuera del vaso y dejar que los cubreobjetos secos.
- Repita el paso 1.1.2 con la solución de NaOH 5 M en vez de cloroformo.
- Tome el soporte de cubreobjetos en un nuevo vaso de precipitados y lavar tres veces con agua destilada. Ponga cubreobjetos limpios en una placa de cultivo celular de vidrio lleno de etanol al 100%.
- Preparación de muestras de calibración
- Disolver colorante fluorescente en disolvente apropiado.
- Preparar una dilución en serie 1:10 del colorante fluorescente que vande 13:00 a 1 nM en esterilizó por filtración (0,22 micras) del agua.
- Tome cubreobjetos limpios almacenados en etanol al 100% y se lava con agua esterilizada por filtración. Punto 20 l de cada dilución tinte fluorescente en un cubreobjetos limpio separado. Deje que los portaobjetos se secan bajo una campana estéril. Proteja los cubreobjetos de la luz y el polvo hasta su uso. Utilice estas muestras para estimar la intensidad de moléculas fluorescentes individuales (ver paso 3).
- Transfección
- Células de cultivo de CHO en 01:01 mezcla de medio / nutriente Eagle modificado de Dulbecco F-12 (DMEM/F12) suplementado con 10% de suero bovino fetal (FBS), 100 U / ml de penicilina y 100 mg / ml de estreptomicina a 37 ° C, en . 5% de CO 2 NOTA: Utilice medios de comunicación libres de fenol rojo durante todo el experimento para minimizar la autofluorescencia.
- Tome cubreobjetos limpios de solución de etanol al 100%, lávelos con una solución salina estéril tamponada con fosfato (PBS) y coloque un cubreobjetos en cada pocillo de una cultura pl 6 celdas asícomió.
- Trypsinize, contar y células CHO semilla a una densidad de 3 x 10 5 células / pocillo en la placa de cultivo celular de 6 pocillos que contiene los cubreobjetos. Deje que las células crecen en una incubadora (37 ° C, 5% CO 2) durante 24 horas a fin de alcanzar aprox. 80% de confluencia, que es la densidad celular óptima para la transfección.
- Para cada pocillo, diluir 2 g de ADN plásmido deseado (p. ej., SNAP-etiquetados β 2-adrenérgicos) y 6 l Lipofectamine 2000 en dos tubos separados que contenían 500 medio OptiMem l. Incubar a TA durante 5 min.
- Se combinan las soluciones de la etapa 1.3.4 en un tubo y mezclar para obtener una mezcla de transfección. Incubar la mezcla de transfección a temperatura ambiente durante 20 min.
- Durante la incubación (1.3.5), tome las células CHO y lavar dos veces con pre-calentado (37 ° C) PBS. Reemplazar PBS con 1 ml / pocillo de fenol medio DMEM/F12 libre de rojo suplementado con 10% de FBS, pero sin antibióticos.
- Añadir todo el transfectmezcla de iones (1 ml) de la Etapa 1.3.5 gota a gota a cada pocillo, y suavemente la placa de roca de ida y vuelta para asegurar una mezcla completa.
- Incubar durante 2 a 4 horas a 37 ° C, en 5% de CO 2 y proceder inmediatamente después para el siguiente paso NOTA:. Estas condiciones de transfección se han optimizado para conseguir densidades de receptores de partícula <0,45 / 2 micras, que son adecuados para un solo de formación de imágenes molécula. Los ajustes podrían ser necesarios cuando se utilizan diferentes células, construcciones o reactivos.
- El etiquetado de proteínas
- Diluir 1 l de solución madre de fluoróforo-BG en 1 ml de medio DMEM/F12 suplementado con FBS al 10% para obtener una concentración final de 1 mM. Tome las células transfectadas de la incubadora y se lava dos veces con precalentado (37 ° C) de PBS. Reemplazar PBS con 1 ml de 1 M solución de fluoróforo-BG y se incuba durante 20 min a 37 º C 5% de CO 2 en una incubadora.
- Después de la incubación, se lavan las células tresveces con medio DMEM/F12 suplementado con FBS al 10%, cada vez seguido de 5 min de incubación a 37 ° C. Tome un cubreobjetos (con células marcadas) con unas pinzas y colocarlo en una cámara de imágenes.
- Lavar dos veces con 300 l buffer de imágenes. Añadir 300 l de buffer de imágenes frescas y proceder de inmediato a las imágenes (Parte 2).
2. Adquisición de imágenes
NOTA: Use un total de fluorescencia de reflexión interna (TIRF) microscopio, equipado con un objetivo de inmersión en aceite de alta apertura numérica (por ejemplo, apertura numérica 100X magnification/1.46.), Los láseres adecuados (por ejemplo, 405 nm, 488 nm, 561 nm y 645 láseres de diodo nm), un dispositivo de acoplamiento de carga del electrón multiplicando (EMCCD) de la cámara, una incubadora y un control de temperatura para visualizar moléculas fluorescentes individuales.
- Establezca los parámetros de microscopio deseados, es decir., Línea de láser, el ángulo TIRF (este parámetro controla la penetraciónprofundidad ción del campo evanescente), tiempo de exposición, la velocidad de fotogramas y el número de imágenes por película 10. Mantenga el control de la calefacción / de la incubadora y la temperatura siempre a evitar las derivas de temperatura y la condensación de humedad.
- Ponga una gota de aceite de inmersión sobre el objetivo 100X del microscopio. Coloque la cámara de formación de imágenes con las células marcadas en el portamuestras del microscopio, y traer las células en foco usando iluminación de campo claro.
- Cambie a la iluminación TIRF. Mantener la potencia del láser tan bajo como sea posible para permitir la búsqueda de la célula deseada, pero al mismo tiempo se minimiza fotoblanqueo.
- Seleccione la celda deseada y ajustar con precisión el enfoque. Establecer la potencia del láser a un nivel que permite la visualización de fluoróforos individuales. Adquirir secuencia de imágenes y guardar el archivo de secuencia de imágenes en bruto como. Tiff.
3. Calibración (fluoróforos individuales sobre Vidrio y monómeros / Controles receptor dimérico)
- Montar cada sampl calibracióne preparado como se describe en 1.2 en la cámara de formación de imágenes. Colocar cada muestra en el microscopio y elegir la muestra que contiene manchas de difracción limitada bien separados que el cloro en un solo paso NOTA:. Estos puntos representan moléculas individuales del colorante fluorescente.
- Adquirir secuencias de imágenes TIRF como se describe en el paso 2 Importante:. Los mismos parámetros de la imagen se deben utilizar para todos los experimentos, incluidos los de calibración.
- Lleve a cabo la detección y análisis de seguimiento tal como se detalla en el apartado 4.1 - 4.2. Extraer la intensidad de cada partícula como se describe en 4.2.6. A partir de estos datos, calcular la media (μ) y la desviación estándar (σ) de la intensidad de fluoróforos individuales.
- Opcional: realizar el mismo análisis en células transfectadas con un receptor de superficie celular monomérico (. Por ejemplo, CD86), N-terminalmente etiquetado con ya sea una o dos copias de la SNAP 10 y se marcó con el derivado de fluoróforo-BG. FolloW el procedimiento descrito anteriormente para la solo fluoróforo en el vidrio. Estimar el etiquetado de eficiencia como se describe en Calebiro, D. et al. 10
4. Análisis de Imágenes
- Preparación de secuencia de imagen
- Utilice un software de procesamiento de imágenes (por ejemplo., ImageJ) para recortar las imágenes.
- Guardar imágenes individuales por separado. Imágenes TIFF en una carpeta nueva, indicando en cada imagen el número de fotograma.
- Medir el área de la superficie celular mediante la elaboración de una región de interés (ROI) a lo largo del contorno de una célula y el uso de la herramienta de medida en ImageJ o una herramienta similar en otro software. Utilice este valor para calcular la densidad de partículas dividiendo el número total de partículas en el principio de la película por el área de superficie de la célula.
- La detección de partículas y de seguimiento
NOTA: Utilice el software no comercial, como u-pista 13, trabajando en Matlabmedio ambiente, para detectar automáticamente y realizar un seguimiento de las partículas de los receptores individuales.
NOTA: El algoritmo u-pista se basa en el enfoque de seguimiento de varias hipótesis. Este enfoque vincula partículas entre bastidores mediante la construcción de matrices de costos, donde se asignan las probabilidades individuales de que una partícula dada en un cuadro corresponde a una partícula dada en el siguiente cuadro, aparece, desaparece o se fusiona / divide a / de otras partículas. La solución que minimiza los costos a nivel global, es decir., La una con la probabilidad más alta, se seleccionó finalmente. Esto también permite el seguimiento de una partícula de desaparecer temporalmente, un fenómeno típico causado por fluoróforo parpadeo. La última versión de u-track (2.1.0) tiene interfaces gráficas de usuario que facilitan la ejecución de estos análisis.- Desde la línea de comandos del Matlab, escriba "movieSelectorGUI" para abrir la interfaz de selección de películas. Siga las instrucciones para crear unanueva base de datos de películas a partir de las imágenes separadas previamente guardados (ver 4.1.2).
- Proporcionar el tamaño de píxel en nm, intervalo de tiempo en segundos, apertura numérica, la profundidad de bits de la cámara y la longitud de onda de emisión del fluoróforo, requerido para la detección de partículas y de seguimiento. Guardar la base de datos de películas.
- Desde la interfaz de selección de películas, ejecutar el análisis, la elección de "partículas individuales" como tipo de objetos. Una nueva ventana aparecerá donde los parámetros utilizados para la detección de las partículas y de seguimiento pueden ser definidos. Comience con los parámetros por defecto. Más tarde, ajustar estos parámetros si la calidad de la detección y / o el seguimiento no es satisfactoria (por ejemplo, algunas partículas no se detectan o pistas están fragmentados) Opcional:. En la configuración de seguimiento, comprobar "los resultados de rastreo Exportar a formato de la matriz" para almacenar las coordenadas y las amplitudes de todas las partículas en una sola matriz (campo llamado "trackedFeaturedInfo"). Para una descripción detallada de estos parámetros, consulte la documentación u-pista.
- Ejecute el algoritmo de detección. Este algoritmo de determinar automáticamente la ubicación y la intensidad por encima del fondo de cada punto de difracción limitada (es decir., Complejos de receptores individuales / receptor) por el ajuste de una función de Gauss de dos dimensiones con una desviación estándar igual a la función de dispersión de punto del microscopio alrededor de máximos de intensidad local de . A continuación, ejecute el algoritmo de seguimiento. Guarde los resultados de los análisis en un archivo colchoneta..
- Utilice la rutina "movieViewer", contenida en el paquete de u-pista, o los personalizados similares a visualizar las pistas y comprobar la calidad de la detección y el seguimiento.
- Abrir el archivo de estera. Para ver la posición y la amplitud (es decir., Intensidad) de las partículas de orugas en cada marco. Los datos generados en el paso 4.2.4 se encuentran en el campo tracksCoordAmpCG del ar tracksFinalRay y / o en trackedFeaturedInfo. A partir del número total de partículas detectado calcular la densidad de partículas dividiendo este valor por el área de superficie de la célula medido a 4.1.3.
- Opcional: Utilice la partícula coordina a través del tiempo (véase 4.2.6) para analizar el movimiento de las partículas de los receptores. Calcular los desplazamientos cuadráticos medios (MSD) y coeficientes de difusión (D) usando Matlab o software similar. Para cada partícula y cada intervalo de tiempo (Δ T) considerado, calcular el desplazamiento cuadrático medio (MSD) utilizando la siguiente fórmula:
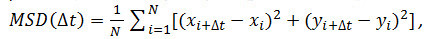
donde Δ t es el intervalo de tiempo en tramas, N es el número de pasos analizados, X e Y son X e Y de las coordenadas de la partícula en la trama indicada por el índice. Utilice el MSD sobre parcelas de tiempo para evaluar el tipo de movimiento de una partícula dada: relaciones linealesindican difusión libre (es decir., el movimiento browniano), una curvatura positiva (es decir., la curva parece una parábola) sugiere movimiento dirigido, una curvatura negativa es indicativa de movimiento limitado 14. En caso de partículas que se difunden libremente, calcular el coeficiente de difusión (D) de cada partícula ajustando los datos MSD obtenidos con la ecuación siguiente: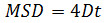
- Cálculo del tamaño de partícula con método de ajuste de Gauss
NOTA: Una vez que se conoce la distribución de intensidad de muestras de calibración (fluoróforos individuales sobre el vidrio y / o los receptores monoméricos marcados con fluorescencia), realizar un ajuste gaussiano mixta en la distribución de intensidades de partículas en el comienzo de una secuencia de imágenes para determinar el tamaño de los complejos receptores (es decir., el número de receptores por partícula) 10. Lleve a cabo estos análisis con Matlab o ssoftware imilar.- Calcular la intensidad de cada partícula por media de la intensidad de la partícula desde el primer fotograma en el bastidor antes de que el primero se produjo cambio en la intensidad (en la mayoría de los casos una disminución debido a photobleaching).
- Realizar un ajuste gaussiano mixta según la siguiente ecuación:
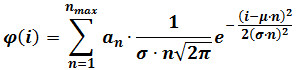
donde φ (i) es la frecuencia de partículas que tiene una intensidad i, n es el número de componentes, α n es un parámetro que contribuye a la altura del componente N, μ y σ son la media y la desviación estándar de la intensidad de fluoróforos individuales de referencia . (calculado según se describe en el paso 3) NOTA: Determine tél número máximo de componentes (n max) para cada secuencia de imágenes mediante el aumento progresivamente n max hasta que la adición de un componente no ya no producen un estadísticamente mejor ajuste, como se juzga por una prueba F. - Opcional (por ej., Los últimos 60 fotogramas de una secuencia de fotogramas 400) Realice un ajuste gaussiano mixta en la distribución de la intensidad obtenida en los últimos fotogramas de la película. Reemplace μ y σ con los valores obtenidos después de este ajuste, que proporcionan estimaciones refinadas para esos parámetros, y repita el paso 4.3.2.
- Calcular el área bajo la curva (AUC) de cada componente de la forma gaussiana mixta. Calcular la abundancia relativa de las partículas de los receptores de diferente tamaño (es decir, monómero, dímero, trímero, etc) dividiendo el valor de AUC de cada componente por la AUC de toda la distribución.
- Opcional: Use datos de diferentes células y las densidades de las partículas correspondientes (calculados como se describe en 4.2.6) para generar parcelas donde la distribución de partículas con diferente tamaño se correlaciona con la densidad de la partícula 10.
- Cálculo del tamaño de partícula con el método de ajuste de paso
NOTA: Utilice un análisis paso a montar como un método alternativo para determinar el tamaño de los complejos de receptores 10. La base de este análisis es que la destrucción inducida por la luz (photobleaching) de una sola resultados fluoróforo en su desaparición instantánea - Se espera por lo tanto, las partículas que contienen n fluoróforos que progresivamente lejía producir un perfil de intensidad con n pasos.- Extracto de perfiles de intensidad de cada partícula del archivo colchoneta. Generada por u-pista o software de detección / seguimiento similar (ver 4.2.6).
- Usa un algoritmo de paso de ajuste, tales como el presentadoen la ref. 10, para contar el número de etapas de blanqueo para cada partícula.
- Opcional: utilizar los resultados para generar distribuciones que muestran la abundancia relativa de las partículas de los receptores de diferente tamaño y correlacionarlos con densidad de las partículas como se describe anteriormente para los resultados de la instalación de Gauss mixto (ver 4.3).
Resultados
El protocolo descrito se puede aplicar a una variedad de diferentes proteínas de la membrana. Como un ejemplo, los resultados representativos obtenidos con β 2-adrenérgicos y GABA B se reportan 10. Dado que las señales fluorescentes de las moléculas individuales son débiles, la minimización de la fluorescencia de fondo es el primer paso clave para obtener resultados exitosos. Por lo tanto, es importante utilizar cubreobjetos ampliamente limpiadas (Figura 2A), así...
Discusión
El protocolo descrito permite el análisis de la disposición espacial, la movilidad y el tamaño de los complejos receptores de la superficie celular a nivel de una sola molécula. En comparación con el uso de proteínas fluorescentes, etiquetado con fluoróforos orgánicos pequeños, que son más brillantes y más fotoestable, tiene la ventaja de permitir la visualización ampliada de las partículas de los receptores individuales. Dado que los niveles de expresión extremadamente bajos se consiguen (<0,45 de los ...
Divulgaciones
The authors declare that they have no competing financial interests.
Agradecimientos
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
Materiales
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
Referencias
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados