Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Etiquetado específico de secuencia de ácidos nucleicos y proteínas con metiltransferasas y el cofactor Análogos
* Estos autores han contribuido por igual
En este artículo
Resumen
ADN y las proteínas se marcan secuencia específicamente con afinidad o grupos indicadores fluorescentes usando ADN o de proteínas metiltransferasas y análogos sintéticos de cofactor. Dependiendo de la especificidad de cofactor de las enzimas, aziridina o dobles análogos de cofactores activados se emplean para el etiquetado de uno o dos pasos.
Resumen
S -Adenosyl-L-metionina (AdoMet o SAM) metiltransferasas dependientes (MTase) catalizan la transferencia del grupo metilo activado a partir de AdoMet a posiciones específicas en el ADN, ARN, proteínas y pequeñas biomoléculas. Esta reacción de metilación natural puede ampliarse a una amplia variedad de reacciones de alquilación utilizando análogos cofactor sintéticos. La sustitución del centro de sulfonio reactiva de AdoMet con un anillo de aziridina conduce a cofactores que se pueden acoplar con el ADN por varios MTases de ADN. Estos cofactores de aziridina pueden ser equipados con grupos informadores en diferentes posiciones del resto de adenina y se utilizan para S-equence específica M ethyltransferase- I nduced L Abel ING de ADN (DNA sonriendo). Como un ejemplo típico damos un protocolo para la biotinilación de ADN del plásmido pBR322 en la secuencia 5'-ATCG A T-3 'con el MTase M.BseCI ADN y el cofactor aziridina en 6BAzun solo paso. Extensión del grupo metilo activado con grupos alquilo insaturados resultados en otra clase de análogos de AdoMet que se utilizan para m ethyltransferase dirigida TRANSFERENCIA DE UN ctivated rupos G (MTag). Dado que las cadenas laterales largos son activados por el centro de sulfonio y el enlace insaturado, estos cofactores son llamados análogos de AdoMet doble activadas. Estos análogos no sólo funcionan como cofactores para MTases de ADN, como los cofactores de aziridina, sino también para el ARN, proteínas y pequeños MTases molécula. Se utilizan típicamente para la modificación enzimática de sustratos MTase con grupos funcionales únicos que se etiquetan con grupos informadores en una segunda etapa química. Esto se ejemplifica en un protocolo para el marcaje de fluorescencia de la proteína histona H3. Un grupo propargilo pequeña se transfiere desde el SeAdoYn análogo cofactor de la proteína por la histona H3 lisina 4 (H3K4) MTase Set7 / 9 seguido por clic etiquetado de lahistona H3 alkynylated con TAMRA azida. Etiquetado mediada MTase con análogos cofactor es una tecnología que permite muchas aplicaciones interesantes, como la identificación y el estudio funcional de sustratos MTase así como el genotipado de ADN y detección de metilación.
Introducción
Un etiquetado específico de los ácidos nucleicos y proteínas 1,2 3,4 es de gran interés para las caracterizaciones funcionales, diagnóstico médico y (nano) biotecnología. Aquí presentamos un método de marcaje enzimático para estos biopolímeros que se basa en S -adenosyl-l-metionina (AdoMet o SAM) metiltransferasas dependientes (MTases). Esta clase de enzimas (EC 2.1.1.) Se dirige a posiciones individuales nucleófilos (nitrógeno, oxígeno, azufre y átomos de carbono) dentro de los residuos específicos de ácidos nucleicos y proteínas y, naturalmente, transfiere el grupo metilo activado de la AdoMet cofactor (Figura 1A) 5. Además, MTases pueden utilizar análogos sintéticos cofactor para el etiquetado específico con etiquetas de afinidad, fluoróforos u otras etiquetas (Figura 1B) 6. Dos clases de análogos de AdoMet se han desarrollado: cofactores aziridina para concreto-equence S M ethyltransferase- I Nduced L abel ing (sonriendo) 7 y dobles activados análogos AdoMet para m ethyltransferase dirigida TRANSFERENCIA DE UN ctivated rupos G (MTag) 8.

Figura 1: reacciones catalizadas por metiltransferasas (MTases) A. Metil transferencia de grupo desde el cofactor natural de AdoMet (SAM) a diversos sustratos incluyendo ADN, ARN, proteínas y pequeñas biomoléculas B. Etiquetado / funcionalización de los ácidos nucleicos y proteínas (NNNNN =.. pares de bases de ADN, nucleótidos de ARN para ácidos y aminoácidos para las proteínas; XXXXX = secuencia de reconocimiento de la MTase con objetivo de residuos en verde) con análogos de cofactor sintéticos. Cofactores aziridina contienen un grupo indicador (azul esfera)unido al anillo de adenina son secuencia junto específicamente con el objetivo de residuos (izquierda) y de doble activado análogos de AdoMet llevar a la transferencia de cadenas de alquilo extendidos que llevan un reportero química Y (derecha) que puede ser etiquetado por reacción bioorthogonal clic en un segundo paso. Haga clic aquí para ver una versión más grande de esta figura.
Cofactores aziridina funcionan mejor con ADN MTases. Contienen un anillo de tres miembros con un átomo de nitrógeno 9 (o un N de mostaza 10,11) en lugar del centro de sulfonio como grupo reactivo. La protonación de este átomo de nitrógeno activa el anillo de aziridina para el ataque nucleofílico por el nucleótido diana que conduce a Acoplamiento covalente de todo el cofactor con el ADN. Al conectar grupos informadores al anillo de adenina los cofactores de aziridina se pueden utilizar en combinación con ADN MTases para etiquetar ADN en un solo paso ( g> Figura 1B, izquierda) 7,12. Esto se demuestra en detalle para la biotinilación de ADN con 6BAz 13-15 (cofactor de aziridina con biotina unido a la posición 6 del anillo de adenina) y la adenina específica de ADN MTase de Bacillus stearothermophilus (M.BseCI) 16 (Figura 2, véase la sección 2 del protocolo: un solo paso etiquetado de ADN a través de aziridina cofactores). Además de M.BseCI ('secuencia de reconocimiento, la MTases ADN de Thermus aquaticus (M.TaqI, 5'-TCG A -3 5'-ATCG A T-3)'), a partir de Haemophilus heamolyticus (M.HhaI, 5 '-G C GC-3') y desde Spiroplasma (M.SssI, 5'-C G-3 ') se han utilizado con éxito para biotinilar ADN con 6BAz 17. Además, cofactores de aziridina pueden emplearse para un solo paso de fluorescencia de ADN etiquetado 18,19.
ontenido "fo: keep-together.within-page =" always ">
Figura 2:. Biotinilación específica de secuencia de un solo paso de ADN con M.BseCI y 6BAz El ADN MTase M.BseCI reconoce la secuencia de doble cadena de ADN 5'-ATCG A T-3 'y, naturalmente, metila el grupo amino de la segunda adenina residuo (verde) usando AdoMet. Con el cofactor aziridínico 6BAz el curso de la reacción se cambia y M.BseCI conduce a la secuencia de ADN específica biotinylation acoplando todo el cofactor incluyendo biotina (azul) con la adenina objetivo. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Doble análogos de AdoMet activados contienen extendieron cadenas laterales insaturadas en lugar de un grupo metilo en el centro de sulfonio (Figura 1B , derecha) 20. El doble o triple enlace insaturado en β-posición al centro de sulfonio compensa electrónicamente efectos estéricos desfavorables en el estado de transición de la estabilización conjugativo. Dado que tanto el centro de sulfonio y el enlace insaturado activan la cadena lateral para la transferencia enzimática, estos cofactores fueron nombrados análogos AdoMet doble activados. Típicamente, se utilizan para transferir cadenas laterales con grupos únicos químicos (reporteros químicos), como los grupos amino, alquino y azida, para el etiquetado quimioselectivo en un segundo paso 8,21. En general, de doble activado análogos AdoMet puede no sólo funcionan como cofactores de ADN MTases 8,20,21 sino también para ARN MTases 22,23 y MTases proteínas 24-28 permitiendo etiquetado adicional de ARN y proteínas. Sin embargo, las cadenas laterales largos son estéricamente más exigente que un grupo metilo y ampliando las MTase sitios activos de proteínas de ingeniería es oftes necesario para obtener las velocidades de transferencia eficientes. Otra solución a este problema es utilizar un análogo AdoMet con un grupo pequeño de propargilo (tres carbonos) donde el alquino terminal sirve para dos funciones: 1. estabilización del estado de transición durante la transferencia enzimática y 2. mango reactiva para el seguimiento de las modificaciones químicas de cobre catalizada cicloadición-azida alquino (CuAAC) haga clic en la química. Resultó que la propargílico resultante AdoMet analógico 29 es bastante inestable en condiciones neutras o ligeramente básicas y sólo de uso limitado. Este inconveniente se puede solucionar mediante la sustitución del átomo de azufre con el selenio. El cofactor resultante 5 '- [(Se) [(3 S) -3-amino-3-carboxipropil] prop-2-ynylselenonio] -5'-desoxiadenosina (SeAdoYn, Figura 3) que es aceptado por el ADN de tipo salvaje, el ARN y MTases proteína 30-32 que abroga la necesidad de la ingeniería de proteínas en muchos casos. Esto se ejemplifica por fluorescencia pro etiquetado proteína con la histona H3 lisina 4 (H3K4) MTase Set7 / 9 33 (Figura 3, véase la sección 3 del protocolo: el etiquetado de proteínas en dos pasos a través de cofactores dobles activados).
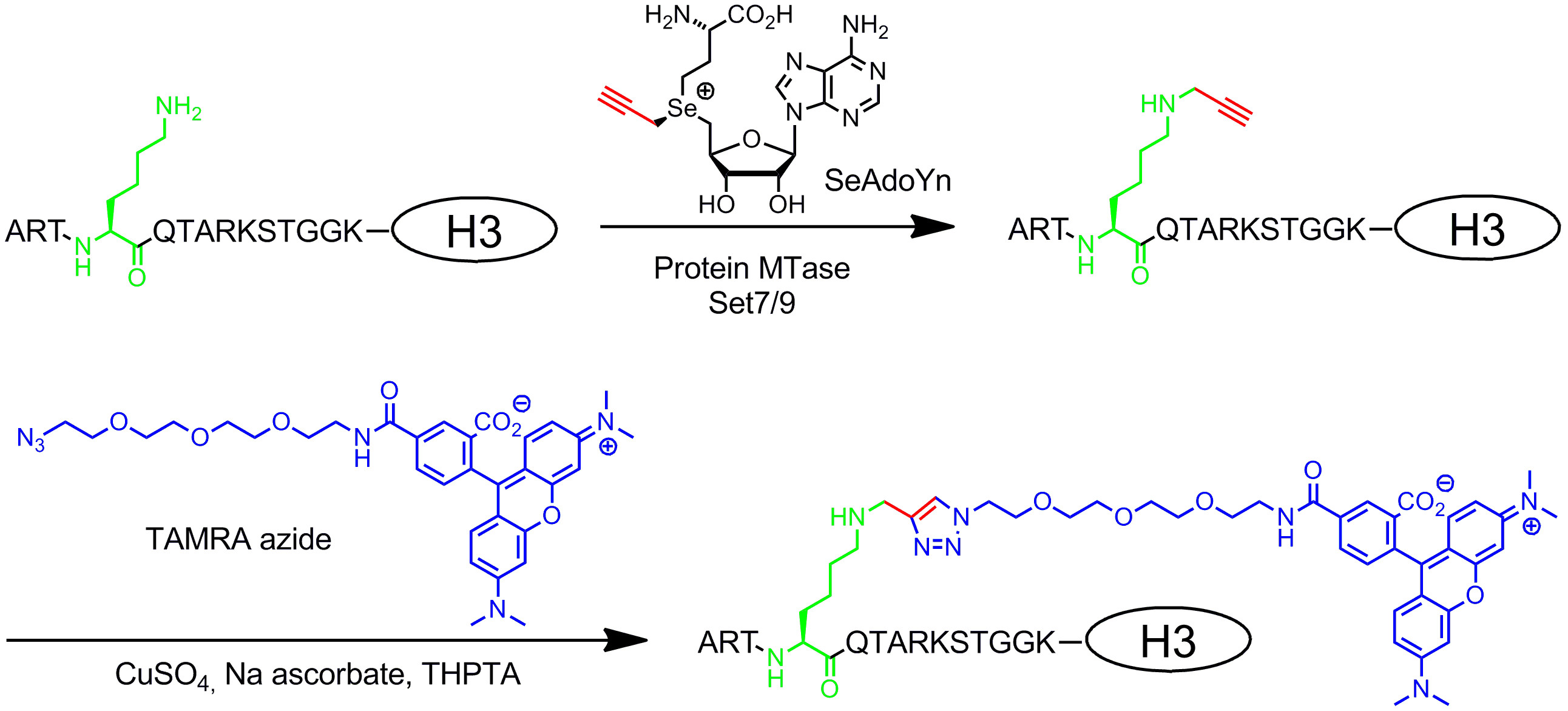
Figura 3:. De dos pasos de fluorescencia etiquetado específico de secuencia de la histona H3 con Set7 / 9, SeAdoYn y TAMRA azida La proteína MTase Set7 / 9 metila naturalmente el grupo amino de la lisina 4 en la histona H3 (H3K4, verde) usando AdoMet. Con el cofactor de doble activado SeAdoYn la MTase transfiere un grupo propargilo pequeña (rojo) al residuo de lisina. El triple enlace terminal conectado es luego modificado selectivamente en un clic reacción bioorthogonal (azida alquino cicloadición catalizada por cobre, CuAAC) con (tetrametilrodamina, azul) fluoróforo azida derivatizado TAMRA.carga / 52014 / 52014fig3highres.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Instrucciones generales
- Aziridínico tienda cofactor 6BAz (en DMSO) y la proteína MTase Set7 / 9 a -80 ° C y todos los demás reactivos incluidos cofactor doble activado SeAdoYn y ADN MTase M.BseCI (en el 50% de glicerol) a -20 ° C.
- Determinar la concentración de 6BAz y SeAdoYn mediante espectroscopia UV / Vis utilizando los coeficientes de extinción ε 269 nm (6BAz) = 16,000 cm -1 M -1 y ε 260 nm (SeAdoYn) = 15,400 cm -1 M -1 en agua desionizada. Determinar la concentración de MTases por el ensayo de Bradford o, si el coeficiente de extinción está disponible, a través de la absorción directa a 280 nm.
- Trate de evitar la creación de burbujas pipeteando intensiva o vórtex para evitar la pérdida de la actividad enzimática. En su lugar, se mezcla con la pipeta suavemente hacia arriba y hacia abajo.
- Cuando la adición de cofactores de aziridina partir de soluciones madre en DMSO asegurarse de que la concentración final de DMSO en el ensayo es menosal 5%. Siempre incluya 10 iones de magnesio mM en el tampón de ensayo para evitar reacciones inespecíficas con ADN.
- Cuando la adición de cofactores dobles activados partir de soluciones madre ácidas usar pequeños volúmenes (soluciones madre altamente concentradas) para evitar cambios de pH y asegúrese de que el pH de la solución de ensayo no cambia significativamente. Evitar tioles, por ejemplo, β-mercaptoetanol o ditiotreitol (DTT), en el tampón de ensayo, ya que pueden interferir con la reacción de clic por complejación de los iones de cobre requeridas.
2. Un paso Etiquetado de ADN a través de aziridina cofactores
- Etiqueta ing Metiltransferasa inducido-secuencia específica (sonriendo) de ADN plásmido con M.BseCI ADN MTase y cofactor aziridínico 6BAz.
- Descongelar la solución de cofactor a 20 ° C y preparar las mezclas de reacción en hielo.
- Además del ensayo de realizar un control "cofactor", para visualizar las modificaciones no específicas, y un & #8220; enzima "control, para asegurarse de que la preparación MTase está libre de la AdoMet cofactor natural.
- Para la mezcla de ensayo de 2 l de tampón 10X modificación (que contiene 100 mM Tris-HCl, 100 mM MgCl2, 20 mM β-mercaptoetanol, pH 7,4), 2 l de pBR322 (0,5 g / l), 10 eq. M.BseCI por secuencia de reconocimiento en el ADN (secuencia 1 reconocimiento en pBR322) y el cofactor aziridina 6BAz a una concentración final de 60 micras dentro de un volumen total de 20 l. Añadir cofactor y el ADN MTase pasado.
NOTA: β-mercaptoetanol es tóxico, corrosivo y perjudicial para el medio ambiente. - Para el control "cofactor" añadir agua desionizada en lugar de M.BseCI y para el control "enzima" añadir agua desionizada en lugar de 6BAz.
- Mezclar las soluciones pipeteando suavemente hacia arriba y hacia abajo.
- Incubar los tubos a 55 ° C durante 1 hr.
- Centrifugar brevemente para recoger todo el líquido en la parte inferior de los tubos.
- Ensayo de restricción-modificación para verificar la modificación del ADN.
- Preparar una solución mediante la mezcla de 10 l de 10x tampón R.TaqI (que contiene 100 mM de Tris-HCl, 50 mM de MgCl 2, 1 M NaCl, 1 mg / ml de albúmina de suero bovino, pH 8,0), 80 l de agua desionizada y 3,3 l de la endonucleasa de restricción (REase) a partir de Thermus aquaticus (R.TaqI, 10 U / l). Asegúrese de añadir el REase en el último paso.
- A cada tubo de 2.1.7 añadir 2 l de tampón 10X Taq I y R. 28 l de la solución desde arriba (2.2.1).
- Mezclar las soluciones pipeteando suavemente hacia arriba y hacia abajo.
- Incubar los tubos a 65 ° C durante 30 min.
- Centrifugar brevemente para recoger todo el líquido en la parte inferior de los tubos.
- Ensayo de cambio de movilidad eléctrica (EMSA) con estreptavidina para verificar modificación funcional.
- Retire 25 l de cada tubo (2.2.5) y añadir 2,4 l de una solución de estreptavidina (1 mM con respecto a la estreptavidina monomer estreptavidina en tampón que contiene 100 mM Na 2 HPO 4, NaCl 100 mM, pH 7,5; 4 equivalentes de biotina total). Añadir 2,4 l de tampón de estreptavidina a los tubos restantes.
- Incubar todos los tubos a 37 ° C durante 1 hr.
- Análisis mediante electroforesis en gel de agarosa.
- Añadir 5 l de tampón de carga 6x (0,25% azul de bromofenol, 30% de glicerol) a cada tubo.
- Mezclar las soluciones suavemente.
- Cargar 10 l de cada muestra en los pocillos de un gel de agarosa (1% de agarosa en tampón TBE 0,5x contiene 1x GelRed de una solución 10.000x de valores).
- Ejecute el gel en tampón TBE 0,5x con 80 V durante aprox. 1 hr.
- Visualizar bandas de ADN en un vector de UV (312 nm) con una cámara CCD equipada con un filtro (540 ± 50 nm).
NOTA: La luz UV es perjudicial para los ojos y la piel.
3. Dos etapas de etiquetado de proteínas a través de cofactores dobles Activado
- MethyltransfTransferencia de grupos activados (MTag) con Set7 / 9 y cofactor de doble activado SeAdoYn para la histona H3 lisina 4 etiquetado (etapa de modificación) borrar-Dirigida.
- Descongele los componentes y preparar las mezclas de reacción en hielo. NOTA: Mantenga siempre SeAdoYn enfría para evitar la degradación.
- Además del ensayo de realizar un control "cofactor", para visualizar las modificaciones no específicas, y un control "enzima", para excluir reacciones no específicas de la sonda fluorescente.
- Preparar una solución de ensayo (20 l) que contenía tampón de modificación (mM Tris-HCl 50, 5% de glicerol, pH 8,5), 10 mM de histona H3, 10 mM Set7 / 9 y 600 mM SeAdoYn (mezcla de ambos epímeros en selenio). En los últimos pasos añadir cofactor y luego MTase.
- Para el control "cofactor" preparar una solución de ensayo como en 3.1.3 y añadir 60 mM AdoMet para competir con el cofactor sintético. Para el control "enzima" añadir agua desionizada en lugar de SeAdoYn.
- Mezclar las soluciones con la pipeta lentamente hacia arriba y hacia abajo. Comprobar el pH mediante la adición de 1 l de cada solución para el campo superior de una tira de pH (intervalo de pH de 5 - 10).
- Incubar a 37 ° C durante 2 hr.
- En el gel Mientras tanto preparar un gel de poliacrilamida SDS 12% (que se ejecuta: 357 Bis-Tris pH 6.5 a 6.8 mM, 0,1% (w / v) APS, 0,04% (v / v) TEMED y 12% de acrilamida / bisacrilamida 37,5: 1 ; gel de carga: 357 Bis-Tris pH 6.5 a 6.8 mM, 0,1% (w / v) APS, 0,04% (v / v) TEMED y 5% de acrilamida / bisacrilamida 37,5: 1).
NOTA: La acrilamida / bisacrilamida es tóxico y la salud peligrosos. Use guantes durante este procedimiento.
- Etiquetado química de lisina alkinylated 4 en la histona H3 mediante cicloadición-azida alquino (CuAAC) (etapa de marcaje) catalizada por cobre.
- Justo antes del final de la reacción de modificación preparar una mezcla 5x clic que contiene 3 mM CuSO 4, 3 mM tris (3-hidroxipropil-triazolilmetilo) amina (THPTA), ascorbato de sodio 250 mM y 6 mM TAMRA azida con unavolumen total de 20 l.
- Añadir 5 l de la mezcla 5x clic recién preparado a cada tubo para iniciar la CuAAC y apagar la reacción de modificación.
- Mezclar suavemente con la pipeta hacia arriba y hacia abajo.
- Proteger a todos los tubos con papel de aluminio de la luz para evitar la foto-blanqueo del fluoróforo.
- Incubar a 20 ° C durante 1 hr.
- Precipitación de proteínas para eliminar el exceso de libre fluoróforo TAMRA.
- Para evitar resplandor de la etiqueta fluorescente histona H3 por intensa fluorescencia en gel de libre fluoróforo TAMRA, eliminar el exceso de fluoróforo por precipitación de proteínas (3.3.2 - 3.3.4) 34.
- Añadir 75 l de metanol, 18,8 l de cloroformo y 50 l de agua desionizada a cada tubo y vórtice brevemente después de cada adición. Centrifugar a 16.000 xg durante 5 min. Eliminar la fase superior sin molestar a la capa de interfaz, que contiene la proteína.
- Añadir 56,3 l de metanol a la fase restante in cada tubo, vórtice y se centrifuga a 16.000 xg durante 5 min para sedimentar la proteína. Eliminar el sobrenadante. Repita este paso para lavar el pellet.
- Cubra los tubos abiertos con un pañuelo de papel libre de pelusa y deje que se sequen durante 15 - 30 min.
- Análisis mediante SDS PAGE.
- Disolver las proteínas precipitadas a partir de 3.3.4 en 20 l de tampón SDS de carga (50 mM Tris-HCl, 2,5% (w / v) SDS, 10% (v / v) de glicerol, 320 mM β-mercaptoetanol y 0,05% (w / v) de azul de bromofenol, pH 6,8). Asegúrese de que para disolver completamente el pellet enjuagando las paredes de los tubos con una pipeta.
- Incubar las muestras a 95 ° C durante 10 minutos y dejar enfriar a 20 ° C.
- Centrifugar brevemente para recoger todo el líquido en la parte inferior de los tubos.
- Cargue la cantidad total de cada muestra en los pocillos de un gel de poliacrilamida SDS (3.1.7). Utilice MOPS 50 mM, 50 mM Tris-X (Tris-base), EDTA 5 mM, 0,1% (w / v) SDS como tampón para electroforesis.
- Ejecute el gel con 120 V durante aprox. 90 min.
- Visualizar la fluorescencia en gel sobre una mesa de UV (312 nm) con una cámara CCD equipada con un filtro (540 nm ± 50 nm).
NOTA: La luz UV es perjudicial para los ojos y la piel.
Resultados
Un paso Etiquetado de ADN a través de aziridina cofactores
Esta reacción se lleva a cabo ejemplo con el ADN MTase M.BseCI, que se modifica el segundo residuo de adenina en el extremo 5 'ATCG A T-3' secuencia de doble cadena y tiene un sitio de reconocimiento en el plásmido pBR322 (Figura 4A). Para probar etiquetado plásmido, pBR322 es desafiado con la endonucleasa de restricción (REase) R.TaqI (5'-TCGA-3 '). R.TaqI tiene siete s...
Discusión
Un paso etiquetado de ADN con MTases ADN y cofactores aziridina (ADN sonriendo) es un método robusto pero algunos aspectos se debe considerar al planear el experimento.
Cofactor Aziridine: La concentración 6BAz para el etiquetado de ADN con M.BseCI fue de 60 mM. Cuando se utilizan otros MTases la concentración de ADN cofactor debe optimizarse, por ejemplo, concentraciones tan bajas como 20 mM han sido empleados con el ADN MTase M.TaqI 19. Las bajas concentraci...
Divulgaciones
The authors disclose the following competing financial interest: E.W. is inventor on related patents.
Agradecimientos
The authors thank Kerstin Glensk for preparing the MTases M.BseCI and Set7/9 and gratefully acknowledge funding by the Excellence Initiative of the German Federal and State Governments and RWTH Aachen University. The authors are happy to provide 6BAz and SeAdoYn or other cofactor analogues for collaborative research.
Materiales
| Name | Company | Catalog Number | Comments |
| 6BAz | Synthesized according to Weinhold et al., Patent number US 8,129,106, published March 6, 2012. | ||
| β-Mercaptoethanol | Serva | 28625 | |
| Acetic acid | Fisher Scientific | 10304980 | |
| Acrylamide/Bis Solution, 37.5:1 | Serva | 10688 | |
| UltraPure Agarose | Invitrogen | 16500100 | |
| Ammonium persulfate (APS) | Serva | 13375 | |
| Bis-Tris | Gerbu | 1304 | |
| Boric acid | Gerbu | 1115 | |
| Bromophenol blue Na salt | Serva | 15375 | |
| Copper(II) sulfate | Aldrich | C1297 | |
| Chloroform | Fisher Scientific | 10020090 | |
| Coomassie Brilliant Blue | Serva | 17525 | |
| EDTA disodium salt | Gerbu | 1034 | |
| Ethanol | Merck | 100983 | |
| GelRed (10,000x in water) | Biotium | 41003 | |
| Glycerol (99.5%) | Gerbu | 2006 | |
| FastRuler Low Range DNA Ladder | Thermo Scientific | SM1103 | |
| Histone H3 | Expression plasmid obtained from Dr. Philipp Voigt and Prof. Danny Reinberg; expression and isolation according to T. J. Richmond et al., J. Mol. Biol. 1997, 272, 301-311. | ||
| M.BseCI | Expression plasmid obtained from Dr. Michael Kokkinidis; expression and isolation according to Kapetaniou et al., Acta Cryst. 2006, F63, 12-14. | ||
| Methanol | Fisher Scientific | 10675112 | |
| Magnesiumchloride hexahydrate | J.T. Baker | 4003 | |
| MOPS | Gerbu | 1081 | |
| Sodium chloride | Gerbu | 1112 | |
| pH strip (Neutralit) | Merck | 1,095,330,001 | |
| pBR322 | Thermo Scientific | SD0041 | |
| R.TaqI (10 u/µl) | Thermo Scientific | ER0671 | |
| SeAdoYn | Synthesized according to Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| Set7/9 | Expression plasmid obtained from Prof. Danny Reinberg, expression and isolation according to D. Reinberg et al., Genes Dev.2002, 16, 479-489. | ||
| Streptavidin | Gerbu | 3058 | |
| (+)-Sodium L-ascorbate | Sigma Life Science | A7631 | |
| SDS Granular | Gerbu | 1833 | |
| di-Sodium hydrogenphosphate | Merck | 106,586 | |
| TAMRA azide | Synthesized according to reference 30: Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| TaqI buffer (10x) | Thermo Scientific | B28 | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) | Acros Organics | 42058 | |
| Tris-HCl | Gerbu | 1028 | |
| Tris-X (TRIS-base) | Gerbu | 1018 | |
| Tris(3-hydroxypropyltriazolyl-methyl)amine (THPTA) | Sigma-Aldrich | 762342 |
Referencias
- Gottfried, A., Weinhold, E. Sequence-specific covalent labelling of DNA. Biochem. Soc. Trans. 39, 623-628 (2011).
- Zohar, H., Muller, S. J. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. Nanoscale. 3, 3027-3039 (2011).
- Hinner, M. J., Johnsson, K. How to obtain labeled proteins and what to do with them. Curr. Opin. Biotechnol. 21, 766-776 (2010).
- Wua, Y. -. W., Goody, R. S. Probing protein function by chemical modification. J. Pept. Sci. 16, 514-523 (2010).
- Struck, A. -. W., Thompson, M. L., Wong, L. S., Micklefield, J. S-Adenosyl-methionine-dependent methyltransferases: Highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. ChemBioChem. 13, 2642-2655 (2012).
- Klimasauskas, S., Weinhold, E. A new tool for biotechnology: AdoMet-dependent methyltransferases. Trends Biotechnol. 25, 99-104 (2007).
- Pljevaljcic, G., Schmidt, F., Weinhold, E. Sequence-specific Methyltransferase-Induced Labeling of DNA (SMILing DNA). ChemBioChem. 5, 265-269 (2004).
- Lukinavicius, G., Lapiene, V., Stasevskij, Z., Dalhoff, C., Weinhold, E., Klimasauskas, S. Targeted labeling of DNA by methyltransferase-directed Transfer of Activated Groups (mTAG). J. Am. Chem. Soc. 129, 2758-2759 (1021).
- Pignot, M., Siethoff, C., Linscheid, M., Weinhold, E. Coupling of a nucleoside with DNA by a methyltransferase. Angew. Chem. Int. Ed. 37, 2888-2891 (1998).
- Weller, R. L., Rajski, S. R. Design, synthesis, and preliminary biological evaluation of a DNA methyltransferase-directed alkylating agent. ChemBioChem. 7, 243-245 (2006).
- Du, Y., Hendrick, C. E., Frye, K. S., Comstock, L. R. Fluorescent DNA Labeling by N-Mustard Analogues of S-adenosyl-l-methionine. ChemBioChem. 13, 2225-2233 (2012).
- Pljevaljcic, G., Schmidt, F., Scheidig, A. J., Lurz, R., Weinhold, E. Quantitative labeling of long plasmid DNA with nanometer precision. ChemBioChem. 8, 1516-1519 (1002).
- Wilkinson, S., et al. Molecular scale architecture: engineered three- and four-way junctions. Bioconjugate Chem. 19, 470-475 (2008).
- Braun, G., et al. Enzyme-directed positioning of nanoparticles on large DNA templates. Bioconjugate Chem. 19, 476-479 (2008).
- Kim, S., et al. Enzymatically incorporated genomic tags for optical mapping of DNA binding proteins. Chem. Int. Ed. 51, 3578-3581 (2012).
- Rina, M., Bouriotis, V. Cloning purification and characterization of the BseCI DNA methyltransferase from Bacillus stearothermophilus. Gene. 133, 91-94 (1993).
- Weinhold, E., Meier, T., Düfel, H., Markert-Hahn, C., Schmuck, R. Sequence-specific detection of methylation in biomolecules. US Patent. , (2012).
- Pljevaljcic, G., Pignot, M., Weinhold, E. Design of a new fluorescent cofactor for DNA methyltransferases and sequence-specific labeling of DNA. J. Am. Chem. Soc. 125, 3492-3410 (2003).
- Schmidt, F. H. -. G., Hüben, M., Gider, B., Renault, F., Teulade-Fichou, M. -. P., Weinhold, E. Sequence-specific Methyltransferase-Induced Labelling (SMILing) of plasmid DNA for studying cell transfection. Bioorg. Med. Chem. 16, 40-48 (2008).
- Dalhoff, C., Lukinavicius, G., Klimasauskas, S., Weinhold, E. Direct transfer of extended groups from synthetic cofactors by DNA methyltransferases. Nat. Chem. Biol. 2, 31-32 (2006).
- Lukinavicius, G., Tomkuviene, M., Masevicius, V., Klimasauskas, S. Enhanced chemical stability of AdoMet analogues for improved methyltransferase-directed labeling of DNA. ACS Chem. Biol. 8, 1134-1139 (2013).
- Motorin, Y., et al. Expanding the chemical scope of RNA:methyltransferases to site-specific alkynylation of RNA for click labeling. Nucleic Acids Res. 39, 1943-1952 (1943).
- Schulz, D., Holstein, J. M., Rentmeister, A. A chemo-enzymatic approach for site-specific modification of the RNA cap. Angew. Chem. Int. Ed. 52, 7874-7878 (2013).
- Peters, W., et al. Enzymatic site-specific functionalization of protein methyltransferase substrates with alkynes for click labeling. Angew. Chem. Int. Ed. 49, 5170-5173 (2010).
- Islam, K., Zheng, W., Yu, H., Deng, H., Luo, M. Expanding cofactor repertoire of protein lysine methyltransferase for substrate labeling. ACS Chem. Biol. 6, 679-684 (2011).
- Wang, R., Zheng, W., Yu, H., Deng, H., Luo, M. Labeling substrates of protein arginine methyltransferase with engineered enzymes and matched S-adenosyl-l-methionine analogues. J. Am. Chem. Soc. 133, 7648-7651 (2011).
- Islam, K., et al. Bioorthogonal profiling of protein methylation using azido derivative of S-adenosyl-l-methionine. J. Am. Chem. Soc. 134, 5909-5915 (2012).
- Islam, K., et al. Defining efficient enzyme-cofactor pairs for bioorthogonal profiling of protein methylation. Proc. Natl. Acad. Sci. U.S.A. 110, 16778-16783 (2013).
- Binda, O., Boyce, M., Rush, J. S., Palaniappan, K. K., Bertozzi, C. R., Gozani, O. A chemical method for labeling lysine methyltransferase substrates. ChemBioChem. 12, 330-334 (2011).
- Willnow, S., Martin, M., Lüscher, B., Weinhold, E. A selenium-based click AdoMet analogue for versatile substrate labeling with wild-type protein methyltransferases. ChemBioChem. 13, 1167-1173 (2012).
- Bothwell, I. R., et al. Se-Adenosyl-l-selenomethionine cofactor analogue as a reporter of protein methylation. J. Am. Chem. Soc. 134, 14905-14912 (2012).
- Tomkuviene, M., Clouet-d’Orval, B., Cerniauskas, I., Weinhold, E., Klimasauskas, S. Programmable sequence-specific click-labeling of RNA using archaeal box C/D RNP methyltransferases. Nucleic Acids Res. 40, 6765-6773 (2012).
- Nishioka, K., et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation. Genes Dev. 16, 479-489 (2002).
- Clark, P. M., et al. Direct in-gel fluorescence detection and cellular imaging of O-GlcNAc-modified proteins. J. Am. Chem. Soc. 130, (2008).
- Lukinavicius, G., Lapinaite, A., Urbanaviciute, G., Gerasimaite, R., Klimasauskas, S. Engineering the DNA cytosine-5 methyltransferase reaction for sequence-specific labeling of DNA. Nucleic Acids Res. 40, 11594-11602 (2012).
- Neely, R. K., Dedecker, P., Hotta, J., Urbanaviciute, G., Klimasauskas, S., Hofkens, J. DNA fluorocode: A single molecule, optical map of DNA with nanometre resolution. Chem. Sci. 1, 453-460 (2010).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Res. 38, 234-236 (2010).
- Petrossian, T. C., Clarke, S. G. Uncovering the human methyltransferasome. Mol. Cell. Proteomics. 10, 1-12 (2011).
- Kriukiene, E., et al. DNA unmethylome profiling by covalent capture of CpG sites. Nat. Commun. 4, 2190 (2013).
- Wang, R., et al. Profiling genome-wide chromatin methylation with engineered posttranslation apparatus within living cells. J. Am. Chem. Soc. 135, 1048-1056 (2013).
- Zhang, C., Weller, R. L., Thorson, J. S., Rajski, S. R. Natural product diversification using a non-natural cofactor analogue of S-adenosyl-l-methionine. J. Am. Chem. Soc. 128, 2760-2761 (2006).
- Stecher, H., et al. Biocatalytic Fiedel-Crafts alkylation using non-natural cofactors. Angew. Chem. Int. Ed. 48, 9546-9548 (2009).
- Lee, B. W. K., Sun, H. G., Zang, T., Kim, B. J., Alfaro, J. F., Zhou, Z. S. Enzyme-catalyzed transfer of a ketone group from an S-adenosylmethionine analogue: A tool for the functional analysis of methyltransferases. J. Am. Chem. Soc. 132, 3642-3643 (2010).
- Winter, J. M., et al. Expanding the structural diversity of polyketides by exploring the cofactor tolerance of an inline methyltransferase domain. Org. Lett. 15, 3774-3777 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados