Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La detección de biomarcadores exosomal por inducida por campo eléctrico de lanzamiento y Medición (Efirm)
En este artículo
Erratum Notice
Resumen
Exosomes are microvesicular structures found within biofluids that potentially carry important disease discriminatory biomarkers. Here, a novel method is used to specifically extract exosomes and rapidly test the exosomal cargo for both RNA/protein targets following the disruption of exosomes using non-uniform electric cyclic square waves.
Resumen
Los exosomas son estructuras microvesicular que juegan un papel mediador en la comunicación intercelular. Es de interés para el estudio de la carga interna de exosomas para determinar si llevan biomarcadores discriminatorias de enfermedades. Para la realización de análisis exosomal, es necesario desarrollar un método para la extracción y el análisis de los exosomas de biofluidos objetivo sin dañar el contenido interno.
Liberación inducida por el campo eléctrico y medición (Efirm) es un método para extraer específicamente exosomas de biofluidos, descargando su carga, y prueba de su contenido interno de ARN / proteína. El uso de un anticuerpo micropartícula magnética específica anti-CD63 humano, los exosomas son primero precipitan a partir de biofluidos. Después de la extracción, las ondas de baja tensión eléctricos cíclicos cuadrados (CSW) se aplican a romper la membrana vesicular y causar desembarque de la carga. El contenido de la exosome se hibrida con cebadores de ADN o anticuerpos inmovilizados sobre una superficie de electrodo para quantification del contenido molecular.
El método Efirm es ventajoso para la extracción de los exosomas y descargando una carga para el análisis sin tampón de lisis. Este método es capaz de realizar la detección específica de ambas dianas de ARN y proteína de biomarcadores en el exosome. Efirm extrae exosomas específicamente en base a sus marcadores de superficie en comparación con las técnicas de tamaño basada en acciones.
Microscopía electrónica de transmisión (TEM) y el ensayo demuestran la funcionalidad del método para la captura y análisis exosome. El método se aplicó a Efirm exosomal análisis de 9 ratones inyectados con células de pulmón humano H640 cáncer (una línea celular transfectada para expresar el marcador exosome humano CD63-GFP) con el fin de poner a prueba su perfil exosome contra 11 ratones que recibieron los controles salinos. Se hallaron niveles elevados de biomarcadores exosomal (referencia GAPDH gen y la proteína marcador de superficie CD63 humano-GFP) para el H640 ratones inyectados en ambas muestras de suero y saliva. Además, saliva y muestras de suero se demostró tener linealidad (R = 0,79). Estos resultados son indicativos para la viabilidad de los biomarcadores de exosoma salivales para la detección de enfermedades distales.
Introducción
Investigación exosome es un campo emergente de investigación que examina microvesículas lípidos que llevan ARN 1, DNA 2, y la proteína 3 de carga. Investigaciones anteriores de la biología exosome han conducido a la identificación de los exosomas en biofluidos tales como sangre 4, 5 orina, leche materna 6, 7 y la saliva. Los estudios han demostrado que los exosomas juegan un papel en diferentes vías celulares, meditando remotamente la comunicación entre diferentes sistemas del cuerpo 8. Debido al papel exosomas desempeñan en la comunicación intercelular, se plantea la hipótesis de que pueden empaquetar objetivos de biomoléculas (proteínas, ARN y ADN) correlacionados con estados de enfermedad. In vitro 3 y animal modelo de 9 estudios parecen corroborar esta hipótesis. En la investigación de contenido exosomal para el descubrimiento de biomarcadores, es necesario el desarrollo de una metodología para el aislamiento selectivo de exosome biofluidos, expulsi inducidaen la carga de los exosomas, y cuantificación de biomoléculas de exosoma. En la medida de este trabajo, los exosomas se definirán como una estructura que tiene un diámetro de aproximadamente 70-100 nm y poseyendo la superficie marcador CD63.
Los investigadores típicamente primera purifican exosomas por ultracentrifugación 10 y luego procesar el contenido exosomal través de la utilización de kits de tampón de lisis. El uso de métodos de tampón de lisis requiere tiempos de incubación que van desde minutos a horas. Este proceso puede potencialmente dañar carga exosome y conducir a probar la degradación. Por ejemplo, el ARN exosome salival lanzado a través de tampón de lisis en el entorno extracelular que rodea posee una vida media de menos de 1 min, por lo que la medición de RNA exosomal post-Tampón de Lisis una tarea particularmente difícil sin la adición de reactivos de estabilización 11. El efecto combinado de la adición de diversos reactivos para la lisis y la estabilización puede introducir agentes que complican e interferir con la Analysis de contenido exosomal. Un enfoque alternativo puede ser útil para la descarga rápidamente contenido exosomal y conservar de forma segura la carga para su caracterización.
En este trabajo, se propone el uso de un campo eléctrico no uniforme para la liberación de contenido exosomal. Eléctricos campos han sido conocidos por llevar la capacidad de polarizar y perturbar la bicapa lipídica que forma las membranas celulares. Nuestro trabajo experimental explora el uso de ondas cuadradas cíclicos no uniformes (CSW) para interrumpir la estructura microvesícula de exosomas y la liberación de la carga transportada. Este método utiliza tensiones en el rango de varios cientos de milivoltios, lo que significa que la mayoría de las biomoléculas no serán interrumpidos. Se demuestra que el uso de una onda cíclica cuadrados es capaz de accionar la liberación de exosome salival contenido de mRNA en el medio ambiente fluido circundante. Esta versión de contenido exosomal se integra perfectamente con un sistema de electrodos que puede ser utilizado para cuantificar los niveles de expresión de biomarcador 12,13. Este método propuesto permite una rápida sensible, y el tampón de lisis análisis, libres de contenidos exosome.

Figura 1. Visión general de Efirm de flujo de trabajo.. El método Efirm se divide a grandes rasgos en las tres fases principales que son necesarios para la purificación y el análisis de los exosomas.
Este método de liberación contenido exosomal y el análisis basado CSW se utiliza en conjunción con microperlas magnéticas-CD63 específico para el aislamiento exosome. Estas perlas CD63-de afinidad permiten el aislamiento selectivo de los exosomas de las muestras salivales (y otros fluidos biológicos). Después de la incubación y la extracción de los exosomas utilizando las perlas magnéticas, las cuentas se migran al sistema de sensor electroquímico para la CSW basado comunicado de contenido y parte de análisis del experimento. Figura 1 ofrece una visión general de la obrade flujo del método Efirm.
Protocolo
1. magnética exosome extracción basado en perlas
- Pipetear una solución bien mezclada de 5 l de micropartículas magnéticas recubiertas con estreptavidina en 495 l de solución salina tamponada (PBS) tampón de fosfato en un tubo de microcentrífuga para resuspender las perlas. Lavar y resuspender las perlas con 500 l de PBS tres veces usando una rejilla magnética. El bastidor es un conjunto de imanes en el lado de una unidad de vivienda que puede sostener los tubos de muestra de microcentrífuga.
- Para cada lavado, primero deje que los tubos se sientan en el estante durante 1 minuto, y luego usar una punta de pipeta para retirar con cuidado el tampón sobrenadante sin perturbar las cuentas.
- Colocar los tubos en un estante regular sin imanes en el lado. Añadir 500 l de PBS en los tubos, y el uso de la pipeta para mezclar la solución y los granos juntos. A continuación, poner los tubos de nuevo en la gradilla magnética para separar de nuevo las perlas de la solución.
- Realizar esta eliminación de tampón a través de la magnetización y resuspensión en PBS unatotal de tres veces. Esto realiza un lavado inicial de las partículas magnéticas.
- Resuspender las perlas en 490 l de tampón de PBS, con el tubo colocado en la parte no magnetizada de la gradilla magnética. Pipetear 5 l de ratón biotinilado anticuerpo anti-CD63 humano en 1,0 mg / ml de concentración de stock en la mezcla de perlas. Use la pipeta para mezclar las perlas y el anticuerpo en solución.
- Coloque los tubos de microcentrífuga con el grano y la mezcla de anticuerpos con biotina en un rotador de muestras. Establezca los parámetros de los rotadores para el rotador de la muestra para la rotación recíproca a 90 ° de inclinación durante 5 segundos y vibrando a 5 ° durante 1 seg. Rotar los tubos de mezcla de muestra de perlas en estos parámetros para 30 min a TA.
- Eliminar el anticuerpo no unido después de la conjugación.
- Después de 30 min de rotación a TA, coloque los tubos de nuevo en la gradilla magnética durante 5 min.
- Realizar tres lavados de los granos mediante la eliminación de la fase líquida utilizando una micropipeta y se lava con 500 μ; L de PBS. Después de la triple lavado, volver a suspender las perlas en 490 l de caseína-PBS y colocar en la parte no magnetizado de la cremallera.
- Extracción exosome usando perlas recubiertas de anticuerpo.
- Marque cada tubo con específica identificación de la muestra. Pipetear una muestra de 10 l de suero o saliva en el tubo de microcentrífuga. Use la pipeta para mezclar la muestra y perlas magnéticas pipeteando varias veces.
- Colocar los tubos con la muestra y anti-humanos perlas de anticuerpo CD63 en los rotadores y rotar durante 2 horas a RT. Utilice los mismos parámetros de los rotadores, como se describe en el paso 1.2.
- Después de 2 hr de la muestra de rotación, realizar un lavado triple de magnetización a perlas separadas de la solución, la eliminación de fase líquida con micropipeta, y resuspensión de perlas en 500 l de tampón Tris-HCl. Las perlas resultantes están ligados a los exosomas y están listos para la liberación del campo eléctrico y la medición.
2. Electric Field inducida publicó unnd Medición de exosomal contenido
- Inicial PRECOATING de Electrodo con GADPH Primer
- Aplicar un plástico bien a un conjunto de electrodos para evitar la contaminación cruzada de los electrodos individuales. Para este experimento, utilizar un conjunto de electrodos 16-sensor con cada electrodo unidad en la matriz que consiste en un trabajo, contador, y electrodo de referencia de oro desnudo.
- Preparar una mezcla madre de 100 Nm sonda de ADN, 0,3 M KCl, y pirrol 10 mM pipeteando reactivos de valores en un tubo con agua destilada ultrapura. Mezclar bien por agitación.
NOTA: Para este estudio, la sonda de ADN seleccionada corresponde a la referencia de genes GAPDH, que se sabe que existe dentro de los exosomas. La secuencia de la sonda utilizada es: 5'-Biotina-AGGTCCACCACTGACACGTTG-3 '. Utilice esta mezcla sobre todos los electrodos. - Pipetear 60 l de la mezcla de sondas de ADN-monómero sobre la superficie de cada electrodo de oro. Examinar los electrodos para garantizar que hay una cobertura adecuada de la de trabajo, counter, y de referencia de la mezcla líquida.
- Electropolymerize mezcla de monómeros-sonda para crear una capa de polímero conductor en la superficie del electrodo mediante la aplicación de una onda cuadrada (CSW) el perfil de campo eléctrico cíclico a la superficie del electrodo. Este campo eléctrico consiste en aplicar 350 mV durante 9 s e inmediatamente cambiar a 950 mV durante 1 seg. Aplicar este perfil cíclico de onda cuadrada al electrodo durante 10 ciclos, para un total de 100 seg de campo eléctrico aplicado.
- Enjuague superficie del sensor 3 veces con agua destilada y se seca con gas nitrógeno para eliminar el líquido de la superficie del electrodo. Asegúrese de que el líquido se elimina correctamente del electrodo.
- Descarga exosome Cargo
- Cargar 5 l de 1 M de una sonda detector en 495 l de la mezcla de complejo de grano exosome y utilizar una pipeta para mezclar.
NOTA: La sonda detector es una secuencia de cebador de ADN conjugado con una molécula de fluoresceína en el extremo 3 '. El prob detectorsecuencia e utilizado para este estudio corresponde al ARNm GAPDH se encuentra dentro de los exosomas. La secuencia de la sonda detector conjugado con fluoresceína es: 5'-GCAGTGGGGACACGGAAGGCC-fluoresceína-3 '. - Pipeta 60 l de la sonda y compleja mezcla grano-exosome sobre una superficie de electrodos de oro con una selección de imanes debajo. Esta matriz imán consta de dieciséis 2,54 mm imanes de neodimio diámetro alineadas para corresponder a los electrodos de trabajo del sensor. La Figura 2A ilustra la colocación de los imanes y solución de grano exosome.
- Una vez que la muestra se carga en la superficie del electrodo, se aplican 20 ciclos del campo eléctrico CSW con 9 segundos a -300 mV y 1 seg a 200 mV (200 sec total). La carga exosomal que se libera se hibridará con los cebadores en la superficie del electrodo. Si los marcadores de superficie de la exosome son objeto de investigación, omitir esta parte del experimento. La Figura 2B ilustra este proceso.
- Wash-off los analitos no unidos en la superficie del electrodo con un triple enjuague la superficie del electrodo con agua destilada. Secar el electrodo con gas nitrógeno.
- Cargar 5 l de 1 M de una sonda detector en 495 l de la mezcla de complejo de grano exosome y utilizar una pipeta para mezclar.
- Reportero de anticuerpos y de lectura
- Añadir 60 l de 150 unidades / anticuerpo anti-fluoresceína ml conjugado con peroxidasa de rábano (HRP en 1: 1000 dilución) diluidas en caseína / PBS.
- Utilice una conjugación impulsado campo eléctrico a lo complejo HRP anti-fluoresceína para el bocadillo de la sonda. Aplicar -200 mV durante 1 segundo y 500 mV durante 1 segundo durante 5 ciclos a la superficie del electrodo. La Figura 2A muestra los complejos de sonda de captura y detector para tanto una proteína y sistema de ácido nucleico.
- Triple superficie del sensor de lavado usando agua destilada y se seca con gas nitrógeno.
- Después de la eliminación por lavado el exceso de anticuerpo no unido de anti-fluoresceína, añadir 60 l de 3,3 ', 5,5'-tetrametilbenzidina (TMB). Cargar este sustrato a cada superficie del sensor usando una pipeta multicanal.
- Realizar la lectura amperométrica de la corriente mediante la medición de la corriente del electrodo en -200 mV durante 60 segundos usando un potenciostato electroquímico capaz de la medición simultánea de 16 canales. Figura 2C es un ejemplo de perfil actual durante la lectura.
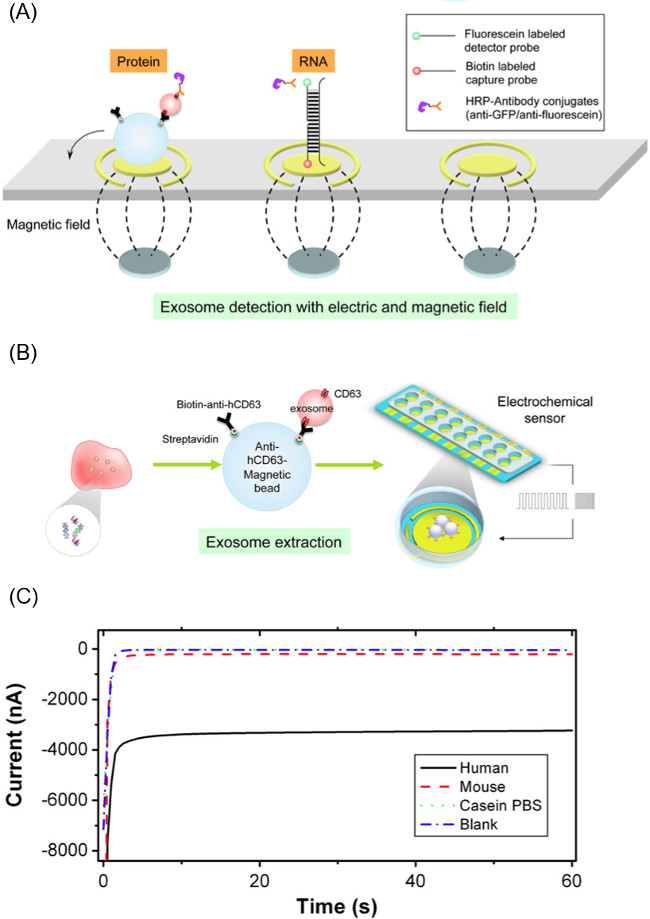
Figura 2. Componentes del Método Efirm. (A) método de extracción de exosomas de biofluido usando micropartículas magnéticas CD63 anti-humano recubierto y luego la descarga de la carga exosome usando ondas cuadradas cíclicos aplicados al complejo de partícula exosome. (B) Esquema de biosensor electrodo utilizado para la detección de objetivos de ARN / ADN / proteína de la exosome liberado. (C) Ejemplo representativo de lectura amperométrica de la metodología Efirm, donde la magnitud de corriente más grande corresponde to niveles más altos de una biomolécula. Esta cifra es de Wei et al. 14 Por favor haga clic aquí para ver una versión más grande de esta figura.
Resultados
Validación de exosome captura de perlas usando TEM
El aislamiento de los exosomas de saliva usando perlas magnéticas CD63 anti-humano fue validado siguiendo el protocolo de extracción mediante el uso de microscopía electrónica de transmisión (TEM). TEM muestra perlas magnéticas con 70-100 nm gránulos inmediatamente adyacentes (ver Figura 3A, y 3B), en consonancia con el perfil conocido de exosomas. No se observaron gránulos 70-100 n...
Discusión
Como indican los resultados, las nanopartículas magnéticas CD63 anti-humano recubierto son capaces de capturar específicamente pequeñas partículas que tienen un tamaño que varía desde 70 hasta 100 nm. Esta partícula capturado es consistente con el perfil observado previamente de exosomas. Además, se muestra el uso de la CSW de baja tensión después de la captura de las partículas para eliminarlos de la superficie de la perla y causar perfiles de degradación de ADN similares a la de un método tradicional bas...
Divulgaciones
David Wong es co-fundador de RNAmeTRIX Inc., una compañía de diagnóstico molecular. PeriRx LLC sublicenciada propiedad intelectual relacionados con el diagnóstico molecular de RNAmeTRIX. David Wong es un consultor para PeriRx.
Agradecimientos
Este trabajo fue apoyado por el Centro Nacional de Recursos para la Investigación y el Centro Nacional para el Avance de las Ciencias de traslación, los Institutos Nacionales de Salud, a través de subvención UL1TR000124 (FW); Felix y Mildred Yip Endowed Cátedra y el Fondo de la Familia Barnes (a DTWW), el Instituto Nacional de Odontología y de Investigación Craneofacial de los Institutos Nacionales de Salud, bajo el premio número T90DE022734 (MT). El contenido es responsabilidad exclusiva de los autores y no representa necesariamente las opiniones oficiales de los Institutos Nacionales de Salud.
Materiales
| Name | Company | Catalog Number | Comments |
| Helios 16-Channel Reader System with Chip Interface | Genefluidics, USA | RS-1000-16 | |
| 16x Sensor Chip, Bare Gold, pack of 5 chips | Genefluidics, USA | SC1000-16X-B | |
| Biotinylated anti-human CD63 Antibody | Ancell, USA | 215-030 | |
| Dynabeads MyOne Streptavidin T1 | Invitrogen, USA | 65601 | |
| Neodynium Magnetics (1/10" dia. x 1/32" thick) | K&J Magnetics, USA | DH101 | |
| Ultrapure Distilled Water | Life Technologies, USA | 10977-023 | |
| Mettler Toldeo 3 M KCl Solution | Fisher Scientific, USA | 1911512 | |
| Pyrrole | Sigma-Aldrich, USA | W338605-100g | |
| Anti-Fluorescein-POD, Fab fragments | Roche, Germany | 11426346910 | |
| 3,3′,5,5′-tetramethylbenzidine substrate (TMB/H2O2, low activity) | Neogen, Usa | 330175 | |
| Phosphate Buffered Saline Solution | Life Technologies, USA | 10010023 | |
| Casein/PBS | Fisher Scientific, USA | 37532 |
Referencias
- Rabinowits, G., Gerçel-Taylor, C., Day, J. M., Taylor, D. D., Kloecker, G. H. Exosomal MicroRNA: A Diagnostic Marker for Lung Cancer. Clinical Lung Cancer. 10 (1), 42-46 (2009).
- Thakur, B. K., et al. Double-stranded DNA in exosomes: a novel biomarker in cancer detection. Cell Research. 24 (6), 766-769 (2014).
- Lau, C. S., Wong, D. T. W. Breast Cancer Exosome-like Microvesicles and Salivary Gland Cells Interplay Alters Salivary Gland Cell-Derived Exosome-like Microvesicles In. Vitro. PLoS ONE. 7 (3), e33037 (2012).
- Bala, S., et al. Circulating microRNAs in exosomes indicate hepatocyte injury and inflammation in alcoholic, drug-induced, and inflammatory liver diseases. Hepatolog. 56 (5), 1946-1957 (2012).
- Dear, J. W., Street, J. M., Bailey, M. A. Urinary exosomes: A reservoir for biomarker discovery and potential mediators of intrarenal signalling. Proteomics. 13 (10-11), 1572-1580 (2013).
- Lässer, C., et al. Human saliva, plasma and breast milk exosomes contain RNA: uptake by macrophages. Journal of Translational Medicine. 9 (1), 9 (2011).
- Palanisamy, V., et al. Nanostructural and Transcriptomic Analyses of Human Saliva Derived Exosomes. PLoS ONE. 5 (1), e8577 (2010).
- Camussi, G., et al. Exosomes/microvesicles as a mechanism of cell-to-cell communication. Kidney International. 78 (9), 838-848 (2010).
- Lau, C., et al. Role of pancreatic cancer-derived exosomes in salivary biomarker development. The Journal of Biological Chemistry. 288 (37), 26888-26897 (2013).
- Théry, C., Amigorena, S., Raposo, G., Clayton, A. Isolation and Characterization of Exosomes from Cell Culture Supernatants and Biological Fluids. Current Protocols in Cell Biology. 3 (22), (2001).
- Park, N. J., Li, Y., Yu, T., Brinkman, B. M. N., Wong, D. T. Characterization of RNA in Saliva. Clinical Chemistry. 52 (6), 988-994 (2006).
- Wei, F., et al. Bio/Abiotic Interface Constructed from Nanoscale DNA Dendrimer and Conducting Polymer for Ultrasensitive Biomolecular Diagnosis. Small. 5 (15), 1784-1790 (2009).
- Wei, F., et al. Electrochemical Sensor for Multiplex Biomarkers Detection. Clinical Cancer Research. 15 (13), 4446-4452 (2009).
- Wei, F., Yang, J., Wong, D. T. W. Detection of exosomal biomarker by electric field-induced release and measurement (EFIRM). Biosensors and Bioelectronics. 44, 115-121 (2013).
Erratum
Formal Correction: Errata: Detection of Exosomal Biomarker by Electric Field-induced Release and Measurement (EFIRM)
Posted by JoVE Editors on 1/01/1970. Citeable Link.
An erratum was issued for Detection of Exosomal Biomarker by Electric Field-induced Release and Measurement (EFIRM). The disclosures were updated.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados