Method Article
PCR en tiempo real basada en sondeos Enfoques para la medición cuantitativa de microRNAs
En este artículo
Resumen
MicroRNAs circulantes han surgido recientemente como biomarcadores prometedores y novedosos para varios tipos de cáncer y otras enfermedades. El objetivo de este artículo es discutir tres plataformas basados en sondas diferentes en tiempo real PCR y métodos que están disponibles para cuantificar y determinar la abundancia de microRNAs en circulación.
Resumen
Basado en la sonda de PCR cuantitativa (qPCR) es un método preferido para medir la abundancia de transcripción, ya que es uno de los métodos de detección más sensibles que proporciona un análisis preciso y reproducible. Química basada-Probe ofrece la fluorescencia de fondo por lo menos en comparación con otras químicas (base de colorante). Actualmente, hay varias plataformas disponibles que la química basada en sonda de uso para cuantificar la abundancia de transcripción. qPCR en un 96 y placa es el método más utilizado habitualmente, sin embargo, sólo un máximo de 96 muestras o miRNAs se puede probar en una sola carrera. Esto consume tiempo y tedioso si un gran número de muestras / miRNAs se van a analizar. Las plataformas basadas en la sonda de alto rendimiento como microfluídica (por ejemplo, tarjetas de TaqMan Array) y matrices nanofluidos (por ejemplo OpenArray) ofrecen la facilidad para detectar de manera reproducible y eficiente la abundancia de múltiples microRNAs en un gran número de muestras en poco tiempo. Aquí, nos demuestran la configuración experimental unnd protocolo para miARN cuantificación de muestras de suero o plasma EDTA, utilizando la química basada en la sonda y tres plataformas diferentes (placa de 96 pocillos, microfluídica y matrices nanofluidos) ofreciendo mayores niveles de rendimiento.
Introducción
Los microARN (miRNA) son ~ 22 nucleótidos no codificante (nc) RNAs, funcionando como reguladores de la expresión génica 3.1. La mayoría de los miRNAs en animales funcionan a través de la secuencia específica de emparejamiento de bases con un ARNm, apuntando a la 3 'UTR, que conduce a la regulación negativa de la expresión del gen 2-4. Esto ocurre generalmente a través de la inhibición de la traducción de ARNm o ribosomal de entrega. miRNAs en circulación han sido demostrado ser nuevos biomarcadores en la investigación y campos clínicos para una variedad de enfermedades, como la diabetes 5-7, 8 de ovario, de próstata y cáncer de mama 9 10,11, hepatitis B 12 y otras enfermedades autoinmunes 13. Se han realizado investigaciones para identificar abundantes miRNAs en diferentes células o tejidos, así como en la circulación del plasma humano y las muestras de suero, que es más accesible y menos invasivo 9,11-15.
Diferentes métodos de cuantificación miRNA han sido eEstablecido el uso de múltiples plataformas, como la plataforma estándar de placas de 96 pocillos 4,12,16-18, la plataforma de tarjetas de microfluidos 12,18-23 y la plataforma nanofluidos matriz 17,24. Cuantitativo en tiempo real PCR (qPCR) ofrece la posibilidad de medir los números relativos o absolutos de las transcripciones utilizando químicas múltiples (basados en sondas de tinte o). En tiempo real la química basada en PCR-Probe ofrece el beneficio de baja fluorescencia de fondo y alta sensibilidad para detectar una sola copia transcripción. Es relativamente es rentable, fácil de usar y altamente reproducible, por lo que es un método preferido para la cuantificación y determinación de la expresión de miRNA 25. Método qPCR basada en sondeos generalmente implica dos pasos: la transcripción (RT) y qPCR 4,26,27 revertir. RT es donde el cebador de tallo-bucle RT se hibrida a una molécula de miARN maduro o primaria y se convierte en (c) ADN complementario. Cuantificación del producto de ADNc se lleva a cabo entonces usando cebadores de PCR específicos de los genes miARN-26-28. El principio de qPCR basado sonda se basa en la detección de extensión complementaria hebra en tiempo real, lo que implica la hidrólisis de la sonda-marcado con fluorescencia. Estas sondas están diseñadas para contener un reportero fluorescente y un inhibidor de la fluorescencia que son sólo de separación para permitir FRET (Transferencia de Energía de Resonancia Fluorescente). La detección de la emisión de la fluorescencia reportero (emisor) está enmascarado por la estrecha proximidad de la molécula de extintor. Cuando Taq polimerasa (pol) se extiende desde el cebador aguas arriba y llega a un tenedor (el extremo 5 'de la sonda), la actividad exonucleasa pol Taq hidroliza la sonda, que conduce a una disociación física / separación del emisor fluorescente del extintor. Esta versión de una sola molécula de emisor de fluorescencia es registrado por el detector y se presenta como un aumento incremental en la señal de fluorescencia de ese pozo / reacción. El aumento de la fluorescencia es proporcional a la cantidad de producto de PCR generado, lo que permite una precisa quantification de la 26,28 diana amplificado.
Con el aumento de la demanda en miARN cuantificación, a medio y tecnologías de alto rendimiento se han desarrollado para permitir que un mayor número de muestras a procesar en un corto período de tiempo. TaqMan Low Density Arrays (TLDA) es un medio de rendimiento de diseño microfluídico innovador basado en la química qPCR basada en sonda que ofrece un aumento en el número de miRNAs analizados en un plato. TLDA implica el uso de una piscina predefinido de RT-cebadores que se utilizan para sintetizar el ADNc. Estos ADNc se centrifugaron en una tarjeta bien personalizado 384 micro-fluidos para determinar la expresión de múltiples miRNAs utilizando qPCR 22,26,29. Cada pocillo de la tarjeta contiene cebadores y sondas se seca para amplificar miRNA específico (s), por lo tanto, hasta 384 reacciones pueden ser procesados en una sola tarjeta TLDA 26.
La matriz nanofluidos es una plataforma de alto rendimiento que se utiliza para la detección de transcritos de genes 24 usando la misma química basada en sondeos. Se utiliza una matriz patentada que ofrece interacciones hidrofóbicas hidrófilo para facilitar la carga fácil de la mezcla de reacción 33 nanolitros en una matriz de 3.072 orificios pasantes en una diapositiva de acero inoxidable 24. Este artículo se centra en la demostración de cómo se llevan a cabo estos métodos para cuantificar miRNAs en suero / plasma y los factores críticos que deben ser considerados al realizar e interpretar estos datos. Llevado a cuenta, sus beneficios individuales y limitaciones serán discutidos en este artículo.
Protocolo
El ARN total se puede aislar a partir de suero usando un protocolo establecido en nuestro laboratorio 30 o el uso de otros kits disponibles comercialmente.
NOTA: hojas de cálculo interactivas complementarias para el cálculo de los volúmenes de reacción en cada experimento (con el exceso de volumen 5% representaron pipeteo incluidos) se proporcionan.
1. basada en sondeos en tiempo real qPCR Usando un 96 pocillos Plataforma Placa Standard
- síntesis de ADNc (transcripción reversa) en muestras de ARN de suero / plasma de miRNA
- Calcular y sacar 10 ng de ARN para cada reacción de síntesis de ADNc. Añadir agua libre de nucleasa para llevar el volumen final de 10 ng de ARN de 1,67 l (para una reacción de 5 l). Mantener las muestras en hielo.
- Seleccione el miARN y descongelar sus primers RT.
- Descongele los componentes de la mezcla de reactivos RT: RT Buffer (10X), dNTPs (100 mM), RNasa Inhibidor (20 U / l). Nunca mantenga lugar enzima virus de la leucemia (recombinante murina de Moloney(RMoMuLV)) (virus de leucemia murina de Moloney recombinanat (rMoMuLV) de la transcriptasa inversa (50 U / l) en hielo. Almacénelo a -20 ° C hasta que se necesite o en un bloque congelador.
- Prepare la mezcla de reactivos RT en hielo como se describe en la Tabla 1A. Se recomienda preparar al menos 5% de exceso de volumen para compensar el error de pipeteado.
- Añadir 2,33 l de la mezcla de reactivos RT cada uno en un 0,2 ml tubo de PCR / placa.
- Vortex los tubos de cebadores de RT para mezclar, a continuación brevemente centrifugar a 10.000 xg durante 10 seg. Añadir 1 l de la imprimación RT específicos miARN a sus respectivos tubos de PCR o placa. Añadir 1,67 l de la ARN diluido a sus respectivos tubos de PCR o pocillos de la placa. Realice todas las adiciones en el hielo.
- Centrifugar el tubo de reacción o de placas a 1.950 g durante 5 min a 4 ° C.
- Configure el programa de síntesis de cDNA miRNA en el termociclador con las siguientes condiciones de ajuste: 16 ° C durante 30 min, 42 ° C durante 30 min, 85 °C durante 5 min y 4 ° C en espera.
- Ajuste de volumen de reacción a 10 l. Cargue los tubos de reacción o placa en el termociclador. Iniciar la carrera RT. Almacenar las reacciones de ADNc (RT) a -20 ° C, si la amplificación por PCR en tiempo real no se procedió inmediatamente.
- Sonda basados en tiempo real qPCR para la detección de miRNAs maduros
- Descongelar los ensayos seleccionados basados en sondas qPCR (20x) para el producto de RT respectivo.
- Mezclar la única solución conveniencia de PCR en tiempo real (mezcla de reactivos qPCR) por turbulencia de la botella.
- Preparar la mezcla de reactivos qPCR para cada miARN para ser analizados. Para preparar la qPCR obtener un tubo de microcentrífuga de 1,5 ml estéril para cada muestra de miRNA y agregar los componentes en cada tubo como se describe en la Tabla 1B. Se recomienda preparar al menos 5% de exceso de volumen para compensar el error de pipeteado.
- Añadir 4,2 l de la respectiva mezcla de reactivos qPCR a cada pocillo de una placa de 96 pocillos óptica (o tubos). Añadir 0,8 lde la reacción de ADNc respectivo (sintetizado en la etapa 1.1 para cada miRNAs) para así respectiva.
- Sellar la placa con la cubierta óptica apropiada. Centrifugar la placa (o tubos) a 1950 xg durante 5 min a 4 ° C
- Encienda el ordenador, 96 pocillos lector de placa array / microfluídica y finalmente iniciar el software. Asegúrese de que las máquinas están conectadas correctamente y correcta bloque y tapa térmica (para placa de rápido 96) están en su lugar.
- Seleccionar experimento como de 96 pocillos bloque rápida (0,1 ml), curva estándar, reactivos TaqMan y el modo rápido. Configure qPCR en el sistema de PCR en tiempo real utilizando las siguientes condiciones de ciclo del programa: 95 ° C durante 20 segundos, 50 ciclos (95 ° C durante 1 seg, 60 ° C durante 30 segundos).
- Ajuste de volumen de reacción a 10 l. Cargar el tubo de reacción o de la placa en el instrumento. Pulse el botón "Iniciar plazo". El programa se llevará a aproximadamente 1 hora en completarse.
2. Tarjeta de matriz microfluídica basada en sondeos (Card A y B)
NOTA: basada en sondeos panel de miARN se presenta como un conjunto de dos tarjetas de 384 pozos de microfluidos (tarjeta matriz A y matriz de la tarjeta B). Cada tarjeta contiene cebadores secos y sondas para hasta 380 miRNAs y controles. producto de ADNc (con o sin pre-amplificación) específica a la tarjeta A o B tarjeta se carga en la matriz respectiva para PCR en tiempo real.
- Transcripción inversa (RT)
- Utilice este protocolo para una entrada de ARN total de 1-1.000 ng. Si la entrada es de entre 1 a 350 ng, lleve a cabo una etapa de pre-amplificación (pre-amplificador). Para entradas superiores a 350 ng, cargar ADNc directamente en las tarjetas de matriz sin pre-amplificador.
NOTA: Para los perfiles de miARN suero, comience con 100 ng de ARN total. Para un perfil completo miRNA, ejecutar dos piscinas predefinidos de conjuntos de cebadores de RT-RT reacciones (Grupo A y Grupo B) por muestra. - Descongele los siguientes reactivos en hielo. RT Primers (10x): Grupo A y Grupo B, dNTPs con dTTP (100 mM), RT Buffer (10x), MgCl2 (25 mM), inhibidor de RNasa (20 U / Y# 181; l). No guarde la enzima (virus de la leucemia murina de Moloney recombinante (rMoMuLV)) de la transcriptasa inversa (50 U / l) en el hielo. Guárdelo a -20 ° C hasta que se necesite.
- Vórtice suavemente todos los reactivos, excepto la enzima, y centrifugar brevemente los tubos a 10.000 xg durante 10 seg.
- Combinar reactivos, como se describe en la Tabla 2A, en dos tubos; un tubo para el Grupo A, el otro para el Grupo B. Cada tubo contendrá sólo una piscina predefinido de conjunto de cebadores RT, ya sea desde el Grupo A o del Grupo B. Se recomienda preparar al menos 5% de exceso de volumen para compensar el error de pipeteado .
- Invertir para mezclar, y brevemente después centrifugar a 10.000 xg durante 10 seg. Alícuota de 100 ng de cada muestra de ARN en un nuevo tubo, a continuación, añadir un volumen apropiado de agua libre de nucleasa para hacer un total de 3 l.
- Alícuota de 4,5 l de la mezcla de reactivos RT apropiada en los respectivos tubos. Invertir para mezclar, y luego centrifugar brevemente a 10.000 xg durante 10 seg. Incubar en hielodurante 5 min.
- Colocar las muestras en un termociclador e iniciar la RT usando las siguientes condiciones: 40 ciclos (16 ° C durante 2 min, 42 ° C durante 1 min, 50 ° C durante 1 segundo), 85 ° C durante 5 min, mantienen a 4 ° C. CDNA tienda generado mediante el uso de estos piscina predefinido de RT-cebadores de -15 a -25 ° C o utilizarse inmediatamente.
- Utilice este protocolo para una entrada de ARN total de 1-1.000 ng. Si la entrada es de entre 1 a 350 ng, lleve a cabo una etapa de pre-amplificación (pre-amplificador). Para entradas superiores a 350 ng, cargar ADNc directamente en las tarjetas de matriz sin pre-amplificador.
- Pre-amplificación
- Descongele la piscina predefinido de cebadores pre-amp en hielo. Suavemente cebadores vórtice, y centrifugar brevemente a 10.000 xg durante 10 seg. Mezcla de reactivos TaqMan PreAmp Remolino (2X) golpeando ligeramente, para mezclar.
- Combinar reactivos, como se describe en la Tabla 2B, en dos tubos; un tubo para el Grupo A, el otro para el Grupo B.
NOTA: Como se describió anteriormente, piscina cebadores A pre-amplificador irán a tubo de cebadores A y Grupo B deben ir al tubo B. Se recomienda preparar al menos 5% de exceso de volumen para compensar el error de pipeteado. - Invertir para mezclar, y luego centrifugar brevemente a 10.000 xg durante 10 seg.Alícuota 22,5 l de la mezcla de reactivos PreAmp apropiada en nuevos tubos. Alícuota de 2,5 l de la muestra de ADNc (preparado en la etapa RT) en el tubo respectivo.
- Invertir para mezclar, y luego centrifugar brevemente a 10.000 xg durante 10 seg. Incubar en hielo durante 5 minutos. Colocar las muestras en un termociclador e iniciar el ciclo previo. Ciclismo condiciones: 95 ° C durante 10 min, 55 ° C durante 2 min, 72 ° C durante 2 min, 12 ciclos (95 ° C durante 15 segundos, 60 ° C durante 4 min), 99,9 ° C durante 10 minutos, mantenga a 4 ° C.
- Invertir pre-amplificado de ADNc para mezclar, y después centrifugar brevemente a 10.000 xg durante 10 seg. Añadir 75 l de tampón TE 0,1x (pH 8,0) para pre-amplificado de ADNc (dilución 1: 4). Invierta muestras pre-amp diluidas para mezclar y centrifugar brevemente a 10.000 xg durante 10 seg. Tienda diluye pre-amplificado ADNc de -15 a -25 ° C durante un máximo de una semana, o utilizarse inmediatamente.
- Cargando tarjetas de microfluidos y la realización de qPCR
- Mantenga la microfluídicamiARN tarjetas fuera durante al menos media hora para llegar a la temperatura ambiente. Descongelar diluido cDNA pre-amplificada en hielo y mezclar invirtiendo los tubos seguido por breve centrifugado. Combina la mezcla de reactivos qPCR agitando la botella.
- En el nuevo tubo de añadir 450 l de mezcla de reactivos qPCR a 9 l de cDNA diluido pre-amplificado. Para la tarjeta A, use pre-amplificado producto preparado usando el cebador Grupo A. Añadir 441 l de agua libre de nucleasa para compensar el volumen final a 900 l. Invertir el tubo para mezclar, y luego centrifugar brevemente a 10.000 xg durante 10 seg.
- Retire la tarjeta TLDA de su envoltorio (una vez que alcance la temperatura ambiente) y colocarlo en un lugar limpio y con la cara de la hoja hacia abajo. Añadir 100 l de mezcla de reacción PCR en cada uno de los 8 puertos de llenado en la tarjeta. Hay 2 puertos en cada uno de los depósitos de (8 embalses en total en cada tarjeta).
NOTA: El puerto de llenado es el agujero más grande, donde se añade la mezcla de reacción, mientras que el orificio de ventilación es el agujero más pequeño. Cada tarjeta miARN TLDA tiene 8 depósitos, cada uno llevando a 48 pocillos (24 pocillos en una columna x 2 columnas), cargando de este modo todos los 384 pozos en la tarjeta con la misma mezcla de reacción PCR. Asegúrese de que el Grupo A y el Grupo B de cada muestra se cargan sobre las respectivas tarjetas TLDA. Coloque la tarjeta matriz de la matriz de microfluidos cubos titular de la tarjeta especializados en la centrífuga. - Para las tarjetas de matriz de microfluidos, usar una centrífuga adecuado (tal como Heraeus Multifuge 3SR, centrifugar 230V) y los compartimientos específicos (como los titulares "Tarjeta TaqMan Array"). Cada cubo puede contener hasta 3 tarjetas (cargado / vacío). Asegúrese siempre de que los 3 slots de un cubo son ocupados y el cubo se equilibra mediante la colocación de cubo similar (este cubo debe contener también 3 cartas, vacíos o llenos) en la ranura opuesto de la centrífuga. Al colocar la tarjeta en el soporte de cubeta, asegúrese de que los 8 embalses proyectan hacia arriba y pocillos de reacción frente a la pared exterior de la centrífuga.
- Haga girar las tarjetas en 331 xg durante 1 minuto a temperatura ambiente. Despuésprimera vuelta, abrir la centrifugadora y asegurar visualmente la mezcla de reacción se ha dispensado a través de los 384 pozos. Repita el lanzamiento en la misma configuración para 1 hora más. Retire la tarjeta de la cubeta y asegúrese de que el nivel de mezcla de reacción en cada uno de los 8 embalses es uniforme. Cualquier inconsistencia en los volúmenes de líquido que quedan en los embalses hacen la tarjeta inapropiado utilizar más.
- Tarjetas de matriz microfluídica necesitan un sellador especializado que tiene un conjunto de aguja de precisión (carro) para sellar los canales de distribución de fluidos de la matriz y distribuir por igual la mezcla de reacción en todos los pocillos (volumen total de reacción de 1 l / pocillo).
- Llevar el carro a la posición de inicio e introduzca la tarjeta cargada en el sellador con la cara de papel de aluminio y se alinearon a los pines del lápiz sobre el sellador. En un golpe lento y uniforme, constante y única, empujar el carro a través de la tarjeta hasta que se alcanza el punto final de la sellador. Retire la tarjeta de gama selladas y luego cortar el depósitos de la tarjeta utilizando tijeras.
- Asegúrese de que el soporte de bloque correcto, tapa calentada y la muestra se instala en la matriz de la microfluídica en tiempo real sistema de PCR / máquina.
- Encienda el ordenador, entonces el sistema de arreglo de microfluidos y finalmente iniciar el software. Asegúrese de que las máquinas están conectadas correctamente. Seleccione experimento como tarjeta matriz, curva estándar, reactivos TaqMan y modo estándar. Importe los archivos de instalación para cualquiera de la tarjeta A y tarjeta B. Guarde el archivo.
- Coloque la tarjeta sellada en la bandeja del instrumento con el pocillo A1 en la esquina superior izquierda y el código de barras hacia la parte delantera del instrumento. Pulse el botón "Ejecutar". El programa se llevará a aproximadamente 2 horas en completarse.
Panel nanofluidos miARN humana basada en sondeos 3.
- Transcripción inversa (RT)
- Utilice este protocolo para una entrada de ARN total de 50 a 200 ng, sin embargo 100 ng es óptimo para la mayoría de las muestras. Para un perfil completo miRNA, ejecute dos predefipiscinas nidos de conjuntos de cebadores de RT-RT reacciones (Grupo A y Grupo B) por muestra.
- Descongelar los reactivos sobre hielo: piscina predefinido de conjuntos de cebadores de RT (10X), dNTPs con dTTP (100 mM), RT Buffer (10x), MgCl 2, (25 mM), inhibidor de ARNasa (20 U / l). No mantenga la enzima (virus de la leucemia murina de Moloney recombinante (rMoMuLV) de la transcriptasa inversa (50 U / l) en hielo. Almacénelo a -20 ° C hasta que se necesite. Vórtice suavemente todos los reactivos, excepto la enzima, y centrifugar brevemente ellos en 10000 xg durante 10 seg.
- Combinar reactivos, como se describe en la Tabla 3A, en dos tubos; un tubo para el Grupo A, el otro para el Grupo B. Cada tubo contendrá sólo una piscina predefinido de conjunto RT-cartilla, ya sea desde el Grupo A o del Grupo B. Se recomienda preparar al menos 5% de exceso de volumen para compensar el pipeteo error.
- Pipeta para mezclar y brevemente después centrifugar a 10.000 xg durante 10 seg. Alícuota de 100 ng de cada muestra de ARN en un nuevo tubo, a continuación, añadir un volumen apropiadode agua libre de nucleasa para hacer un total de 3 l. Alícuota de 4,5 l de la mezcla de reactivos RT apropiada en el tubo respectivo.
- Invertir para mezclar, y luego centrifugar brevemente a 10.000 xg durante 10 seg. Incubar en hielo durante 5 min. Colocar las muestras en un termociclador e iniciar el programa de RT. Ciclismo condiciones: 40 ciclos (16 ° C durante 2 min, 42 ° C durante 1 min, 50 ° C durante 1 segundo), 85 ° C durante 5 min, mantienen a 4 ° C. CDNA tienda generado utilizando estos piscina predefinido de RT-conjunto de cebadores de -15 a -25 ° C o usar inmediatamente.
- Pre-amplificación (pre-amp)
- Descongele la piscina predefinido de imprimación preamplificador establecido en el hielo. Suavemente cebadores vórtice, centrifugar brevemente a 10.000 xg durante 10 seg. Mezcla de reactivos TaqMan PreAmp Remolino (2X) para mezclar.
- Combinar reactivos, como se describe en la Tabla 3B, en dos tubos; un tubo para el Grupo A, el otro para el Grupo B. Cada tubo contendrá sólo una piscina predefinido de conjuntos RT-imprimación, ya sea from Grupo A o del Grupo B. Se recomienda preparar al menos 5% de exceso de volumen para compensar el error de pipeteo.
- Pipeta para mezclar, y luego centrifugar brevemente ellos a 10.000 xg durante 10 seg. Alícuota 22,5 l de la mezcla de reactivos PreAmp apropiada en nuevos tubos. Alícuota de 2,5 l de la muestra de ADNc en el tubo respectivo. Invertir para mezclar, y luego centrifugar brevemente los tubos a 10.000 xg durante 10 seg. Incubar en hielo durante 5 min.
- Colocar las muestras en un termociclador y ejecute el pre-amplificador. Volumen de reacción Ajuste a 25 l. Ciclismo condiciones: 95 ° C durante 10 min, 55 ° C durante 2 min, 72 ° C durante 2 min, 12 ciclos (95 ° C durante 15 segundos, 60 ° C durante 4 min), 99,9 ° C durante 10 minutos, mantenga a 4 ° C.
- Invertir pre-amplificado de ADNc para mezclar, y después centrifugar brevemente a 10.000 xg durante 10 seg. En los nuevos tubos de añadir 4 l de ADNc pre-amplificada a 156 l de 0,1x TE Buffer pH 8,0 (dilución 1:40). Invierta muestras diluidas de preamplificador para mezclar, y luego ce brevemententrifuge a 10.000 xg durante 10 seg. Tienda diluye y cDNA preamplificada sin diluir de -15 a -25 ° C durante un máximo de una semana, o el uso inmediatamente.
- Cargando nanofluidos Arrays y escénicas qPCR
- Descargue el archivo de la placa correspondiente (.tpf) desde el sitio web 31 utilizando el número de serie de diapositivas gama nanofluidos. Este contiene la información de ejecución para la diapositiva específica matriz nanofluidos.
- Descongele la mezcla de reactivos diluido previamente amplificado ADNc y TaqMan en tiempo real qPCR (si se utiliza por primera vez) en el hielo. Combina la mezcla de reactivos qPCR agitando la botella. En los nuevos tubos de añadir 22,5 l de mezcla de reactivos qPCR a 22,5 l de cDNA diluido pre-amplificado. Mezclar suavemente, y centrifugar brevemente a 10.000 xg durante 10 seg.
- Alícuota de 5 l de cada muestra en 8 pozos (2 columnas, 4 filas) de la placa de 384 pocillos de muestras de flujo de trabajo nanofluidos matriz. Asegúrese de que el Grupo A y el Grupo B de cada muestra se encuentran en bloques de 8 pocillos adyacentes (ver Figure 1 o complementario sobresalen la hoja de diseño).
- Cada placa de la muestra puede contener hasta ocho diapositivas por valor de muestras. Sin embargo, el sistema de nanofluidos array sólo puede procesar 4 arrays nanofluidos en una sola carrera. Si hay más de cuatro diapositivas por valor de las muestras se van a cargar en una placa de muestra, asegúrese de que los tramos restantes están sellados. Selle con OpenArray placa de muestras sellador.
NOTA: Es recomendable precortado, el sellador en las secciones necesarias, por lo que las secciones se puede sellar / sin sellar de forma individual para reducir la evaporación. Alternativamente, la placa se puede sellar con un sellador intacto, y luego se puede cortar secciones individualmente a cabo cuando se carga.
- Cada placa de la muestra puede contener hasta ocho diapositivas por valor de muestras. Sin embargo, el sistema de nanofluidos array sólo puede procesar 4 arrays nanofluidos en una sola carrera. Si hay más de cuatro diapositivas por valor de las muestras se van a cargar en una placa de muestra, asegúrese de que los tramos restantes están sellados. Selle con OpenArray placa de muestras sellador.
- Placa Centrifugar a 490 xg durante 1 min a 4 ° C. Cargue los nanofluidos diapositivas de matriz dentro de 1 hora. Debido al escaso tiempo permitido para sellar las diapositivas, por favor, sólo cargar una diapositiva a la vez. Retire la diapositiva nanofluidos gama del congelador y dejar que se adapte a la temperatura ambientebibliografía (~ 15 min).
- Asegúrese de que el soporte de bloque correcto, tapa térmica y la muestra está instalada en el sistema nanofluidos matriz. Encienda el ordenador y el sistema de PCR en tiempo real y el sistema de carga. Acceso al software respectivo y garantizar que las máquinas están conectadas. Retire los consumibles del sistema de carga (array tapa deslizante, enchufe y fluidos de inmersión) de su embalaje.
- Tire suavemente del émbolo de la jeringa de fluido de inmersión para aflojar. Retire la tapa, coloque la punta en el aire y al ras de la punta. Coloque la punta del sistema de carga dentro de la máquina y retire la tapa. Coloque la placa de la muestra dentro del sistema de PCR.
- Póngase los guantes. Asegúrese de que estén herméticamente cerradas para minimizar el riesgo de marcar accidentalmente la tapa deslizante. Envases de diapositivas cuidadosamente abierta. Poco a poco la punta de diapositivas en la mano. No toque la parte superior de la diapositiva.
- Coloque la diapositiva en el sistema de PCR, con el código de barras de la izquierda. Eliminar sellador de la porción de la placa de muestra destinada para la carga. Utilice el sistema de cargasoftware para introducir el código de barras de diapositivas, deslice posición, posición de la muestra y la configuración de la punta.
- Cuando se hayan completado todas las comprobaciones pertinentes, corredera de la prensa de la carga. Mientras que el sistema de PCR se está cargando la diapositiva, retire el plástico claro y rojo de la parte inferior de la tapa deslizante. Cuando haya terminado la carga, retire con cuidado y sellar la diapositiva dentro de 90 seg.
- Colocar el portaobjetos en la mordaza de plancha. Coloque la tapa deslizante sobre el portaobjetos. Pinza para 30 seg. Asegúrese de que la tapa se coloca de modo que el código de barras se muestra correctamente. Retire el conjunto de la abrazadera de la placa.
- Posición de la jeringa de fluido de inmersión dentro de la diapositiva de manera que la punta está presionando contra la tapa. Lentamente llenar de diapositivas con fluido de inmersión, garantizando las carreras de fluido a lo largo de la tapa. Una vez lleno, selle la diapositiva con el enchufe, girando el tornillo hasta que el mango se interrumpe.
- Retire la cubierta de plástico en la parte superior de la tapa deslizante, y luego colocar cuidadosamente en el soporte de diapositivas del sistema de PCR en tiempo real. Asegúrese de que haya Dopport en la parte inferior de la diapositiva como se hace descender, por lo que no cae de repente, y no toque la parte superior de la diapositiva. Está bien tocar los lados del sistema / cassette.Initialize la PCR de diapositivas e iniciar el programa para qPCR dentro de 1 hr.
- Seleccione "OpenArray" dentro del programa de PCR-sistema. Pulse el botón "Buscar ID de diapositivas". Esto tomará unos minutos. Si el software no puede encontrar el ID de placa, se le preguntará para que pueda ser introducida manualmente.
- Pulse el botón "Confirmar Centros Plate". Una vez más, esto llevará unos pocos minutos. Compruebe que el punto rojo se encuentra dentro del centro y que no haya huellas digitales / marcas en la parte superior de la diapositiva. Cargue el archivo .tpf respectivo para cada diapositiva y especificar un nombre de archivo de resultados y la ubicación. Pulse el botón "Ejecutar". El programa se llevará a aproximadamente 2 horas en completarse.
Resultados
El volumen recomendado para una reacción qPCR ensayo basado sonda-miRNA es de 20 l. NOTA: Hemos confirmado que un volumen de reacción de 5 l es capaz de producir resultados similares a los obtenidos utilizando 20 l de volumen 4,7,30. La reducción del volumen de reacción a 5 l permite una disminución del 75% en los costes de reactivos sin pérdida apreciable en la sensibilidad. Tal como se presenta en la Figura 2, los volúmenes de reacción de 20 l y 5 l muestran una fuerte correlación hasta 39 ciclos (con r 2 de 0,92, p = 0,0002).
Array microfluídica proporciona una herramienta para la obtención de datos sobre 754 miRNAs expresadas en una muestra en alrededor de 5 horas (para la tarjeta de A y B de tarjeta), que es una forma más eficiente de analizar múltiples muestras en comparación con convencionales 96 PCRs placa de pocillos. Comparamos miARN tarjetas de matriz de microfluidos para la misma muestra (muestra A y la muestra A repetición) Figura 3A -. B muestra un gráfico de Bland-Altman ( 3A) y la trama de correlación (3B) para todos los 754 miRNAs ensayadas para estas muestras. Hay 3 miRNAs control diferentes (U6, RNU44 y RNU48) colocados al azar en ambos las tarjetas (Card A y B) en varias ubicaciones. Cuando los valores de umbral ciclo U6 (MA) son comparados entre los 2 carreras, no observamos diferencias significativas entre los valores (Tabla 4). También es importante señalar aquí que U6 se expresa en mayor abundancia (menor valor Ct) en la muestra evaluada. Por último, comparamos todos los miRNAs que tienen valores de Ct entre 0-19,99 tanto en las carreras (n = 150), que había expresión similar de miRNAs general con un coeficiente de determinación de 98% (Figura 3C-D). De todos los 277 miRNAs que tienen valores de Ct de 20 a 29.99 en tanto las carreras, 16 miRs difirieron significativamente entre los originales y ensayos repetidos (Figura 3E - F) El número de miRNAs con diferencia significativa betwee.n las carreras aumentaron (89 de 327) cuando se seleccionan los valores de Ct entre 30-40 para ambas carreras (Figura 3G - H).
La plataforma nanofluidos matriz proporciona datos para 754 miRNAs de cada muestra de suero / plasma ensayadas, tal como se representa en la figura 4A Es importante examinar estas curvas de amplificación -. Como lo es con todo qPCR - para asegurar que el resultado es indicativo de la verdadera amplificación. Cada una de las 48 subarrays (Figura 1) contiene también un ensayo para las tres ncRNAs de "mantenimiento" más populares:. U6, RNU44 y RNU48 Figura 4B ilustra una agrupación típica de U6 replica de una sola muestra. Estas repeticiones muestran baja desviación estándar (SD <0,5) y así son un indicador de fiabilidad. Alternativamente, la Figura 4C demuestra el aumento de la variabilidad de repeticiones U6 (SD> 0,5) en una segunda muestra. Esto no niega la validez of los ensayos restantes, aunque sí requeriría una crítica más a fondo. U6, como con la mayoría de miRNAs "limpieza" en fluidos biológicos, puede tener una expresión variable. Cabe señalar que una de las muestras, se describen en la Figura 4C, muestra 4 veces menor contenido U6 que la presentada en la Figura 4B. Dado que el nivel de U6 en la muestra presentada en 4C es un 75% menos, para empezar que el presentado en el panel 4B, una mayor variabilidad técnica se espera debido a la distribución de Poisson de las transcripciones, que se ve agravada por el volumen de reacción pequeño 17.
Otra herramienta útil es que las imágenes de control de calidad (CC), disponible para la exportación una vez que la carrera ha terminado. Una selección de estos utiliza la fluorescencia de ROX, el colorante pasiva que se encuentra en la mezcla de reactivos qPCR, para confirmar que cada orificio pasante se ha cargado correctamente (Figura 5). A-agujero a través, o incluso un cuarto subarreglo neumático, no se puede cargar debido al volumen de muestra insuficiente, la evaporación, las burbujas presentes en los pocillos de la placa de muestra de 384 pocillos, la falta de eliminar completamente la junta de la placa de la muestra, o defectos en el sistema de Accufill o sus puntas. Cualquier descargado orificios pasantes deben ser identificados para evitar miRNAs etiquetado como "no detectable", cuando en realidad el ensayo no estaba cargada. Si se produce este problema, asegúrese de que al menos 5 l de muestra / mezcla maestra se carga en cada pocillo de la placa de la muestra de 384 pozos, la placa muestra se centrifuga correctamente antes de la carga, el sello de aluminio se elimina por completo, y la OpenArray cargado diapositiva se sella y se ejecuta dentro del tiempo asignado para todas las carreras sucesivas. Si todavía persisten problemas de carga, estos pueden ser más probable que pertenecen a lote específico o lote de las matrices o consumibles relacionados y más ayuda debe buscarse a través del fabricante.
"Src =" / files / ftp_upload / 52586 / 52586fig1.jpg "/>
Figura 1: Distribución de las muestras para el flujo de trabajo de matriz nanofluidos: (A) Cada placa de muestras de 384 pozos puede contener muestras para un máximo de 8 matrices nanofluidos. (B) Diluido, ADNc pre-amplificado se coloca en 8 pozos (2 columnas por 4 filas), con el Grupo A y Grupo B en grupos de 8 pocillos adyacentes. Cada círculo representa un pozo. (C) Cada pocillo de la placa de la muestra podrá ser cargada en un subconjunto de la matriz nanofluidos. Cada pequeño cuadrado representa un subconjunto.
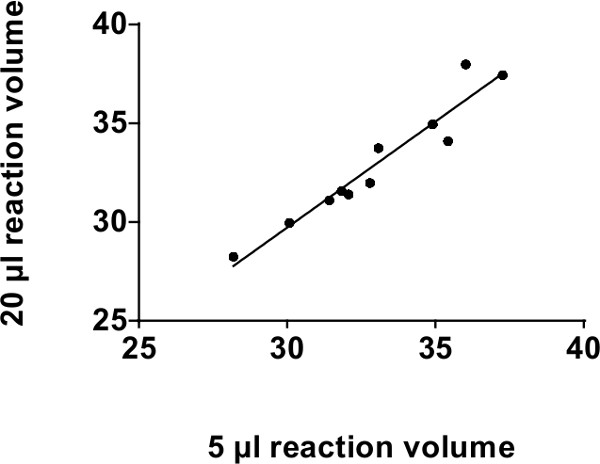
Figura 2: Análisis de Co-relación para convencionales de 96 pocillos plataformas de PCR: Co-relación entre 20 l y 5 volúmenes de reacción l en tiempo real TaqMan qPCR utilizando una plataforma estándar de placa de 96 pocillos en valores de CT (39 ciclos). Comparamos 4 microRNAs diferentes (miR-375, miR-30c, miR-30d y miR-7) en 4 muestras de suero y plasma humanos diferentes. Sólo 11 puntos de datos se representan ya que los demás fueron indetectables. R 2 = 0,92, p = 0,0002.
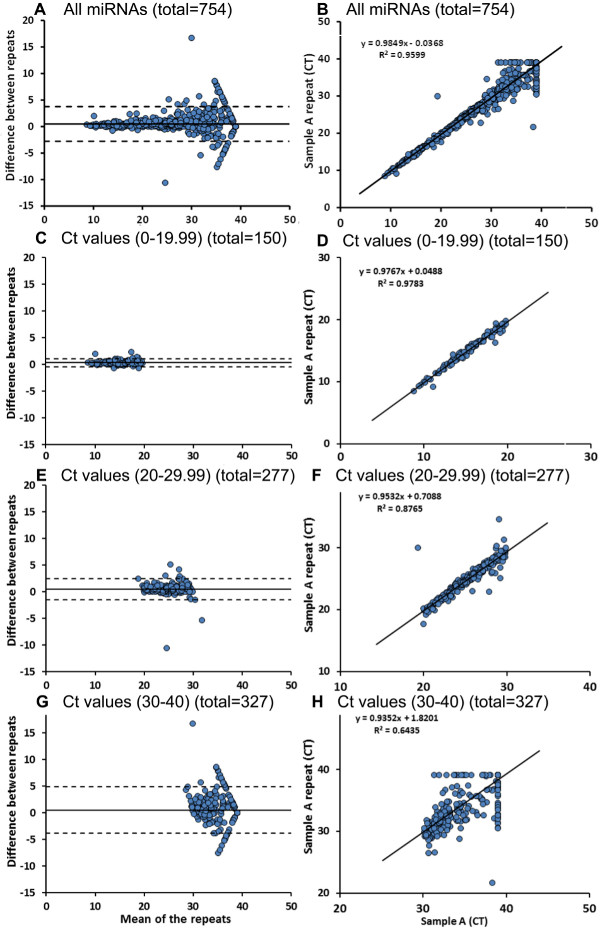
. Figura 3: Circulación miARN perfiles de uso de tarjetas de microfluidos matriz usando 2 tarjetas de microfluidos (tarjeta-A y tarjeta-B), se genera un perfil de 754 miRNAs (A - B). Como se muestra aquí, se utilizó una misma muestra de 2 matriz microfluídica corre para comprobar la reproducibilidad de los resultados de la tarjeta microfluídica. Se ha observado una expresión similar de miRNAs en general, con los valores de Ct entre 0-19,99 (C - D). Hay pocos miRNAs (16 de 277) con los valores de Ct entre 20 a 29,99 y significativas diferencias entre ensayos repetidos (E - F). Ochenta y nueve de 327microRNAs con los valores de Ct más altas (30-40) mostraron diferencias significativas entre ambas carreras (G - H). Los datos se analizaron mediante la prueba T pareada. Por favor, haga clic aquí para ver una versión más grande de esta figura.
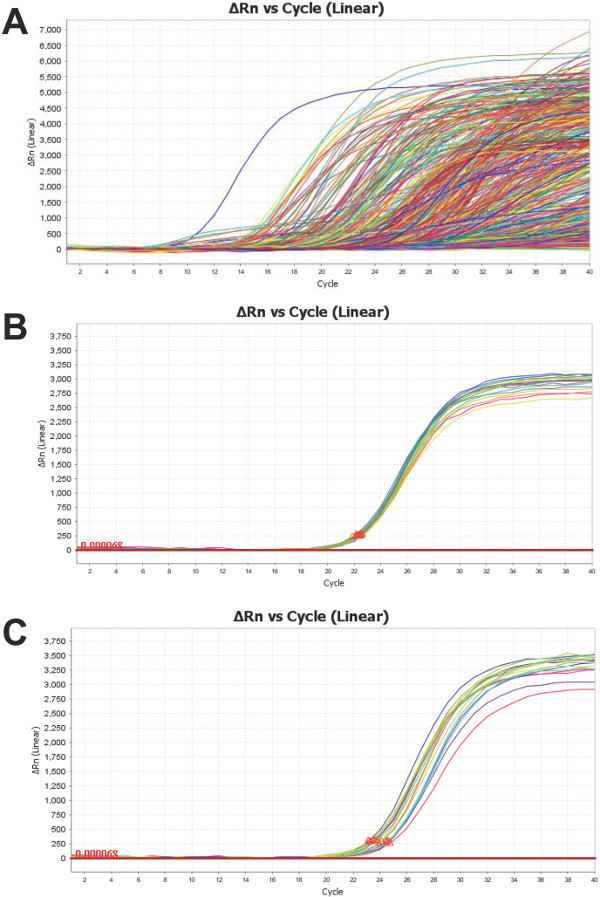
Figura 4: Representante del perfil de las curvas de amplificación por PCR del producto: Es una figura representativa de los combinados curvas (A) de amplificación de todos los genes miARN objetivos para una muestra de plasma humano. Un ensayo para U6 (un control común ncRNA) se coloca en cada subconjunto. La muestra en (B) demuestra una baja variabilidad (SD <0,5), mientras que (C) muestra alta desviación estándar (SD> 0,5) dentro de U6 se replica. Ambas muestras son total ARN aislado a partir de plasma humano. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Análisis del control de calidad de nanofluidos Arrays: control de calidad (QC) imágenes de un array nanofluidos cargado correctamente (izquierda) y una serie nanofluidos cargado incorrectamente (a la derecha). El tinte pasiva, ROX (presente en la mezcla de reactivos qPCR), fluorescente para indicar un agujero pasante cargado correctamente. La matriz de la derecha tiene varios subconjuntos / orificios pasantes que no estén cargados con la mezcla de reactivos qPCR y estos deben ser identificadas como reacciones falsos negativos de PCR.
| Componentes | Volumen por 5 l de reacción (l) |
| Tampón de RT (10x) | 0.5 |
| dNTPs (100 mM) | 0.05 |
| RNasa inhibidor | 0.6 |
| Agua libre de nucleasa | 1.39 |
| Transcriptasa Inversa | 0.33 |
| Volumen total | 2.33 |
Tabla 1A: RT componentes de la mezcla de reactivos en una preparación de reacción de RT 5 l de TaqMan en tiempo real qPCR utilizando una plataforma de placa de 96 pocillos estándar.
| Componentes | Volumen por 5 l de reacción (l) | Volumen por 20 l de reacción (l) |
| Mastermix PCR rápida (2x) | 2.5 | 10 |
| Ensayo TaqMan qPCR (20x) * | 0.25 | 1 |
| Libre de nucleasaagua | 1.45 | 5.8 |
| Volumen Total: | 4.2 | 16.8 |
* Componente de ensayo basado en el miARN seleccionado probado.
Tabla 1B: qPCR componentes de la mezcla de reactivos en una preparación de reacción qPCR 5 o 20 l de TaqMan en tiempo real qPCR utilizando una plataforma de placa de 96 pocillos estándar.
| Componentes | Volumen por reacción (l) |
| Megaplex RT Primers (10x) | 0.8 |
| dNTPs con dTTP (100 mM) | 0.2 |
| Multiscribe transcriptasa inverso (50 U / l) | 1.5 |
| 10X RT Buffer | 0.8 |
| MgCl2 (25 mM) | 0.9 |
| RNasa Inhibidor & # 160; (20 U / l) | 0.1 |
| Agua libre de nucleasa | 0.2 |
| Total | 4.5 |
Tabla 2A: RT componentes de la mezcla de reactivos en una preparación de reacción RT 5 l para el panel TLDA miRNA.
| Componentes | Volumen por reacción (l) |
| TaqMan PreAmp Mastermix (2x) | 12.5 |
| Megaplex preamplificador Primers (10x) | 2.5 |
| Agua libre de nucleasa | 7.5 |
| Total | 22.5 |
Tabla 2B: preamplificador componentes de la mezcla de reactivos en una preparación de reacción de 25 l de pre-amplificación para el panel TLDA miRNA.
"Cellspacing =" 0 "> Componentes Volumen por reacción (l) Megaplex RT Primers (10x) 0.75 dNTPs con dTTP (100 mM) 0.15 Multiscribe transcriptasa inverso (50 U / l) 1.5 10X RT Buffer 0.75 MgCl2 (25 mM) 0.9 RNasa Inhibidor (20 U / l) 0.09 Agua libre de nucleasa 0.35 Total 4.5Tabla 3A: RT componentes de la mezcla de reactivos en una preparación de reacción RT 5 l para el panel nanofluidos miARN basada en sondeos.
| Componentes | Volumen por reacción (l) |
| TaqMan PreAmp Mastermix (2x) | 12.5 |
| Megaplex preamplificador Primers (10x) | 2.5 |
| Agua libre de nucleasa | 7.5 |
| Total | 22.5 |
Tabla 3B:. Preamplificador componentes de la mezcla de reactivos en una preparación de reacción de 25 l de pre-amplificación para el panel nanofluidos miARN basado en la sonda NOTA: El complementario interactivo excel hojas de cálculo previstas para las tres plataformas, ya representa la adición de 5% para compensar el error de pipeteo.
| Muestra A | Muestra A repetición |
| 15.535 | 16.156 |
| 15.471 | 15.652 |
| 15.623 | 16.063 |
| 15.963 | 15.889 |
| 14.006 | 13.993 |
| 14.502 | 14.623 |
| 14.907 | 14.384 |
| 13.732 | 14.946 |
Tabla 4: Circulación miARN perfiles U6 usando microfluidos tarjeta matriz. Uso de dos tarjetas de matriz de microfluidos (A y B). Valores Ct de miRNA de control U6 de la muestra A (total = 8). El control U6 Consistente miARN abundancia observada entre las carreras.
Discusión
Los pasos críticos dentro de los protocolos de qPCR basados en sondas para obtener resultados exactos y reproducibles son para asegurarse de que 1) el mismo volumen y concentración de RT-producto se carga en cada reacción qPCR, 2) las proporciones correctas y los volúmenes de los componentes necesarios para qPCR reacción se preparó y se mezcló bien, 3) los volúmenes correctos y consistentes se añaden a cada reacción qPCR, y 4) la preparación y la carga de cada muestra y se mezcla de reacción se completa en el menor tiempo posible, mientras que todavía consciente de los pasos críticos antes mencionados antes.
Las tarjetas de matriz microfluídica necesitan centrífuga adecuada y cubos específicos. Cada cubo puede contener hasta 3 tarjetas (cargado / vacío). Asegúrese siempre de que los 3 slots de un cubo son ocupados y el cubo se equilibra mediante la colocación de cubo similar (este cubo debe contener también 3 cartas, vacíos o llenos) en la ranura opuesto de la centrífuga. Al colocar la tarjeta en la bucket titular, asegúrese de que los 8 embalses proyectan hacia arriba y pocillos de reacción frente a la pared exterior de la centrífuga. Haga girar las tarjetas en 331 xg durante 1 minuto a temperatura ambiente. Después de la primera vuelta, abrir la centrífuga y asegurar visualmente la mezcla de reacción se ha dispensado a través de los 384 pozos. Repita el lanzamiento en la misma configuración para 1 hora más. Retire la tarjeta de la cubeta y asegúrese de que el nivel de mezcla de reacción en cada uno de los 8 embalses es uniforme. Cualquier inconsistencia en los volúmenes de líquido que quedan en los embalses hacen la tarjeta inapropiado utilizar más.
Para tener la misma concentración del producto RT, se añadió misma concentración de entrada de ARN en cada reacción de RT. La concentración de ARN total se mide utilizando un micro-volumespectrophotometer. La relación observada 260/280 puede ser tan baja como 1,3 para el ARN aislado a partir de plasma / suero; esto no parece tener un efecto en proceso relacionado qPCR-aguas abajo o datos generados 30. Del mismo modo, la relación 260/280 delas muestras de RNA ensayadas en el presente documento fueron entre 1.3 a 1.7 sin efectos anormales en el qPCR observados.
Cuando se utilizan muestras de contenido de ARN bajos, tales como los de biofluidos, puede ser difícil de cuantificar ARN antes del procesamiento. Recomendamos el uso de sintéticos pico-in en el aislamiento de ARN, así como las etapas de transcripción inversa. En nuestra experiencia, los candidatos Arabidopsis thaliana miARN (ath-miR-159a y ath-miR-172a) son preferibles a Caenorhabditis elegans miRNAs (como cel-miR-39 o cel-miR-54), que en nuestra experiencia puede tener mayor homología que los de A. thaliana. El uso de tal etapa específica de espiga-en puede dar cuenta de la normalización de los datos a través de múltiples genes miARN muestras ensayadas en diferentes momentos. El uso de un volumen de entrada fija de ARN para la reacción de síntesis de ADNc también se recomienda 32,33.
Los tres protocolos basados en sondas para miARN cuantificación describen aquí requieren cantidades variablesde entrada de ARN total, los diferentes flujos de trabajo y los costos. Cada uno de los flujos de trabajo están diseñados para atender a los diferentes caudales en función del número de genes miARN objetivos y el número de muestras a analizar. Con el aumento de rendimiento (96 rxn 384 rxn 3072 rxn), el coste por reacción disminuye con un aumento en la cantidad de datos obtenidos durante una unidad de tiempo. Dado que todas estas plataformas utilizan la química TaqMan, la calidad de los datos obtenidos se puede esperar a ser similares. TaqMan qPCR es un método bien establecido para la identificación de la abundancia de miRNAs en muestras de suero / plasma 9,11,27. Aunque las tres plataformas discutidos aquí comparten la misma química, una disminución en el volumen de reacción conduce a una disminución en el rango dinámico de detección de la transcripción (Farr RJ et al., Datos no publicados). La plataforma qPCR 96 pocillos es un rendimiento más bajo, pero la plataforma de alta sensibilidad y en nuestra opinión, el "estándar de oro" para todos (o tinte a base) basada en sondeos plataformas de PCR. Sin embargo, esto puedeno ser la plataforma más económica o eficiente si varios cientos o miles de muestras están siendo analizadas y para múltiples microRNAs. Microfluídica (TLDA) y nanofluidos (OA) plataformas son plataformas de alto rendimiento / contenidos diseñados para permitir la adquisición de datos más grandes en un tiempo más corto. Aunque las diferencias de proceso por lotes se han observado en las tarjetas de TLDA, esto puede ser minimizado mediante la solicitud de TLDA tarjetas del mismo lote. Observamos que la plataforma TLDA (Figura 3) mostró una variación significativa en el 17% de las medianas - miRNAs baja abundancia cuando se prueba utilizando la misma muestra en diferentes lotes de TLDA tarjetas. Por lo tanto, recomendamos el uso de los mismos números de lote para el análisis utilizando TLDA tarjetas. Esta variación también podría ser debido a la variabilidad técnica incluyendo cualquier potenciales errores de carga y de pipeteo. Sin embargo, se recomienda ordenar / solicitar el mismo lote de TLDA tarjetas. Ninguna variación lote significativa se observó en la plataforma de OA. A pesar de esto, TaqMan experimentalmente basadotal qPCR se acerca ofrecen facilidad para medir la abundancia de miRNAs en muestras de plasma / suero. Los bajo, medio y alto rendimiento enfoques discutidos aquí ofrecen la flexibilidad para analizar una serie de muestras y miRNAs utilizando una (bajo nivel de ruido de fondo) química altamente eficiente, reproducible y limpio.
Divulgaciones
Los costos de procesamiento / publicación Artículo estaban cubiertos por Life Technologies, tras la aceptación de este manuscrito para su publicación.
Agradecimientos
Todos los autores reconocen el apoyo a la infraestructura de la CTC NHMRC, Universidad de Sydney, la Fundación de Investigación de Diabetes Juvenil (JDRF), Australia y la Fundación Rebecca Cooper, Australia. Esta investigación fue financiada por subvenciones del Consejo Australiano de Investigación (FT110100254) y la JDRF, Australia (CRN201314) a AAH WW, RJF y MVJ lleva a cabo toda la experimentación laboratorio húmedo, ASJ llevó a cabo el análisis de datos. WW escribió el primer borrador. AAH previsto el estudio y analizó los datos. Todos los autores leído y coincidieron en la versión final del manuscrito, figuras y hojas de trabajo presentados para su publicación.
Materiales
| Name | Company | Catalog Number | Comments |
| 96 well-platform | |||
| For Reverse transcription | |||
| TaqMan MicroRNA Assays INV SM | Applied Biosystems | 4427975 | https://www.lifetechnologies.com/au/en/home/life-science/pcr/real-time-pcr/real-time-pcr-assays/mirna-ncrna-taqman-assays/single-tube-mirna-taqman-assays.html?ICID=search-4427975 The assays comes as a pack of RT primers and PCR primer. |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366597 or 4366596 | https://www.lifetechnologies.com/order/catalog/product/4366597?ICID=search-4366597 or http://www.lifetechnologies.com/order/catalog/product/4366596?ICID=search-4366596 The TaqMan MicroRNA Reverse Transcription Kit is the same kit used for reverse transcription in all the threeTaqMan platform- 96-well platform, TLDA and OpenArray. |
| For qPCR run on a 96-well platform | |||
| 2x TaqMan Fast Universal PCR Master Mix, no AmpErase UNG | Applied Biosystems | 4367846 | https://www.lifetechnologies.com/order/catalog/product/4367846?ICID=search-4367846 The 2X TaqMan Fast Universal PCR Master Mix is used for qPCR on the two TaqMan platforms-96-well platform and TLDA. |
| MicroAmp Optical Adhesive Film | Applied Biosystems | 4311971 | https://www.lifetechnologies.com/order/catalog/product/4311971?ICID=search-product |
| MicroAmp Fast Optical 96-Well Reaction Plate, 0.1 ml | Applied Biosystems | 4346907 | https://www.lifetechnologies.com/order/catalog/product/4346907?ICID=search-product |
| Microfluidics platform (TLDA) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | https://www.lifetechnologies.com/order/catalog/product/4399966?ICID=search-4399966 The Megaplex RT Primers, Human Pool A v2.1 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex RT Primers, Human Pool B v3.0 | Applied Biosystems | 4444281 | https://www.lifetechnologies.com/order/catalog/product/4444281?ICID=search-4444281 The Megaplex RT Primers, Human Pool B v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| OR | OR | ||
| Megaplex Primer Pools, Human Pools Set v3.0 | Applied Biosystems | 4444750 | http://www.lifetechnologies.com/order/catalog/product/4444750?ICID=search-4444750 The Megaplex Primer Pools, Human Pools Set v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| For Pre-amplification | |||
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 or 4488593 | https://www.lifetechnologies.com/order/catalog/product/4391128?ICID=search-4391128 or http://www.lifetechnologies.com/order/catalog/product/4488593 The TaqMan PreAmp Master Mix is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool B v3.0 | Applied Biosystems | 4444303 | https://www.lifetechnologies.com/order/catalog/product/4444303?ICID=search-4444303 The Megaplex PreAmp Primers, Human Pool B v3.0 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399233 | https://www.lifetechnologies.com/order/catalog/product/4399233?ICID=search-4399233 The Megaplex PreAmp Primers, Human Pool A v2.1 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| 1x TE Buffer (100 ml) | Invitrogen | 12090-015 | https://www.lifetechnologies.com/order/catalog/product/12090015?ICID=search-product The 1x TE Buffer is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| To load and run the 384 well microfluidics (TLDA) card | |||
| TaqMan Array Human MicroRNA A+B Cards Set v3.0 | Applied Biosystems | 4444913 | https://www.lifetechnologies.com/order/catalog/product/4444913 |
| Nanofluidics platform (OpenArray) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| For Pre-amplification | |||
| Refer to TLDA pre-amplification reagents (as shown above) | |||
| To load and run the slides | |||
| TaqMan OpenArray Human MicroRNA Panel, QuantStudio 12K Flex (1 panel) | Applied Biosystems | 4470187 | https://www.lifetechnologies.com/order/catalog/product/4470187?ICID=search-4470187 |
| QuantStudio 12K Flex OpenArray Accessories Kit | Applied Biosystems | 4469576 | https://www.lifetechnologies.com/order/catalog/product/4469576?ICID=search-4469576 |
| OpenArray 384-well Sample Plates | Applied Biosystems | 4406947 | https://www.lifetechnologies.com/order/catalog/product/4406947?ICID=search-4406947 |
| TaqMan OpenArray Real-Time PCR Master Mix | Applied Biosystems | 4462159 | https://www.lifetechnologies.com/order/catalog/product/4462159?ICID=search-4462159 |
| OpenArray AccuFill System Tips | Applied Biosystems | 4457246 | https://www.lifetechnologies.com/order/catalog/product/4457246?ICID=search-4457246 |
| Others | |||
| Nuclease-Free Water | Qiagen | 129117 | http://www.qiagen.com/products/catalog/lab-essentials-and-accessories/nuclease-free-water |
Referencias
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Alvarez-Garcia, I., Miska, E. A. MicroRNA functions in animal development and human disease. Development. 132, 4653-4662 (2005).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-297 (2004).
- Joglekar, M. V., Wei, C., Hardikar, A. A. Quantitative estimation of multiple miRNAs and mRNAs from a single cell. Cold Spring Harb Protoc. 2010, pdb prot5478(2010).
- Joglekar, M. V., Parekh, V. S., Hardikar, A. A. Islet-specific microRNAs in pancreas development, regeneration and diabetes. Indian J Exp Biol. 49, 401-408 (2011).
- Farr, R. J., Joglekar, M. V., Taylor, C. J., Hardikar, A. A. Circulating non-coding RNAs as biomarkers of beta cell death in diabetes. Pediatr Endocrinol Rev. 11, 14-20 (2013).
- Joglekar, M. V., Joglekar, V. M., Hardikar, A. A. Expression of islet-specific microRNAs during human pancreatic development. Gene Expr Patterns. 9, 109-113 (2009).
- Iorio, M. V., Croce, C. M. MicroRNA profiling in ovarian cancer. Methods Mol Biol. 1049, 187-197 (2013).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. P Natl Acad Sci USA. 105, 10513-10518 (2008).
- Mattie, M. D., et al. Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies. Mol Cancer. 5, 24(2006).
- Zhu, W., Qin, W., Atasoy, U., Sauter, E. R. Circulating microRNAs in breast cancer and healthy subjects. BMC Res Notes. 2, 89(2009).
- Zhu, H. T., et al. Identification of suitable reference genes for qRT-PCR analysis of circulating microRNAs in hepatitis B virus-infected patients. Mol Biotechnol. 50, 49-56 (2012).
- Yamada, H., Itoh, M., Hiratsuka, I., Hashimoto, S. Circulating microRNAs in autoimmune thyroid diseases. Clin Endocrinol (Oxf). , (2014).
- Liu, R., et al. Serum MicroRNA Expression Profile as a Biomarker in the Diagnosis and Prognosis of Pancreatic Cancer. Clinical Chemistry. 58, 610-618 (2012).
- Gilad, S., et al. Serum microRNAs are promising novel biomarkers. PLoS One. 3, e3148(2008).
- Joglekar, M. V., Parekh, V. S., Mehta, S., Bhonde, R. R., Hardikar, A. A. MicroRNA profiling of developing and regenerating pancreas reveal post-transcriptional regulation of neurogenin3. Dev Biol. 311, 603-612 (2007).
- Hardikar, A. A., Farr, R. J., Joglekar, M. V. Circulating microRNAs: understanding the limits for quantitative measurement by real-time PCR. J Am Heart Assoc. 3, e000792(2014).
- Wu, C., et al. Diagnostic and Prognostic Implications of a Serum miRNA Panel in Oesophageal Squamous Cell Carcinoma. PLoS One. 9, e92292(2014).
- McDonald, M. K., Capasso, K. E., Ajit, S. K. Purification and microRNA profiling of exosomes derived from blood and culture media. J Vis Exp. , e50294(2013).
- Joglekar, M. V., et al. The miR-30 family microRNAs confer epithelial phenotype to human pancreatic cells. Islets. 1, 137-147 (2009).
- Genda, Y., et al. microRNA changes in the dorsal horn of the spinal cord of rats with chronic constriction injury: A TaqMan(R) Low Density Array study. Int J Mol Med. 31, 129-137 (2013).
- Wang, B., et al. Systematic evaluation of three microRNA profiling platforms: microarray, beads array, and quantitative real-time PCR array. PLoS One. 6, e17167(2011).
- Cuk, K., et al. Circulating microRNAs in plasma as early detection markers for breast cancer. Int J Cancer. 132, 1602-1612 (2013).
- Morrison, T., et al. Nanoliter high throughput quantitative PCR. Nucleic Acids Res. 34, e123(2006).
- Git, A., et al. Systematic comparison of microarray profiling, real-time PCR, and next-generation sequencing technologies for measuring differential microRNA expression. RNA. 16, 991-1006 (2010).
- Benes, V., Castoldi, M. Expression profiling of microRNA using real-time quantitative PCR, how to use it and what is available. Methods. 50, 244-249 (2010).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res. 33, e179(2005).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44, 31-38 (2008).
- Kodani, M., et al. Application of TaqMan low-density arrays for simultaneous detection of multiple respiratory pathogens. J Clin Microbiol. 49, 2175-2182 (2011).
- Taylor, C. J., Satoor, S. N., Ranjan, A. K., Pereirae Cotta,, V, M., Joglekar, M. V. A protocol for measurement of noncoding RNA in human serum. Exp Diabetes Res. 2012, 168368(2012).
- TPF & SPF File Download Options [Internet]. , Available from: http://www.appliedbiosystems.com/absite/us/en/home/products/tpf-spf-download.html (2014).
- Turchinovich, A., Weiz, L., Langheinz, A., Burwinkel, B. Characterization of extracellular circulating microRNA. Nucleic Acids Res. 39, 7223-7233 (2011).
- Li, Y., Kowdley, K. V. Method for microRNA isolation from clinical serum samples. Anal Biochem. 431, 69-75 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados