Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Mapeo de todo el genoma de la droga-ADN interacciones en células con COSMIC (reticulación de pequeñas moléculas para aislar la cromatina)
En este artículo
Resumen
La identificación de los blancos directos de moléculas del genoma de metas sigue siendo un reto importante. Para entender cómo las moléculas de unión al ADN involucran el genoma, hemos desarrollado un método que se basa en la reticulación de moléculas pequeñas para aislar la cromatina (cósmica).
Resumen
El genoma es el objetivo de algunos de los agentes quimioterapéuticos más eficaces, pero la mayoría de estos fármacos carecen de especificidad de secuencia de ADN, que conduce a la toxicidad limitante de la dosis y muchos efectos secundarios adversos. Targeting el genoma con pequeñas moléculas específicas de secuencia puede permitir moléculas con el aumento del índice terapéutico y menos efectos fuera de objetivo. -methylpyrrole N / N poliamidas metilimidazol son moléculas que pueden ser racionalmente diseñados para dirigir las secuencias de ADN específicas con exquisita precisión. Y a diferencia de la mayoría de los factores de transcripción naturales, poliamidas pueden unirse al ADN metilado y cromatinizados sin una pérdida en la afinidad. La especificidad de secuencia de poliamidas ha sido ampliamente estudiado in vitro con la identificación del emplazamiento cognado (CSI) y con los enfoques bioquímicos y biofísicos tradicionales, pero el estudio de poliamida unión a dianas genómicas en las células sigue siendo difícil de alcanzar. Aquí mostramos un método, la reticulación de moléculas pequeñas a Isolate cromatina (cósmica), que identifica los sitios de unión de poliamida en todo el genoma. Cósmico es similar a inmunoprecipitación de cromatina (ChIP), pero difiere en dos aspectos importantes: (1) un fotoentrecruzador se emplea para permitir la captura selectiva, controlada temporalmente de eventos de unión de poliamida, y (2) el asa de afinidad biotina se utiliza para purificar poliamida conjugados -ADN en condiciones semi-desnaturalizante para disminuir ADN que es no covalentemente. Cósmico es una estrategia general que puede ser utilizado para revelar los eventos de unión en todo el genoma de poliamidas y otros agentes quimioterapéuticos del genoma de metas.
Introducción
La información para hacer cada célula en el cuerpo humano está codificado en el ADN. El uso selectivo de la información que gobierna el destino de una célula. Los factores de transcripción (TFS) son proteínas que se unen a secuencias específicas de ADN para expresar un subconjunto particular de los genes en el genoma, y el mal funcionamiento de TFS está vinculada a la aparición de una amplia gama de enfermedades, incluyendo defectos de desarrollo, el cáncer y la diabetes 1,2. Hemos estado interesados en el desarrollo de moléculas que pueden unirse selectivamente al genoma y modular las redes de regulación de genes.
Poliamidas compuestos de N -methylpyrrole y N metilimidazol son moléculas que pueden diana de ADN con especificidades y afinidades que los factores de transcripción naturales rivales. 3-6 Estas moléculas se unen a secuencias específicas en el surco menor del ADN racionalmente diseñados. 4,5,7 -11 Las poliamidas se han empleado tanto para reprimir y activar la expresión de g específicaenes. 4,12-19 También tienen interesantes antivirales 20-24 y anticancerígenas 12,13,25-30 propiedades. Una característica atractiva de poliamidas es su capacidad para acceder a secuencias de ADN que están metilados 31,32 y envuelven alrededor de las proteínas histonas 9,10,33.
Para medir las especificidades de unión integrales de moléculas de unión al ADN, nuestro laboratorio creó el método de identificador de sitio de reconocimiento (CSI). 34-39 La aparición prevista de sitios de unión basado en in vitro especificidades (genomescapes) se puede visualizar en el genoma, ya que el in vitro intensidades de unión son directamente proporcionales a las constantes de asociación (K a). 34,35,37 Estos genomescapes proporcionan información sobre la ocupación de poliamida en todo el genoma, pero poliamida medir la unión en células vivas ha sido un reto. ADN está estrechamente empaquetado en el núcleo, que podría influir en la accesibilidad de los sitios de unión. La Accesibilidad de estas secuencias de ADN cromatinizados a poliamidas sigue siendo un misterio.
Recientemente, muchos métodos para estudiar las interacciones entre pequeñas moléculas y ácidos nucleicos han surgido. 40-48 La captura por afinidad química y la secuenciación del ADN masivamente paralelo (chem-ss) es una de estas técnicas. Chem-ss utiliza formaldehído para reticular moléculas pequeñas a una diana genómica de interés y un derivado biotinilado de una pequeña molécula de interés para capturar la interacción ligando-diana. 48,49
Reticulación formaldehído conduce a interacciones indirectas que pueden producir falsos positivos. 50 Hemos desarrollado un nuevo método, la reticulación de moléculas pequeñas para aislar la cromatina (cósmica), 51 con un fotoentrecruzador para eliminar estos llamados picos "fantasma". 50 Para empezar, hemos diseñado y sintetizado derivados trifuncionales de poliamidas. Estas moléculas contienen una pol unión al ADNyamide, un fotoentrecruzador (psoraleno), y un asa de afinidad (biotina, Figura 1). Con poliamidas trifuncionales, podemos capturar covalentemente interacciones de ADN de poliamida con irradiación UV 365 nm, una longitud de onda que no daña el ADN o inducir no psoraleno-basado de reticulación. 51 A continuación, fragmentar el genoma y purificar el ADN capturado bajo estrictas, semi condiciones -denaturing para disminuir ADN que es no covalentemente. Por lo tanto, visión cósmica como un método relacionado con chem-SEQ, pero con una lectura más directa de ADN de direccionamiento. Es importante destacar que los débiles (K a 10 3 -10 4 M -1) afinidad de psoraleno para el ADN no detectable de poliamida de impacto especificidad. 51,52 Los fragmentos de ADN enriquecidos pueden ser analizados por cualquiera de reacción en cadena de la polimerasa cuantitativa 51 (COSMIC-qPCR) o mediante la secuenciación de próxima generación 53 (COSMIC-ss). Estos datos permiten un diseño imparcial genoma guiada de ligandos que entreactuar con sus loci genómica deseada y minimizar los efectos fuera de objetivo.
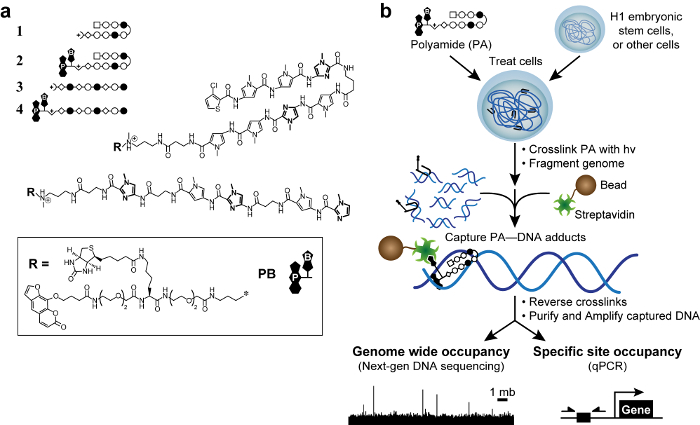
Figura 1. poliamidas bioactivos y esquema cósmico. (A) poliamidas horquilla 1-2 diana la secuencia de ADN 5'-WACGTW-3 '. Poliamidas lineales 3 - 4 diana 5'-AAGAAGAAG-3 '. Anillos de N-metilimidazol están en negrita para mayor claridad. Los círculos vacíos y llenos representan N -methylpyrrole y N metilimidazol, respectivamente. Plaza representa 3-clorotiofeno y diamantes representan β-alanina. Psoralen y la biotina se denotan por P y B, respectivamente. (B) esquema cósmico. Las células se tratan con derivados trifuncionales de poliamidas. Después de la reticulación con 365 nm de la irradiación UV, las células son lse corta ADN genómico y ysed. Se añaden perlas magnéticas recubiertas con estreptavidina para capturar aductos de ADN-poliamida. El ADN se libera y puede ser analizado mediante PCR cuantitativa (qPCR) o mediante la secuenciación de próxima generación (NGS). Por favor, haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. La reticulación en células vivas
- Comience con ~ 2.5x10 7 células H1, u otras células cultivadas.
NOTA: Este número de células H1 corresponde a cinco platos de 10 cm (aproximadamente 40% de confluencia). - Cultivar las células en medios E8 en placas revestidas en una superficie que soporta las células madre pluripotentes (ver Lista de Materiales), y se incuba a 37 ° C en ambiente humidificado del 5% de CO 2. Células Cosecha enzimáticamente (ver Lista de Materiales). Nota: No permita que las células H1 excedan el 90% de confluencia; confluencia induce la diferenciación espontánea de células H1. Para contar las células, crecen un extra de 10 cm de placa de células, levantar las células enzimáticamente como se describe en las secciones 1.8-1.10, y contar con un hemocitómetro.
- Preparar E8 medios de cultivo como se describe en Chen et al. 54
- Antes de la adición de poliamida, extraiga el medio de cultivo gastado con una pipeta Pasteur unido a una trampa de vacío. Añadir medio fresco con la mayorpipeta erological y dispensador de pipeta (8 ml por 10 cm de plato).
NOTA: Agregue los medios de comunicación para el lado del plato con el fin de evitar la interrupción de las células. Desde este punto en adelante proteger a las células de la luz para evitar la prematura foto-reticulación. - Añadir poliamida con una pipeta (8 l de 400 mM de poliamida en DMSO, 400 nM de concentración final) directamente a los medios de cultivo de cada plato. Agitar el plato para dispersar la poliamida uniformemente en los medios de comunicación.
NOTA: La concentración puede ser variada, pero asegurar que ninguna toxicidad celular se observa para el tratamiento seleccionado. - Se incuban las células 24 horas 37 ° C en un ambiente humidificado del 5% de CO 2, y asegurarse de que están protegidos de la luz.
NOTA: El tiempo de incubación puede variarse para medir la unión de poliamida a través del tiempo. - Lave cada placa de 10 cm con 4 ml de PBS (1,05 mM fosfato monobásico de potasio, cloruro de sodio 155,17 mM, 2,97 mM fosfato dibásico de sodio) con una pipeta y pipetas serológicas disdispensador. Aspirar PBS y añadir 3 ml E8 medios de cultivo.
- Con las luces se apagaron, retire la tapa de 5 placas de cultivo y colocar las células en una superficie plana exterior de la campana. Coloque un filtro de vidrio sobre las 5 placas de cultivo para filtrar la luz con λ <300 nm. Coloque la fuente de UV en la parte superior del filtro. Muestras de reticulación 30 min con 365 nm de la irradiación UV (2,4 mW / cm 2).
NOTA: El tiempo de reticulación se debe determinar empíricamente. - Traslado placas de cultivo de vuelta a la campana. Aspirar los medios de comunicación con una pipeta Pasteur unido a una trampa de vacío. Lave cada placa de 10 cm con 4 ml de PBS. Aspirar el PBS. Añadir 3 ml de enzima de disociación celular (ver Lista de Materiales) por 10 cm plato de disociar las células. Incubar 5 minutos a 37 ° C.
NOTA: No precalentar la enzima. - Se inactiva la enzima con 3 ml E8 medios de comunicación por plato. Transferencia de células disociada en un tubo cónico de 15 ml.
NOTA: Coloque las células en hielo desde este punto en adelante. - Centrifuge disociada células 5 min, 500 xga 4 ° C. Aspirar el sobrenadante para eliminar la enzima y medios de comunicación.
NOTA: Punto Pausa. Las células pueden ser-flash congelado en nitrógeno líquido, se almacena a -80 ° C para su posterior procesamiento en un momento posterior.
2. Aislamiento de la cromatina
- Añadir 1,2 ml de tampón cósmicos (20 mM Tris-HCl [pH 8,1], EDTA 2 mM, NaCl 150 mM, 1% Triton-X100, 0,1% SDS) y la pipeta arriba y abajo varias veces para resuspender el sedimento de células para cada muestra en 1,2 ml de tampón cósmica.
- Añadir 133 l de cada uno de 100 mM fluoruro de fenilmetilsulfonilo (PMSF), benzamidina 100 mM y 150 mM, pepstatina inhibidores de la proteasa fresca a una concentración final de 1 mM PMSF y para benzamidina y 1,5 M durante pepstatina. Solución de Split de lisado celular en dos tubos de microcentrífuga de 1,7 ml de color ámbar.
- Sonicar con ultrasonidos 35 minutos (10 segundos en 10 segundos apagado, la alimentación 60%) para fragmentar el genoma a entre 100nd 500 pb.
- Mantener el nivel de la solución de la cromatina en el tubo de microcentrífuga paralelo al nivel de agua en el depósito. Confirmar este nivel mediante inspección visual.
NOTA:. Confirmar que el ADN se cortó con un gel de agarosa al 1,5% 55 - Optimizar el tiempo de sonicación empíricamente. Utilice una cantidad mínima de hielo en el depósito para enfriar las muestras, y asegurar el hielo no está interfiriendo entre las muestras y el cuerno taza.
- Mantener el nivel de la solución de la cromatina en el tubo de microcentrífuga paralelo al nivel de agua en el depósito. Confirmar este nivel mediante inspección visual.
- Centrifugar la muestra 12.000 xg 10 min. Guardar la solución acuosa que contiene la cromatina soluble mediante la transferencia a un nuevo tubo de microcentrífuga de color ámbar con una pipeta. Deseche la pastilla.
- Transfiera 110 l (10%) de la muestra a un nuevo tubo de microcentrífuga y la etiqueta de ADN de entrada. Almacenar a -80 ° C. Guarde el resto de la muestra de la cromatina en hielo para su uso en el paso 3.3.
3. Captura de ligando de ADN Crosslinks
- Utilice una pipeta para dispensar 60 l streptavirecubierto-din perlas magnéticas en un tubo de microcentrífuga. Añadir 1 ml de tampón COSMIC y mezclar en un nutator 5 minutos a temperatura ambiente. Coloque perlas magnéticas en la separación magnética estante 2 min para capturar los granos. Retire búfer cósmica con una pipeta.
NOTA: No permita que las perlas se sequen. Proceda inmediatamente al siguiente paso. - Añadir la muestra de la cromatina (~ 1 ml) a las perlas (60 l) y resuspender las perlas. Incubar la cromatina con perlas magnéticas recubiertas con estreptavidina al menos 4 h en un mezclador oscilante que gira a 4 ° C. Incubar las muestras S / N, si se desea.
4. Aislamiento de ADN purificado por afinidad
- Lavar los granos con intervalo de 7 min a TA con los siguientes tampones de lavado para eliminar las interacciones no específicas. Utilice un rack de separación magnética para capturar los granos después de cada lavado. Resuspender las perlas después de cada cambio de tampón de lavado.
- Preparar tampones de lavado en agua destilada desionizada y el filtro (0,2 micras) antes de su uso. Los tampones de lavado tienda 1 y 2 a 4 ° C para smes arios. Preparar el tampón de lavado 3 fresco cada día. Agregar y quitar tampones de lavado de la muestra con una pipeta. Para 2 y 4, añadir 1 y 3 (5 mM), respectivamente, en los lavados. Las muestras pueden ser adicionalmente lavaron dos veces con tampón de COSMIC (una vez 12 hr, una vez 4 horas) antes de los lavados se enumeran a continuación.
- Lavar una vez con tampón de lavado 1 (10 mM Tris-Cl [pH 8,0], EDTA 1 mM, 3% de SDS). Lavar una vez con tampón de lavado 2 (10 mM Tris-Cl [pH 8,0], 250 mM LiCl, EDTA 1 mM, 0,5% NP40, 1% de desoxicolato de sodio). Lavar dos veces con tampón de lavado 3 (4 M urea, 10 mM Tris-Cl [pH 7,5], EDTA 1 mM, 0,1% NP-40). Lavar dos veces con Tris EDTA (TE) tampón (10 mM Tris-Cl [pH 8,0], EDTA 1 mM).
- Resuspender las perlas en tampón TE 200 l con una pipeta. Esta muestra de ADN capturado se conoce como ADN purificado por afinidad (AP).
- Complementar el ADN de entrada y AP con 10x Crosslink Reversión Buffer (100 mM Tris-Cl [pH 8,0], 1 M KOH, 4 mMEDTA) a 1x concentración final. 56,57 Incubar 30 min a 90 ° C.
NOTA: 254 nm irradiación UV también se puede usar para invertir la reticulación de psoraleno, 58 pero dímeros de pirimidina ciclobutilo pueden formarse a partir irradiación a esta longitud de onda e interferir con el procesamiento aguas abajo del ADN. - Para el ADN AP, coloque el tubo de microcentrífuga que contiene la muestra en la separación magnética rack de 2 min a TA y aislar líquido (ADN) con una pipeta. Transferir el ADN AP (que se ha liberado de las perlas) a un nuevo tubo de microcentrífuga ámbar.
NOTA: El ADN AP deseado en el líquido, ya no se une a las perlas. - Neutralizar de entrada y el ADN AP con HCl concentrado hasta pH 7 mediante la adición de aproximadamente 1 l de HCl 6 N por 100 l de la muestra con una pipeta. Confirme las muestras se neutralizan mediante la adición de ~ 0,5 l en papel pH con una pipeta ADVERTENCIA:. HCl concentrado es un ácido corrosivo fuerte. Manipular de acuerdo a su institución217; s directrices para el equipo de protección personal adecuado.
- Añadir RNasa A (100 mg / ml) a 0,2 g / l de concentración final tanto de entrada como de ADN AP. Incubar 1 hora a 37 ° C. Añadir a la proteinasa K (20 mg / ml) a 0,2 g / l de concentración final tanto de entrada como de ADN AP, agregar. Incubar 1 hora a 55 ° C.
- Se purifica el ADN con un kit de limpieza de la columna de ADN (ver Lista de Materiales). 55 Eluir el ADN en 58 l de ADN de grado H 2 O. Muestras de entrada de la tienda y AP a -20 o -80 ° C
- Analizar ADN AP mediante la reacción en cadena de polimerasa cuantitativa (qPCR) con cebadores específicos del locus, y / o por secuenciación de próxima generación. Para qPCR, utilizar 2 l de ADN AP por locus con los siguientes parámetros: 1 ciclo de 95 ° C 10 min y 40 ciclos de 95 ° C 20 seg, 54 ° C o 56 ° C 20 seg, 72 ° C 40 seg.
NOTA: La temperatura de hibridación debe ser modificado de acuerdo con la temperatura de fusión de los pares de cebadores usados. Seleccione una temperatu recocidore que minimiza el ciclo de cuantificación sin amplificación no específica.
NOTA: Para el análisis de secuenciación de próxima generación, el objetivo de al menos 10 millones de dólares asignada lee. El número de mapeado lee puede aumentarse mediante el aumento de la cantidad de ADN AP y mediante la combinación de menos muestras a correr para la secuenciación. Más lee mejorar la sensibilidad. Los paquetes estándar para el análisis de chip siguientes (por ejemplo, Homero, 59 MACS, 60 y 61 SPP) trabajan con datos COSMIC ss. Al igual que con otros métodos que se basan en la secuenciación de próxima generación, incluyendo la secuenciación del genoma y chip-ss, regiones repetitivas crear ambigüedades en la alineación y montaje y por lo tanto siguen siendo un desafío técnico.
Resultados
Para tener en cuenta la fragmentación del genoma no uniforme y otras variables, el ADN purificado siempre debe ser normalizado contra una referencia de ADN de entrada. Los cebadores específicos de un locus de interés pueden ser utilizados. Es útil también para analizar un locus donde no se espera que la molécula para unirse, como un control negativo. Vemos un incremento> 100 veces en la ocupación de poliamida tras la irradiación con luz de 365 nm (Figura 2).
Enriq...
Discusión
One of the primary challenges with conventional ChIP is the identification of suitable antibodies. ChIP depends heavily upon the quality of the antibody, and most commercial antibodies are unacceptable for ChIP. In fact, the Encyclopedia of DNA Elements (ENCODE) consortium found only 20% of commercial antibodies to be suitable for ChIP assays.50 With COSMIC, antibodies are replaced by streptavidin. Because polyamides are functionalized with biotin, streptavidin is used in place of an antibody to capture polyam...
Divulgaciones
A.Z.A. is the sole proprietor of Vista Motif, LLC and WINStep Forward.
Agradecimientos
We thank members of the Ansari lab and Prof. Parameswaran Ramanathan for helpful discussions. This work was supported by NIH grants CA133508 and HL099773, the H. I. Romnes faculty fellowship, and the W. M. Keck Medical Research Award to A.Z.A. G.S.E. was supported by a Peterson Fellowship from the Department of Biochemistry and Molecular Biosciences Training Grant NIH T32 GM07215. A.E. was supported by the Morgridge Graduate Fellowship and the Stem Cell and Regenerative Medicine Center Fellowship, and D.B. was supported by the NSEC grant from NSF.
Materiales
| Name | Company | Catalog Number | Comments |
| Phenylmethylsulfonyl fluoride (PMSF) | any source | ||
| Benzamidine | any source | ||
| Pepstatin | any source | ||
| Proteinase K | any source | ||
| Dynabeads MyOne Streptavidin C1 | Life Technologies | 65001 | |
| PBS, pH 7.4 | Life Technologies | 10010-023 | Other sources can be used |
| StemPro Accutase Cell Dissociation Reagent | Life Technologies | A1110501 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | We have tried other manufacturers of DNA columns with success. |
| TruSeq ChIP Sample Prep Kit | Illumina | IP-202-1012 | This Kit can be used to prepare COSMIC DNA for next-generation sequencing |
| Matrigel Basement Membrane Matrix | BD Biosciences | 356231 | Used to coat plates in order to grow H1 ESCs |
| pH paper | any source | ||
| amber microcentrifuge tubes | any source | ||
| microcentrifuge tubes | any source | ||
| pyrex filter | any source | Pyrex baking dishes are suitable | |
| qPCR master mix | any source | ||
| RNase | any source | ||
| HCl (6 N) | any source | ||
| 10-cm tissue culture dishes | any source | ||
| Serological pipettes | any source | ||
| Pasteur pipettes | any source | ||
| Pipette tips | any source | ||
| 15-ml conical tubes | any source | ||
| centrifuge | any source | ||
| microcentrifuge | any source | ||
| nutator | any source | ||
| Magnetic separation rack | any source | ||
| UV source | CalSun | B001BH0A1A | Other UV sources can be used, but crosslinking time must be optimized empirically |
| Misonix Sonicator | Qsonica | S4000 with 431C1 cup horn | Other sonicators can be used, but sonication conditions must be optimized empirically |
| Humidified CO2 incubator | any source | ||
| Biological safety cabinet with vacuum outlet | any source |
Referencias
- Lee, T. I., Young, R. A. Transcriptional Regulation and Its Misregulation in Disease. Cell. 152, 1237-1251 (2013).
- Vaquerizas, J. M., Kummerfeld, S. K., Teichmann, S. A., Luscombe, N. M. A census of human transcription factors: function, expression and evolution. Nat Rev Genet. 10, 252-263 (2009).
- Meier, J. L., Yu, A. S., Korf, I., Segal, D. J., Dervan, P. B. Guiding the Design of Synthetic DNA-Binding Molecules with Massively Parallel Sequencing. J. Am. Chem. Soc. 134, 17814-17822 (2012).
- Dervan, P. B. Molecular recognition of DNA by small molecules. Bioorg. Med. Chem. 9, 2215-2235 (2001).
- Wemmer, D. E., Dervan, P. B. Targeting the minor groove of DNA. Curr. Opin. Struct. Biol. 7, 355-361 (1997).
- Eguchi, A., Lee, G. O., Wan, F., Erwin, G. S., Ansari, A. Z. Controlling gene networks and cell fate with precision-targeted DNA-binding proteins and small-molecule-based genome readers. Biochem. J. 462, 397-413 (2014).
- Mrksich, M., et al. Antiparallel side-by-side dimeric motif for sequence-specific recognition in the minor groove of DNA by the designed peptide 1-methylimidazole-2-carboxamide netropsin. Proc Natl Acad Sci U S A. 89, 7586-7590 (1992).
- Edayathumangalam, R. S., Weyermann, P., Gottesfeld, J. M., Dervan, P. B., Luger, K. Molecular recognition of the nucleosomal "supergroove". Proc Natl Acad Sci U S A. 101, 6864-6869 (2004).
- Suto, R. K., et al. Crystal structures of nucleosome core particles in complex with minor groove DNA-binding ligands. J. Mol. Biol. 326, 371-380 (2003).
- Gottesfeld, J. M., et al. Sequence-specific Recognition of DNA in the Nucleosome by Pyrrole-Imidazole Polyamides. J. Mol. Biol. 309, 615-629 (2001).
- Chenoweth, D. M., Dervan, P. B. Structural Basis for Cyclic Py-Im Polyamide Allosteric Inhibition of Nuclear Receptor Binding. J. Am. Chem. Soc. 132, 14521-14529 (2010).
- Raskatov, J. A., et al. Modulation of NF-κB-dependent gene transcription using programmable DNA minor groove binders. Proc. Natl. Acad. Sci. U.S.A. 109, 1023-1028 (2012).
- Yang, F., et al. Antitumor activity of a pyrrole-imidazole polyamide. Proc. Natl. Acad. Sci. U.S.A. 110, 1863-1868 (2013).
- Mapp, A. K., Ansari, A. Z., Ptashne, M., Dervan, P. B. Activation of gene expression by small molecule transcription factors. Proc. Natl. Acad. Sci. U.S.A. 97, 3930-3935 (2000).
- Ansari, A. Z., Mapp, A. K., Nguyen, D. H., Dervan, P. B., Ptashne, M. Towards a minimal motif for artificial transcriptional activators. Chem. Biol. 8, 583-592 (2001).
- Arora, P. S., Ansari, A. Z., Best, T. P., Ptashne, M., Dervan, P. B. Design of artificial transcriptional activators with rigid poly-L-proline linkers. J. Am. Chem. Soc. 124, 13067-13071 (2002).
- Nickols, N. G., Jacobs, C. S., Farkas, M. E., Dervan, P. B. Modulating Hypoxia-Inducible Transcription by Disrupting the HIF-1–DNA Interface. ACS Chemical Biology. 2, 561-571 (2007).
- Pandian, G. N., et al. A synthetic small molecule for rapid induction of multiple pluripotency genes in mouse embryonic fibroblasts. Sci. Rep. 2, 544 (2012).
- Pandian, G. N., et al. Synthetic Small Molecules for Epigenetic Activation of Pluripotency Genes in Mouse Embryonic Fibroblasts. Chem Bio Chem. 12, 2822-2828 (2011).
- He, G., et al. Binding studies of a large antiviral polyamide to a natural HPV sequence. Biochimie. 102, 83-91 (2014).
- Edwards, T. G., Vidmar, T. J., Koeller, K., Bashkin, J. K., Fisher, C. DNA Damage Repair Genes Controlling Human Papillomavirus (HPV) Episome Levels under Conditions of Stability and Extreme Instability. PLoS ONE. 8, e75406 (2013).
- Edwards, T. G., Helmus, M. J., Koeller, K., Bashkin, J. K., Fisher, C. HPV Episome Stability is Reduced by Aphidicolin and Controlled by DNA Damage Response Pathways. Journal of Virology. , (2013).
- Edwards, T. G., et al. HPV episome levels are potently decreased by pyrrole-imidazole polyamides. Antiviral Res. 91, 177-186 (2011).
- Dickinson, L. A., et al. Inhibition of RNA polymerase II transcription in human cells by synthetic DNA-binding ligands. Proc. Natl. Acad. Sci. U.S.A. 95, 12890-12895 (1998).
- Dickinson, L. A., et al. Arresting Cancer Proliferation by Small-Molecule Gene Regulation. Chem. Biol. 11, 1583-1594 (2004).
- Nickols, N. G., et al. Activity of a Py–Im Polyamide Targeted to the Estrogen Response Element. Molecular Cancer Therapeutics. 12, 675-684 (2013).
- Raskatov, J. A., Puckett, J. W., Dervan, P. B. A C-14 labeled Py–Im polyamide localizes to a subcutaneous prostate cancer tumor. Bioorg. Med. Chem. 22, 4371-4375 (2014).
- Jespersen, C., et al. Chromatin structure determines accessibility of a hairpin polyamide–chlorambucil conjugate at histone H4 genes in pancreatic cancer cells. Bioorg. Med. Chem. Lett. 22, 4068-4071 (2012).
- Chou, C. J., et al. Small molecules targeting histone H4 as potential therapeutics for chronic myelogenous leukemia. Molecular Cancer Therapeutics. 7, 769-778 (2008).
- Nickols, N. G., Dervan, P. B. Suppression of androgen receptor-mediated gene expression by a sequence-specific DNA-binding polyamide. Proc. Natl. Acad. Sci. U.S.A. 104, 10418-10423 (2007).
- Minoshima, M., Bando, T., Sasaki, S., Fujimoto, J., Sugiyama, H. Pyrrole-imidazole hairpin polyamides with high affinity at 5CGCG3 DNA sequence; influence of cytosine methylation on binding. Nucleic Acids Res. 36, 2889-2894 (2008).
- Warren, C. L., et al. Fabrication of duplex DNA microarrays incorporating methyl-5-cytosine. Lab on a Chip. 12, 376-380 (2012).
- Dudouet, B., et al. Accessibility of nuclear chromatin by DNA binding polyamides. Chem. Biol. 10, 859-867 (2003).
- Carlson, C. D., et al. Specificity landscapes of DNA binding molecules elucidate biological function. Proc. Natl. Acad. Sci. U.S.A. 107, 4544-4549 (2010).
- Warren, C. L., et al. Defining the sequence-recognition profile of DNA-binding molecules. Proc. Natl. Acad. Sci. U.S.A. 103, 867-872 (2006).
- Tietjen, J. R., Donato, L. J., Bhimisaria, D., Ansari, A. Z., Voigt, C. Chapter One - Sequence-Specificity and Energy Landscapes of DNA-Binding Molecules. Methods Enzymol. 497, 3-30 (2011).
- Puckett, J. W., et al. Quantitative microarray profiling of DNA-binding molecules. J. Am. Chem. Soc. 129, 12310-12319 (2007).
- Keles, S., Warren, C. L., Carlson, C. D., Ansari, A. Z. CSI-Tree: a regression tree approach for modeling binding properties of DNA-binding molecules based on cognate site identification (CSI) data. Nucleic Acids Res. 36, 3171-3184 (2008).
- Hauschild, K. E., Stover, J. S., Boger, D. L., Ansari, A. Z. CSI-FID: High throughput label-free detection of DNA binding molecules. Bioorg. Med. Chem. Lett. 19, 3779-3782 (2009).
- Lee, M., Roldan, M. C., Haskell, M. K., McAdam, S. R., Hartley, J. A. . In vitro Photoinduced Cytotoxicity and DNA Binding Properties of Psoralen and Coumarin Conjugates of Netropsin Analogs: DNA Sequence-Directed Alkylation and Cross-Link. 37, 1208-1213 (1994).
- Wurtz, N. R., Dervan, P. B. Sequence specific alkylation of DNA by hairpin pyrrole–imidazole polyamide conjugates. Chem. Biol. 7, 153-161 (2000).
- Tung, S. -. Y., Hong, J. -. Y., Walz, T., Moazed, D., Liou, G. -. G. Chromatin affinity-precipitation using a small metabolic molecule: its application to analysis of O-acetyl-ADP-ribose. Cell. Mol. Life Sci. 69, 641-650 (2012).
- Rodriguez, R., Miller, K. M. Unravelling the genomic targets of small molecules using high-throughput sequencing. Nat Rev Genet. 15, 783-796 (2014).
- Guan, L., Disney, M. D. Covalent Small-Molecule–RNA Complex Formation Enables Cellular Profiling of Small-Molecule–RNA Interactions. Angew. Chem. Int. Ed. 52, 10010-10013 (2013).
- White, J. D., et al. Picazoplatin, an Azide-Containing Platinum(II) Derivative for Target Analysis by Click Chemistry. J. Am. Chem. Soc. 135, 11680-11683 (2013).
- Rodriguez, R., et al. Small-molecule–induced DNA damage identifies alternative DNA structures in human genes. Nat Chem Biol. 8, 301-310 (2012).
- Bando, T., Sugiyama, H. Synthesis and Biological Properties of Sequence-Specific DNA-Alkylating Pyrrole−Imidazole Polyamides. Acc. Chem. Res. 39, 935-944 (2006).
- Anders, L., et al. Genome-wide localization of small molecules. Nat. Biotechnol. 32, 92-96 (2014).
- Jin, C., et al. Chem-seq permits identification of genomic targets of drugs against androgen receptor regulation selected by functional phenotypic screens. Proc. Natl. Acad. Sci. U.S.A. 111, 9235-9240 (2014).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
- Erwin, G. S., Bhimsaria, D., Eguchi, A., Ansari, A. Z. Mapping Polyamide–DNA Interactions in Human Cells Reveals a New Design Strategy for Effective Targeting of Genomic Sites. Angew. Chem. Int. Ed. 53, 10124-10128 (2014).
- Hyde, J. E., Hearst, J. E. Binding of psoralen derivatives to DNA and chromatin: influence of the ionic environment on dark binding and photoreactivity. Biochemistry. 17, 1251-1257 (1978).
- Erwin, G. S., Bhimsaria, D., Rodríguez-Martínez, J. A., Grieshop, M. P., Ansari, A. Z. Genome-wide localization of polyamide-based genome readers reveals sequence-based binding to repressive heterochromatin. In preparation. , (2015).
- Chen, G., et al. Chemically defined conditions for human iPSC derivation and culture. Nat Meth. 8, 424-429 (2011).
- Deliard, S., Zhao, J., Xia, Q., Grant, S. F. A. Generation of High Quality Chromatin Immunoprecipitation DNA Template for High-throughput Sequencing (ChIP-seq). J Vis Exp. (74), e50286 (2013).
- Shi, Y. B., Spielmann, H. P., Hearst, J. E. Base-catalyzed reversal of a psoralen-DNA cross-link. Biochemistry. 27, 5174-5178 (1988).
- Kumaresan, K. R., Hang, B., Lambert, M. W. Human Endonucleolytic Incision of DNA 3′ and 5′ to a Site-directed Psoralen Monoadduct and Interstrand. J. Biol. Chem. 270, 30709-30716 (1995).
- Cimino, G. D., Shi, Y. B., Hearst, J. E. Wavelength dependence for the photoreversal of a psoralen-DNA crosslink. Biochemistry. 25, 3013-3020 (1986).
- Heinz, S., et al. Simple Combinations of Lineage-Determining Transcription Factors Prime cis-Regulatory Elements Required for Macrophage and B Cell Identities. Mol. Cell. 38, 576-589 (2010).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biol. 9, R137 (2008).
- Kharchenko, P. V., Tolstorukov, M. Y., Park, P. J. Design and analysis of ChIP-seq experiments for DNA-binding proteins. Nat Biotech. 26, 1351-1359 (2008).
- Diamandis, E. P., Christopoulos, T. K. The biotin-(strept)avidin system: principles and applications in biotechnology. Clin. Chem. 37, 625-636 (1991).
- Martinson, H. G., True, R. J. On the mechanism of nucleosome unfolding. Biochemistry. 18, 1089-1094 (1979).
- Gloss, L. M., Placek, B. J. The Effect of Salts on the Stability of the H2A−H2B Histone Dimer. Biochemistry. 41, 14951-14959 (2002).
- Jackson, V. Formaldehyde Cross-Linking for Studying Nucleosomal Dynamics. Methods. 17, 125-139 (1999).
- Kasinathan, S., Orsi, G. A., Zentner, G. E., Ahmad, K., Henikoff, S. High-resolution mapping of transcription factor binding sites on native chromatin. Nat Meth. 11, 203-209 (2014).
- Teytelman, L., Thurtle, D. M., Rine, J., van Oudenaarden, A. Highly expressed loci are vulnerable to misleading ChIP localization of multiple unrelated proteins. Proc. Natl. Acad. Sci. U.S.A. 110, 18602-18607 (2013).
- . Phantompeakqualtools home page Available from: https://www.encodeproject.org/software/phantompeakqualtools/ (2010)
- Wang, D., Lippard, S. J. Cellular processing of platinum anticancer drugs. Nature Reviews Drug Discovery. 4, 307-320 (2005).
- Hurley, L. H. DNA and its associated processes as targets for cancer therapy. Nat. Rev. Cancer. 2, 188-200 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados