Method Article
La secuenciación profunda imparcial de virus de ARN de muestras clínicas
En este artículo
Resumen
This protocol describes a rapid and broadly applicable method for unbiased RNA-sequencing of viral samples from human clinical isolates.
Resumen
Aquí describimos un protocolo de secuenciación de ARN de última generación que permite a los conjuntos de novo y variantes llamadas intra-anfitriones de los genomas virales obtenidos de fuentes clínicas y biológicas. El método es imparcial y universal; que utiliza cebadores aleatorios para la síntesis de cDNA y no requiere conocimiento previo de la contenido de la secuencia viral. Antes de la construcción de bibliotecas, selectiva digestión a base de RNasa H se utiliza para agotar no deseada de ARN - vehículo que incluye poli (rA) y el ARN ribosomal - a partir de la muestra de ARN viral. depleción selectiva mejora tanto la calidad de los datos y el número de única lee en las bibliotecas de secuenciación del ARN viral. Por otra parte, el paso de una a base de transposasa 'tagmentation' se utiliza en el protocolo, ya que reduce el tiempo total de construcción de la biblioteca. El protocolo ha permitido profunda secuenciación rápida de más de 600 muestras, incluyendo el virus de Lassa y Ebola colecciones de sangre y tejido aislados y es ampliamente aplicable a otros estudios de genómica microbiana.
Introducción
secuenciación de próxima generación de virus a partir de fuentes clínicas puede informar a la transmisión y la epidemiología de infecciones, así como el apoyo ayuda novela de diagnóstico, y desarrollo de vacunas terapéuticas. la síntesis de ADNc usando cebadores aleatorios ha permitido la detección y el conjunto de los genomas de divergente, co-infectante o incluso nuevos virus 1,2. Al igual que con otros métodos imparciales, los contaminantes no deseados ocupan muchos secuenciación lee y un impacto negativo en los resultados de secuenciación. Anfitrión y poli (rA) ARN transportador son contaminantes presentes en muchas colecciones de muestras virales existentes.
El protocolo describe una forma eficiente y rentable de los genomas de virus de ARN de la secuencia de profundidad sobre la base de imparcial RNA-seq total. El método utiliza una RNasa H paso depleción selectiva 3 para eliminar ribosomal de acogida no deseada y el ARN portador. Depleción selectiva enriquece para el contenido viral (Figura 1) y mejora la calidad general de los datos de secuenciación(Figura 2) a partir de muestras clínicas. Por otra parte, tagmentation se aplica al protocolo, ya que reduce significativamente el tiempo de construcción de la biblioteca. Estos métodos se han utilizado para generar rápidamente grandes conjuntos de datos de Ébola y Lassa genomas de virus 2,4,5 y se pueden utilizar para estudiar una amplia gama de virus de ARN. Por último, el enfoque no se limita a las muestras humanas; la utilidad de la depleción selectiva se demostró en muestras de tejido recogidas de roedores infectados por Lassa y modelos de enfermedad de primates no humanos 5,6.

Figura 1. El ARN total contenido refleja Enriquecimiento del contenido de Lassa virus mediante depleción selectiva. A partir contenido global (entrada de ARN) y el enriquecimiento del virus de Lassa única (LASV) lee (contenido de la Biblioteca) en el agotamiento del ARNr a partir de nueve aislados clínicos diferentes. Esta cifra ha sido modificado a partir de 6 . Haga clic aquí para ver una versión más grande de esta figura.
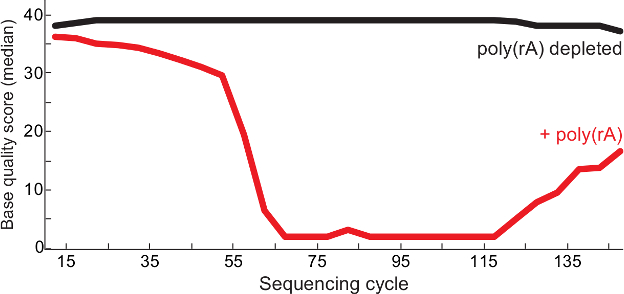
Figura 2. Calidad Superior Secuenciación Después Carrier RNA agotamiento. Calidades de base mediana de secuenciación por ciclo de poli (rA) contaminadas con el virus de Lassa bibliotecas (rojo) y el control (sin soporte observado en la biblioteca, negro) del informe de control de calidad 13. Ambos leen 1 y 2 de leer extremo emparejado lecturas se fusionan en el archivo de BAM biblioteca y las puntuaciones de calidad se muestran en cada base. Esta cifra ha sido modificado a partir de 6. Por favor, haga clic aquí para ver una versión más grande de esta figura.
El ARN viral siguientes detalles del protocolo de construcción de bibliotecas directamente desde extraídaARN recogido de muestras clínicas y biológicas. Para garantizar la seguridad personal, todas las muestras de suero, plasma y tejidos virales deben ser inactivados en tampones apropiados antes de la extracción de RNA. En algunos kits de inactivación y de extracción, se incluye poli portador (rA) RNA; este será eliminado durante la etapa inicial de agotamiento selectivo de RNasa H. Sobre la base de la recuperación completa, la concentración esperada de ARN portador es 100 ng / l. En el protocolo, 110 ng / l oligo dT ARN (1,1x concentración de portadores) se utiliza para el agotamiento. Si poli (rA) portadora no está presente en la muestra, a continuación, oligo (dT) no se debe añadir antes de agotamiento.
El siguiente protocolo está diseñado para 24 reacciones en formato de placa de PCR (hasta 250 l de volumen). Una versión anterior de este protocolo se informó en Matranga, et al. 6.
Protocolo
declaración de la ética: los pacientes con fiebre de Lassa fueron reclutados para este estudio utilizando los protocolos aprobados por los comités de los sujetos humanos en la Universidad de Tulane, Universidad de Harvard, Instituto Broad, Irrua Especialista Hospital Universitario (ISTH), Hospital del Gobierno de Kenema (KGH), Oyo Ministerio de Salud, Ibadan Estado , Nigeria y Sierra Leona Ministerio de Salud. Todos los pacientes fueron tratados con un nivel similar de la atención y se les ofreció el fármaco ribavirina, ya sea o no que decidieron participar en el estudio. Para los pacientes con fiebre de Lassa (LF), el tratamiento con ribavirina a las directrices recomendadas actualmente y en general fue ofrecido tan pronto como LF se sospecha fuertemente.
Debido a la grave brote de virus del Ébola de Enfermedades (EVD), pacientes no pudieron ser consintieron a través de nuestros protocolos estándar. En cambio el uso de muestras clínicas de pacientes exceso EVD fue evaluado y aprobado por las Juntas de Revisión Institucional en Sierra Leona y en la Universidad de Harvard. el OffiCE de la Comisión Sierra Leona Ética y Revisión Científica, el ministerio de Sierra Leona de Salud y Saneamiento, y el Comité de Harvard sobre el Uso de Sujetos Humanos han concedido una exención del consentimiento para secuenciar y hacer secuencias virales públicamente disponibles obtenidos a partir de muestras de pacientes y datos de contacto recogida durante el brote de Ébola en Sierra Leona. Estos organismos también concedió el uso de datos clínicos y epidemiológicos para muestras sin identificación recogidos de todos los pacientes que reciben atención EVD sospechosos durante la respuesta a los brotes. La Leona Ministerio de Salud y Sanidad de Sierra también aprobó los envíos de no infecciosas, las muestras no biológicas desde Sierra Leona a Broad Institute y la Universidad de Harvard para estudios genómicos de muestras de brotes.
1.-DNasa tratamiento de ARN de la muestra (hasta 55 l extraídos ARN total, ~ 4 h)
- Configurar la reacción de DNasa en una placa de 96 pocillos PCR en hielo en una cabina de bioseguridad como se describe en la Tabla 1 , Etapa 1.1 (volumen total, 70 l / pocillo). Nota: Un maestro de la mezcla puede ser preparada.
- Vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min.
- Incubar a 37 ° C durante 30 min.
- La limpieza usando perlas de ARN en Fase sólida reversible Inmovilización (SPRI).
- perlas de ARN calentar a ta durante 30 min.
- Agitar suavemente la botella de perlas de ARN para volver a suspender las partículas magnéticas que se haya asentado. Añadir un volumen 1,8 veces (126 l) de perlas de ARN a ARN tratado con DNasa (70 l), mezclar con una pipeta de 10 veces y se incuba durante 5 minutos a temperatura ambiente (volumen total en el pozo, 196 l).
- Coloque la mezcla en la estación magnético. Espere a que la solución para limpiar (de 5 - 10 min).
- Eliminar la solución de aclarado, mientras que en la estación mediante una pipeta y el descarte. Mientras que en la estación, lavar los granos cubriendo pellet con etanol al 70% y se incuba durante 1 min. Eliminar el etanol con la pipeta y descarte. Repita para un total de dos lavados.
Nota: El uso y precisión del 70% recién preparadaHanol es crítico, ya que un porcentaje más alto dará como resultado en el lavado ineficiente de moléculas de menor tamaño, mientras que <70% de etanol podría causar la pérdida de la muestra 7. - Mantenga la placa en la estación y dejar abierta secar al aire. Nota: Asegúrese de permitir que las perlas se sequen por completo hasta que los granos comienzan a agrietarse.
- Añadir 55 l de agua libre de nucleasa a la placa para eluir el ARN. Retire la placa de la estación de mezclar los granos y el agua al vaso a fondo. Nota: Como alternativa, utilizar menos agua (≤ 10 l) con el fin de concentrar el ARN total.
- Coloque la placa posterior de la estación. Espere hasta que la solución limpia para transferir mediante una pipeta a un nuevo tubo con tapón de rosca para el almacenamiento a largo plazo (-80 ° C). Coloca 5 l de ARN en una nueva placa de 96 pocillos de PCR para el agotamiento (. El paso 2.4).
- Opcional:. Guardar y diluir 1 l en 19 l de agua (1:20) para QRT-PCR de rRNA (por ejemplo, 18S, 28S rRNA) (Tabla 2) y los marcadores virales 5
2. El agotamiento selectivo de ARN ribosomal y Carrier de ARN viral de la muestra (~ 4 h)
- Hacer hibridación 5x y 10x tampones de reacción de RNasa H, y agua libre de nucleasa con el portador de acrilamida lineal como se describe en la Tabla 1.
- Configurar reacción de hibridación mediante la combinación de ARN con oligos de agotamiento de rRNA (Tabla 3) y oligo (dT) en hielo en una placa de 96 pocillos de PCR como se describe en la Tabla 1.
Nota: Un maestro de la mezcla puede ser preparada. 50 femtogramos (fg) de un ARN sintético único (ERCCs 8) se pueden agregar para el seguimiento tanto del proceso de secuenciación viral y potencial leer índice de contaminación cruzada.- Vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min.
Incubar a 95 ° C durante 2 min, rampa lenta a 45 ° C a -0,1 ° C por segundo. Una pausa en el termociclador a 45 ° C.
- Vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min.
- Configurar la RNasa H de la mezcla de reacción en hielo como se ha descritod en la Tabla 1, a continuación, precalentar a 45 ° C durante 2 min. Nota: Un maestro de la mezcla puede ser preparada.
- Añadir la mezcla de la RNasa H precalentado a la reacción de hibridación en placa, manteniendo la placa en el termociclador a 45 ° C.
- Mezclar bien pipeteando suavemente 6 - 8 veces. Incubar a 45 ° C durante otros 30 min. Colocar sobre hielo.
- Configurar la mezcla de reacción en hielo DNasa tal como se describe en la Tabla 1 Nota:. Un maestro de la mezcla se puede preparar.
- Añadir a la reacción de la RNasa H en la placa, vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min. Incubar a 37 ° C durante 30 min.
- Detener la reacción mediante la adición de DNasa 5 l 0,5 M EDTA. Vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min.
- La limpieza usando perlas de ARN (véase el paso 1.3) utilizando un volumen de 1,8 veces (144 l) perlas. Eluir en 11 l de agua libre de nucleasa. Nota: Para el almacenamiento en frío seguro, almacenar agota RNA las muestras a -80 ° C O / N.
3. Síntesis de ADNc (~ 6 hr)
- Mix rRNA / ARN carrier-empobrecido con cebadores aleatorios en hielo en una placa de 96 pocillos de PCR como se describe en la Tabla 1, vórtice suave y completamente, a continuación, se centrifuga a 280 xg a TA durante 1 min.
- Se calienta la mezcla a 70 ° C durante 10 minutos en un termociclador. Inmediatamente después de desnaturalización por calor, coloque el ARN en hielo durante 1 - 5 min. No permita que el ARN a pie (incluso en hielo) durante más de 5 minutos antes de la primera cadena de reacción.
- Configurar el primer capítulo de la síntesis de la mezcla de reacción en hielo como se describe en la Tabla 1.
Nota: Una mezcla maestra puede ser preparado.- Agregar a la mezcla de cebadores de ARN / azar en la placa, vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min. Se incuba a 22-25 ° C durante 10 min.
- Incubar a 55 ° C en una incubadora de aire para 60 min. Colocar la placa en el hielo para terminar la reacción. Noe: El uso de una incubadora de aire se recomienda para crear el calentamiento gradual de la primera hebra de reacción durante el cual los cebadores de recocido y la primera hebra comienza a alargarse.
- Configurar la segunda cadena de la mezcla de reacción de síntesis sobre hielo como se describe en la Tabla 1.
Nota: Una mezcla maestra puede ser preparado.- Añadir a la primera cadena de reacción de síntesis de la placa, vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min. Incubar durante 2 horas a 16 ° C (mantener la tapa a 25 ° C). No permita que la temperatura se eleve por encima de 16 ° C.
- Colocar la placa en el hielo, entonces inactivar la reacción mediante la adición de 5 l de EDTA 0,5 M, mezclar suavemente y en profundidad, centrifugar a 280 xg a temperatura ambiente durante 1 min.
- De limpieza con los granos de ADN (véase el paso 1.3 para protocolo) utilizando el volumen de 1.8x (153 l) de perlas. Eluir en 9 l de tampón de elución (EB). Guardar 1 l para su cuantificación. Utilice 1 ng de cDNA para subsequepasos NT. Si la concentración de cDNA es demasiado baja para detectar, utilizar 4 l de cDNA para tagmentation (véase el paso 4.1).
- Para el almacenamiento en frío seguro, almacenar doble hebra de ADNc a 4 ° CO / N o -20 ° C para el almacenamiento a largo plazo.
4. Preparación Biblioteca - ADN de construcción de biblioteca (~ 4 h)
- Transferencia de 4 l de cDNA a una placa de 96 pocillos y guardar el cDNA restante para un segundo intento si es necesario.
- Configurar la reacción tagmentation en hielo como se describe en la Tabla 1.
Nota: Una mezcla maestra puede ser preparado. Para reducir el fondo y el coste total, el volumen total de la reacción tagmentation se reduce de 20 a 10 l. Como cDNA es el factor limitante, la cantidad de ATM (es decir, transposome) utilizado en la reacción también se reduce para disminuir el número de sitios de integración.- Añadir la mezcla tagmentation de cDNA en la placa, vórtice suave y completamente y se centrifuga a 280 xg (a temperatura ambiente) durante 1 min.Se incuba a 55 ° C durante 5 min, mantenido a 10 ° C.
- Una vez a 10 ° C, añadir 2,5 l de inmediato Neutralizar Tagment Buffer (NT) para terminar la reacción. Mezclar pipeteando arriba y abajo y centrifugar a 280 xg (a temperatura ambiente) durante 1 min.
- Incubar a temperatura ambiente durante 5 min.
- Configurar reacción de amplificación PCR en hielo como se describe en la Tabla 1.
- Vórtice suave y profundamente, centrifugar a 280 xg a temperatura ambiente durante 1 min.
- Realizar PCR en termociclador usando las condiciones descritas en la Tabla 1.
Nota: 12 ciclos de PCR se sugieren para 1 ng de cDNA tagmented; sin embargo, muestras clínicas virales a menudo tienen cantidades indetectables de cDNA. Para bajas cantidades de cDNA (<1 ng), utilizar hasta 18 ciclos de PCR para crear suficiente biblioteca para la secuenciación.
- Biblioteca preparación - la limpieza y la puesta en común para la secuenciación
- Llevar la muestra hasta 50 l con EB.
- limpieza con los mediosperlas de ADN (véase el paso 1.3 para protocolo) utilizando el volumen de 0.6x (30 l) perlas. Eluir en 15 l EB.
- Determinar la concentración de la biblioteca (Figura 3) mediante la realización de análisis de región (150 a 1000 pb) utilizando software Bioanalyzer 9, con exclusión de los dímeros de cebadores (~ 120 pb) a partir del análisis región. Nota: Como alternativa, qPCR se puede utilizar para cuantificar las bibliotecas 10.
- Las bibliotecas de la piscina en la concentración molar más baja de 1 nm o mayores. Si la biblioteca es inferior a 1 nM, añadir un pequeño volumen de la biblioteca a la piscina (~ 1 x volumen de otras bibliotecas) para capturar información de la secuencia de estas bibliotecas.
- piscina de limpieza con los granos de ADN 0.7x como se indica más arriba (véase el paso 2). Eluir en 15 l EB. Nota: El volumen de los granos dependerá del volumen final de la piscina.
- Analizar la piscina 9. Determinar la concentración molar mediante la realización de análisis de región (150 a 1000 pb) 9. Nota: Como alternativa, qPCR puede ser utilizado para cuantificar la piscina biblioteca 10 </ Sup>.
- Cargar secuenciador a las 10 pm concentración biblioteca para generar 101 pb, de extremo emparejado con doble lee el código de barras lee 11.
Figura 3. bibliotecas construidas a partir de virus Ébola muestras clínicas. Gel de imagen de las bibliotecas 4 representativa del virus del Ébola (EBOV). Se muestran las regiones de biblioteca y de cebadores dímeros. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Resultados
El protocolo descrito permite la generación de secuenciación de alta calidad se lee a partir de muestras de ARN viral de bajos insumos, mientras que para el enriquecimiento de contenido viral única. Como se muestra en la Figura 1, el protocolo enriquecido contenido único virus Lassa al menos cinco veces en todas las muestras (en comparación con los controles no agotado) con al menos un millón de copias de 18S rRNA (~ 100 pg de ARN total). Del mismo modo, el éxito de secuenciación también se correlacionó con la cantidad de virus en una muestra dada. Uso de QRT-PCR como un sustituto para la cantidad viral, las muestras que contenían ~ 1.000 o más copias del genoma viral con más frecuencia creados conjuntos completos (datos no mostrados). Además, el agotamiento de poli portador (rA) reduce secuencias de homopolímero de A y T en las bibliotecas, lo que resulta en las preparaciones más limpias y garantizar una mejor calidad de secuenciación lee (Figura 2). bibliotecas finales a partir de muestras clínicas viral baja de entrada a menudo tienen una amplia longitud de los fragmentos de 150 a 1.000 pb(Figura 3).
Después de la secuenciación, para reducir los errores de identificación de la muestra y la diafonía entre las bibliotecas dentro de una piscina de 12, sólo el índice lee con una puntuación de calidad de base de 25 (q25) y asegúrese de cero desajustes se mantienen durante el proceso de demultiplexación. Los genomas virales se ensamblan usando una tubería de bioinformática específico para los virus divergentes 2,4-6. Estas herramientas están disponibles en https://github.com/broadinstitute/viral-ngs oa través de plataformas en la nube comerciales 4.
| Paso 1.1: Reacción DNasa | |
| Reactivo | Volumen por reacción (l) |
| 10x tampón ADNasa | 7 |
| agua libre de nucleasa | 6 |
| ARN viral extraído | 55 |
| DNasa (2 U / & #181; l) | 2 |
| Volumen total | 70 |
| Paso 2.1: 5x tampón de hibridación | |
| Reactivo | Volumen de 1 ml (l) |
| 5 M NaCl | 200 |
| 1 M Tris-HCl (pH 7,4) | 500 |
| agua libre de nucleasa | 300 |
| Volumen total | 1000 |
| Paso 2.1: 10x tampón de reacción de RNasa H | |
| Reactivo | Volumen de 1 ml (l) |
| 5 M NaCl | 200 |
| 1 M Tris-HCl (pH 7,5) | 500 |
| 1 M MgCl 2 | 200 |
| agua libre de nucleasa | 500 |
| Volumen total | 1000 |
| Paso 2.1: El agua wacrilamida lineal ITH | |
| Reactivo | Volumen de 1 ml de tampón (l) |
| agua libre de nucleasa | 992 |
| acrilamida lineal (5 mg / ml) | 8 |
| Volumen total | 1000 |
| Paso 2.2: reacción de hibridación para el agotamiento selectivo | |
| Reactivo | Volumen por reacción (l) |
| 5x Tampón de Hibridación | 2 |
| rRNA-agotamiento mezcla oligo (100 M) | 1.22 |
| Oligo (d) T (550 ng / l) | 1 |
| Tratado con DNasa ARN total | hasta 5 |
| Pico-a ARN (Esto es opcional) | 0,5 |
| Agua (con acrilamida lineal) | llevar hasta 10 totales |
| volu totalyo | 10 |
| Paso 2.3: RNasa H de reacción para el agotamiento selectivo | |
| Reactivo | Volumen por reacción (l) |
| 10x RNasa H Reaction Buffer | 2 |
| Agua (con acrilamida lineal) | 5 |
| Termoestable RNasa H (5 U / l) | 3 |
| Volumen total | 10 |
| Paso 2.4: DNasa posterior reacción depleción selectiva | |
| Reactivo | Volumen por reacción (l) |
| 10x tampón de ADNasa | 7.5 |
| Agua (con acrilamida lineal) | 44.5 |
| inhibidor de RNasa (20 U / l) | 1 |
| DNasa libre de RNasa I (2,72 U / l) | 2 |
| Volumen total (con la reacción de RNasa H) | 75 |
| Paso 3.1: síntesis de ADNc, la hibridación del cebador aleatorio | |
| Reactivo | Volumen por reacción (l) |
| rRNA / ARN portador-agotado | 10 |
| 3 g de cebador al azar | 1 |
| Volumen total | 11 |
| Paso 3.2: La primera cadena de reacción de síntesis de ADNc | |
| Reactivo | Volumen (l) |
| 5x First-Strand Buffer de Reacción | 4 |
| DTT 0,1 M | 2 |
| mezcla de dNTP 10 mM | 1 |
| inhibidor de RNasa (20 U / l) | 1 |
| La transcriptasa inversa (añadir última) | 1 |
| Volumen total (con ARN arriba) | 20 |
| Paso 3.3: de la segunda cadena de reacción de síntesis de ADNc | |
| Reactivo | Volumen (l) |
| agua libre de RNasa | 43 |
| 10x tampón de reacción de la segunda Strand | 8 |
| mezcla de dNTP 10 mM | 3 |
| E. coli ADN ligasa (10 U / l) | 1 |
| ADN de E. coli polimerasa I (10 U / l) | 4 |
| E. coli RNasa H (2 U / l) | 1 |
| Volumen total (con 1 pt reacción de cadena) | 80 |
| Paso 4.2: Reacción Tagmentation | |
| Reactivo | Volumen (l) |
| Amplicón Tagment Mix (ATM) | 1 |
| Tagment ADN Buffer (TD) | 5 |
| Volumen total (con cDNA) | 10 |
| Paso 4.3: Biblioteca de reacción de PCR | |
| Reactivo | Volumen (l) |
| PCR Master Mix (NPM) | 7.5 |
| Índice 1 imprimación (i7) | 2.5 |
| Índice 2 imprimación (i5) | 2.5 |
| Volumen total (con tagmented cDNA) | 25 |
| Paso 4.3.2: Las condiciones de PCR Biblioteca | |
| 72 ° C, 3 min | |
| 95 ° C, 30 seg | |
| hasta 18 ciclos de 10 segundos a 95 ° C, 30 seg a 55 ° C, 30 seg a 72 ° C | |
| 72 ° C, 5 min | |
| 10 ° C, para siempre | |
Tabla 1:. Reacción puesta a punto y tampones paso a paso tablas con contenido de todos los tampones y las mezclas de reacción.
Tabla 2: QRT-PCR Primers secuencias de los cebadores utilizados para la medición de acogida (18S rRNA) y virales contenidos (Ébola y Lassa).. 'KGH' es el hospital público de Kenema en Sierra Leona, donde los cebadores de Ébola se probaron 2. 'Kulesh' es el investigador que diseñó el conjunto de cebadores 14.
Tabla 3: ARN ribosomal (ARNr) Agotamiento Oligos 195 largas secuencias de 50 nucleótidos complementarios al ARNr humana para el paso depleción selectiva 6.. Por favor, haga clic aquí para descargar este archivo.
| Nombre oligo | Secuencia (5 'a 3') |
| Ébola KGH FW | GTCGTTCCAACAATCGAGCG |
| Ébola KGH RV | CGTCCCGTAGCTTTRGCCAT |
| Ébola kulesh FW | TCTGACATGGATTACCACAAGATC |
| Ébola kulesh RV | GGATGACTCTTTGCCGAACAATC |
| Lassa SL FW | GTA AGC CCA GCD GYA AAB CC |
| Lassa SL RV | AAG CCA CAG AAA ECA SGG AGC A |
| 18S rRNA FW | TCCTTTAACGAGGATCCATTGG |
| 18S rRNA RV | CGAGCTTTTTAACTGCAGCAACT |
Discusión
El enfoque descrito permite la secuenciación robusto, universal, rápido y fue utilizado para secuenciar el virus del Ébola durante el brote de 2014 2,4. Mediante el acoplamiento de depleción selectiva y la síntesis de ADNc con la construcción de la biblioteca tagmentation, el tiempo de proceso total se redujo en un ~ 2 días a partir de métodos adaptador ligadura anteriores. Más recientemente, este protocolo se empleó por colaboradores internacionales y otros con gran éxito 15,16 y será enviado a los laboratorios en África Occidental para apoyar los estudios de investigación basada en la genómica y diagnóstico local 17.
El protocolo descrito aquí utiliza cebadores aleatorios para preparar cDNA para las bibliotecas de ARN-seq virales. A diferencia de los enfoques de ARN-seq virales anteriores, que no requiere un conocimiento a priori de los datos de secuencia o el diseño de cebadores elaborada y requiere mucho tiempo de un virus o clado específico. El método puede aplicarse a cualquier muestra de ARN viral. Por ejemplo, se utiliza para generar el contenido viral de ambos Ebolay muestras de Lassa 6. El protocolo también se puede usar para transcriptómica anfitrión, proyectos de secuenciación metagenomic y descubrimiento patógeno 1.
Un paso crítico del protocolo está dirigido digestión RNasa H, un alto rendimiento, método de bajo coste para la eliminación de portadora no deseada y el ARN huésped a partir de muestras virales. La etapa de depleción selectiva del protocolo utiliza muchos componentes y requiere habilidad y precisión. tiempo extra y se debe tener cuidado durante la configuración inicial.
Como la mayoría de las muestras de suero y plasma clínicos a menudo tienen muy poco material de ácido nucleico, la contaminación y la pérdida de la muestra son comunes. Para evitar estos problemas, especial se debe tener cuidado al usar este protocolo. En primer lugar, el ARN es altamente susceptible a la degradación; Por lo tanto, todas las áreas deben estar limpias y libres de nucleasas. En segundo lugar, para identificar las muestras adecuadas para su uso en este protocolo, los ensayos de QRT-PCR para ambos ARN del virus de acogida y deben utilizarse para la cuantificación 5,6 . Cuando se compara con la entrada asciende resultados de la secuenciación del protocolo, el éxito de secuenciación (es decir, la generación de datos suficientes para el ensamblaje viral completo) se correlacionaron con las muestras que contenían al menos 100 pg de ARN total y 1.000 copias del virus. En tercer lugar, la exposición a las fuentes ambientales de los ácidos nucleicos debe ser evitado. El protocolo descrito aquí está hecho en una cabina de bioseguridad como medida de seguridad y para limitar los contaminantes ambientales. Por otra parte, nuestro grupo y otros han dado cuenta de que las enzimas comerciales pueden ser otra fuente de ácidos nucleicos contaminantes bacterianos en muestras de entrada bajas 6,18. El uso de un espacio de trabajo limpio (por ejemplo, PCR capó, cabina de bioseguridad) y controles negativos (por ejemplo, agua o tampón) ayudarán a aliviar y realizar un seguimiento de la contaminación, respectivamente. Para muestras con <100 pg de ARN total, sólo el poli (rA) ARN portador, no rRNA, deberá ser agotado para garantizar resultados de la secuenciación de alta calidad al tiempo que limita la pérdida de material. por muymuestras de entrada bajas, los métodos de amplificación de cDNA pueden ser más adecuados 19, a pesar de poli (rA) portador debe ser eliminado antes de la síntesis de ADNc.
El agotamiento de anfitrión rRNA enriquece para el contenido viral en las bibliotecas de secuenciación y es aplicable a diferentes colecciones de muestras incluyendo suero o plasma, y múltiples tipos de tejidos de roedores y primates no humanos 5,6. En los organismos no humanos, las lecturas alinear a 28S rRNA se mantuvo después de la depleción, lo que sugiere 28S rRNA es menos conservadas entre los seres humanos y otras especies de 6,20. Al utilizar este método con los aislados no humanos, puede ser necesario complementar con oligos de ADN complementarias a las secuencias de rRNA divergentes del 3,21 host específico.
Dado que el protocolo es imparcial, viral lee puede representar sólo una pequeña fracción del contenido total de biblioteca. Aunque rRNA es la especie más abundante de ARN de acogida y sólo un pequeño porcentaje de ARNr (lee0; 1%) se encuentran después de la depleción selectiva, el resto de ARN huésped (por ejemplo, ARNm) se mantendrá después de agotamiento y puede dar cuenta de que muchos de secuenciación lee de la muestra. Por lo tanto "sobremuestreo" (es decir, oversequencing) bibliotecas individuales se requiere con el fin de tener suficiente cobertura para montaje y variantes virales llamadas. Para nuestros estudios, se intenta secuencia de ~ 20 millones de lecturas por muestra para tener suficiente profundidad para el análisis de variantes genómicas y asociadas a virus, así como el contenido de metagenómica 2,5. Para los estudios de descubrimiento metagenomic y patógenos, es importante tener en cuenta que la contaminación de ADN huésped se retira por digestión con DNasa. Por lo tanto virus y otros patógenos que contienen genomas de ADN se pueden perder durante el proceso, sin embargo intermedios de ARN todavía pueden ser secuenciados.
Divulgaciones
The authors have no competing financial interests.
Agradecimientos
This work has been funded in part with Federal funds from the National Institutes of Health, Office of Director, Innovator (No.: DP2OD06514) (PCS) and from the National Institute of Allergy and Infectious Diseases, National Institutes of Health, Department of Health and Human Services, under Contracts (No:HHSN272200900018C, HHSN272200900049C and U19AI110818).
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well PCR Plates | VWR | 47743-953 | |
| Strips of Eight Caps | VWR | 47745-512 | |
| Nuclease-free water | Ambion | AM9937 | 50 ml bottle |
| TURBO DNase | Ambion | AM2238 | post RNA extraction step, 2 U/µl, buffer included |
| PCR cycler | any PCR cyclers | ||
| Agencourt RNAClean XP SPRI beads | Beckman Coulter Genomics | A63987 | beads for RNA cleanup |
| Real Time qPCR system | any system | ||
| DynaMag-96 Side Skirted Magnet | Invitrogen | 12027 | |
| 70% Ethanol | prepare fresh | ||
| qRT-PCR primers | IDT DNA | see Table 2 | |
| 5 M NaCl | Ambion | AM9760G | |
| 1 M Tris-HCl pH 7.4 | Sigma | T2663-1L | |
| 1 M Tris-HCl pH 7.5 | Invitrogen | 15567-027 | |
| 1 M MgCl2 | Ambion | AM9530G | |
| Linear acrylamide | Ambion | AM9520 | |
| DNA oligos covering entire rRNA region | IDT DNA | see Table 3, order lab-ready at 100 µM | |
| Oligo (dT) | IDT DNA | 40 nt long, desalted | |
| Hybridase Thermostable RNase H | Epicentre | H39100 | |
| RNase-free DNase Kit | Qiagen | 79254 | post selective depletion step |
| SUPERase-In RNase Inhibitor | Ambion | AM2694 | |
| Random Primers | Invitrogen | 48190-011 | mostly hexamers |
| 10 mM dNTP mix | New England Biolabs | N0447L | |
| SuperScript III Reverse Transcriptase | Invitrogen | 18080-093 | with first-strand buffer, DTT |
| Air Incubator | any air incubator cyclers | ||
| NEBNext Second Strand Synthesis (dNTP-free) Reaction Buffer | New England Biolabs | B6117S | 10x |
| E. coli DNA Ligase | New England Biolabs | M0205L | 10 U/μl |
| E. coli DNA Polymerase I | New England Biolabs | M0209L | 10 U/μl |
| E. coli RNase H | New England Biolabs | M0297L | 2 U/μl |
| 0.5 M EDTA | Ambion | AM9261 | |
| Agencourt AMPure XP SPRI beads | Beckman Coulter Genomics | A63881 | beads for DNA cleanup |
| Elution Buffer | Qiagen | 10 mM Tris HCl, pH 8.5 | |
| Quant-iT dsDNA HS Assay Kit | Invitrogen | Q32854 | |
| Qubit fluorometer | Invitrogen | Q32857 | |
| Nextera XT DNA Sample Prep Kit | Illumina | FC-131-1096 | |
| Nextera XT DNA Index Kit | Illumina | FC-131-1001 | |
| Tapestation 2200 | Agilent | G2965AA | |
| High Sensitivity D1000 reagents | Agilent | 5067-5585 | |
| High Sensitivity D1000 ScreenTape | Agilent | 5067-5584 | |
| BioAnalyzer 2100 | Agilent | G2939AA | |
| High Sensitivity DNA reagents | Agilent | 5067-4626 | |
| Library Quantification Complete kit (Universal) | Kapa Biosystems | KK4824 | alternative to tapestation, bioanalyzer for library quantification |
Referencias
- Stremlau, M. H., et al. Discovery of novel rhabdoviruses in the blood of healthy individuals from West Africa. PLoS Negl Trop Dis. 9, e0003631(2015).
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345, 1369-1372 (2014).
- Morlan, J. D., Qu, K., Sinicropi, D. V. Selective depletion of rRNA enables whole transcriptome profiling of archival fixed tissue. PLoS One. 7, e42882(2012).
- Park, D. J., et al. Ebola Virus Epidemiology, Transmission, and Evolution during Seven Months in Sierra Leone. Cell. 161, 1516-1526 (2015).
- Andersen, K. G., et al. Clinical Sequencing Uncovers Origins and Evolution of Lassa Virus. Cell. 162, 738-750 (2015).
- Matranga, C. B., et al. Enhanced methods for unbiased deep sequencing of Lassa and Ebola RNA viruses from clinical and biological samples. Genome Biol. 15, 519(2014).
- Tang, F., et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell. Nat Protoc. 5, 516-535 (2010).
- Jiang, L., et al. Synthetic spike-in standards for RNA-seq experiments. Genome Res. 21, 1543-1551 (2011).
- Agilennt Technologies. , Available from: http://www.agilent.com/cs/library/usermanuals/Public/G2946-90004_Vespucci_UG_eBook_(NoSecPack).pdf. (2015).
- Kapa Biosystems. , Available from: https://www.kapabiosystems.com/product-applications/products/next-generation-sequencing-2/library-quantification/ (2015).
- Illumina Technologies. , Available from: https://support.illumina.com/content/dam/illumina-support/documents/documentation/system_documentation/miseq/preparing-libraries-for-sequencing-on-miseq-15039740-d.pdf. (2015).
- Kircher, M., Sawyer, S., Meyer, M. Double indexing overcomes inaccuracies in multiplex sequencing on the Illumina platform. Nucleic Acids Res. 40, 3(2012).
- Andrews, S. Babraham Bioinformatics. , Available from: http://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2010).
- Trombley, A. R., et al. Comprehensive panel of real-time TaqMan polymerase chain reaction assays for detection and absolute quantification of filoviruses, arenaviruses, and New World hantaviruses. Am J Trop Med Hyg. 82, 954-960 (2010).
- Hu, Y., et al. Serial high-resolution analysis of blood virome and host cytokines expression profile of a patient with fatal H7N9 infection by massively parallel RNA sequencing. Clin Microbiol Infect. 21, e1-4 713(2015).
- Simon-Loriere, E., et al. Distinct lineages of Ebola virus in Guinea during the 2014 West African epidemic. Nature. 524, 102-104 (2015).
- Folarin, O. A., Happi, A. N., Happi, C. T. Empowering African genomics for infectious disease control. Genome Biol. 15, 515(2014).
- Blainey, P. C., Quake, S. R. Digital MDA for enumeration of total nucleic acid contamination. Nucleic Acids Res. 39, 19(2011).
- Malboeuf, C. M., et al. Complete viral RNA genome sequencing of ultra-low copy samples by sequence-independent amplification. Nucleic Acids Res. 41, 13(2013).
- Gonzalez, I. L., Sylvester, J. E., Smith, T. F., Stambolian, D., Schmickel, R. D. Ribosomal RNA gene sequences and hominoid phylogeny. Mol Biol Evol. 7, 203-219 (1990).
- Adiconis, X., et al. Comparative analysis of RNA sequencing methods for degraded or low-input samples. Nat Methods. 10, 623-629 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados