Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La definición de Sustrato especificidades para la lipasa y la fosfolipasa candidatos
En este artículo
Resumen
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
Resumen
Los microorganismos producen un amplio espectro de lipasas (fosfo) que son secretadas en el fin de hacer los sustratos externos disponibles para el organismo. Alternativamente, otros (fosfo) lipasas pueden estar asociadas físicamente con el organismo productor causando una facturación de lípidos intrínsecos y con frecuencia dando lugar a una remodelación de las membranas celulares. Aunque potenciales (fosfo) lipasas se pueden predecir con un número de algoritmos cuando la secuencia de gen / proteína está disponible, no ha sido obtenido con frecuencia prueba experimental de las actividades enzimáticas, especificidades de sustrato, y posibles funciones fisiológicas. Este manuscrito describe la optimización de las condiciones de ensayo de (fosfo) lipasas prospectivos con especificidades de sustrato desconocidos y cómo emplear estas condiciones optimizadas en la búsqueda para el sustrato natural de un respectivo lipasa (fosfo). El uso de sustratos cromogénicos artificiales, tales como derivados de p-nitrofenil, puede ayudar a detectar un menorla actividad enzimática de una lipasa predicho (fosfo) en condiciones estándar. Después de haber encontrado una actividad enzimática tal menor, los parámetros distintos de un ensayo enzimático se pueden variar con el fin de obtener una hidrólisis más eficiente del sustrato artificial. Después de haber determinado las condiciones en las que una enzima funciona bien, una variedad de posibles sustratos naturales deben ser analizadas para su degradación, un proceso que puede ser seguido empleando métodos cromatográficos diferentes. La definición de especificidades de sustrato de nuevas enzimas, a menudo proporciona hipótesis para un posible papel fisiológico de estos enzimas, que a continuación pueden ser probados experimentalmente. Siguiendo estas directrices, hemos sido capaces de identificar una fosfolipasa C (SMc00171) que degrada la fosfatidilcolina a fosfocolina y diacilglicerol, en un paso crucial para la remodelación de las membranas en la bacteria Sinorhizobium meliloti de las condiciones de fósforo limitante del crecimiento. Para dos predijo patatin-como fosfolipasas (SMc00930 y SMc01003) del mismo organismo, podríamos redefinir sus especificidades de sustrato y aclarar que SMc01003 es una lipasa diacilglicerol.
Introducción
Lípidos basados en glicerol tales como triglicéridos y fosfolípidos (glicero) constituyen importantes y probablemente las clases de lípidos más conocidos 1. Triglicéridos (TG) son grasas o aceites, que por lo general funcionan como lípidos de almacenamiento, y por lo tanto, como posibles fuentes de energía y de carbono. Las etiquetas pueden ser degradadas por las lipasas, que con frecuencia son secretadas por el organismo productor de digerir variables externas y ponerlos a disposición como fuentes de carbono. Además, las lipasas se han estudiado ampliamente en los últimos años debido a sus importantes aplicaciones biotecnológicas 2.
Debido a su naturaleza anfifílica y su forma casi cilíndrica, (glicero fosfolípidos) exhiben propiedades de formación de membrana y por lo general constituyen los principales componentes lipídicos de la membrana de dos capas 3. En los microorganismos simples, tales como la bacteria Escherichia coli, sólo tres variantes principales del grupo de cabeza, fosfatidilglicerol (PG), cardiolipina (CL), y phosphatidylethanolamine (PE) se encontró, aunque se debe tener en cuenta que cada uno de ellos puede estar sustituido con un número considerable de diferentes cadenas de acilo graso en la posición sn -1 o sn -2 posición que da lugar a un gran número de diferentes especies moleculares de 4 . Otras bacterias podrían tener otros fosfolípidos, además o en lugar. Por ejemplo, Sinorhizobium meliloti, una bacteria del suelo, que es capaz de formar una raíz simbiosis nódulo de fijación de nitrógeno con la alfalfa leguminosas (Medicago sativa), contiene, además de PE un segundo fosfolípido zwitteriónico, la fosfatidilcolina (PC) 5. Además, los lípidos no contiene fósforo o glicerol pueden ser anfífilo y forma parte de la membrana celular. Por ejemplo, de las condiciones de crecimiento de fósforo limitativo, en S. meliloti, (glicero fosfolípidos) se sustituyen en gran medida por los lípidos de membrana que no contienen fósforo, es decir, sulfolípidos, lípidos ornitina, y diacylglyceryl trimethylhomoserine (DGTS) 6. En las bacterias, DGTS se forma a partir de diacilglicerol (DAG) en una vía de dos pasos 7, pero la fuente para la generación de DAG no estaba claro. Experimentos de pulso-caza sugirieron que PC puede ser un precursor para DGTS 8 y usando la metodología descrita en este manuscrito podríamos identificar una fosfolipasa C (PLCP, SMc00171) que se forma en condiciones de fósforo limitativo y que puede convertir PC en DAG y fosfocolina 8.
En un estudio separado, descubrimos que un acil-CoA sintetasa (FADD) mutante deficiente en S. meliloti o de Escherichia coli acumula ácidos grasos libres al entrar en la fase estacionaria de crecimiento 9. Aunque estos ácidos grasos parecían ser derivados de lípidos de la membrana, no se sabe que la fuente precisa de los ácidos grasos libres o la enzima (s) liberándolos. Una vez más, empleando la estrategia descrita en este manuscrito, dos 10 (fosfo) lipasas patatina-como (SMc00930 y SMc01003) que contribuyó a la formación de ácidos grasos libres en S. meliloti 11 estaba previsto. Sorprendentemente, SMc01003 utiliza DAG como sustrato la conversión a monoacilglicerol y, finalmente, glicerol y ácidos grasos libres 11. Por lo tanto, es una lipasa SMc01003 DAG (DGLA).
A pesar de que existe una serie de algoritmos de predicción del potencial (fosfo) 12,13 lipasas, su función precisa y papel fisiológico por lo general no se conoce. Aquí describimos un protocolo, para clonar y sobreexpresan lipasas (fosfo) predichos o potenciales. Este manuscrito se explica cómo los ensayos enzimáticos pueden ser desarrollados y optimizados para la lipasa sobreexpresa (fosfo) mediante el uso de sustratos cromogénicos artificiales. Proporcionamos ejemplos de cómo en un ensayo enzimático optimizado el (fosfo) sustrato de la lipasa real puede ser encontrado y cómo estos hallazgos podrían enriquecer nuestra comprensión de la fisiología microbiana.
Protocolo
1. Clon y sobreexpresan gen estructural de la lipasa predicha
- El uso de la reacción en cadena de la polimerasa (PCR) 14 y oligonucleótidos específicos (Tabla 1) 15, amplificar el gen de interés (smc01003, smc00930, o smc00171), predijo para codificar una lipasa o fosfolipasa, a partir del ADN genómico del organismo huésped (es decir, , S. meliloti).
- Introducir sitios de restricción específicos (con la secuencia diseñada de los oligonucleótidos). Digerir el fragmento de ADN amplificado con las enzimas de restricción correspondientes y clonar en un vector de expresión tales como plásmidos de la serie pET 16.
- Después de verificar la secuencia de ADN correcta para el gen clonado, transformar el vector en una cepa de expresión, tales como Escherichia coli BL21 (DE3) pLysS 16.
- Preparar un pre-cultivo durante la noche del huésped de expresión E. coli BL21 (DE3) pLYSS, que alberga el vector pET respectivo con el gen clonado o el vector vacío, en matraces de 100 ml de cultivo que contenían 20 ml de caldo Luria Bertani (LB) 17 además de los antibióticos requeridos. Cultivar las células a 30 ° C (o a la temperatura de crecimiento normal de la bacteria de la que se origina la lipasa).
- El uso de los pre-cultivos de una noche, inocular 500 ml de medio LB precalentado (además de los antibióticos requeridos) en matraces de 2 L de cultivo para obtener una densidad óptica inicial a 620 nm (OD 620) = 0,05. Siga crecimiento de cultivos y a una DO 620 = 0,3, añadir isopropil-β-D-tiogalactósido (IPTG) a una concentración final de 100 mM, y se incuba con agitación a 30 ° C durante un período de 4 horas.
- Al final del período de incubación, la transferencia de cada cultivo a un tubo de centrífuga de 500 ml y centrifugar a 5000 xga 4 ° C durante 30 min. Resuspender los sedimentos celulares bacterianas en 5 ml de tampón de suspensión (por ejemplo, SMc00930- y las células que expresan SMc01003 en Tris-HCl 50 mM pH 8,0 y las células SMc00171 expresan en 50 mM dietanolamina-HCl pH 9,8). Almacenar las suspensiones de células a -80 ° C hasta su uso.
2. Preparar los extractos de proteínas libres de células y determinar la concentración de proteínas
- Descongelar las suspensiones de células bacterianas y almacenar en hielo. Pasar las suspensiones de células tres veces a través de una célula de presión en frío a 20.000 libras por 2 en. Eliminar las células intactas y restos celulares por centrifugación a 5000 xg durante 30 min a 4 ° C.
- Después de la centrifugación, se preparan partes alícuotas de 100 y 500 l del sobrenadante para su posterior análisis y almacenar a -80 ° C hasta su uso.
- Utilice una de la parte alícuota de 100 l para determinar la concentración de proteína de los extractos libres de células distintas por un método de elección o como se describe 18.
3. Utilizar sustratos artificiales para la optimización de la enzimaActividades de lipasas (fosfo)
- Para una cobertura inicial de las actividades enzimáticas distintas, utilizar sustratos artificiales que producen un producto coloreado tras la hidrólisis, tales como p-nitrofenol (p -NP).
- Para los ensayos de la enzima ya optimizados con sustratos de éster de p-nitrofenil artificiales (descritos para la fosfolipasa C PLCP (SMc00171), así como para las fosfolipasas patatina-como predichos SMc00930 y SMc01003), esquemas de pipeteado de uso descritas en la Tabla 2.
- Al explorar un nuevo lipasa (fosfo) potencial, preparar una primera ensayo de la enzima estándar que contiene 50 mM Tris-HCl, pH 8,5, NaCl 100 mM, 0,05% de Triton X-100, 0,5 mM p-nitrofenil-compuesto que contiene (p-nitrofenil fosfato , fosfato de bis- p -nitrofenilo, p-nitrofenil decanoato, o p-nitrofenil palmitato), y el extracto de proteínas libre de células (de verificación 1, 3, 10, 30, 100, 300, y 1000 g) en un volumen total de 1 ml en 1 ml de plástico cuveTTES.
NOTA: El uso de pH alcalino (Figura 1) cuando después de p-nitrofenil hidrólisis del éster en un ensayo continuo. Alternativamente, utilizar los ensayos de tiempo de un solo punto para una gama de valores de pH, la adición de NaOH al final del período de incubación para terminar la reacción de la enzima y para asegurar que todos p -NP está presente en la forma fenolato. - Sigue el curso de tiempo para un aumento de la absorbancia a 405 nm, debido a la formación de p -NP, en un espectrofotómetro a 30 ° C durante un período de 5 min. Cuantificar la formación inicialmente lineal de p -NP mediante la determinación de la pendiente inicial de aumento de la absorbancia por unidad de tiempo.
- Calcular la variación de la concentración (? C) para p -NP usando la ley de Lambert-Beer (? A = ε? C d) 1.
NOTA:? A es el cambio lineal de absorbancia determinado, ε es el coeficiente de extinción molar a la longitud de onda respectiva (en unidades de M -1 cm-1), d es la longitud de la trayectoria de la luz (1 cm), y? c es el cambio de la concentración (en unidades de M) que se determine.- Teniendo en cuenta que el volumen de ensayo es de 1 ml, se calcula la cantidad de p -NP formado.
NOTA: La cantidad x = concentración de volumen. - Calcular la actividad de la enzima dividiendo la cantidad de p -NP formado por el tiempo en el que se forma. Determinar la actividad de la enzima específica dividiendo actividad de la enzima por la cantidad de proteína (en mg), que fue responsable de la generación de esta actividad.
- Teniendo en cuenta que el volumen de ensayo es de 1 ml, se calcula la cantidad de p -NP formado.
- Comparar los cambios de absorbancia provocados por los extractos de proteína en la que un gen candidato (smc00171, smc00930, o smc01003) había sido expresadas con extractos que albergan solamente un vector vacío.
NOTA: Con el fin de continuar con los pasos siguientes, las actividades específicas, causadas por los extractos de proteína en la que se habían expresado un gen candidato, deben ser por lo menos dos veces o más de los valores obtenidos para las actividades específicas causadas por extractos de proteínas que albergan solamente un vector vacío. - Para experimentos adicionales, seleccionar aquellas condiciones en las que la hidrólisis del compuesto p-nitrofenil-que contiene es mínima con extractos libres de células (es decir, el vector vacío) y para el que la formación más pronunciada de p -NP y el anión p -nitrophenolate (Figura 1) se puede observar cuando se emplean extractos de proteínas, en el que un gen candidato se ha expresado.
- Después de determinar la actividad de la enzima inicial en 3.1, optimizar las condiciones de ensayo para la enzima respectiva mediante la variación de pH, tipo de tampón, tampón de fuerza, las concentraciones de NaCl, detergentes tales como Triton X-100, y la ausencia o presencia de diferentes cationes bivalentes.
- Para diferentes concentraciones de cada variable, a determinar la actividad enzimática específica (véase 3.1.4.2) (el número más alto obtenido define la condiciónde la actividad enzimática máxima). Utilice la combinación de las condiciones óptimas encontradas para cada variable para definir un ensayo enzimático optimizado en la que cada variable está presente en su concentración óptima.
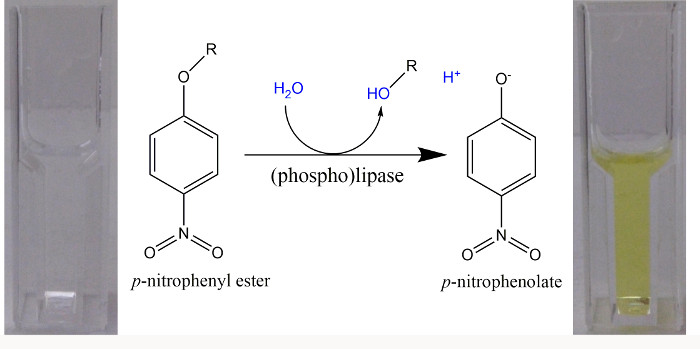
Figura 1. p-nitrofenil ésteres como sustratos artificiales para lipasas (fosfo) en un ensayo espectrofotométrico. Con la hidrólisis de los ésteres de p-nitrofenil, se forman un ácido (R-OH) y p-nitrofenol (p -NP). Debido a la pK a = 7,2 para la disociación de la fenólico H + a partir de p -NP, a un pH> 9,2 más de 99% está en la forma p -nitrophenolate de color amarillo brillante y un coeficiente de extinción molar de 18.000 M -1 cm - 1 puede ser utilizado en una longitud de onda de 405 nm para la cuantificación de la libre p -nitrophenolate 22. Cuando se utilizaron tampones con un pH de 8,5, se determinó la absorbancia a 400 nm y un coeficiente de extinción molar de 14.500 M-1 cm-1 fue empleado 23. Haga clic aquí para ver una versión más grande de esta figura.
NOTA: Después de haber definido las condiciones óptimas para la actividad de la enzima de interés, se embarcan en la búsqueda de la verdadera sustrato / fisiológica de esta lipasa. En principio, tomar dos, a menudo complementaria, los enfoques para lograr este objetivo, un enfoque in vivo o un enfoque in vitro.
4. En Vivo Identificación de la Physiological sustrato de una lipasa
ove_content "> NOTA:. En un enfoque in vivo, expresar la lipasa de interés en un organismo huésped 8,11 con el fin de registrar el paso del tiempo si la expresión de la lipasa altera el perfil de lípidos host's En otro enfoque in vivo, generar una mutante deficiente del gen de interés 8,11 y estudiar si su perfil de lípidos es distinta de la versión de tipo salvaje 6,8,11. con el fin de obtener una evaluación cuantitativa de perfil de lípidos de un organismo, un método simple consiste en radiomarcar compuestos celulares , la extracción de los lípidos, separándolos por cromatografía, y cuantificar los lípidos separadas marcadas radiactivamente.- El radiomarcado de los lípidos.
- Preparar un pre-cultivo de una noche de un organismo de interés (E. coli o S. meliloti) en 5 ml del medio de cultivo deseado (medio complejo o medio mínimo definido) y crecer a 30 ° C.
- Desde el pre-cultivo, inocular en 20 ml de la saen un matraz de cultivo de 100 ml me medio fresco para obtener una DO inicial 620 = 0,3 para el cultivo.
- Tomar una alícuota (1 ml) del cultivo en condiciones estériles y transferir a un tubo de fondo redondo de poliestireno de 14 ml estéril.
- Añadir 1 Ci de acetato de [1- 14 C] (60 mCi por mmol) al cultivo 1 ml.
- Incubar el cultivo líquido con agitación a 30 ° C durante un período de 24 horas.
- Al final del período de incubación, la transferencia de la cultura a un tubo de microcentrífuga de 1,5 ml y centrifugar a 12.000 xg a temperatura ambiente durante 5 min.
- Resuspender el precipitado en 100 l de agua. En este punto, almacenar la suspensión celular a -20 ° C o inmediatamente continuar con la extracción de los lípidos polares (sección 4.2).
- La extracción de lípidos polares.
NOTA: El método descrito aquí esencialmente sigue el procedimiento descrito por Bligh y Dyer 19.- A los 100 l de susp celular acuosaensión, añadir 375 l de metanol: solución de cloroformo (2: 1; vol / vol).
- Vortex durante 30 segundos e incubar durante 5 min a temperatura ambiente.
- Centrifugar 5 min a 12.000 xg a la temperatura ambiente.
- Transferir el sobrenadante a un nuevo tubo de microcentrífuga de 1,5 ml.
- Añadir 125 l de cloroformo y 125 l de agua, vórtice 30 seg.
- Centrifugar 1 min a 12.000 xg a la temperatura ambiente.
- Pasar la fase inferior de cloroformo a un tubo nuevo y se seca con una corriente de gas nitrógeno.
- Disolver los lípidos secos en 100 l de cloroformo: solución de metanol (1: 1; vol / vol).
NOTA: En este punto, una parte alícuota de 5 l de la solución de lípidos se puede cuantificar mediante recuento de centelleo líquido. - Para el análisis de cromatografía en capa fina (TLC), se seca por los 95 l restantes con una corriente de gas nitrógeno y volver a disolver los lípidos secos en 20 l de cloroformo: solución de metanol (1: 1; vol / vol). Utilice una alícuota de 3 lpara el análisis de TLC.
- La separación de los lípidos polares por cromatografía en capa fina (TLC).
NOTA: En función de las clases de lípidos a analizar, diferentes combinaciones de fases sólidas y móviles podrían emplearse para la separación. Aquí una separación típica de lípidos cargados polares y otro, más adecuado para los lípidos polares neutros, utilizando cromatografía en capa fina (HPTLC) láminas de aluminio de gel de sílice de alto rendimiento como la fase sólida, se describen.- La separación de los lípidos polares cargados por TLC bidimensional (2D-TLC).
- Aplicar una alícuota de 3 l de muestra de lípidos en una esquina de una hoja de sílice HPTLC de aluminio en gel (10 x 10 cm), 2 cm del borde de la placa.
- Preparar y mezclar la fase móvil (140 ml de cloroformo, 60 ml de metanol, y 10 ml de agua) para la separación en la primera dimensión.
- Rociar una cámara de revelado TLC internamente con papel de cromatografía.
NOTA: Esto es para asegurar que la fase de gas de la cámara deserá saturada rápidamente (dentro de 30 min) después de la fase móvil para la primera dimensión se ha agregado a la cámara y la cámara se ha cerrado con una placa de vidrio. - Preparar y mezclar la fase móvil (130 ml de cloroformo, 50 ml de metanol, y 20 ml de ácido acético glacial) para la separación en la segunda dimensión y la transferencia a una segunda cámara de revelado TLC internamente recubierto con papel de cromatografía y dejar que la cámara de saturar.
- Transferir con cuidado la lámina de aluminio de gel de sílice HPTLC con la muestra de lípido seco a la primera cámara y desarrollar (es decir, realiza una cromatografía) la placa durante 60 min en la cámara cerrada en la primera dimensión 5.
- Retire la placa de la cámara y dejar que los disolventes se secan en una campana de flujo durante 30 minutos.
- Después de girar la placa de 90 grados con respecto a la cromatografía anterior, la transferencia de la lámina de aluminio de gel de sílice HPTLC, en el que los lípidos han sido separados en una dimensión, a la segundala cámara D y el desarrollo de la placa durante 60 minutos en la segunda dimensión 5.
- Retire la hoja de la cámara y dejar que los disolventes se secan en una campana de flujo durante al menos 2 horas.
- La separación de los lípidos polares neutros.
- Aplicar 3 ml de alícuotas de las muestras de lípidos en una hoja de aluminio de gel de sílice HPTLC de partida 2 cm de los bordes de la placa. Si se analizan múltiples muestras de una cromatografía unidimensional, mantenga una distancia de al menos 1,5 cm entre los diferentes puntos de aplicación de ejemplo.
- Preparar y mezclar la fase móvil (140 ml de hexano, 60 ml de éter dietílico, y 8 ml de ácido acético) y la transferencia a una cámara de revelado internamente TLC recubiertas con papel de cromatografía y cubiertas con una placa de vidrio para permitir que el saturar cámara (30 min).
- La transferencia de la hoja de aluminio de gel de sílice HPTLC con las muestras de lípidos secos a la cámara y desarrollar la placa durante 30 min en la cámara cerrada.
- Retire la placa de la cámaray dejar que los disolventes se secan en una campana de flujo durante 2 horas.
- La separación de los lípidos polares cargados por TLC bidimensional (2D-TLC).
- La cuantificación y visualización de los lípidos polares separadas.
- Una vez que la hoja de TLC desarrollado es seco, se incuban con una pantalla de luminiscencia fotoestimulable (PSL) en un casete cerrado durante 3 días.
- Exponga la pantalla se incuba a un escáner PSL y adquirir una imagen virtual de los lípidos radiomarcados separadas.
- Realizar la cuantificación usando software PSL 20.
- La visualización y el aislamiento de las clases de lípidos polares individuales.
- Incubar hoja TLC desarrollada durante 10 minutos en una cámara de cromatografía en presencia de 1 g de cristales de yodo.
NOTA: Separado compuestos lipídicos se disolverán el yodo y aparecen como manchas de color marrón. - Círculo de los puntos con un lápiz, compararlos con la movilidad relativa (Rf) de compuestos estándar (es decir, 1,2-sn-glicerol dipalmitoyl-, dipalmitoil-L-α-Phosphatidylcholine, DL-α-monopalmitina, o ácido palmítico), e identificar a qué clase de lípidos que podrían pertenecer.
- En una campana de extracción, dejar que el yodo se evapore de la hoja de TLC.
- Con la ayuda de una espátula, raspar el gel de sílice que contiene el compuesto de interés de la hoja, y extraer el compuesto del gel de sílice con una mezcla de 100 l de agua y 375 l de metanol: solución de cloroformo (2: 1; vol / vol).
- Continuar con la extracción de acuerdo con Bligh y Dyer como se indica (4.2.2 en adelante).
- Tienda clase de lípidos purificado en 100 l de cloroformo: solución de metanol (1: 1; vol / vol) a -20 ° C hasta su uso.
- Incubar hoja TLC desarrollada durante 10 minutos en una cámara de cromatografía en presencia de 1 g de cristales de yodo.
5. En Vitro Identificación de la Physiological sustrato de una lipasa
NOTA: En un enfoque in vitro, estudiar si la lipasa de interés puede convertir una mezcla de lípidos aislados o lípidos puros individuales a la hidrol correspondienteproductos analysis en las condiciones definidas como óptimo en 3.2.
- Utilizar esquemas de pipeteado para ensayos enzimáticos según la Tabla 3 para la fosfolipasa C específica de PC-SMc00171 (véase 5.2), la fosfolipasa A (véase 5.3), y DAG lipasa SMc01003 (véase 5.4) la actividad.
- Determinación de la actividad de la fosfolipasa C-PC específica (Tabla 3).
- A un tubo de microcentrífuga de 1,5 ml, añadir 5.000 recuentos por minuto (cpm) del total de 14 PC marcado con C y una solución de Triton X-100.
- Mezclar y seco bajo una corriente de nitrógeno.
- Añadir dietanolamina-HCl, tampón de pH 9,8, así como NaCl y MnCl2 soluciones y agua bidestilada para obtener un volumen final de 99,5 l. Vortex durante 5 seg.
- Añadir 0,5 l de enzima (5 mg de proteína) (es decir, un extracto libre de células en las que sobreexpresa SMc00171 está presente) para iniciar la reacción. Mezclar brevemente.
- Incubar a 30 ° C durante 4 h.
- Detener la reacción por eladición de 250 l de metanol y 125 l de cloroformo.
- Extracto de lípidos como se describió anteriormente (véase 4.2).
- lípidos separados por una sola dimensión--TLC (1D) (ver 4.3.2 y 4.4), y analizarlos mediante imágenes de PSL.
- Determinación de la actividad de la fosfolipasa A (Tabla 3).
- A un tubo de microcentrífuga de 1,5 ml, añadir 5.000 cpm de 14 Total de fosfolípidos marcado con C y una solución de Triton X-100.
- Mezclar y seco bajo una corriente de nitrógeno.
- Para un ensayo final de 100 l, añadir Tris-HCl, 8,5 tampón de pH, solución de NaCl y agua. Vortex durante 5 seg.
- Añadir 5 l de enzima (50 mg de proteína) (es decir, un extracto libre de células en el que sobreexpresa SMc00930 o SMc01003 está presente).
- Incubar a 30 ° C durante 5 hr.
- Detener la reacción mediante la adición de 250 l de metanol y 125 l de cloroformo.
- Extraer los lípidos como se describió anteriormente (véase el punto 4.2), separadosellos por 1D-TLC usando 130 ml de cloroformo, 50 ml de metanol y ácido acético glacial 20 ml como la fase móvil, y analizarlos por imágenes de PSL.
- Determinación de la actividad de la lipasa diacilglicerol (DAG).
- Preparación de DAG marcado con C 14.
- Radiomarcador S. culturas meliloti (ver 4.1) y extraer los lípidos polares (véase el punto 4.2) como se ha descrito. Separada S. meliloti extractos lípidos totales por 1D-TLC en cloroformo: metanol: ácido acético (130: 50: 20; vol / vol) utilizando condiciones descritas para la separación en segunda dimensión en 4.3.1.
- Visualizar PC mediante tinción con yodo y usar un lápiz para marcar la localización de fosfatidilcolina (PC).
- Aislar PC radiomarcado como se describe en 4.5.
- Cuantificar PC extraído por recuento de centelleo.
NOTA: Se espera que alrededor de 320.000 cpm PC. - Tratar PC (250.000 cpm) con 0,1 U de la fosfolipasa C de Clostridium perfringens en 50 mM Tris-HCl, pH 7,2, 0,5% De Triton X-100 y CaCl mM 10 2 durante 2 horas en un volumen total de 100 l y detener la reacción mediante la adición de 250 l de metanol y 125 l de cloroformo.
- Extraer los lípidos como se ha descrito anteriormente y separados por ellos por 1D-TLC (ver 4.3.2).
- Aislar diacilglicerol de la placa de sílice y cuantificar mediante recuento de centelleo (como se describe en 4.2)
- Ensayo de lipasa diacilglicerol (Tabla 3).
- A un tubo de microcentrífuga de 1,5 ml, añadir 5.000 cpm de 14 DAG marcado con C y una solución de Triton X-100.
- Mezclar y seco bajo una corriente de nitrógeno.
- Para un ensayo final de 100 l, añadir Tris-HCl (pH 9,0) tampón, una solución de NaCl y agua bidestilada. Vortex durante 5 seg.
- Iniciar la reacción mediante la adición de 5 l de enzima (50 mg de proteína de extracto libre de células).
- Incubar a 30 ° C durante 4 h.
- Detener la reacción mediante la adición de 250 l de metanol unad 125 l de lípidos en cloroformo y el extracto como se describió anteriormente (véase el punto 4.2).
- Analizar los lípidos polares neutros por 1D-TLC (véase 4.1.3.2) y las imágenes PSL posterior.
- Preparación de DAG marcado con C 14.
Resultados
Actividad de la fosfolipasa-PC específico C SMc00171 con Bis- fosfato de p-nitrofenil
Extractos libres de células obtenidas a partir de E. coli BL21 (DE3) pLysS x, que tenían smc00171 expresaron, fueron estudiados por su capacidad para hidrolizar ésteres de fosfato-nitrofenil bis- p, utilizando un ensayo enzimático espectrofoto...
Discusión
Durante los últimos 20 años, los genomas de muchos organismos se han secuenciado y aunque una gran cantidad de datos de secuencia del genoma se ha generado, la interpretación funcional es la zaga, y por consiguiente, dificulta nuestra comprensión de la función del genoma. las funciones de genes en los genomas son a menudo asignados basándose en la similitud de los genes de función o la aparición de motivos conservados conocido. Sin embargo, la función exacta de un gen dado es a menudo no se conoce. Especialment...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por becas de Consejo Nacional de Ciencias y Tecnología-México (CONACyT-México) (82614, 153998, 253549 y 178359 de Investigación Científica Básica, así como 118 en Investigación en Fronteras de la Ciencia) y de la Dirección General de Asuntos de Personal Académico-Universidad Nacional Autónoma de México (UNAM-DGAPA; PAPIIT IN202616, IN203612).
Materiales
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
Referencias
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados