Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Análisis de citometría de flujo de las células asesinas naturales lítico Actividad en sangre entera humana
En este artículo
Resumen
This work describes an advanced workflow for the accurate and fast determination of NK (Natural Killer) cell count and NK cell cytotoxicity in human blood samples.
Resumen
la citotoxicidad de células NK es una medida ampliamente utilizada para determinar el efecto de la intervención exterior en función de las células NK. Sin embargo, la precisión y reproducibilidad de este ensayo pueden considerarse inestable, ya sea debido a errores del usuario o debido a la sensibilidad de las células NK a la manipulación experimental. Para eliminar estos problemas, un flujo de trabajo que les reduce al mínimo fue establecido y se presenta aquí. Para ilustrar, se obtuvieron muestras de sangre, en diversos momentos, desde corredores (n = 4) que fueron sometidos a una intensa sesión de ejercicio. En primer lugar, las células NK se identifican de forma simultánea y aislados mediante el etiquetado CD56 y la clasificación basada en magnético, directamente de la sangre entera y de tan poco como un mililitro. Las células NK según se retiran de cualquier anticuerpo reactivo o de tapado. Se puede contar con el fin de establecer un recuento de células NK precisa por mililitro de sangre. En segundo lugar, las células NK según (efectores células o E) se pueden mezclar con 3,3'-Diotadecyloxacarbocyanine perclorato (DIO) etiquetada células K562 (células diana o T) en un ensayo-óptima 1: 5 T: relación de E, y se analizó usando un citómetro de flujo de imágenes que permite la visualización de cada evento y la eliminación de cualquier falso positivo o falsos negativos (tales como dobletes o células efectoras). Este flujo de trabajo se puede completar en aproximadamente 4 h, y permite obtener resultados muy estables incluso cuando se trabaja con muestras humanas. Cuando esté disponible, los equipos de investigación pueden probar varias intervenciones experimentales en sujetos humanos, y comparar las mediciones a través de varios puntos de tiempo sin poner en peligro la integridad de los datos.
Introducción
Las células asesinas naturales son un elemento esencial del sistema inmune innato. Mientras que están muy regulados, que tienen la capacidad de reconocer y eliminar las células anormales a través del contacto de célula a célula y sin activación previa 1. Como tales, forman una línea rápida de defensa contra las infecciones. El ejercicio, especialmente intenso, se ha demostrado que deprimen de forma transitoria el sistema inmune 2, 3, 4, 5. Las células NK son particularmente propensos a este efecto 4, 6, 7, creando una ventana de aumento de la sensibilidad a la enfermedad. Por lo tanto, el estudio de las intervenciones antes, durante o después del ejercicio intenso, con el objetivo de reducir su impacto sobre la función de las células NK es de particular interés para el bienestar de los atletas después de los concursos.
Sin embargo, el estudio de este tipo de intervenciones se complica por varios factores: 1) las células NK son escasos 8, en torno a 1% del compartimiento de glóbulos blancos; 2) Las células NK son muy sensibles al estrés y se basan en la exposición constante a las condiciones fisiológicas para seguir siendo viable y estable durante la experimentación; y 3) ensayos estándar para determinar la citotoxicidad de células NK, tales como gradientes de Ficoll 9 y liberan los ensayos 10, son poco fiables e inconsistente. La variabilidad inherente de las muestras de humanos sólo agravan estos problemas. muestras humanas frescas recogidas durante las intervenciones son bastante regulado y difíciles de conseguir, al menos en comparación con muestras de animales o líneas celulares inmortalizadas. Esto reduce las oportunidades para repetir los experimentos o añadir participantes a la cohorte del estudio para llegar a los umbrales estadísticos significativos. En conjunto, estas cuestiones apoyan la necesidad de un protocolo optimizado que permite for tanto de alto rendimiento y un análisis de alta fiabilidad de la actividad lítica de las células NK en muestras humanas.
Hemos establecido un flujo de trabajo que acorta el tiempo necesario para identificar, aislar y células NK prueba de la sangre entera humana y reducir al mínimo la exposición a factores externos. El método optimiza el uso de dos instrumentos, un clasificador basado en magnética celular y un flujo de imágenes citómetro, y un, T optimizado ensayo específico: relación E para permitir la detección de las disminuciones o incrementos de la citotoxicidad de células NK.
Protocolo
NOTA: Todos los procedimientos de recolección de sangre se llevaron a cabo de acuerdo con las directrices establecidas por la Universidad Estatal de los Apalaches (ASU) Junta de Revisión Institucional (IRB).
1. Extracción de sangre entera
- Tiene un flebotomista certificado extraer la sangre según las directrices de la Organización Mundial de la Salud.
- Extraer la sangre en un tubo de recogida de sangre de 4 ml que contenía ácido etilendiaminotetraacético Di-Potasio (K 2 EDTA). Invertir tubo de recogida de sangre de acuerdo con las instrucciones del fabricante. Mantenga el tubo de recogida de sangre a temperatura ambiente en un agitador de sobremesa hasta la separación.
2. Preparación de células diana marcadas con DiO
- Crecer células K562 en completa Modificado de Iscove medio de Dulbecco (IMDM) suplementado con 10% de suero bovino fetal (FBS) y 1% penicilina estreptomicina durante varias semanas antes del ensayo para garantizar la salud de las células. Ajuste el concentration de las células a 3 x 10 5 células / ml diariamente a través de la realización de un recuento de células y la subsiguiente paso de las células. Realizar un cambio de medio completo cada 2 a 3 días.
- En el día del ensayo, realizar un recuento de células usando un 1: 1 hemocitómetro.
- Retire 10 l del matraz K562 y ponerlo en un tubo de 1,5 ml.
- Añadir 10 l de colorante azul de tripano en el mismo tubo de 1,5 ml para un factor de dilución de 1: 1.
- Agitar suavemente el tubo para mezclar las células K562 y colorante azul tripano.
- Permitir K562 colorante azul de tripano de células en incubación durante 1 min a temperatura ambiente.
- Retire 15 l de células K562 manchadas de tubo.
- Pipeta sobre hemocitómetro para el recuento de células.
- Recuento de células en las cuatro esquinas, así como el cuadrado del medio para un total de cinco plazas.
- Calcular el recuento de células mediante el uso de la ecuación:
células totales / mL = Total de contado X (factor de dilución / Número de plazas) X 10.000 células / ml
- Resuspender las células diana K562 a una densidad final de 1 x 10 6 células / ml en medio de cultivo IMDM libre de suero.
- Para las células diana K562 no teñidas, añadir 10 ml de células diana K562 a un tubo de 15 ml a una densidad de 1 x 10 6 células / ml para un total de 10 x 10 6 células K562. Colocar en una incubadora a 37 ° con 5% de CO 2 hasta que esté listo para su uso.
- Para las células diana K562 DiO manchada, agregar 10 ml de células K562 se dirige a un tubo de 15 ml a una densidad de 1 x 10 6 células / ml para un total de 10 x 10 6 células K562.
- Añadir 1 l de DiO solución de etiquetado de células por ml de suspensión celular en 15 ml de tubo designado para la tinción DiO y suavemente vórtice. Por ejemplo, añadir 10 l de solución de etiquetado celular DiO a 10 x 10 6 células / ml K562 en un volumen de 1 x 10 6 células / ml.
- Incubar solución K562-DiO durante 20 min a 37 ° C con 5% de CO 2 en un tubo de 15 ml.
- Seguiring incubación, añadir 3 ml de solución salina tamponada con fosfato temperatura ambiente (PBS) a la solución K562-DIO que contiene 15 ml tubo.
- Centrifugar durante 10 min a 135 xg a temperatura ambiente.
- Retirar con cuidado el sobrenadante sin alterar el sedimento celular con una pipeta de 1000 l de volumen ajustable.
- Añadir 10 ml de IMDM fresco a la celda que contiene 15 ml tubo de pellets.
- Suavemente el tubo de vórtice para resuspender las células.
- Repita los pasos 2.7 a 2.10 dos veces más.
- Almacenar células en una incubadora a 37 ° con 5% de CO 2 hasta que esté listo para su uso.
NOTA: Las células pueden ser almacenados en la incubadora durante hasta 24 h, pero es preferible usarlos en el mismo día.
3. Preparación de los controles
- Transferir el siguiente en tubos de 1,5 ml separadas y debidamente etiquetados:
- Añadir 500 l de IMDM fresco que contiene volvió a suspender dio-etiquetados células K562 en el "doble positivo" marcado tubo de 1,5 ml.
- Añadir 500 l de IMDM fresco que contiene volvió a suspender las células K562 marcadas con DiO en el "DiO única" etiquetados tubo de 1,5 ml.
- Añadir 500 l de IMDM fresco que contiene células K562 no marcados se volvieron a suspender en el "yoduro de propidio (PI) sólo se" etiquetan tubo de 1,5 ml.
- Ponga el cable positivo y doble chispa solamente tubos en un baño de agua a 55 ° C durante 5 min.
- Una vez transcurrido el 5 minutos, retirar y limpiar los tubos con un 70% de etanol.
- Añadir 10 l de PI a la doble positivo y PI sólo tubos de 1,5 mL.
- Coloque los tres controles de células diana K562 en la incubadora a 37 ° C durante 30 min.
- Después de la incubación de 30 min ha transcurrido, de centrífuga de los tres controles de células diana K562 durante 2 minutos a 163 x g.
- Retirar con cuidado el sobrenadante sin alterar el sedimento celular.
- Resuspender cada control con 20 l de medio de cultivo fresco de células IMDM, y dejar en el incubador a 37ºC con 5% de CO 2 durante al menos30 min para la señal de DiO óptima.
NOTA: Los controles son ahora listo para ser ejecutado a través del flujo de imágenes citómetro.
4. Separación de Células NK automatizada
- A su vez en el separador de células y permitir que el ciclo de arranque hasta el final.
- Asegúrese de que todas las botellas de líquido iluminaciones son de color verde y que la botella de residuos está vacía.
- Obtener una temperatura de 15 ml soporte de tubos de habitación.
NOTA: La selección se basa en los volúmenes de muestra. Por ejemplo, para un volumen de menos de 3 ml utilizan un soporte de tubos de 15 ml y para un volumen de más de 3 ml utilizan un bastidor 50 ml tubo. - tubos de muestra de la etiqueta en consecuencia (por muestra Repetir / participante): Participante 1 muestra de sangre entera; Participante 1 fracción negativa; Participante 1 fracción positiva.
- pipeta suavemente 1,5 ml de sangre total desde el paso 1.2 en tubo de 15 "muestra de sangre" ml.
- Coloque debidamente etiquetado tubo de 15 ml "muestra de sangre" de Paso 4.5 y etiquetado 15 ml "Fracción Negativo" ytubos "fracción positiva" de Paso 4.4 en el bastidor de tubo. Utilice thefollowing muestra estante configuración: Fila (R1) A: muestra de sangre entera, Fila (R2) B: Negativo, la fracción no marcado, Fila (R4) C: Positivo, fracción y marcadas magnéticamente.
- Introduzca la rejilla separadora sobre la MiniSampler para autolabeling.
- Seleccione "Reactivo" en el menú y resaltar la posición donde se colocará el vial en el bastidor separador.
- Seleccione "Leer Reactivo" para activar el lector de códigos 2D.
- Colocar el vial reactivo apropiado delante del lector de códigos 2D entre 0,5-2,5 cm de la cubierta del lector de código.
NOTA: Por ejemplo, el reactivo necesario para la separación de células NK es CD56. - Mantenga vial de reactivo en un ángulo delante del lector de código 2D de lectura óptima.
- Colocar el vial de reactivo en la posición adecuada bastidor separador.
- Poner de relieve las posiciones deseadas en la pestaña de separación en la pantalla.
- En el submenú etiquetado, asignar una autolabeling programa.
- Asignar las microperlas de separación de células CD56 Reactivo para acumular las posiciones 1, 2, 3, y 4.
- Seleccione el programa de separación "posselwb".
- Seleccione el programa "aclarado" lavado.
- Insertar un volumen de muestra de 1.500 l en el submenú "Volumen" con el teclado numérico.
- Seleccionar la opción "Enter" en el teclado.
- Seleccione "Ejecutar" para iniciar la separación de células.
- Seleccione "OK" para confirmar que se dispone de suficiente memoria intermedia para el procesamiento de todas las muestras.
5. Recuento de Células NK Separación Después de la célula
- Inmediatamente después de la separación de células con el separador de células, recuperar la fracción positiva. Dejar a temperatura ambiente. Esta fracción contiene la población de células NK puro deseado.
- Para cada muestra individual, realice un recuento de células usando un hemocitómetro acuerdo con el paso 2.2.
- Después de los cálculos, registrar el recuento de células.
6. Ensayo de citotoxicidad Preparación de la muestra
- Preparar y etiquetar tubos de 1,5 mL de cada muestra / participante en consecuencia.
- Pipeta proporción de células NK y células K562 labled-DiO deseada en cada tubo.
NOTA: Por ejemplo, la relación deseada de las células diana K562 y células efectoras NK es de 1: 5. - Centrifugar durante 5 min a 135 x g.
- Retirar con cuidado el sobrenadante sin alterar el sedimento celular.
- Resuspender NK-DiO etiquetados mezcla de células K562 en 500 l de medio de células NK sin la interleucina-2 (IL-2) y 2-mercaptoetanol (2-ME) (incompleto medios de cultivo de las células NK).
NOTA: El medio de cultivo celular incompleta NK es esencial mínimo de Eagle Medium con bicarbonato de sodio, sin L-glutamina, ribonucleósidos y desoxirribonucleósidos. - Añadir 5 l de PI a cada tubo.
- Centrifugar durante 2 minutos a 163 x g.
- Se incuban las células a 37 ° C durante 2 h.
- Después de 2 h de incubación, centrifuge durante 2 minutos a 163 x g.
- Retirar con cuidado el sobrenadante sin alterar el sedimento celular.
- Resuspender las células en 25 l incompleta medios de cultivo de las células NK.
7. Preparación de espontánea ( "S") Muestra
- Pipeta 500 l de células K562 marcadas con DiO (concentración de 1 x 10 6 células / ml) en 1,5 ml de tubo.
- Añadir 10 l de PI a cada tubo.
- tubo de centrifugación durante 2 minutos a 163 x g.
- Se incuban las células a 37 ° C durante 2 h.
- Después de 2 h de incubación, se centrifuga durante 2 minutos a 163 x g.
- Retirar con cuidado el sobrenadante sin alterar el sedimento celular.
- Resuspender las células en 25 l incompleta medios de cultivo celular alfa medio esencial mínimo (α-MEM).
8. Adquisición de Datos de Imagen con citómetro de flujo
- Pulse el botón verde dentro de la puerta principal de la formación de imágenes citómetro de flujo para encender el instrumento.
- Encienda coll equipos asociados con la formación de imágenes citómetro de flujo.
- Poner en marcha el flujo de imágenes citómetro de software.
- Haga clic en el botón "Inicio" para limpiar el sistema y preparar la línea de muestreo.
- Una vez que el "inicio", cierre la ventana "calibraciones".
- Asignar canales: en la parte superior izquierda, haga clic en cada canal con el fin de asignarlos.
- En el lado derecho, haga clic en el botón gráfico de dispersión para crear diagramas de dispersión 4: Raw Max Pixel _MC_6 vs Area_M06, Crudo Max Pixel _MC_2 vs Area_M02, Crudo Max Pixel _MC_5 vs Area_M05 y FieldArea_M01 vs AspectRatio_M01.
- Comenzar a analizar las muestras utilizando primero el control "Doble positivo".
- Haga clic en "Cargar".
- Coloque el tubo de 1,5 ml con la muestra "Doble positiva" de los pasos 3.4 a 3.9 en el cargador de muestras.
- Seleccione el objetivo de 40X en la pestaña "aumento".
- Encienda el 405 mW y642 láseres MW.
- Activar el canal "campo claro".
- Haga clic en "Seleccionar intensidad."
- Sobre la base de la "doble positivo" muestra de control, determinar la intensidad deseada para el láser 405 mW por lo que el detector no está sobrecargado.
Nota: Por ejemplo, la intensidad óptima para este experimento se fijó en 11 mW.
- Después de lograr la configuración deseada, la adquisición de datos.
- En la pestaña "File Adquisición", introduzca el nombre de archivo en un texto personalizado. Seleccione una carpeta para guardar el archivo (s) de datos.
- Introduce el número de células de adquirir junto a "Suma". Normalmente, este número varía entre 1.000 a 10.000.
- Haga clic en "Adquirir".
NOTA: Una vez que se adquiere el número deseado de células, el archivo de datos se guarda automáticamente en la carpeta seleccionada previamente.
- Una vez finalizada la adquisición, cargue la siguiente muestra de control - DiO único control.
- Haga clic en "Cargar".
- Coloque el tubo de 1,5 ml con la "única DiO" muestra en el cargador de muestras.
- Deje el objetivo de 40X en la pestaña "aumento" seleccionado.
- Deje el láser de 405 mW encendido.
- Deben apagar el láser de 642 mW y el canal "campo claro".
NOTA: Ahora que la configuración deseada se ha logrado, los datos pueden ser adquiridos. - En la pestaña "File Adquisición", introduzca el nombre de archivo en un texto personalizado y seleccione una carpeta para guardar el archivo (s) de datos. Introduce el número de células de adquirir junto a "Collect.". Normalmente este número es de 1.000.
- Haga clic en "Adquirir".
NOTA: Una vez que se adquiere el número deseado de células en el archivo de datos se guarda automáticamente en la carpeta seleccionada previamente.
- Repita el paso 8.10 para el "PI única" muestra de control. Las muestras experimentales ya están listos para ser recogidos.
- Manejar las remaining muestras experimentales, incluyendo los "espontáneos de procedencia 'S'" de la siguiente manera:
- Deje el objetivo de 40X en la pestaña "aumento" seleccionado.
- Encienda el 405 mW y 642 mW láseres.
- Activar el canal "campo claro".
- Haga clic en "Ajuste de intensidad."
- En la pestaña "File Adquisición", introduzca el nombre de archivo en un texto personalizado y seleccione una carpeta para guardar el archivo (s) de datos. Introduzca el nombre de archivo en un texto personalizado.
- Introduce el número de células de adquirir junto a "Suma".
- Haga clic en el botón "Adquirir".
NOTA: Una vez que se adquiere el número deseado de células en el archivo de datos se guarda automáticamente en la carpeta seleccionada previamente.
- Repita el paso 8.12 para todas las muestras experimentales.
- Después se han recogido todos los datos y archivos experimentales, haga clic en el botón de "apagado" para esterilizar el sistema.
9. ImaginAnálisis de las muestras g citómetro de flujo
- Abrir el flujo de imágenes citómetro de aplicación de software de análisis.
- En "Archivo", abrir un archivo .RIF experimental.
- Construir una nueva matriz usando los archivos .RIF un solo color (DIO-únicamente controlan y control PI-solamente, creado durante los pasos de 8.10 y 8.11) seleccionando la opción "Crear una nueva matriz" en la pestaña de Compensación en el flujo de imágenes citómetro de software.
NOTA: El software le pedirá para la selección de los archivos de un solo color y unirlos para crear un archivo de la matriz (extensión de archivo .ctm), que ha de ser seleccionada para aplicar la compensación de canal. - Crear gráficos de puntos mediante el uso de la función "bloques de construcción" del software.
- Crear un punto de parcelas (BrightFieldGradient_RMS vs frecuencias) con puerta de las células enfocadas. Llamar a la puerta "Focus" (Figura 1A).
- El uso de la puerta "Focus", crear un gráfico de puntos de la zona de campo brillante vs Campo Claro Relación de aspecto ag comieron las células singlete. Llamar a la puerta "Single" (Figura 1B).
- El uso de la puerta "individual", crear un gráfico de puntos de Intensity_MC_Ch02 vs Intensity_MC_Ch5. Utilice este diagrama para identificar y células doble positivas (objetivos, muertas) Puerta dio-positivo sólo las células (objetivos, viva) y PI-DIO (Figura 1C).
NOTA: Todas las parcelas descritas en los pasos 9.4.1, 9.4.2, 9.4.3 y se pueden crear mediante el uso de la función "bloques de construcción" del software.
- Haga clic en la función de las estadísticas del gráfico de puntos para acceder a los números de células de cada puerta.
- Calcular el porcentaje de objetivos muertos en la muestra espontánea y las muestras experimentales utilizando la siguiente fórmula:
% blancos muertos en la muestra = (objetivos #DEAD x 100) / (# viven Objetivos + #DEAD objetivos) - Calcular la citotoxicidad utilizando la siguiente fórmula:
% De citotoxicidad = [(Experimental-muertos muertos espontánea) / (100-muertos espontánea)] x100
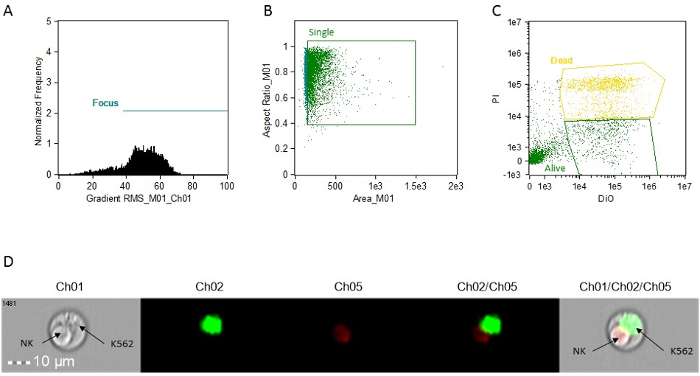
Figura 1: histogramas, gráficos de dispersión representativos e imágenes para el análisis de la actividad citotóxica. Análisis (A) de células de enfoque. (B) Análisis de una sola célula. Análisis (C) la tinción de células objetivo. Todas las determinaciones se realizan con la imagen adjunta a cada evento. Esto se puede acceder en el software de análisis simplemente haciendo clic en el evento en los gráficos. (D) la imagen representativa de un evento doblete, mostrando una célula apoptótica NK y un objetivo K562 vivo. CH01, campo claro. CH02, Dio. CH05, PI. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Determinación del número de células NK
Se midió el efecto de la pesada que se ejecuta en el recuento de células NK en la sangre total, utilizando el protocolo de ejercicio descrito en la figura 2. Las muestras de sangre fueron extraídas antes del ejercicio, inmediatamente después del ejercicio, 1,5 horas después del ejercicio, y, finalmente, 24 y 48 h después de la extracción de sangre inicial. La concentración de las células NK por mililitro de sangre...
Discusión
El método descrito en este estudio mide directamente la actividad específica de las células NK de un individuo en respuesta a estímulos (en este caso particular, el ejercicio prolongado). Típicamente, las células NK están aisladas de la propia sangre usando gradientes de densidad o clasificación de células mediante el uso de una combinación de marcadores. Aunque se utilizan ampliamente estos métodos, que tienen muchos inconvenientes: son mucho tiempo, implican múltiples manipulaciones, y son en gran medida d...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
This project was supported by Agriculture and Food Research Initiative Competitive Grant no. 2100-68003-30395 from the USDA National Institute of Food and Agriculture.
Materiales
| Name | Company | Catalog Number | Comments |
| K-562 lymphoblasts | ATCC | CCL-243 | |
| Iscove's Modified Dulbecco's Media | ATCC | 30-2005 | High glucose, with L-Glutamine, with HEPES, Sterile-filtered |
| Alpha Minimum Essential medium | ATCC | CRL-2407 | Without ribonucleosides and deoxyribonucleosides but with 2 mM L-glutamine and 1.5 g/L sodium bicarbonate |
| Trypan Blue Solution 0.4% | Amresco | K940-100ML | Tissue culture grade |
| Propridium Iodide Staining Solution | BD Pharmingen | 51-66211E | |
| Vybranto DiO cell-labeling solution | Vybranto | V-22886 | |
| autoMACS Pro Separator | Miltenyi Biotec | 130-092-545 | |
| autoMACS Running Buffer | Miltenyi Biotec | 130-091-221 | |
| autoMACS Washing Buffer | Miltenyi Biotec | 130-092-987 | |
| autoMACS Columns | Miltenyi Biotec | 130-021-101 | |
| Whole Blood CD56 MicroBeads, human | Miltenyi Biotec | 130-090-875 | |
| ImageStream X Mark II Imaging Flow Cytometer | EMD Millipore | ||
| Speedbeads | Amnis Corporation | 400030 | |
| 0.4-0.7% Hypochlorite (Sterilizer) | VWR | JT9416-1 | |
| Coulter Clenz | Beckman Coulter | 8546929 | |
| 70% Isopropanol (Debubbler) | EMD Millipore | 1.3704 | |
| D-PBS (Sheath fluid) | EMD Millipore | BSS-1006-B (1X) | No calcium or magnesium |
| INSPIRE Software | EMD Millipore | Version Mark II, September 2013 | |
| Ideas Application Software | EMD Millipore | Version 6.1, July 2014 |
Referencias
- Cerwenka, A., Lanier, L. L. Natural killer cells, viruses and cancer. Nat Rev Immunol. 1 (1), 41-49 (2001).
- Nieman, D. C. Exercise, infection, and immunity. Int J Sports Med. 15, 131-141 (1994).
- Romeo, J., Warnberg, J., Pozo, T., Marcos, A. Physical activity, immunity and infection. Proc Nutr Soc. 69 (3), 390-399 (2010).
- Nieman, D. C. Marathon training and immune function. Sports Med. 37 (4-5), 412-415 (2007).
- Simpson, R. J., Kunz, H., Agha, N., Graff, R. Exercise and the Regulation of Immune Functions. Prog Mol Biol Transl Sci. 135, 355-380 (2015).
- Walsh, N. P., et al. Position statement. Part one: Immune function and exercise. Exerc Immunol Rev. 17, 6-63 (2011).
- Timmons, B. W., Cieslak, T. Human natural killer cell subsets and acute exercise: a brief review. Exerc Immunol Rev. 14, 8-23 (2008).
- Westermann, J., Pabst, R. Distribution of lymphocyte subsets and natural killer cells in the human body. Clin Investig. 70 (7), 539-544 (1992).
- Boyum, A. Isolation of leucocytes from human blood. Further observations. Methylcellulose, dextran, and ficoll as erythrocyteaggregating agents. Scand J Clin Lab Invest Suppl. 97, 31-50 (1968).
- McMillan, R., Scott, J. L. Leukocyte labeling with 51-Chromium. I. Technic and results in normal subjects. Blood. 32 (5), 738-754 (1968).
- Berk, L. S., et al. The effect of long endurance running on natural killer cells in marathoners. Med Sci Sports Exerc. 22 (2), 207-212 (1990).
- McAnulty, L. S., et al. Effect of blueberry ingestion on natural killer cell counts, oxidative stress, and inflammation prior to and after 2.5 h of running. Appl Physiol Nutr Metab. 36 (6), 976-984 (2011).
- Millard, A. L., et al. Brief Exercise Increases Peripheral Blood NK Cell Counts without Immediate Functional Changes, but Impairs their Responses to ex vivo Stimulation. Front Immunol. 4, 125 (2013).
- Janeway, C. A., Travers, P., Walport, M., Shlomchik, M. J. . Immunobiology: The Immune System in Health and Disease. 5th edn. , (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados