Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Diseño experimental para microdisección láser RNA-Seq: Lecciones de un análisis del desarrollo de la hoja de maíz
En este artículo
Resumen
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Resumen
Los genes con funciones importantes en el desarrollo con frecuencia tienen patrones de expresión espacial y / o temporalmente restringidas. A menudo, estas transcripciones de genes no se detectan o no son identificados como diferencialmente expresado (DE) en los análisis transcriptomic de órganos de plantas enteras. La microdisección láser RNA-Seq (LM RNA-Seq) es una poderosa herramienta para identificar los genes que se encuentran en dominios DE específicas de desarrollo. Sin embargo, la elección de los dominios celulares para microdissect y comparar, y la exactitud de los microdisecciones son cruciales para el éxito de los experimentos. A continuación, dos ejemplos ilustran las consideraciones de diseño para los experimentos de transcriptómica; un LM análisis de RNA-seq para identificar los genes que están De lo largo del eje proximal-distal hoja de maíz, y un segundo experimento para identificar los genes que son DE en liguleless1-R (LG1-R) mutantes en comparación con el de tipo salvaje. Los elementos clave que contribuyeron al éxito de estos experimentos se detallan histológico y en SIanaliza tu hibridación de la región a analizar, la selección de los primordios de la hoja en las etapas de desarrollo equivalentes, el uso de puntos de referencia morfológicas para seleccionar regiones para microdisección y microdisección de los dominios medidos con precisión. Este documento proporciona un protocolo detallado para el análisis de las áreas de desarrollo de LM RNA-Seq. Los datos aquí presentados ilustran cómo la región seleccionada para microdisección afectará a los resultados obtenidos.
Introducción
La hoja de maíz es un modelo ideal para estudiar la formación de campos de desarrollo durante la morfogénesis, ya que tiene un límite distinto entre la cuchilla y la vaina que es susceptible a la disección genética (Figura 1A). Durante las primeras etapas de desarrollo de las hojas, una banda lineal de células más pequeñas, la banda preligule (PLB), subdivide el primordio de la hoja en dominios pre-cuchilla y pre-vaina. Un lígula franja similar y aurículas triangulares desarrollan a partir de la PLB (Figura 1 A, C, D). cribados genéticos han identificado mutaciones que alteran la frontera hoja-vaina. Por ejemplo, liguleless1 recesivo (LG1) mutaciones eliminar la lígula y aurículas 1, 2, 3, 4 (Figura 1B). La hibridación in situ reveló que la transcripción lg1 se acumula en el PLB y lígula emergentes, por lo que es un excelente marcador para el desarrollo lígula 5, 6 (Figura 1E).
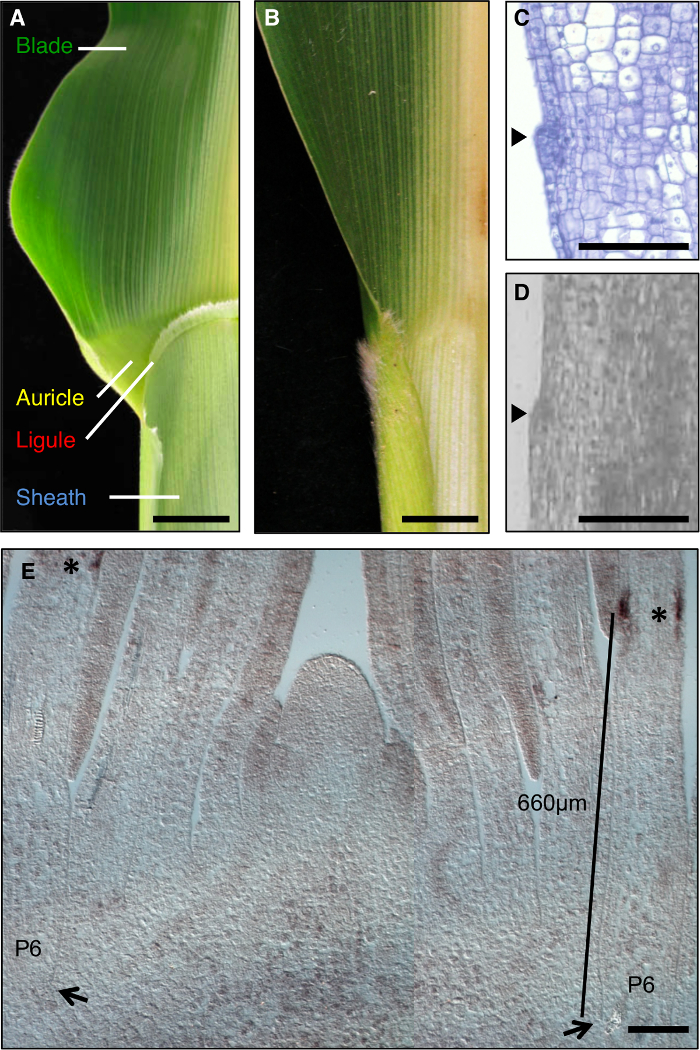
Figura 1: De tipo salvaje y hojas de maíz liguleless1-R. (A) zona de borde de la lámina de la vaina de la hoja de tipo salvaje madura que muestran estructuras lígula y la aurícula. (B) zona de borde de la lámina de las vainas maduras de liguleless1-R hoja que muestra ausencia de estructuras lígula y la aurícula. Hojas en A y B se han reducido a la mitad a lo largo del nervio central. (C) longitudinal a través de la sección primordio de hoja de tipo salvaje. Muestra que se ha procesado y se tiñeron para el análisis histológico. La lígula iniciar es evidente como una protuberancia que sobresale desde el plano de la hoja (punta de flecha). (D) secta Longitudinalprimordio de iones a través de la hoja de tipo salvaje. Muestra que se ha procesado para LM como se describe en el texto. Punta de flecha indica el inicio lígula. (E) lg1 hibridación in situ de rodaje sección longitudinal lateral ápice. Los asteriscos indican la acumulación de transcripción lg1 en el PLB del primordio de hoja P6. Las flechas indican la base de P6 primordio. La barra indica la medición de la base del primordio de la PLB. Las barras de escala en A y B = 20 mm. Las barras de escala en CE = 100 m. Esta cifra se ha modificado de la referencia 6 (Derecho de Autor Sociedad Americana de Biólogos Vegetales). Haga clic aquí para ver una versión más grande de esta figura.
En este estudio, se empleó LM RNA-Seq para identificar un conjunto de genes que se expresan diferencialmente (DE) en el límite de la hoja de la vaina en relación con otras partes del primordio de la hoja y a IDE ntify genes que están en lg1 DE-R mutantes en relación con los hermanos de tipo salvaje. LM RNA-Seq es un método de cuantificación de la acumulación de transcripción en células específicas o dominios celulares 7. sistemas LM combinan un láser y un microscopio con una cámara digital. tejido seccionado se monta en portaobjetos y se ve a través del microscopio. El software LM típicamente incluye herramientas de dibujo que permiten al usuario para delinear cualquier región seleccionada para la microdisección. Los cortes de láser a lo largo de la línea, y el tejido seleccionado es catapultado fuera de la diapositiva y en un tubo suspendido por encima de la diapositiva. LM permite al usuario microdissect dominios precisas, incluyendo capas de células específicas e incluso células individuales 8, 9. ARN se puede extraer del tejido microdissected. Posteriormente, el componente de RNA-Seq utiliza secuenciación de próxima generación para secuenciar bibliotecas de ADNc generados a partir de ARN extraído 10,= "xref"> 11.
Las principales ventajas de LM ARN-ss son la capacidad de cuantificar la acumulación de transcripción en dominios definidos con precisión y la capacidad para perfilar todo el transcriptoma simultáneamente 7. La técnica es particularmente adecuada para el sondeo acontecimientos de desarrollo tempranos en la región de interés es a menudo microscópico. Estudios anteriores han utilizado LM combina con la tecnología de microarrays para estudiar los procesos de desarrollo en plantas 9, 12, 13. RNA-Seq tiene la ventaja de la cuantificación de los transcritos en un amplio rango dinámico, incluyendo genes de bajo expresado, y la información de secuencia anterior no se requiere 10, 11. Por otra parte, LM RNA-Seq tiene el potencial para resaltar genes de desarrollo importantes que pueden perderse en las pantallas de mutagénesis debido a la redundancia genética o a la letalidad de la pérdida-de-función mutante.
Genes importantes para el desarrollo, tales como sheath1 estrecha (NS1) y en forma de copa cotyledon2 (CUC2), a menudo tienen patrones de expresión específica de sólo una o unas pocas células 17, 18, 19, 20. Muchos se expresan solamente durante las etapas tempranas del desarrollo y no en el órgano maduro. Cuando se analizan órganos enteros o grandes dominios, estos transcritos específicos de células se diluyen y no pueden detectarse en los análisis más convencionales. Al permitir que los análisis de dominios definidos con precisión, LM RNA-Seq permite que estos genes específicos de tejido para ser identificados y cuantificados.
factores cruciales para el éxito de los experimentos descritos aquí fueron un análisis histológico exhaustivo que guió a la selección de la etapa de desarrollo apropiado y dominio para el análisis y la medida PA precisant de dominios de células de tejidos para LM. Para asegurar que los dominios equivalentes se tomaron muestras para todas las repeticiones, se recogió tejido de primordios de la hoja en la misma etapa de desarrollo y los dominios microdissected se mide en relación con puntos de referencia morfológicos tales como la lígula emergente (Figura 2). Se sabe que algunos genes se expresan en un gradiente desde la punta hasta la base de la hoja. Mediante la medición de dominios precisos, debido a la variación de muestreo de diferentes lugares a lo largo del eje proximal-distal de la hoja se mantiene al mínimo (Figura 3A). Por microdissecting dominios del mismo tamaño, la variación debido a la diferencia de dilución de los transcritos específicos de células también se redujo (Figura 3B). secciones longitudinales laterales del ápice del brote se utilizaron para todos microdisecciones. Estos son secciones que son perpendiculares al eje nervio central margen (Figura 4). Usando sólo las secciones que incluyen la SAM asegura que las regiones laterales equivalentes deSe analizan primordios foliares.
En las muestras procesadas y seccionados para LM, la primera señal morfológica del crecimiento de lígula es una protuberancia en el lado adaxial debido a las divisiones celulares periclinales en la epidermis adaxial (Figura 1 D, Figura 2). Se determinó que la lígula emergente podría ser fiable identificado en plastocrono 7 primordios foliares etapa. Estábamos interesados en los genes expresados en toda la región lígula, incluyendo la lígula emergentes y las células inmediatamente distales que formarán la aurícula. Con el fin de asegurar que se hicieron selecciones de tejidos equivalentes, la protuberancia lígula se utilizó como un punto de referencia morfológica y un rectángulo 100 micras centrado en la protuberancia lígula fue seleccionado para LM (Figura 2A, 2B). rectángulos de tamaño equivalente de pre-lámina y pre-envoltura fueron seleccionados de las mismas primordios foliares.
Los análisis de las plantas mutantes liguleless presentaron un challe diferenteESN; LG1-R mutantes no forman una lígula, por lo tanto, esta característica morfológica no se podría utilizar para seleccionar la región de LM. En lugar de ello, se determinó el dominio de la acumulación de transcripción lg1 en los primordios de la hoja de tipo salvaje, y se definió una región que abarque este dominio. Estos análisis preliminares se realizaron en plantas de semillero de la misma plantación que se utilizaron para el análisis final, ya que el trabajo previo ha demostrado que la ubicación de la PLB varía dependiendo de las condiciones de crecimiento. La hibridación in situ indicó que los transcritos LG1 se acumulan en el PLB de P6 primordios foliares (Figura 1E). Hemos seleccionado un dominio de 400-900 micras desde la base de la primordios foliares que abarcaba el dominio de la expresión lg1 (rectángulos de color violeta, Figura 2A) y capturó estas regiones equivalentes de tipo salvaje y las plantas lg1-R. Para minimizar la variación en el fondo de crecimiento y condiciones genéticas cuando se comparan trascripciónSe utilizaron t acumulación en lg1-R y plantas de tipo salvaje, la segregación de las familias de los mutantes y de tipo salvaje hermanos.
Protocolo
NOTA: Fijar tejido para el análisis histológico, al mismo tiempo que el tejido se fija por LM. Examinar secciones teñidas para las características morfológicas que orientarán más tarde LM. Al comparar mutante de tipo salvaje, lleve a cabo la hibridación in situ o inmunolocalización para definir el dominio en el que se expresa el gen de interés (en este caso lg1).
1. fijación del tejido y Procesamiento
- Crecer pisos de plántulas de maíz de dos semanas de edad en condiciones estándar 6.
- Diseccionar ápices de las secciones laterales (Figura 4).
- Impuestos Especiales de plántulas justo por debajo de la línea del suelo.
- Utilizando una hoja de afeitar, eliminar rodajas finas de la base del tallo (cortes 1, Figura 4A) hasta un óvalo de caña rodeado por una o dos hojas maduras es visible (Figura 4B).
- Hacer otro corte de aproximadamente 10 mm por encima de la base (corte 2, Figura 4A).Este segmento de 10 mm contendrá el SAM y primordios foliares jóvenes.
- Gire el segmento de 10 mm para que la base quede hacia arriba. Hacer dos cortes paralelos al eje lateral de manera que se obtiene un trozo de tejido de 2-3 mm de espesor (cortes 3, y 4, la Figura 4B). Desechar las dos partes exteriores y retener la rebanada central para la fijación e inclusión.
NOTA: hojas exteriores pueden ser recortados y se descartan.
- Fijar un tejido y un proceso para la incrustación.
- Asegúrese de que todos los materiales que se utilizarán en las etapas subsiguientes son RNasa libre. Tratar soluciones con pirocarbonato de dietilo (DEPC) (1 ml DEPC por litro de solución. Incubar durante la noche con agitación ocasional, y autoclave). cristalería Hornear en un horno a 200 ° C o más durante al menos 6 horas y el tratamiento de artículos de plástico con una solución de descontaminación RNasa.
- Día 1: Sumergir las rebanadas de tejido en ~ 10 ml de solución de Farmer (3: 1 de etanol: ácido acético) en un vial de vidrio sobre hielo. Después de todas las muestras han sido disecados, aplicar vacuum para eliminar las burbujas de aire y la penetración ayuda de fijador. Mantenga al vacío durante 10 minutos y después se liberan lentamente el vacío. Reemplazar fijador y se incuba a 4 ° C durante la noche con agitación suave.
- Día 2: Incubar en la siguiente serie de soluciones, ~ 10 ml cada uno, 1 hr cada uno, todo con agitación suave; 85% de etanol a 4 ° C, 95% de etanol a 4 ° C, 100% de etanol a 4 ° C, 100% de etanol a 4 ° C, 100% de etanol a 4 ° C, 1: 1 etanol: xilenos a temperatura ambiente, 100% xilenos a temperatura ambiente, 100% xilenos a temperatura ambiente.
NOTA: Los xilenos son tóxicos por contacto y la inhalación. Trabajar en una campana de extracción y el uso de guantes apropiados. - Añadir tejido parafina pelets medio a aproximadamente medio volumen de xilenos y se incuba durante la noche a temperatura ambiente con agitación suave.
- Día 3: Traslado del vial a 60 ° C horno hasta que los gránulos se funden. Derrame la solución y reemplazar con fresco medio de inclusión de tejido derretida. Cambiar el medio dos veces másdurante el día.
- Día 4: Cambiar el tejido medio de inclusión vez por la mañana. Regreso a 60 ° C horno hasta la tarde.
- bloques elenco
- Colocar los moldes de incrustación en la placa caliente de la estación de incrustación de tejidos. El uso de fórceps para transferir las muestras de tejido de los moldes de la incrustación con la superficie cortada hacia abajo. Rellenar el molde con parafina derretida y colocar el anillo de incrustación en la parte superior del molde. Pasar a un plato frío hasta que la parafina se ha solidificado. Almacenar los bloques de parafina a 4 ° C en un recipiente hermético con gel de sílice.
2. Preparación de seccionamiento y diapositiva
- Cortar 10 micras secciones en un micrótomo 25.
- Examine las cintas y elegir secciones intermedias. La mediana de secciones son los que incluyen la SAM, que aparece como una cúpula de las células rodeadas por primordios foliares.
- secciones de montaje de diapositivas.
- Colocar los portaobjetos que son adecuados para LM (ya sea libre o RNasaal horno) en 42 ° C deslice más caliente y aplicar varias gotas de solución de etanol al 50% para cubrir la diapositiva.
- Flotar secciones en solución de etanol hasta que las secciones se han ampliado.
NOTA: Flotación de cortes en solución de etanol en vez de agua mantiene en un estado de ARN precipitado reducir la degradación del ARN. - Incline diapositiva y eliminar el exceso de solución de etanol por aspiración con una pipeta de transferencia desechable. Use toallitas sin pelusa para secar la solución de etanol adicionales.
- diapositivas de secado a 42 ° C durante varias horas o toda la noche. Tienda desliza a 4 ° C en un recipiente hermético con gel de sílice.
- Desparafinar desliza sobre el día de uso.
- Preparar tres frascos de vidrio que contienen Coplin; 100% xilenos (xilenos I), 100% xilenos (xilenos II), y 100% de etanol (~ 50 ml de cada solución).
- El uso de pinzas limpias para transferir diapositivas, sumerja diapositivas en xilenos yo por 2 min, xilenos II durante 2 min, y 100% de etanol durante 1 min.
- drenaje deslizadoes sobre toallitas sin pelusa y secar al aire a temperatura ambiente.
3. La microdisección de Blade, lígula y la vaina muestras de plastocrono 7 primordios foliares
- Asegure las diapositivas en el escenario de microscopio LM. Utilice cinco o seis diapositivas para cada réplica, utilizando cinco secciones por portaobjetos.
NOTA: Tissue la puesta en común de una sola réplica se ilustra en la Figura 5. - Examinar los portaobjetos e identificar las cinco secciones más la mediana en cada diapositiva, utilizando el SAM ápice como el punto de referencia central.
NOTA: Esto se puede hacer a bajo aumento, por lo general un objetivo de 5X es suficiente. - Uso de 10X o 20X objetivo, identificar la posición de la lígula en la plastocrono 7 primordio de la hoja de cada sección. La lígula será visible como una protuberancia que sobresale de la superficie adaxial del primordio de hoja. Marque esta posición utilizando la herramienta de dibujo del software LM; seleccione el icono del lápiz, mueva el cursor a la positi apropiadaen y haga clic y arrastre el ratón para dibujar.
NOTA: Una 10X o 20X objetivo es apropiada para esto y los pasos subsiguientes. Al usar secciones laterales, los dos lados de cada primordio de hoja estarán presentes en cada sección (Figura 2A). - Utilizando la herramienta de la regla y la herramienta de dibujo rectangular, medir 100 m rectángulos altos centradas en la lígula de cada sección (rectángulos rojos, la Figura 2A, 2B). Estos serán la muestra "lígula".
- Para utilizar la herramienta regla; seleccione el icono regla, mueva el cursor a un extremo del objeto a medir, hacer clic y arrastrar para medir el objeto. La longitud de la regla se mostrará en la pantalla.
- Para dibujar un rectángulo; seleccione el icono rectángulo, mover el cursor a un punto que será una de las esquinas del rectángulo, haga clic y arrastre para dibujar un rectángulo del tamaño apropiado. Como alternativa, seleccione la herramienta de dibujo de línea recta y dibujar cuatro líneas rectas.
- medida 100 micras rectángulos posicionados 50 m por encima y por debajo del rectángulo "Lígula".
NOTA: Estos serán los "Blade" y "muestras" de la envoltura, respectivamente (verde y azul rectángulos, Figura 2A, 2B). En base a los datos histológicos, un rectángulo 100 micras abarca toda la región lígula. porciones de tamaño equivalente de la hoja y la vaina fueron elegidos de forma que se recogieron cantidades similares de tejido para cada uno. Se usaron espaciadores de 50 micras para garantizar que no tejido región lígula se inadvertidamente incluida en la microdisecciones cuchilla o de la vaina.
- Microdissect medido rectángulos (Figura 2D - 2F) 7, 8, 9, recogiendo muestras Lígula, la cuchilla y la vaina en tubos separados. Utilice la función de corte láser para cortar a través de la sección de tejido a lo largo del contorno del dominio seleccionado. Utilice la catafunción pult para propulsar el rectángulo de tejido fuera de la diapositiva y en la tapa del tubo (Figura 2D - 2F).
4. La microdisección de Blade, lígula y la envoltura adaxial epidérmicas Las muestras de plastocrono 7 primordios foliares
- Seleccionar secciones y utilizar la herramienta de regla para medir 100 micras segmentos altos centrados en la lígula plastocrono 7, tal como se describe en la sección 3 (arriba).
- Seleccionar sólo las células de la epidermis adaxial de cada 100 micras alta "Blade" y "la envoltura" del segmento (verde y azul selecciones, Figura 2C) esbozando con la herramienta de dibujo. La epidermis es la capa celular externa; el lado adaxial es el más cercano a la SAM.
- Para la muestra "lígula", seleccionar sólo las células de la protuberancia emergente lígula como se describe en la Sección 3.3 (selección de color rojo, Figura 2C).
- Microdissect regiones seleccionadas, la recogida de la hoja, lígula y Sheaº epidermis muestras en tubos separados, tal como se describe en la sección 3.5.
5. La microdisección de plastocrono 6 primordios foliares de lg1-R y de tipo salvaje Hermanos
- Crecer la segregación de las familias de los mutantes (LG 1-R) y plantas de tipo silvestre.
- Fijar y disparar proceso de ápices para LM, como se describe en las secciones 1.2-1.4. Fijar de tipo salvaje y mutantes ápices en viales separados con el fin de mantenerlos separados. Las muestras de la misma plantación deben ser fijados y procesados para hibridación in situ.
- Determinar dónde lg1 se transcribe en los hermanos de tipo salvaje, mediante la realización de lg1 hibridación in situ 6, 26, 27. La posición de la acumulación de transcripción lg1 desde la base del primordio de la hoja en múltiples muestras (Figura 1E) medir.
- Sobre la base de los datos de hibridación in situ, elegir porción de la hoja primordium que abarca la región en donde se transcribe lg1. En este caso, 400 a 900 micras desde la base de plastocrono 6 primordios foliares (rectángulos de color violeta, Figura 2A).
- Microdissect parte de primordios foliares seleccionada, como se describe en la sección 3.5, la recogida de muestras de tipo salvaje lg1-R y en tubos separados.
6. Aplicar RNA Extraction Buffer
- Aplique 50 l de tampón de extracción de RNA para el tejido microdissected y proceder con la extracción de RNA. Continuar con la extracción de RNA, la amplificación del ARN, la construcción de bibliotecas, secuenciación y análisis bioinformático como se describe en la referencia 6.
Resultados
Usando el esquema LM indica en la Figura 2, aproximadamente 1,000,000-1,500,000 m 2 de tejido se recogió para cada replican en el todos-Cell-capas LM (Figura 5), y 200.000 m 2 por réplica para la epidermis adaxial LM. Aproximadamente 2.500.000 m 2 de tejido se recogió para cada réplica en el LM de lg1-R y primordios foliares de tipo salvaje. Dos rondas de amplificación de ARN lineal produjeron cantidades de ...
Discusión
Diseño experimental es un factor crítico en los experimentos de RNA-seq. Las principales consideraciones son el dominio preciso (s) y la etapa de desarrollo (s) para ser analizados, y se hará lo que las comparaciones. Es crucial para pensar en términos de comparaciones, ya que la salida es normalmente una lista de genes que se de entre dos o más condiciones. Al igual que con todos los experimentos, es importante para alterar sólo una variable a la vez. Por ejemplo, al comparar los diferentes dominios de la hoja, l...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
Los autores agradecen a S. Hake para la colaboración en curso y estimular los debates sobre el desarrollo lígula. Este trabajo es apoyado por la National Science Foundation Grants MCB 1052051 y IOS-1848478.
Materiales
| Name | Company | Catalog Number | Comments |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials - 22 ml | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 mm x 15 mm x 5 mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60 °C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
Referencias
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados