Method Article
Una de alto rendimiento plataforma de microarrays de células para análisis correlativo de la diferenciación celular y las fuerzas de tracción
En este artículo
Resumen
La diferenciación celular está regulada por una serie de factores microambientales, incluyendo tanto la composición de la matriz y de material de sustrato propiedades. Se describe aquí una técnica utilizando microarrays de células en combinación con microscopía de fuerza de tracción para evaluar tanto la diferenciación celular y las interacciones célula-sustrato biomecánicas como una función del contexto microambientales.
Resumen
microarrays celulares microfabricados, que consisten en combinaciones de contactos impresos de biomoléculas sobre una superficie elástica hidrogel, proporcionan un alto rendimiento sistema de ingeniería estrechamente controlado para medir el impacto de las señales bioquímicas dispuestas sobre la diferenciación celular. Los esfuerzos recientes utilizando microarrays de células han demostrado su utilidad para estudios combinatorios en los que muchos factores microambientales se presentan en paralelo. Sin embargo, estos esfuerzos se han centrado principalmente en la investigación de los efectos de las señales bioquímicas en las respuestas de células. A continuación, presentamos una plataforma de microarrays de células con propiedades materiales sintonizables para evaluar tanto la diferenciación celular mediante inmunofluorescencia y célula-sustrato interacciones biomecánico mediante microscopía de fuerza de tracción. Para ello, hemos desarrollado dos formatos diferentes que utilizan hidrogeles de poliacrilamida de diferentes módulo de Young fabricado en cualquiera de portaobjetos de microscopio o platos de Petri con fondo de cristal. Proporcionamos best prácticas y reparación para la fabricación de microarrays de estos sustratos de hidrogel, el posterior cultivo de células en microarrays, y la adquisición de datos. Esta plataforma está bien adaptado para su uso en investigaciones de procesos biológicos para los que tanto bioquímica (por ejemplo, composición de la matriz extracelular) y biofísico (por ejemplo, sustrato de rigidez) señales pueden jugar significativa, la intersección de roles.
Introducción
Las interacciones entre las células y factores microambientales rodean mediar una gran variedad de procesos biológicos en todo el desarrollo, la homeostasis, y la patogénesis 1, 2, 3, 4 enfermedad. Estas interacciones microambientales incluyen la entrega de factores solubles a las células, la unión célula-matriz, y las interacciones célula-célula a través de la unión del ligando-receptor. Además de las consideraciones bioquímicas anteriores, los parámetros biofísicos, como las propiedades mecánicas de sustrato (por ejemplo, el módulo de Young, la porosidad) y forma de la célula, y mechanotransduction aguas abajo asociados han ganado cada vez más reconocimiento como mediadores clave de la diferenciación de células 5, 6, 7, 8, 9 10. Las señales resultantes de estas interacciones microambientales sirven como entradas a las redes de genes y vías de señalización. Por otra parte, estos componentes de células intrínseca también proporcionan retroalimentación al microambiente a través de factores secretados y enzimas que degradan la matriz, que completaron un complejo bucle co-regulación entre los programas genéticos de células intrínseca y factores microambientales de células extrínseca 5, 11, 12.
El uso de sistemas de ingeniería para la presentación controlada de factores microambientales ha demostrado ser útil en una variedad de diferentes contextos 13, 14, 15. Sistemas microfabricados en particular, han facilitado patrón espacial precisa de las proteínas y células, así como análisis altamente paralelizado a través de la miniaturización 13, 16, 17, 18, 19, 20, 21, 22. Microarrays de células representan un sistema de este microfabricado en el que las combinaciones de biomoléculas son el contacto-impreso sobre un sustrato de poliacrilamida elástico de hidrogel 23, 24, 25. La inclusión de componentes de células de adhesivo (es decir, proteínas de la matriz) permite la adhesión sostenida cultivo de células y en microarrays, que es seguido a menudo por el análisis de aguas abajo a través de inmunocitoquímica y reporteros fluorescentes. Células microarrays han sido productiva dirigida hacia el logro de una mejor comprensión del fenotipo de células hepáticas 23, 26, neural diferenciación precursora 27, Mammardecisiones y destino progenitora 28, embrionaria de células madre de mantenimiento / diferenciación 23, 29, 30, 31 metástasis del cáncer de pulmón, y la respuesta terapéutica en el melanoma 32. Recientemente hemos demostrado el uso de células microarrays para definir el papel de la matriz extracelular (ECM) composición de proteínas en la especificación endodermo 33, el hígado progenitora diferenciación 34, 35, y la respuesta a los fármacos de células tumorales de pulmón 36. En estas obras, nos hemos centrado en la ampliación de las capacidades combinatorias de la gama plataforma y explorar las intersecciones de señalización de las células intrínseca con la composición de la matriz extracelular y la biomecánica. Además, hemos puesto en marcha lecturas biofísicos en esta gama plataforma para proporcionar cuantitativamente la capacidad de caracrizar el papel de la contractilidad celular en la diferenciación de los procesos 35. Para ello, hemos integrado la microscopía de fuerza de tracción (TFM) con microarrays de células para permitir la evaluación de alto rendimiento de la tracción generada por células. TFM es un método ampliamente utilizado para medir las fuerzas de tracción generada por células y se ha proporcionado información significativa con respecto a la coordinación de una sola célula y la función a nivel de tejido con la composición y la biomecánica del microambiente local de 37, 38, 39, 40. Por lo tanto, la combinación de TFM con microarrays de células proporciona un sistema de alto rendimiento para la medición de parámetros biofísicos clave, fisiológicamente relevantes.
La plataforma de microarrays de células descrito aquí consiste en cuatro secciones: la fabricación de sustratos de poliacrilamida, fabricación de matrices, cultivo de células y lectura del ensayo y análisis de los datos. VerFigura 1 para un resumen esquemático de las tres primeras secciones experimentales; véase la Figura 2 para un resumen esquemático de la sección final con un enfoque en el análisis de los datos de inmunofluorescencia. Con el fin de adaptar la plataforma de microarrays de células para estudios de las interacciones célula-sustrato biomecánicos, hemos utilizado sustratos de poliacrilamida de módulo de Young sintonizable pero porosidad similares, por Wen et al. 41. Para habilitar las mediciones de TFM fuerzas ejercidas por las células en su sustrato, se implementó un formato de placa de Petri con fondo de vidrio, además del microscopio de vidrio grueso portaobjetos de frecuencia utilizada por otros grupos. Por lo tanto, esta plataforma de microarrays célula es capaz de mediciones paralelas de diferenciación celular a través de inmunofluorescencia en portaobjetos de microscopio y las fuerzas generadas por células a través de TFM en los platos con fondo de vidrio separados. También hemos aplicado varias mejoras en el enfoque analítico empleado normalmente en los microarrays de células. ESPECÍFICOSLy, en lugar de Z-paramétrica de puntuación de la intensidad global isla, que mide la intensidad de una sola célula y aplicar cuantil normalización con el fin de dar cuenta de las distribuciones no normales y con mayor precisión describir el comportamiento celular. Creemos que estas mejoras proporcionan una utilidad particular hacia la investigación de procesos biológicos en los que las señales bioquímicas y biofísicas tanto juegan papeles importantes, que se cruzan. Además, nuestras mejoras analíticos permiten la aplicación de microarrays de células para estudios de una serie de funciones celulares para los que una sola célula y el comportamiento a nivel de población divergen.
Protocolo
1. La fabricación de poliacrilamida Sustratos
- sustratos de vidrio limpias - ya sea portaobjetos de microscopio estándar de inmunofluorescencia de punto final o de 35 mm placas de Petri para el TFM con fondo de vidrio - con el fin de garantizar la óptima fabricación de poliacrilamida y la integridad de hidrogel durante el cultivo celular. Como alternativa, el uso de pre-limpiado sustratos de vidrio.
- Sumergir sustratos de vidrio de 0,25% v / v Triton X-100 en agua destilada (dH2O). Coloque sustratos en un agitador orbital durante 30 min.
- Retire la solución de Triton X-100 y enjuague sustratos 5 veces con dH2O Dejar sustratos sumergidos en el enjuague final y el lugar en un agitador orbital durante 30 minutos.
- Retire dH2O y sumergir los sustratos en acetona. Coloque sustratos en un agitador orbital durante 30 min.
- Eliminar la acetona y sumergir los sustratos en metanol. Coloque sustratos en un agitador orbital durante 30 min.
- Eliminar el metanol y enjuague sustratos 5 veces con dH 2 O. sustratos sumergirán en NaOH 0,05 N y colocar en un agitador orbital durante 1 h.
PRECAUCIÓN: NaOH es altamente cáustico y puede causar quemaduras en la piel y lesiones oculares graves. Use guantes protectores, ropa adecuada y protección para los ojos. - Retire la solución de NaOH y enjuague sustratos 5 veces con dH 2 O. El uso de aire comprimido filtrado para secar substratos y hornear a 110 ° C en una placa caliente hasta que esté seco (5 - 15 min). sustratos limpiados se pueden almacenar a temperatura ambiente indefinidamente.
- Silanizar sustratos de vidrio limpias con el fin de asegurar la fijación del hidrogel de poliacrilamida.
- Sumergir sustratos de vidrio limpias en 2% v / v de metacrilato de 3- (trimetoxisilil) propil recién preparado (3-TPM) en etanol. Coloque sustratos en un agitador orbital durante 30 min.
PRECAUCIÓN: 3-TPM es un líquido combustible. Mantener alejado del calor, chispas, llamas abiertas y superficies calientes y utilizar sólo en una campana de humos química. - Eliminar la solución de 3-TPM y sumergirse en sustratos ethanol. Coloque sustratos en un agitador orbital durante 5 min.
- Use aire comprimido filtrado para secar los sustratos y hornear a 110 ° C en una placa caliente hasta que esté seco (5 - 15 min). silanized sustratos se pueden almacenar a temperatura ambiente hasta por 1 mes.
- Sumergir sustratos de vidrio limpias en 2% v / v de metacrilato de 3- (trimetoxisilil) propil recién preparado (3-TPM) en etanol. Coloque sustratos en un agitador orbital durante 30 min.
- Opción 1: Fabrique hidrogeles de poliacrilamida en portaobjetos de microscopio de inmunofluorescencia para silanizados punto final.
- Preparar una solución de pre-polímero en dH 2 O con la acrilamida / porcentaje bisacrilamida deseada (w / v) de relación para fabricar sustratos con módulos de 4 kPa (4% de acrilamida, 0,4% de bisacrilamida), 13 kPa (6% de acrilamida, 0,45 de Young % de bisacrilamida), o 30 kPa (8% de acrilamida, 0,55% de bisacrilamida) y la porosidad similares, por Wen et al. 41. agitar hasta disolución clara y filtro con una jeringa de 0,2 micras. soluciones pre-polímero se pueden almacenar a 4 ° C durante 3 meses.
PRECAUCIÓN: La exposición a la acrilamida o bisacrilamida puede dar lugar a toxicidad aguda, neurotoxicity, y la irritación. Use guantes protectores, ropa adecuada y protección para los ojos. - Preparar una solución de fotoiniciador de 20% w / v Irgacure 2959 en metanol. Esta solución fotoiniciador no se puede almacenar y debe prepararse de nuevo cada vez.
- Mezclar el pre-polímero y soluciones de fotoiniciador en una relación de 9:: 1 (fotoiniciador pre-polímero). desgasificar opcionalmente con una cámara de vacío durante 15 min para eliminar las burbujas.
- Colocar los portaobjetos silanizados en una bandeja de secado de vidrio y pipetear 100 l de 9: 1 pre-polímero: solución de fotoiniciador en cada diapositiva. cubierta con cuidado cada diapositiva con un cubreobjetos de 22 mm × 60 evitando al mismo tiempo la creación de burbujas. Tenga en cuenta que el cubreobjetos impide la inhibición de la reacción de polimerización por el oxígeno.
- Bandeja de secado lugar en un agente de reticulación UV y exponer diapositivas a 365 nm UV A para 10 min (4 W / m 2). Optimizar el tiempo de polimerización según sea necesario. Ya las exposiciones de riesgo dificultad de retirar el cubreobjetos debido a overpolymerization. exposiciones más cortas runderpolymerization riesgo y aplicar una baja estabilidad de hidrogel.
- Sumergir los hidrogeles en dH2O durante 5 min. Retire cubreobjetos con una navaja, teniendo cuidado de no dañar los hidrogeles polimerizados.
- Deja hidrogeles en dH2O a temperatura ambiente por 1 - 3 d, cambiando dH2O diaria. Deshidratar hidrogeles a 50 ° C en una placa caliente hasta que se seque (15 - 30 min) y se almacena a temperatura ambiente durante un máximo de 3 meses.
- Preparar una solución de pre-polímero en dH 2 O con la acrilamida / porcentaje bisacrilamida deseada (w / v) de relación para fabricar sustratos con módulos de 4 kPa (4% de acrilamida, 0,4% de bisacrilamida), 13 kPa (6% de acrilamida, 0,45 de Young % de bisacrilamida), o 30 kPa (8% de acrilamida, 0,55% de bisacrilamida) y la porosidad similares, por Wen et al. 41. agitar hasta disolución clara y filtro con una jeringa de 0,2 micras. soluciones pre-polímero se pueden almacenar a 4 ° C durante 3 meses.
- Opción 2: Fabrique hidrogeles de poliacrilamida que contiene a perlas fluorescentes en los platos con fondo de vidrio de 35 mm silanizados Petri para la evaluación en vivo de las interacciones célula-sustrato utilizando TFM.
- Sonicar una solución madre de 1 m perlas fluorescentes durante 15 min para dispersar los agregados.
- Preparar una solución de pre-polímero en dH 2 O con la acrilamida / porcentaje bisacrilamida deseada (w / v) de relación para fabricar sustratos con módulos de 4 kPa (4% de acrilamida, 0,4% de bisacrilamida), 13 kPa (6% de acrilamida, 0 de Young0,45% de bisacrilamida), o 30 kPa (8% de acrilamida, 0,55% de bisacrilamida) y la porosidad similares, por Wen et al. 41. agitar hasta disolución clara y filtro con una jeringa de 0,2 micras. soluciones pre-polímero se pueden almacenar a 4 ° C durante 3 meses.
PRECAUCIÓN: La exposición a la acrilamida o bisacrilamida puede dar lugar a toxicidad aguda, neurotoxicidad, y la irritación. Use guantes protectores, ropa adecuada y protección para los ojos. - Añadir perlas fluorescentes a la solución de pre-polímero a una concentración final de 0,2% v / v y agitar para mezclar.
- Preparar una solución de fotoiniciador de 20% w / v Irgacure 2959 en metanol. Esta solución fotoiniciador no se puede almacenar y debe prepararse de nuevo cada vez.
- Mezclar las soluciones pre-polímero / grano y fotoiniciadores en una mezcla 9: 1 (pre-polímero / perlas: fotoiniciador) relación. desgasificar opcionalmente con una cámara de vacío durante 15 min para eliminar las burbujas.
- Colocar placas de Petri de 35 mm silanizada con fondo de cristal en una bandeja de secado de vidrio y pipet 20 l de 9: solución de fotoiniciador en el centro de cada plato: 1 pre-polímero / perlas. cubierta con cuidado cada portaobjetos con un cubreobjetos circular de 12 mm, evitando la creación de burbujas. Tenga en cuenta que el cubreobjetos impide la inhibición de la reacción de polimerización por el oxígeno.
- Con el fin de distribuir las perlas fluorescentes a la superficie del hidrogel, invertir los platos y dejar a temperatura ambiente durante 20 min, por Knoll et al. 42.
- Mientras que aún invertida, exponer los platos a 365 nm UV durante 10 minutos (4 W / m 2). Optimizar el tiempo de polimerización según sea necesario. Ya las exposiciones de riesgo dificultad de retirar el cubreobjetos debido a overpolymerization. Underpolymerization más corta exposición al riesgo y baja estabilidad de hidrogel.
- Sumergir hidrogeles en 0,1 M de 4- (2-hidroxietil) -1-piperazinetanosulfónico (HEPES) buffer y dejar a temperatura ambiente en la oscuridad durante la noche. Retire cubreobjetos con cuidado con una navaja de afeitar, teniendo cuidado de no dañar la de poLos hidrogeles son las autorizadas.
- Deshidratar hidrogeles a 50 ° C en una placa caliente hasta que se seque (15 - 30 min). Los hidrogeles se pueden almacenar a temperatura ambiente en la oscuridad durante 3 meses.
2. La fabricación de matrices
- Preparar los tampones para imprimir biomoléculas. Utilice la memoria intermedia de impresión adecuado a las biomoléculas de interés. factor de crecimiento (GF) tampón de impresión es en general adecuado para otras clases de moléculas, tales como ligandos de célula-célula.
- Para preparar 2 × ECM tampón de impresión proteína, añadir 164 mg de acetato de sodio y 37,2 mg de ácido etilendiaminotetraacético (EDTA) a 6 ml dH 2 O. Vortex y se incuba a 37 ° C para solubilizar completamente. Después de la solubilización, se añaden 50 l de pre-calentado Triton X-100 y 4 ml de glicerol. Vortex e incubar a 37 ° C para disolver de nuevo. Añadir 40 - 80 l de ácido acético glacial, titulando para ajustar el pH a 4,8. 2 x tampón de impresión de proteínas ECM se puede almacenar a 4° C durante 1 mes.
PRECAUCIÓN: El ácido acético es inflamable y corrosivo. Use guantes protectores, ropa adecuada y protección para los ojos. - Para preparar la impresión tampón GF 2 ×, añadir acetato de sodio 105,5 mg y 37,2 mg de EDTA al 6 de buffer fosfato salino ml (PBS). Vortex y se incuba a 37 ° C para solubilizar completamente. Después de la solubilización, añadir 100 mg 3 - [(3-colamidopropil) dimetilamonio] -1-propanosulfonato (CHAPS) y 4 ml de glicerol. tampón de impresión proteína GF 2 × se puede almacenar a 4 ° C durante 1 mes.
- Para preparar 2 × ECM tampón de impresión proteína, añadir 164 mg de acetato de sodio y 37,2 mg de ácido etilendiaminotetraacético (EDTA) a 6 ml dH 2 O. Vortex y se incuba a 37 ° C para solubilizar completamente. Después de la solubilización, se añaden 50 l de pre-calentado Triton X-100 y 4 ml de glicerol. Vortex e incubar a 37 ° C para disolver de nuevo. Añadir 40 - 80 l de ácido acético glacial, titulando para ajustar el pH a 4,8. 2 x tampón de impresión de proteínas ECM se puede almacenar a 4° C durante 1 mes.
- Preparar la placa fuente.
- En una microplaca de 384 pocillos con fondo en V, combinar volúmenes iguales de 2 x tampón de impresión con cada solución biomolécula al doble de la concentración objetivo.
NOTA: Una concentración objetivo apropiado para las proteínas ECM más comunes es 250 mg / ml mientras que las concentraciones de destino para otros tipos de factores dispuestos varían en función de la retención en el hidrogel y la función biológica. El volumen total en cada pocillo puede sertan bajo como 5 l y no necesita ser más de 15 l. Además de las combinaciones de biomoléculas de interés, incluir un marcador fluorescente dispuestos en el fin de facilitar el análisis de imágenes de aguas abajo. Utilice dextrano conjugado con rodamina (2,5 mg / ml). - Mezclar a fondo cada pocillo con la pipeta, teniendo cuidado de no generar burbujas. Centrifugar la microplaca fuente de 1 min a 100 x g. Fabricar utilizando microarrays de placas preparadas de origen en el mismo día y se almacenaron a 4 ° C hasta la fabricación de microarrays.
- En una microplaca de 384 pocillos con fondo en V, combinar volúmenes iguales de 2 x tampón de impresión con cada solución biomolécula al doble de la concentración objetivo.
- pasadores limpias de acuerdo con las instrucciones del fabricante antes de cada ejecución de microarrays de fabricación. Cargar pasadores limpias directamente en el cabezal de impresión de la microarrayer.
- Preparar microarrayer y programa utilizando el software del fabricante. Aunque los pasos siguientes son parte específica de la microarrayer particular usado aquí, el funcionamiento de la mayoría de microarrayers es similar.
- Encienda la unidad de humidificador, ajustar el punto de ajuste a 65% HR (sin-condensing), y esperar hasta que el reómetro coincide con el punto de ajuste. Coloque la placa de la fuente en el adaptador apropiado.
- Deshidratar sustratos de hidrogel a 50 ° C durante 15 minutos y colocar en el adaptador apropiado. El microarrayer tiene adaptadores para ambos portaobjetos de microscopio y microplacas. Para poner en orden placas de Petri con fondo de vidrio 35 mm, la carga de las vajillas en una microplaca de 6 pocillos y colocar la microplaca en el adaptador de microplacas en el arrayer.
- Ajustar los parámetros del programa para reflejar con precisión el diseño de la placa de fuente, los diseños de matriz, y el formato deseado (por ejemplo, portaobjetos de microscopio o microplaca que contenía 35 mm placas de Petri). Incluir etapas de lavado utilizando tanto el agua como el dimetilsulfóxido (DMSO) entre cada condición con el fin de evitar la mezcla y la contaminación cruzada.
- Iniciar la gama de fabricación, control de frecuencia no inferior a una vez cada hora que la humedad no ha caído por debajo de 65% de humedad relativa (sin condensación) y que las patillas no están obstruidos. Si el humidity ha caído de forma inesperada, poner en orden una pausa para llenar el humidificador y limpiar los tubos asociados de condensación. Si se obstruyen las clavijas, hacer una pausa en poner en orden para limpiar los pines o si no, sustituir con los pernos pre-limpiado. Tenga en cuenta que es posible de matriz múltiples tipos de biomoléculas secuencialmente sobre los mismos sustratos proporcionados suficiente tiempo de secado (es decir, 4 h a toda la noche).
- Una vez que el programa se ha completado, colocar las matrices fabricadas en una caja de diapositivas o microplaca cubierta con papel de aluminio a temperatura ambiente y 65% de humedad relativa (sin condensación) durante la noche. Tenga en cuenta que puede ser necesario para evaluar la calidad de matriz y la retención usando manchas de proteína en general o de inmunofluorescencia; ver Brafman y col. 25 para más detalles.
3. Cultivo celular y ensayo de lectura
- El día después de la fabricación, coloque sustratos dispuestos en la recámara 4-platos (portaobjetos de microscopio) o 6-así microplacas (placas de Petri) y sumergirse en el 1%v / v de penicilina / estreptomicina en PBS; utilizar 4 ml de diapositivas y 3 ml de platos. Exponer a la radiación UV C durante 30 min. Cambio de penicilina / estreptomicina solución para medios de cultivo celular.
- Recoger y contar las células. Semilla en matrices a 500 × 10 3 - 2 x 10 6 células / matriz en 4 ml por portaobjetos de microscopio y 3 ml por 35 mm placa de Petri. Se incuban los cultivos de matriz a 37 ° C y 5% de CO2 durante 2 - 24 horas o hasta que la formación de islas de células bien pobladas. Ajustar tanto la densidad de siembra y el tiempo que sea necesario para sus células y aplicación en particular. Underseeding (es decir, de baja densidad o el tiempo de la siembra) puede provocar una disminución de la población de matriz y los resultados biológicos sesgadas. Resiembra (es decir, de alta densidad o el tiempo de la siembra), podría disminuir la integridad matriz debido al desprendimiento de la isla.
- Después de permitir para la formación de islas de células, lavar los cultivos de matriz dos veces con medio de cultivo celular pre-calentada; utilizar de nuevo 4 ml de diapositivas y 3 ml de platos. optionaLLY agregar controles y tratamientos adecuados (por ejemplo, inhibidores de moléculas pequeñas, factores de crecimiento, etc.) de interés para el sistema biológico. Cambiar el soporte de las matrices de cada 1 - 2 d con el fin de mantener la concentración de cualquier tratamiento. Evaluar la expresión de marcadores de células y la función celular mediante inmunofluorescencia o célula-sustrato interacciones por TFM dentro de 1 - 5 días de iniciar cultivos de matriz - ver las Opciones 1 y 2 a continuación.
- Opción 1: Realizar inmunofluorescencia de punto final. Tenga en cuenta que la inmunofluorescencia de algunas proteínas puede requerir permeabilización más rigurosas utilizando metanol, etanol, o HCl. Debido al daño potencial para arrays, evaluar y optimizar cada protocolo de permeabilización antes de su uso en experimentos de mayor escala.
- Aspirar los medios de cultivo celular a partir de diapositivas de matriz en la recámara 4-platos y añadir 4 ml / diapositivas de recién preparada 4% v / v paraformaldehído (PFA) en PBS. Incubar durante 15 min a temperatura ambiente.
PRECAUCIÓN: ExposUre de PFA puede dar lugar a toxicidad aguda y también puede irritar o corroer la piel al contacto. Use guantes protectores, ropa adecuada y protección para los ojos y usar solamente en una campana de humos química. - Aspirar solución de PFA y lavar cada diapositiva 3 veces con 4 ml de PBS. En este punto, los portaobjetos fijados se pueden almacenar a 4 ° C durante 1 semana. Es aconsejable, sin embargo, para continuar a través immunolabeling y de montaje en el mismo día como la fijación con el fin de asegurar la integridad de la matriz.
- Aspirar PBS y añadir 4 ml / slide de 0,25% v / v Triton X-100 en PBS. Incubar durante 10 min a temperatura ambiente.
- Aspirar la solución de Triton X-100 y lavar cada diapositiva 3 veces con 4 ml de PBS. Añadir 4 ml / slide de 5% v / v de suero adaptado a las especies del anticuerpo secundario (por ejemplo, suero de burro para burro anticuerpos secundarios) en PBS y se incuba a temperatura ambiente durante 1 h.
- eliminar completamente la solución de bloqueo de cada diapositiva. Añadir 500 l / slide de anticuerpo primario diluido en 5% v / v de suero en PBS. Este volumen es suficiente para cubrir las matrices para ambos 1 h incubaciones a temperatura ambiente, así como las incubaciones durante la noche a 4 ° C.
- Lavar cada serie de diapositivas 3 veces con 4 ml de PBS. Eliminar completamente el lavado final y añadir 500 l / diapositivas del anticuerpo secundario apropiado diluido en 5% v / v de suero en PBS.
- Lavar cada serie de diapositivas 3 veces con 4 ml de PBS. Lavar brevemente con dH2O antes de retirar cuidadosamente diapositivas de la matriz de la solución utilizando pinzas. Utilice un pañuelo de papel para absorber el laboratorio o DH residual seca 2 O.
- Pipetear 100 l de solución con DAPI de montaje a través de la corredera mientras se confirma visualmente la cobertura completa de toda la matriz.
- Colocar un cubreobjetos de 22 mm × 60 sobre el portaobjetos para montar. Sellar los bordes del cubreobjetos con esmalte de uñas transparente. Almacenar en la oscuridad a 4 ° C hasta el momento de formación de imágenes, no antes del día siguiente.
- toda la imagen matrices usando ya sea un escáner de microarrays o invertida equipp microscopio de fluorescenciaed con una etapa de robótica. Escáneres de microarrays proporcionan lectura más rápida, pero pueden requerir Cy3 o Cy5 compatibles con fluoróforos y son a menudo de la resolución limitada en el orden de las celdas individuales (es decir, 1 - 10 micras). microscopios fluorescentes proporcionan la opción de utilizar una variedad de canales fluorescentes y una resolución más alta (<1 m, ~ 100 × ampliación total) pero proporcionan lectura más lenta dependiendo de la calidad de la etapa de robótica y de la ampliación / objetivo.
- Guardar las imágenes capturadas de arrays enteros desde cualquiera de los métodos como archivos TIFF con el fin de evitar la compresión de datos o pérdida asociada a otros formatos de archivo (por ejemplo, JPG).
- Aspirar los medios de cultivo celular a partir de diapositivas de matriz en la recámara 4-platos y añadir 4 ml / diapositivas de recién preparada 4% v / v paraformaldehído (PFA) en PBS. Incubar durante 15 min a temperatura ambiente.
- Opción 2: Realizar la evaluación en vivo de las interacciones célula-sustrato utilizando TFM.
- Preparar una solución de 1% v / v de albúmina de suero bovino (BSA) y 1% v / v de dodecilsulfato de sodio (SDS) en PBS para disociar las células de los sustratos durante TFM.
- Mover 35 mm placas de Petri que contienen cultivos de matriz deun incubaron (37 ° C, 5% de CO 2), microscopio de fluorescencia invertida con una etapa de robótica para las mediciones de TFM.
- En un plato, marcar las posiciones (coordenada X, coordenada Y) y se centran planos (coordenada Z) de las islas de células individuales utilizando microscopía de contraste de fase.
- Cambiar a la microscopía de fluorescencia roja lejana para visualizar las perlas. Volver a cada una de las posiciones guardadas en el paso anterior y corregir la coordenada Z del plano de enfoque de manera que sólo la primera capa de perlas por debajo de la isla de células está en el foco. Guardar las nuevas coordenadas y proceder a la imagen automatizada de todas las islas de células para captar contraste de fase previa a la disociación y las imágenes fluorescentes rojas lejanas.
- Añadir con cuidado 150 ml de solución de BSA / SDS al plato y esperar 5 minutos para permitir la disociación completa de las células del sustrato; monitorear la disociación de células mediante microscopía de contraste de fase.
- Después de las islas celulares se han disociado del sustrato, volver a tmarcó posiciones y compruebe que la primera capa de perlas están aún en el enfoque. Si estas perlas están fuera del plano debido a la deformación inducida por la tracción generada por células, a continuación, corregir la coordenada Z del plano de enfoque de modo que sean de nuevo en el foco. Guardar las coordenadas Z corregidos y repetir automatizado de imágenes de todas las islas para capturar posterior a la disociación de imágenes fluorescentes rojos lejos.
- Repita los pasos 3.5.1 - 3.5.4 para los platos restantes.
4. Análisis de los Datos
- Análisis de los datos de inmunofluorescencia.
- Proceso obtuvo imágenes de la matriz. Dividir serie de imágenes compuestas en archivos que contienen canales individuales (es decir, rojo, azul o verde) y convertir a las imágenes TIFF de 8 bits 43, 44. Aplicar hurgar en la basura (por ejemplo, 2 × 2 o 4 × 4) para reducir el tamaño de la imagen a ~ 32 megapíxeles por canal para reducir los requisitos de memoria durante el análisis de una sola célula de aguas abajo de la entire serie de imágenes. Ver archivo de código suplementario titulado "array_processing.ijm" para una macro de aplicación ImageJ de estos pasos de procesamiento de sensores.
- Tenga en cuenta las coordenadas en píxeles de la parte superior izquierda, abajo a la izquierda, y los marcadores de rodamina conjugada con la parte inferior derecha de dextrano o condiciones dispuestas. Utilice estas coordenadas para girar las imágenes de 8 bits TIFF para ser perfectamente vertical y, más tarde, para anotar la salida a partir del análisis de una sola célula con las condiciones dispuestas específicos. Ver archivos de código suplementario titulado "rb_array_rotater.ijm", "rg_array_rotater.ijm", "rgb_array_rotater.ijm", y "array_gridding.ijm" para las implementaciones de estos matriz de rotación y gridding pasos.
- Realizar el análisis de una sola célula de compresión igual, girar imágenes TIFF de 8 bits en CellProfiler (versión 2.1.1) 45 usando los siguientes módulos: IdentifyPrimaryObjects, IdentifySecondaryObjects, y MeasureObjectIntensity. IdentifyPrimaryObjects identifica núcleos, IdentifySecondaryObjects identifica immunolabels asociados a cada núcleo de la célula, y MeasureObjectIntensity proporciona cuantificaciones de las etiquetas y immunolabels nucleares.
- La salida de datos de una sola célula de los tres módulos como un archivo CSV por el canal utilizando el módulo de ExportToSpreadsheet para facilitar el análisis posterior aguas abajo. Ver archivos de código suplementario titulado "b_array_image_analysis.cppipe", "gb_array_image_analysis.cppipe", "rb_array_image_analysis.cppipe", y "rgb_array_image_analysis.cppipe" para las tuberías de ejecución CellProfiler estos pasos para conjuntos de imágenes que contienen canales rojo, verde o azul.
- Para transformar los datos para tener en cuenta la variabilidad experimental y distribuciones unicelulares no gaussiana, aplicar cuantil normalización biológica replicar por 46. Este proceso genera una distribución compartida a través de repeticiones y permite comparaciones imparciales de cambios en la intensidad inmunomarcador. Además, unlIke Z-puntuación y otros métodos paramétricos, cuantil normalización es no paramétrico y no supone una distribución particular de datos, lo que permite más representativa de análisis de la conducta de una sola célula en función del estado dispuestos.
- Trazar datos e interpretar. Dependiendo del sistema biológico y la hipótesis, calcular y trazar una o más de las siguientes medidas de conjunto para cada condición dispuestos:
- Calcular y trazar células por isla como una medida combinada de la adhesión y la supervivencia durante el transcurso del experimento.
- Calcular y representar gráficamente la intensidad inmunomarcador cuantil-normalizado como una medida del destino celular o función.
- Calcular y representar gráficamente el porcentaje de células positivas para un inmunomarcador como se determina por la intensidad por encima de un umbral constante, por lo general 2 SD por encima de la intensidad media de un control negativo.
- Alternativamente, las distribuciones de intensidad de la trama inmunomarcador el fin de examinar y clasificar el comportamiento de una sola célulacomo una función de la condición dispuestos. Estas distribuciones se pueden caracterizar aún más el uso de medidas de tendencia central (media, mediana, moda) y la variación (varianza, coeficiente de variación, el factor de Fano) y los métodos de prueba de hipótesis, como la prueba de Kolmogorov-Smirnov.
- Análisis de los datos TFM. Aquí se describe un enfoque que incorpora un algoritmo desarrollado previamente por Butler et al. y Wang et al. 40, 47.
- ImageJ utilizar para convertir por lotes las imágenes para archivos TIFF de 8 bits. Aplicar pixel binning promediada (por ejemplo, 2 × 2) para reducir el coste computacional y el tiempo de análisis de aguas abajo. Como algoritmos para TFM se han centrado en gran medida en el análisis de una sola célula, la gran interfase celular sustrato de las islas (~ 17,5 × 10 3 m 2) en comparación con la interfaz célula-sustrato de una sola célula (75 m 2) necessistados el paso hurgar en la basura.
- Introduzca el contraste de fase capturados y las imágenes fluorescentes rojas la fecha (tanto pre-y post-disociación de disociación) en un entorno de programación científica, tales como MATLAB y el proceso utilizando los algoritmos desarrollados anteriormente de Butler et al. y Wang et al. 40, 47.
- Seleccione tres regiones distantes de la isla de células. Estas regiones se usan para tener en cuenta los desplazamientos debidos a la imagen o la deriva de la muestra.
- Proporcionar el factor para convertir de píxeles para micrómetros (por ejemplo, 0.454 pixels / M), el módulo de Young del sustrato (por ejemplo, 13 kPa), y la relación de Poisson (por ejemplo, 0,48 para los geles de poliacrilamida que se describen aquí).
- Para cada isla, trazar una frontera en torno a la periferia para definir las limitaciones geométricas; todas las fuerzas fuera de este límite se ponen a cero. Este sistema restringido es razonable dada la gran distana vez (es decir, 450 micras) entre las islas.
- Calcular el esfuerzo de tracción de la raíz cuadrada de la media y el momento de contracción para cada isla. El momento contráctil es una medida de la tensión residual a través de la isla de células y se ha demostrado para reflejar la fuerza de las interacciones célula-célula 48. Para cada condición dispuestos, los valores de la raíz cuadrada de la media promedio de más de múltiples islas y repeticiones biológica y calcular asociados varianza para la prueba de hipótesis. También es posible promediar la distribución de tensiones o momentos sobre muchas islas para proporcionar un mapa representativo de las dos medidas como una función de la geometría, por ejemplo, la distancia desde el centro de la isla.
Resultados
El uso de esta plataforma, se investigó el papel de ambas señales bioquímicas y biofísicas en la especificación de destino de células progenitoras del hígado 34, 35. Ligandos / Notch G conjugada con proteína A mostraron una mejor retención y la agrupación en el hidrogel de poliacrilamida (Figura 3A) y además están capaz de impulsar la diferenciación de las células progenitoras del hígado hacia un destino celular de las vías biliares (Figura 3B). Utilizando el análisis de una sola célula, que cuantifica la respuesta a los ligandos de Notch para las proteínas ECM colágeno I, colágeno III, colágeno IV, fibronectina y laminina (Figura 3C), encontrando que la respuesta de las células progenitoras del hígado al ligando depende también de la contexto ECM. Por último, hemos utilizado shRNA desmontables para generar células progenitoras del hígado sin los ligandos Dll1 y Jag1. La respuesta al ligando de Notch dispuestos variaba dependiendo del presencia de cualquiera de ligando, lo que confirma que la capacidad de respuesta al ligando de células extrínseca es también una función de la expresión de ligando de células intrínseca (Figura 3D). Además, se observó una subpoblación distinta de doble positivo (+ ALB / OPN +) células en la caída Dll1 (Figura 3D). En conjunto, estos resultados muestran representativas: (1) las capacidades combinatorias del formato de matriz, como se ejemplifica por el emparejamiento de múltiples dispuestos proteínas ECM y ligandos Notch con la caída de los ligandos individuales; (2) la funcionalidad de no sólo vistió proteínas ECM, sino también vistió ligando célula-célula a través de Proteína A conjugación / mediada por G; y (3) la aplicación de nuestro análisis de una sola célula y su capacidad de discernir las subpoblaciones únicas.
También se observó que la diferenciación de células progenitoras del hígado depende tanto de la rigidez del sustrato y la composición de la MEC (Figura 4A ong>), encontrar específicamente que el colágeno IV es de apoyo de la diferenciación en ambos sustratos blandos y rígidos, mientras que la fibronectina sólo es compatible con la diferenciación en sustratos rígidos (Figura 4B). Mapas de calor representativas de las mediciones TFM sugieren que el estrés sostenido de tracción a baja rigidez sustrato sobre el colágeno IV promovió la diferenciación en células de los conductos biliares (Figura 4C), un hallazgo confirmado por los valores promedio de la raíz cuadrada media (Figura 4D). En conjunto, estos resultados muestran representativas: (1) la integración exitosa de TFM con microarrays de células en sustratos con una rigidez sintonizable para evaluar tanto el fenotipo celular y el esfuerzo de tracción; (2) la coordinación del destino de las células progenitoras hepáticas tanto con la composición de la matriz y la rigidez del sustrato; y (3) la aplicación de nuestros análisis y típicos perfiles de esfuerzo de tracción TFM en células microarrays.
e 1 "src =" / files / ftp_upload / 55362 / 55362fig1.jpg "/>
Figura 1: Descripción esquemática que muestra los primeros tres secciones experimentales. En la Sección 1, sustratos de vidrio se limpian y silanizada para facilitar la fabricación de hidrogeles de poliacrilamida. En la Sección 2, las combinaciones de biomoléculas de interés se preparan en una microplaca de fuente de 384 pocillos. Un arrayer robótico se carga entonces con alfileres limpias, la microplaca fuente, y los hidrogeles de poliacrilamida y se inicializa, la fabricación de matrices en los hidrogeles. En la sección 3, las células se siembran en los dominios dispuestos y permite que se adhieran, después de lo cual se realiza el protocolo de cultivo de interés. En el punto final, las células se fijan ya sea para inmunocitoquímica / inmunofluorescencia o analizados mediante TFM. Las barras de escala son 75 micras. Haga clic aquí para ver una versión más grande de esta figura.
dentro-page = "1"> 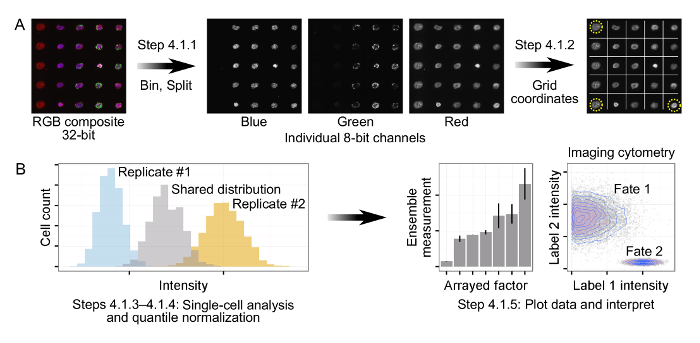
Figura 2: Procesamiento y Análisis de inmunofluorescencia de datos de matrices. (A) de baldosa, compuestas imágenes RGB de 32 bits se han agrupado primero y luego divide en canales individuales de 8 bits. Usando una combinación de marcadores fluorescentes dispuestos e islas de células, se identifican tres esquinas de la matriz para permitir la orientación automatizado y cuadriculación de las matrices. (B) los datos de celda única se genera para cada canal de las matrices de entrada. Con el fin de dar cuenta de la deriva experimental, cuantil normalización se aplica mediante biológica replicar, produciendo una única distribución compartida a través de todas las repeticiones. Cuantil normalizado de datos se representa posteriormente y se interpreta a través de cálculo de las mediciones del conjunto (por ejemplo, células / isla, intensidad media, porcentaje de células positivas para una etiqueta) o el análisis directo de la distribución de una sola célula.m / archivos / ftp_upload / 55362 / 55362fig2large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
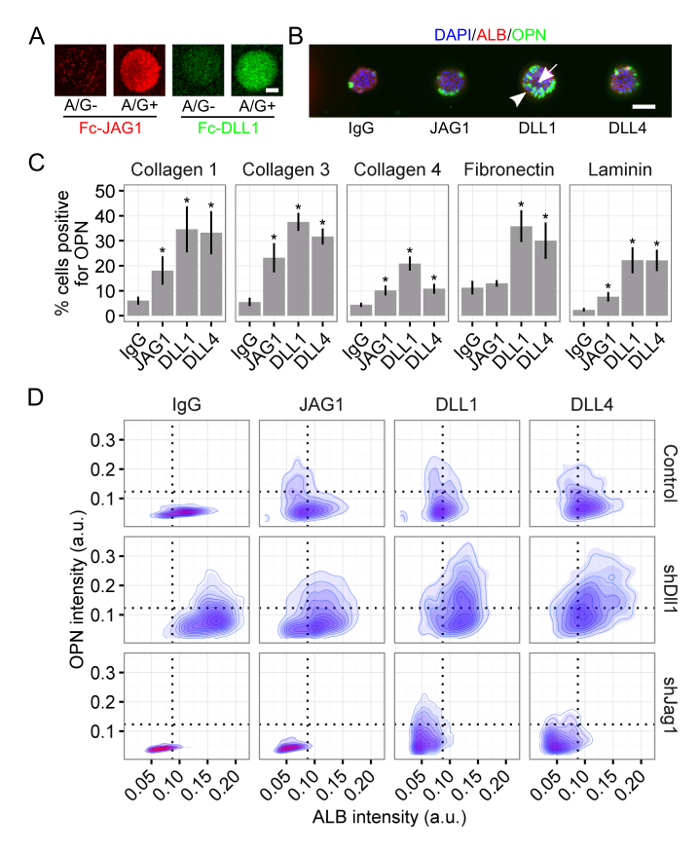
Figura 3: Presentación Notch ligando media la diferenciación hepática Progenitor. (A) ligandos de Notch-Fc recombinante Jagged-1 (JAG1) y Delta tipo 1 (DLL 1) exhibió una mejor retención y la agrupación cuando se vistió con proteína A / G. La barra de escala es de 50 micras. Progenitores (B) del hígado diferenciarse en células del conducto biliar mediante la presentación con el ligando de Notch. 4 ', 6-diamidino-2-fenilindol (DAPI) es una etiqueta nuclear, albúmina (ALB) es un marcador de células hepáticas, y osteopontina (OPN) es un marcador de células del conducto biliar. La barra de escala es de 150 micras. (C) Cuantificación de porcentaje de células positivas para OPN para los ligandos de Notch JAG1, DLL1, y Delta-como 4 (Dll4) en las proteínas ECM colágeno I, collagen III, colágeno IV, fibronectina y laminina. T de Student pruebas se llevaron a cabo contra IgG de control para cada ligando Notch dispuestos dentro de cada proteína de ECM con P-valores indicados para p <0,05 (*). (D) Imagen citometría de ALB y OPN para las células de colágeno III presentados con los ligandos Notch JAG1, Dll1, y Dll4. Células progenitoras del hígado sin la Notch ligandos Dll1 y Jag1 (es decir, shDll1 y shJag1) se generaron utilizando shRNA desmontables. Los datos en (C) presentan como media ± SEM Esta cifra ha sido modificado a partir de Kaylan et al. 34. Haga clic aquí para ver una versión más grande de esta figura.
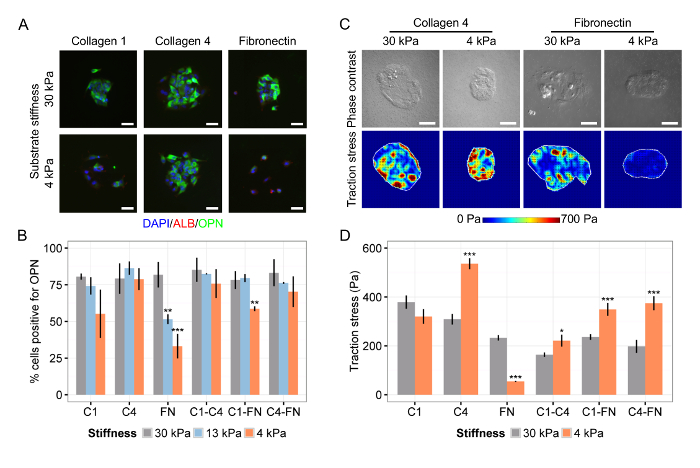
Figura 4: composición de la matriz y el sustrato Rigidez Coordinate hígado Progenitor diferenciación. (A) la diferenciación de progenitores de hígado a las células del conducto biliar depende tanto de composición de la MEC y la rigidez del sustrato. DAPI es una etiqueta nuclear, ALB es un marcador de células hepáticas, y de OPN es un marcador de células del conducto biliar. (B) Cuantificación de porcentaje de células positivas para OPN sobre sustratos de módulo de Young 30 kPa, 13 kPa, y 4 kPa para el colágeno I (C1), colágeno IV (C4), fibronectina (FN), y todos de dos vías combinaciones de aquellas proteínas de ECM. (C) la tensión de tracción de la célula depende tanto de la rigidez del sustrato y la composición ECM. (D) La cuantificación de los valores de la raíz cuadrada de la media de la tensión de tracción sobre sustratos de módulo de Young de 30 kPa y 4 kPa para el colágeno I (C1), colágeno IV (C4), fibronectina (FN), y todas las combinaciones binarias de aquellos proteínas ECM. En (B) y (D), los datos se presentan como media ± SEM y t de Student-pruebasse llevaron a cabo contra 30 kPa para cada combinación de ECM con P-valores indicados para p <0,05 (*), P <0,01 (**), y P <0,001 (***). Las barras de escala son 50 micras. Esta cifra ha sido modificado a partir de Kourouklis et al. 35. Haga clic aquí para ver una versión más grande de esta figura.
| Sección | Problema | Causas potenciales | Solución |
| 1. La fabricación de poliacrilamida sustrato. | Cubreobjetos no se puede quitar de hidrogel. | Overpolymerization. | Reducir el tiempo de polimerización a <10 minutos (4 W / m 2). Compruebe que crossli UVnker de salida está dentro del rango esperado. |
| polimerización hidrogel de poliacrilamida pobres. | Underpolymerization. | Aumentar el tiempo de polimerización para> 10 minutos (4 W / m 2). Compruebe que la salida de agente de reticulación UV está dentro del rango esperado. | |
| hidrogeles de poliacrilamida se dañan después de la eliminación de cubreobjetos. | hidrogeles de poliacrilamida suaves son fáciles de dañar. | Observamos la disminución de rendimiento de hidrogel de fabricación (~ 50%) para la más suave (es decir, 4 kPa) hidrogeles en particular. Manejar hidrogeles suavemente y aumentar los números de partida para alcanzar el rendimiento deseado. | |
| 2. La fabricación de matrices. | la morfología del terreno pobre o inconsistente. | función de humectación inconsistente. | Compruebe que humidificador y un reómetro funcional a través de cada tirada y mantener un 65% de humedad relativa. |
| Alfileres clavados en el cabezal de impresión o estorboGED. | Limpiar el cabezal de impresión para permitir el movimiento libre de pasador. Limpiar bien los pasadores antes o después de cada tirada para eliminar los agregados de canales de espiga. | ||
| 3. Cultivo de células y Ejecución del ensayo. | desprendimiento o muerte celular en matrices después de la colocación inicial. | Resiembra y la proliferación excesiva. | Reducir la densidad de siembra inicial y el tiempo. Utilice el "mantenimiento" o los medios de comunicación "diferenciación" durante el cultivo conjunto para reducir la proliferación celular. |
| La liberación de monómero de acrilamida tóxica de hidrogel. | Remojar hidrogeles en dH 2 O durante al menos 3 d para permitir la difusión / liberación de monómero de acrilamida y reducir la toxicidad celular. | ||
| Las células no se adhieren a las matrices. | Underseeding. | Aumentar la densidad de siembra inicial y el tiempo. Use un tipo de célula más fuertemente adherente. | |
| Poor deposición dematriz o condición biomolécula. | Pasadores limpias de partículas y agregados, confirman los parámetros de impresión, y evaluar la localización de los marcadores fluorescentes, por ejemplo, dextrano conjugado con rodamina. | ||
| La especificidad de las interacciones célula-matriz. | Diferentes tipos de células se adhieren específicamente a algunos pero no a otras proteínas ECM. Prueba de múltiples proteínas de la ECM con sus células. | ||
| almacenamiento de matrices subóptima después de la fabricación. | Recomendamos almacenar matrices fabricadas durante la noche a 65% de humedad relativa y temperatura ambiente, en parte para evitar cambios de fase durante la congelación. La adhesión celular es sensible tanto a la humedad, la temperatura y tiempo de almacenamiento; asegurarse de que estos parámetros son consistentes / optimizado para sus experimentos. | ||
| Desprendimiento de hidrogel de sustrato de vidrio durante el cultivo celular. | limpieza de diapositivas pobres y silanización. | Reemplazar las soluciones de trabajo para la limpieza de diapositivas ysilanización. | |
| Overdehydrated hidrogel. | No deje hidrogeles de deshidratación en un plato caliente durante más de 15-30 minutos. | ||
| 4. Análisis de Datos. | Alta variabilidad entre los puntos en paralelo y diapositivas. | La variabilidad en la matriz de fabricación. | Compruebe que los terminales y los cabezales de impresión están limpias. Confirmar la función humidificador. Visualizar y cuantificar lugar y la matriz calidad utilizando marcadores fluorescentes. matrices de las tiendas a las recomendaciones anteriores. |
Tabla 1: Solución de problemas.
Discusión
En nuestros experimentos, hemos encontrado que los fallos más comunes están relacionados con la calidad de las matrices fabricadas y pobremente caracterizados respuesta en el sistema biológico de interés. Remitimos al lector a la Tabla 1 para los modos de fallo comunes en los experimentos de microarrays de células y pasos para solucionar problemas asociados. En cuanto a la calidad de las matrices, en particular, se recomienda lo siguiente. Confirmar la calidad técnica y la solidez de poner en orden los programas, parámetros y tampones que usan moléculas marcadas con fluorescencia, tales como dextrano conjugado con rodamina. pasadores limpiar a fondo, ya sea antes o después de poner en orden según las instrucciones del fabricante, y aún más visualmente comprobar que los canales de espiga son libres de desechos utilizando un microscopio de luz. Confirmar la retención biomolécula puso en orden el uso de manchas de proteínas generales o inmunomarcaje. Tenga en cuenta que las biomoléculas con un peso molecular inferior a 70 kDa con frecuencia no se retienen en el hidrogel 23, sup> 31. Validar puso en orden el biomolécula célula funcionalidad mediante múltiples tipos de células. Tenga en cuenta que sólo las células adherentes son compatibles con las matrices; Además, la adherencia a las matrices depende tanto de las propiedades de células específicas (por ejemplo, perfil de expresión de integrina) y las proteínas de la MEC seleccionados.
Debido al espacio limitado, no hemos proporcionado un extenso tratamiento de la gama de diseño, el diseño y la fabricación aquí y remitir al lector a trabajos previos 23, 25. Generalmente usamos 100 subarreglos punto (150 micras de diámetro punto, 450 m de centro a centro de distancia) compuestas de 10-20 condiciones de biomoléculas únicos (es decir, 5-10 puntos / condición). El número de subconjuntos en una matriz varía en función del número de condiciones de biomoléculas de interés, que se puede escalar cómodamente hasta 1280 en un 25 × 75 mm portaobjetos de microscopio (~ 6.400 puntos en 64 submatrices)xref "> 25, 31 de los parámetros anteriores variarán más dependiendo del tamaño del patrón de interés;. pines capaces de generar patrones de 75 a 450 micras están fácilmente disponibles.
serie de experimentos se complementan con mejor validación de alta puntuación condiciones dispuestas de interés utilizando otros formatos de cultivo, las lecturas de ensayo, y sistemas de modelos biológicos. Específicamente, sugerimos validar aún más los efectos de las condiciones dispuestas seleccione utilizando cultivos en masa en conjunción con técnicas de biología molecular estándar (por ejemplo, QRT-PCR, inmunotransferencia) o estándar de TFM. La manipulación genética (por ejemplo, desmontables o sobreexpresión) del factor de interés en un sistema modelo biológico adecuado también puede servir para confirmar los efectos observados en las matrices. En modelos animales in vivo representan otro medio de validación y se utilizaron recientemente, por ejemplo, para confirmar el papel central de la galectina-3 y la galectina-8 enel cáncer de pulmón metastásico nicho, como se identificó inicialmente a través de microarrays de células 31, 49.
Un número de otros métodos se han utilizado para investigar la regulación microambientales de funciones celulares, incluyendo una variedad de dos dimensiones sistemas microfabricados 18, 50, 51, 52, 53, 54, 55 y tridimensionales sistemas de biomateriales de ingeniería 56, 57, 58 , 59, 60, 61. En comparación con otros métodos, las ventajas particulares de la plataforma de microarrays de células descritas aquí consisten en: (1) un rendimiento de hastacientos o miles de diferentes combinaciones de factores, lo que permite el análisis de los efectos de interacción; (2), de formación de imágenes automatizado accesible y análisis; (3) la integración de las dos lecturas bioquímicas y biofísicas con la presentación controlada de factores desplegadas; (4) capacidad de variar las propiedades del material de sustrato; y (5) de alto contenido de análisis de una sola célula de destino celular y la función.
En resumen, la combinación de microarrays de células con TFM sobre sustratos de rigidez sustrato sintonizable permite la caracterización exhaustiva de las dos señales bioquímicas y biofísicas. Tal como se presenta aquí, esta plataforma es generalizable y se puede aplicar fácilmente a una variedad de tipos de células adherentes y contextos de tejido hacia una mejor comprensión de la regulación microambientales combinatoria de la diferenciación celular y mechanotransduction.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Reconocemos Austin Cyphersmith y Mayandi Sivaguru (Carl Woese Instituto de Biología Genómica R., Universidad de Illinois en Urbana-Champaign) para obtener ayuda con la microscopía y para la pantalla y captura de vídeo con capacidad generosamente en el centro de microscopía.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | Pall Corporation | 4433 | Match with appropriately-sized Luer lock plastic syringes. |
| 100 × penicillin–streptomycin solution | Fisher Scientific | SV30010 | |
| 22 × 60 mm coverglasses | Electron Microscopy Sciences | 63765 | |
| 3-(trimethoxysilyl)propyl methacrylate (3-TPM) | Sigma-Aldrich | 440159 | Store under inert gas per manufacturer's instructions. Exposure of 3-TPM to air could compromise silanization of glass substrates. CAUTION: 3-TPM is a combustible liquid. Keep away from heat, sparks, open flames, and hot surfaces and use only in a chemical fume hood. |
| 3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate hydrate (CHAPS) | Sigma-Aldrich | C3023 | |
| 35 mm glass-bottom Petri dishes | Cell E&G | GBD00002-200 | 13 mm well consisting of #1.5 coverglass. Enables TFM and live-cell imaging. |
| 384-well polypropylene V-bottom microplate, non-sterile | USA Scientific | 1823-8400 | |
| 6-well polystyrene microplates | Fisher Scientific | 08-772-1B | 35 mm glass-bottom Petri dishes fit into wells of microplate, easing array fabrication. |
| Acetone | Sigma-Aldrich | 179973 | |
| Acrylamide | Sigma-Aldrich | A3553 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Collagen I, rat tail | EMD Millipore | 08-115MI | |
| Collagen III, human | EMD Millipore | CC054 | |
| Collagen IV, human | EMD Millipore | CC076 | |
| Crosslinker, 365 nm | UVP | CL-1000 | |
| Dextran, rhodamine B-conjugated, 70 kDa | ThermoFisher Scientific | D1841 | Used as a marker for array location. |
| Dimethyl sulfoxide | Fisher Scientific | BP231 | |
| Dulbecco's phosphate-buffered saline (PBS) | Fisher Scientific (HyClone) | SH3001302 | |
| Ethyl alcohol | Decon Labs | 2701 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | ED | |
| Fc-recombinant DLL1, mouse | R&D Systems | 5026-DL-050 | |
| Fc-recombinant DLL4, mouse | AdipoGen | AG-40A-0145-C050 | |
| Fc-recombinant JAG1, rat | R&D Systems | 599-JG-100 | |
| Fibronectin, human | Sigma-Aldrich | F2006 | |
| Fluorescent microscope, inverted | Zeiss | Axiovert 200M | Ensure microscope is equipped with a robotic stage for both automated fluorescent imaging and TFM. Environmental control (i.e., 37 °C and 5% CO2) is highly advisable for TFM. |
| Fluoromount G with DAPI | SouthernBiotech | 0100-20 | |
| Glacial acetic acid | Sigma-Aldrich | 695092 | CAUTION: Acetic acid is flammable and corrosive. Wear protective gloves, clothing, and eye protection. |
| Glycerol | Sigma-Aldrich | M6145 | |
| Irgacure 2959 | BASF Corporation | 55047962 | |
| Laminin, mouse | EMD Millipore | CC095 | |
| Methanol | Sigma-Aldrich | 179957 | |
| Microarray scanner | GenePix | 4000B | Fluorophores must be Cy3- or Cy5-compatible. |
| Microarrayer | Digilab | OmniGrid Micro | Other microarrayers of similar or greater capability can readily be substituted. |
| Microscope slides, 25 × 75 mm | Sigma-Aldrich | CLS294775X25 | ~0.9 – 1.1 mm thickness. |
| N,N′-Methylenebisacrylamide (bisacrylamide) | Sigma-Aldrich | M7279 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Paraformaldehyde (PFA), 16% v/v | Electron Microscopy Sciences | RT15710 | Prepare PFA fresh (do not store) for optimal fixation. CAUTION: Exposure to PFA can result in acute toxicity and can also irritate or corrode skin on contact. Wear protective gloves, clothing, and eye protection and use only in a chemical fume hood. |
| Protein A/G, recombinant | ThermoFisher Scientific | 21186 | |
| Pyrex drying tray, 2,000 mL | Fisher Scientific | 15-242B | |
| Rectangular 4-chambered culture dish | Fisher Scientific (Nunc) | 12-565-495 | For cell culture on arrayed microscope slides. |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | 415413 | CAUTION: NaOH is highly caustic and can cause severe skin burns and eye damage. Wear protective gloves, clothing, and eye protection. |
| Stealth pin for arraying | ArrayIt | SMP3 | Clean pins after each array run using the instructions of the manufacturer. Produces 150 micron domains; purchase other pin sizes (75–450 microns) as suited to your particular application. |
| Triton X-100 | Sigma-Aldrich | X100 |
Referencias
- Joyce, J. A., Pollard, J. W. Microenvironmental regulation of metastasis. Nat Rev Cancer. 9 (4), 239-252 (2009).
- Hsu, Y. C., Fuchs, E. A family business: stem cell progeny join the niche to regulate homeostasis. Nat Rev Mol Cell Biol. 13 (2), 103-114 (2012).
- Whiteside, T. L. The tumor microenvironment and its role in promoting tumor growth. Oncogene. 27 (45), 5904-5912 (2008).
- Jones, D. L., Wagers, A. J. No place like home: anatomy and function of the stem cell niche. Nat Rev Mol Cell Biol. 9 (1), 11-21 (2008).
- Discher, D. E., Mooney, D. J., Zandstra, P. W. Growth factors, matrices, and forces combine and control stem cells. Science. 324 (5935), 1673-1677 (2009).
- Trappmann, B., et al. Extracellular-matrix tethering regulates stem-cell fate. Nat Mater. 11 (7), 642-649 (2012).
- Ivanovska, I. L., Shin, J. W., Swift, J., Discher, D. E. Stem cell mechanobiology: diverse lessons from bone marrow. Trends Cell Biol. 25 (9), 523-532 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Chaudhuri, O., et al. Hydrogels with tunable stress relaxation regulate stem cell fate and activity. Nat Mater. 15 (3), 326-334 (2016).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev Cell. 6 (4), 483-495 (2004).
- Legate, K. R., Wickstrom, S. A., Fassler, R. Genetic and cell biological analysis of integrin outside-in signaling. Genes Dev. 23 (4), 397-418 (2009).
- Kessenbrock, K., Plaks, V., Werb, Z. Matrix metalloproteinases: regulators of the tumor microenvironment. Cell. 141 (1), 52-67 (2010).
- Underhill, G. H. Stem cell bioengineering at the interface of systems-based models and high-throughput platforms. Wiley Interdiscip Rev Syst Biol Med. 4 (6), 525-545 (2012).
- Underhill, G. H., Galie, P., Chen, C. S., Bhatia, S. N. Bioengineering methods for analysis of cells in vitro. Annu Rev Cell Dev Biol. 28, 385-410 (2012).
- Zorlutuna, P., et al. Microfabricated biomaterials for engineering 3D tissues. Adv Mater. 24 (14), 1782-1804 (2012).
- Ruiz, S. A., Chen, C. S. Microcontact printing: A tool to pattern. Soft Matter. 3 (2), 168-177 (2007).
- Guillotin, B., Guillemot, F. Cell patterning technologies for organotypic tissue fabrication. Trends Biotechnol. 29 (4), 183-190 (2011).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. J. Cell Sci. 123 (Pt 24), 4201-4213 (2010).
- Ranga, A., Lutolf, M. P. High-throughput approaches for the analysis of extrinsic regulators of stem cell fate. Curr. Opin. Cell Biol. 24 (2), 236-244 (2012).
- Kobel, S., Lutolf, M. High-throughput methods to define complex stem cell niches. Biotechniques. 48 (4), ix-xxii (2010).
- Fernandes, T. G., Diogo, M. M., Clark, D. S., Dordick, J. S., Cabral, J. M. S. High-throughput cellular microarray platforms: applications in drug discovery, toxicology and stem cell research. Trends Biotechnol. 27 (6), 342-349 (2009).
- Montanez-Sauri, S. I., Beebe, D. J., Sung, K. E. Microscale screening systems for 3D cellular microenvironments: platforms, advances, and challenges. Cell Mol Life Sci. 72 (2), 237-249 (2015).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat Methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Flaim, C. J., Bhatia, S. N., Parekkadan, B., Yarmush, M. . Methods in Bioengineering: Stem Cell Bioengineering Artech House Methods in Bioengineering. , 63-73 (2009).
- Brafman, D. A., Chien, S., Willert, K. Arrayed cellular microenvironments for identifying culture and differentiation conditions for stem, primary and rare cell populations. Nat Protoc. 7 (4), 703-717 (2012).
- Brafman, D. A., et al. Investigating the role of the extracellular environment in modulating hepatic stellate cell biology with arrayed combinatorial microenvironments. Integr Biol. 1 (8-9), 513-524 (2009).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol Syst Biol. 2, 37 (2006).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integr Biol. 1 (1), 70-79 (2009).
- Anderson, D. G., Levenberg, S., Langer, R. Nanoliter-scale synthesis of arrayed biomaterials and application to human embryonic stem cells. Nat Biotechnol. 22 (7), 863-866 (2004).
- Brafman, D. A., Shah, K. D., Fellner, T., Chien, S., Willert, K. Defining long-term maintenance conditions of human embryonic stem cells with arrayed cellular microenvironment technology. Stem Cells Dev. 18 (8), 1141-1154 (2009).
- Reticker-Flynn, N. E., et al. A combinatorial extracellular matrix platform identifies cell-extracellular matrix interactions that correlate with metastasis. Nat Commun. 3, 1122 (2012).
- Wood, K. C., et al. MicroSCALE screening reveals genetic modifiers of therapeutic response in melanoma. Sci Signal. 5 (224), rs4 (2012).
- Braga Malta, D. F., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kaylan, K. B., Ermilova, V., Yada, R. C., Underhill, G. H. Combinatorial microenvironmental regulation of liver progenitor differentiation by Notch ligands, TGFbeta, and extracellular matrix. Sci Rep. 6 (23490), 23490 (2016).
- Kourouklis, A. P., Kaylan, K. B., Underhill, G. H. Substrate stiffness and matrix composition coordinately control the differentiation of liver progenitor cells. Biomaterials. 99, 82-94 (2016).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integr. Biol. , (2016).
- Mann, C., Leckband, D. Measuring Traction Forces in Long-Term Cell Cultures. Cellular and Molecular Bioengineering. 3 (1), 40-49 (2010).
- Heisenberg, C. P., Bellaiche, Y. Forces in tissue morphogenesis and patterning. Cell. 153 (5), 948-962 (2013).
- Schwarz, U. S., Soine, J. R. Traction force microscopy on soft elastic substrates: A guide to recent computational advances. Biochim Biophys Acta. 1853 (11 Pt B), 3095-3104 (2015).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology-Cell Physiology. 282 (3), C595-C605 (2002).
- Wen, J. H., et al. Interplay of matrix stiffness and protein tethering in stem cell differentiation. Nat Mater. 13 (10), 979-987 (2014).
- Knoll, S. G., Ali, M. Y., Saif, M. T. A novel method for localizing reporter fluorescent beads near the cell culture surface for traction force microscopy. J Vis Exp. (91), e51873 (2014).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Bolstad, B. M., Irizarry, R. A., Astrand, M., Speed, T. P. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics. 19 (2), 185-193 (2003).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. Am J Physiol Cell Physiol. 282 (3), C606-C616 (2002).
- Krishnan, R., et al. Substrate stiffening promotes endothelial monolayer disruption through enhanced physical forces. Am J Physiol Cell Physiol. 300 (1), C146-C154 (2011).
- Reticker-Flynn, N. E., Bhatia, S. N. Aberrant glycosylation promotes lung cancer metastasis through adhesion to galectins in the metastatic niche. Cancer Discov. 5 (2), 168-181 (2015).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Kilian, K. A., Bugarija, B., Lahn, B. T., Mrksich, M. Geometric cues for directing the differentiation of mesenchymal stem cells. Proc Natl Acad Sci USA. 107 (11), 4872 (2010).
- Nelson, C. M., Chen, C. S. Cell-cell signaling by direct contact increases cell proliferation via a PI3K-dependent signal. FEBS Lett. 514 (2-3), 238-242 (2002).
- Hui, E. E., Bhatia, S. N. Micromechanical control of cell-cell interactions. Proc. Natl. Acad. Sci. U. S. A. 104 (14), 5722-5726 (2007).
- Lutolf, M. P., Blau, H. M. Artificial stem cell niches. Adv Mater. 21 (32-33), 3255-3268 (2009).
- Gobaa, S., et al. Artificial niche microarrays for probing single stem cell fate in high throughput. Nat. Methods. 8 (11), 949-955 (2011).
- DeForest, C. A., Anseth, K. S. Cytocompatible click-based hydrogels with dynamically tunable properties through orthogonal photoconjugation and photocleavage reactions. Nat Chem. 3 (12), 925-931 (2011).
- Nelson, C. M., VanDuijn, M. M., Inman, J. L., Fletcher, D. A., Bissell, M. J. Tissue geometry determines sites of mammary branching morphogenesis in organotypic cultures. Sci. STKE. 314 (5797), 298 (2006).
- Liu Tsang, V., et al. Fabrication of 3D hepatic tissues by additive photopatterning of cellular hydrogels. FASEB J. 21 (3), 790-801 (2007).
- Albrecht, D. R., Underhill, G. H., Mendelson, A., Bhatia, S. N. Multiphase electropatterning of cells and biomaterials. Lab. Chip. 7 (6), 702-709 (2007).
- Chan, V., Zorlutuna, P., Jeong, J. H., Kong, H., Bashir, R. Three-dimensional photopatterning of hydrogels using stereolithography for long-term cell encapsulation. Lab. Chip. 10 (16), 2062-2070 (2010).
- Boghaert, E., et al. Host epithelial geometry regulates breast cancer cell invasiveness. Proc. Natl. Acad. Sci. U. S. A. 109 (48), 19632-19637 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados