Method Article
Discriminación y Caracterización de las Poblaciones Heterocelulares Usando Técnicas de Imagen Cuantitativa
En este artículo
Resumen
Hemos desarrollado un nuevo protocolo para estudiar la dinámica de la población heterocelular en respuesta a las perturbaciones. Este manuscrito describe una plataforma basada en imágenes que produce conjuntos de datos cuantitativos para la caracterización simultánea de múltiples fenotipos celulares de poblaciones heterocelulares de una manera robusta.
Resumen
Los procesos celulares son complejos y resultan de la interacción entre múltiples tipos de células y su entorno. Las técnicas existentes de biología celular a menudo no permiten una interpretación precisa de esta interacción. Utilizando un enfoque basado en imágenes cuantitativas, presentamos un protocolo de alto contenido para caracterizar las respuestas fenotípicas dinámicas ( es decir , cambios morfológicos, proliferación, apoptosis) de poblaciones de células heterogéneas a cambios en los estímulos ambientales. Destacamos nuestra capacidad para distinguir entre tipos de células basadas en la intensidad de fluorescencia o características morfológicas inherentes dependiendo de la aplicación. Esta plataforma permite una caracterización más completa de la respuesta de la subpoblación a la perturbación mientras que utiliza tiempo más corto, cantidades más pequeñas de reactivos, y menor probabilidad de error que los ensayos tradicionales de biología celular. Sin embargo, en algunos casos, las poblaciones celulares pueden ser difíciles de identificar y cuantificar basándose en células complejasY requerirá una solución de problemas adicional; Destacamos algunas de estas circunstancias en el protocolo. Demostramos esta aplicación usando la respuesta al fármaco en un modelo de cáncer; Sin embargo, se puede aplicar con mayor facilidad a otros procesos fisiológicos. Este protocolo permite identificar subpoblaciones dentro de un sistema de co-cultivo y caracterizar la respuesta particular de cada uno a estímulos externos.
Introducción
Ensayos basados en células han sido un caballo de batalla en la investigación básica y la configuración del desarrollo de drogas. Sin embargo, las limitaciones de estos ensayos estándar se han hecho cada vez más evidentes con la discordancia entre los datos in vitro y clínicos y el fracaso de la mayoría de los fármacos para recibir la aprobación de la FDA. Aquí, presentamos un nuevo método para la utilización de imágenes cuantitativas para analizar simultáneamente los fenotipos heterocelulares en respuesta a los estímulos ambientales pertinentes co-occurring.
Los ensayos basados en células tradicionales que se usan para medir la viabilidad celular incluyen: ensayos de exclusión de azul de tripán, tinción con citometría de flujo con MTT / MTS y anexina V-FITC. Trypan azul exclusión ensayos, mientras que simple y barato, requieren un gran número de células, son mucho tiempo, y son a menudo influenciados por el usuario sesgo 1 . MTT y MTS indirectamente medir la viabilidad celular a través de mediciones de la tasa metabólica mitocondrial. Sin embargo, la actividad metabólica(Por ejemplo, los medios de comunicación o la concentración de oxígeno), lo que conduce a resultados imprecisos y evita la estandarización entre los tipos y condiciones de las células 2 , 3 . Otra desventaja principal de estas técnicas es su incapacidad para distinguir entre tipos de células múltiples - la mayoría de los sistemas biológicos son heterocelulares. Aunque los métodos de citometría de flujo tienen la capacidad de distinguir entre poblaciones de células múltiples, se requieren etiquetas de células, el muestreo dinámico es desafiante y cuando se usan células adherentes, esta aplicación se convierte en mucho tiempo y propensa a errores.
Otros importantes fenotipos celulares, incluyendo los cambios morfológicos, se producen en respuesta a los estímulos ambientales, pero no son capturados por los ensayos basados en células tradicionales. La elaboración de perfiles de los estados de las células a través de la caracterización morfológica y las similitudes de mapeo entre muestras es una herramienta poderosa e imparcial con el aBility para proveer nuevas ideas sobre muchos aspectos de la investigación básica y traslacional, incluyendo la biología celular básica y el descubrimiento de fármacos 4 . Además, se ha demostrado que la morfología de las células tumorales se correlaciona con los subtipos tumorales 5 y la agresividad 6 . Por lo tanto, es de gran interés estudiar estas características celulares y cómo se relacionan con perturbaciones ambientales específicas. Además, se pueden utilizar diferencias en las características morfológicas para discriminar entre subpoblaciones en sistemas de co-cultivo. Las células marcadoras fluorescentes tienen caídas ( es decir, alteran las propiedades celulares inherentes, consumen tiempo) y, por lo tanto, son ventajosos métodos adicionales para clasificar tipos celulares.
La imagen basada en microscopía es un método alternativo para perfilar los fenotipos celulares de una manera multiplexada, cuantitativa y robusta. En este manuscrito, aplicamos nuestra tubería de imagen cuantitativa para destacar la evoluciónDinámica de las poblaciones de células heterogéneas dentro de un tumor. Nos centramos en la interacción entre células de cáncer de pulmón de células no pequeñas (NSCLC) y Fibroblastos Asociados al Cáncer (CAF), el tipo de células estromales más prevalente en tumores. Las CAF han sido implicadas en la iniciación tumoral, la progresión y la respuesta terapéutica; Por lo tanto, la realización de ensayos fenotípicos en las células tumorales en ausencia de CAF puede ser engañosa [ 7 , 8 , 9] . Específicamente, evaluamos los efectos de las CAFs en las células tumorales en respuesta al erlotinib, una molécula pequeña dirigida al Receptor del Factor de Crecimiento Epidérmico (EGFR) que se usa a menudo en el tratamiento clínico del NSCLC. Utilizamos una plataforma de cribado de alto contenido y su software de análisis de imagen que acompaña a la evaluación; Sin embargo, en un intento de hacer accesible esta metodología a otros investigadores, también hemos desarrollado un protocolo comparable de downstream usando el software de código abierto:CellProfiler 10 y CellProfiler Analyst 11 . La mayoría de los ensayos de detección de alto contenido basados en imágenes se analizan con software comercializado específico para un modelo de instrumento dado. Los resultados son difíciles de replicar en otros laboratorios con software diferente porque los algoritmos subyacentes son a menudo propietarios. Usando este oleoducto basado en imágenes, se midió la proliferación celular, la muerte y la morfología de cada subpoblación de un cultivo heterocelular en respuesta al tratamiento con fármaco utilizando tanto la clasificación basada en fluorescencia como en la morfología. El siguiente protocolo proporciona una metodología robusta para probar procesos celulares complejos.
Protocolo
1. Cultivo celular
NOTA: Aquí se investigaron las respuestas fenotípicas de células NSCLC (H3255) al agente de direccionamiento de EGFR, erlotinib, cuando se co-cultivaron con fibroblastos pulmonares (CCD-19Lu GFP). Para el mismo conjunto de datos, basada en la fluorescencia y la clasificación basada en la morfología de las dos poblaciones de células (H3255 y CCD-19Lu GFP) para ejemplificar su concordancia se realizó. Sin embargo, sólo se debe utilizar un método de clasificación y debe elegirse sobre la base de la aplicación.
- Preparar 500 ml de RPMI-1640 suplementado con FBS inactivado por calor al 10% y penicilina / estreptomicina al 1%.
NOTA: El medio de crecimiento y los suplementos se pueden reemplazar según sea necesario para otros tipos de células. - Cultivar células en placas de 10 cm 3 como poblaciones puras en CO 2 al 5% a 37 ° C y pasar a una proporción apropiada cada 3-4 días.
2. Preparación de células
- Para preparar una suspensión celular, eliminar cEll y las células de lavado con 5 ml de solución salina tamponada con fosfato 1X (PBS).
- Aspirar fuera del PBS, añadir 1 mL de 0,05% de tripsina calentada a 37 ° C e incubar durante 5 min (o hasta que las células ya no se adhieren a la placa) a 37 ° C.
- Para neutralizar la tripsina, añadir 4 ml de RPMI a las placas H3255 y CCD-19Lu GFP y pipetear las soluciones celulares en tubos cónicos de 15 ml.
- Centrifugar las células a 150 xg durante 5 minutos a RT.
- Aspirar el sobrenadante y resuspender los sedimentos celulares en 10 ml de medio RPMI en tubos cónicos para preparar la siembra.
3. Revestimiento de células
- Cuente las células para estandarizar la siembra.
- Invertir tubos para mezclar las células en solución. Pipetear 10 μl de cada suspensión de células en un tubo de microcentrífuga y añadir 10 μl de azul de tripano.
- Pipetee 10 μL de esta solución en una diapositiva de conteo de células e inserte en un contador de células automatizado.
NOTA: Otros métodos de recuento celular ( es decir,. Hemocitómetro). - Repita el recuento de células por lo menos tres veces, o hasta que los recuentos obtenidos sean consistentes.
- Las células de semilla en una placa de varios pocillos
NOTA: El número de placas a sembrar depende del número de puntos de tiempo que se visualizarán. Alternativamente, una placa se puede volver a imaginar con el tiempo si las células se transducen con histona-2B-GFP (u otras tinciones nucleares lentivirales estables que proporcionan una segmentación nuclear adecuada).- Semilla de un total de 1.500 células / pocillo en 100 μ l de RPMI. Preparar tres suspensiones de células para la placa de tres poblaciones de células: H3255, CCD-19Lu GFP, y 50% H3255 + 50% CCD-19Lu GFP. Realice cada uno por triplicado con configuraciones de placa idénticas para cada punto de tiempo.
NOTA: Las densidades de siembra iniciales se deben optimizar para cada línea celular y tamaño de placa. - Seed células en placas de 3 x 96 pocillos utilizando una pipeta multicanal. Incubar las células O / N a 37 ° C, 5% de CO 2 .
- Semilla de un total de 1.500 células / pocillo en 100 μ l de RPMI. Preparar tres suspensiones de células para la placa de tres poblaciones de células: H3255, CCD-19Lu GFP, y 50% H3255 + 50% CCD-19Lu GFP. Realice cada uno por triplicado con configuraciones de placa idénticas para cada punto de tiempo.
4. Dosificación de fármacos
- Añadir DMSO a erlotinib en polvo para hacer una solución madre de erlotinib final de 10 mM de concentración.
NOTA: El fármaco puede ser sustituido como se desee. - Diluir el fármaco con medio de cultivo celular a 2x la concentración final con la concentración final más alta a 10 μM y diluir en serie cuatro veces a una relación 1:10, para un total de cinco concentraciones de fármaco y un control sin fármaco.
NOTA: Si la concentración de DMSO es igual o superior a 0,1% v / v a la dosis de fármaco más alta, el control de fármaco no debe contener una cantidad equivalente de DMSO para asegurar que los efectos observados observados no se deban a la toxicidad de DMSO. - Pipetear 100 μl de solución de fármaco en el pocillo apropiado para una concentración final de fármaco de 1x diluido en medio.
5. Adquisición de imágenes
NOTA: Las imágenes fueron adquiridas en los días 0, 2 y 3. Dependiendo de los tipos celulares y de los procesos celulares estudiados, oSe pueden estudiar los puntos temporales deseados.
- Mancha las células para preparar la imagen.
- Preparar la solución de colorante a las siguientes concentraciones finales.
- Para la clasificación basada en fluorescencia, preparar tinción nuclear de 5 μg / mL y tinción de células muertas 5 μM en PBS (ver Tabla de Materiales ).
- Para la clasificación basada en la morfología, preparar tinción nuclear de 5 μg / mL, tinción de células muertas 5 μM y tinción celular 5 μM en PBS (ver Tabla de Materiales ).
- Añadir 20 μL de solución de colorante a cada pocillo. Incubar durante 30 minutos a 37 ° C protegido de la luz.
- Preparar la solución de colorante a las siguientes concentraciones finales.
- Optimizar y adquirir imágenes.
- Retire la placa del pozo de la incubadora, limpie la parte inferior de la placa con EtOH al 70% y coloque la placa en la cámara de imagen.
- En la pestaña 'Configuración', haga clic en el botón '+' en 'Selección de canales' para agregar canales(Es decir , campos brillantes, tinción nuclear, tinción de células muertas, GFP y señales RFP).
- Haga clic en 'Layout Selection' (selección de disposición) y tome imágenes de prueba empiladas en z cada 2 μm comenzando en 0 μm y terminando en 20 μm para identificar el plano de enfoque. Introduzca esta distancia para cada canal bajo 'Altura'.
- Haga clic en "Instantánea" en cada canal para evaluar las intensidades y optimizar los tiempos de exposición. Ingrese valores más altos o más bajos en "Hora" según sea necesario.
- En el lado derecho de la pantalla, resalte los pozos apropiados (que se van a visualizar) en el esquema de la placa. En el esquema de pozos a continuación, resalte los veinticinco campos que se van a tomar imágenes de una manera similar.
- En "Ejecutar experimento", identifique la placa en "Nombre de placa" en la pestaña izquierda y haga clic en el botón "Inicio" (debajo) para ejecutar el protocolo de adquisición de imágenes con un objetivo 10X para generar imágenes TIFF en escala de grises en los canales mencionados.
NOTA: paso a paso instRucciones para el protocolo de imagen pueden diferir entre los instrumentos, pero los valores de los parámetros deben ser optimizados. - Repita la proyección de imagen en los días dos y tres.
6. Análisis de imágenes
NOTA: Todo el análisis de imagen se realizó utilizando software propietario. Sin embargo, debido a que esto no es disponible públicamente, los análisis comparables también fueron diseñados en CellProfiler 2.2 y CellProfiler Analyst 2.0, con un breve protocolo listado a continuación y un protocolo detallado proporcionado como material complementario (las imágenes de prueba ya están cargadas en las tuberías para probar el flujo de trabajo). Las imágenes de este experimento se agruparon en una base por pozo para que cada pocillo pudiera cargarse individualmente. Los pipelines de CellProfiler a continuación contienen una expresión regular que analiza la información de metadatos de cada nombre de archivo de imagen y permite que las imágenes se agrupan aún más por canal.
- Descargue e instale el software de código abierto: 'CellProfiler9; Y 'CellProfiler Analyst'. Acepte todas las opciones y complementos predeterminados durante la instalación.
- Clasificar los cultivos heterocelulares en subpoblaciones.
- Abra el software 'CellProfiler', haga clic en 'Archivo' | 'Pipeline de importación' | 'From File' y seleccione el archivo apropiado ("Fluorescence_Classification.cppipe" para la clasificación basada en fluorescencia , o "Morphology_Classification.cppipe" para la clasificación basada en morfología).
NOTA: Estos archivos contienen protocolos para procesamiento básico de imágenes y segmentación de núcleos / células. Debido a la irregular mancha Hoechst presente a través de estas células, los núcleos se segmentaron y agrandaron, y luego se utiliza para enmascarar la imagen para permitir la segmentación de núcleos con intensidades más bajas.- Seleccione el gasoducto de clasificación basado en fluorescencia para clasificar las células como H3255 o CCD-19Lu sobre la base de la intensidad eGFP y para calcular las características morfológicas.
NOTA: CCD-19Lu células fueron transducidas con GFP. - Seleccione el gasoducto de clasificación basado en morfología para segmentar núcleos y células y para calcular características morfológicas.
NOTA: Para la clasificación es necesario realizar análisis adicionales en el 'CellProfiler Analyst'. Las células muertas se clasifican mediante clasificación basada en fluorescencia.
- Seleccione el gasoducto de clasificación basado en fluorescencia para clasificar las células como H3255 o CCD-19Lu sobre la base de la intensidad eGFP y para calcular las características morfológicas.
- Haga clic en "Ver configuración de salida" en la parte inferior izquierda de la pantalla. Seleccione la ubicación de los archivos de imagen para análisis ('Default Input Folder') y el destino de los datos extraídos ('Default Output Folder').
- Haga clic en 'Analizar imágenes' para comenzar el análisis. Una vez finalizado el análisis, haga clic en "Aceptar" y vaya a la "Carpeta de salida predeterminada" para ver los datos calculados.
NOTA: Los valores de los parámetros de ejemplo y las imágenes están disponibles en el archivo suplementario 1-CellProfilerProtocol.pdf . - Para la clasificación basada en morfología (solamente) haga lo siguiente.
- Abra el software 'CellProfiler Analyst', seleccione el archivo de propiedades de la base de datos generado en CellProfiler y haga clic en 'Abrir'.
- Haga clic en la pestaña 'Clasificador' en la parte superior izquierda de la pantalla. Para llamar a las imágenes celulares aleatorias del experimento, haga clic en 'Buscar'; Las imágenes aparecerán en la ventana sin clasificar.
- Clasifique manualmente las células como positivas (H3255) o negativas (CCD-19Lu) seleccionando y arrastrando las celdas a la casilla correspondiente (vea el archivo suplementario 2-Pipeline.pdf : Figura B ). Después de clasificar al menos 50 células por subpoblación, haga clic en 'Train Classifier' y, a continuación, en 'Check Progress'.
NOTA: Las celdas se clasifican a través de un algoritmo de aprendizaje de máquina forestal aleatoria supervisada por el usuario [ 12] . Si la precisión no está por encima del umbral aprobado (> 90%), puede ser necesario retroceder y optimizar la segmentación celular en 'CellProfiler', o las células pueden no ser ideales para classiFying por la morfología. - Haga clic en "Puntuación completa" para generar una tabla con recuentos de celdas para cada subpoblación.
- Abra el software 'CellProfiler', haga clic en 'Archivo' | 'Pipeline de importación' | 'From File' y seleccione el archivo apropiado ("Fluorescence_Classification.cppipe" para la clasificación basada en fluorescencia , o "Morphology_Classification.cppipe" para la clasificación basada en morfología).
- Añadir DMSO a erlotinib en polvo para hacer una solución madre de erlotinib final de 10 mM de concentración.
Resultados
Se generó un conjunto de imágenes compuesto por 25 campos / pocillo, 54 pocillos / placa (3 poblaciones de células x 6 concentraciones de fármaco x 3 repeticiones), a través de tres placas para un total de 4.050 imágenes individuales. Los conjuntos de imágenes generadas a lo largo del experimento se analizaron utilizando un software propietario (véase tabla de materiales) para extraer varias propiedades cuantitativas de células ( es decir , morfología, fluorescencia) que podrían utilizarse para clasificar subpoblaciones celulares. Sin embargo, debido a que el software comercial utilizado tiene acceso limitado, se crearon ductos descendentes comparables en CellProfiler y CellProfiler Analyst.
Clasificación heterocelular en subpoblaciones
Los núcleos fueron identificados y segmentados en base a la tinción de ADN (aquí Hoechst) y las poblaciones de células se clasificaron basados en fluorescencia o mo( Figura 1 ). Para la clasificación basada en la fluorescencia, los fibroblastos (CCD-19Lu) se transdujeron previamente con GFP-lentivirus. Los niveles de intensidad GFP se midieron para cada núcleo, y los que se calcularon por encima del umbral aceptado (basado en la señal de fondo) se clasificaron como CCD-19Lu mientras que los siguientes fueron identificados como células tumorales (H3255). Para la clasificación basada en morfología, las células se tiñeron previamente con una tinción celular no tóxica (véase la tabla de materiales) y se utilizó para identificar y segmentar el citoplasma. Un algoritmo de aprendizaje de la máquina fue entrenado con ~ 50-100 células de cada población. Se identificaron características morfológicas que eran significativamente diferentes entre las poblaciones, que se utilizaron entonces para diseñar un clasificador lineal para distinguir entre células CCD-19Lu y H3255. Los protocolos de clasificación de fluorescencia y morfología fueron 97,4% (n = 1403) concordantes en distinguir entre las dos poblaciones de célulasEn condiciones no tratadas y 92,5% (n = 916) concordante en las condiciones tratadas con fármacos (erlotinib 1 μM) ( Figura 2 ).
Análisis fenotípicos de subpoblaciones
Además de la discriminación entre los tipos de células, se trató de caracterizar las propiedades fenotípicas de cada subpoblación. Los ensayos de multiplexación ahorran tiempo y reactivos, aportan consistencia y proporcionan información adicional sobre el sistema que se está estudiando. Hay muchos potenciales resultados fenotípicos y uno debe elegir basados en las preguntas de interés. Aquí, se investigaron los cambios en la morfología celular y el estado de viabilidad en respuesta al tratamiento con erlotinib. Después de tres días de tratamiento con fármaco, se observó una disminución en el área nuclear y un aumento en el área celular de las células H3255 ( Figura 3A ). La diferencia media en el área nuclear entreSe encontró que las poblaciones tratadas con "fármaco sin fármaco" y "fármaco" eran estadísticamente significativas a través de una prueba t de dos caras tipo 2 (varianza igual) ( p = 7,92 x 10 -16 ). Se plantea la hipótesis de que esta observación es una respuesta celular al estrés impuesto por el tratamiento farmacológico.
También es de interés estudiar si un fármaco tiene un efecto citotóxico ( es decir, aumento del número de células muertas en el tiempo) o citostático ( es decir, disminución del número de nacimientos celulares en el tiempo), ya que esto tiene un impacto clínico profundo. Por ejemplo, un efecto citostático de fármaco induce la detención del crecimiento pero no elimina las células del tumor, por lo que existe el potencial de que las células cancerosas reinicien la proliferación celular una vez que se elimina el fármaco. Los efectos de la droga a menudo pueden ser el contexto, la concentración y el tipo de células dependientes. Anteriormente se observó erlotinib provocando una respuesta citotóxica en un tipo de célula, mientras que mostrando acRespuesta ytostática en otro 13 .
Los ensayos de viabilidad tradicional producen el número relativo de células y, por tanto, no discriminan entre los arrestos por crecimiento y la muerte celular. En este documento, se identificaron células muertas basadas en tinción de yoduro de propidio ( Figura 3B ). Se observaron efectos citotóxicos y citostáticos del erlotinib sobre las células H3255, con un aumento en el número de muertes y una disminución en el número de nacimientos después del tratamiento con fármacos ( Figura 3C ). Vale la pena señalar que el número de células muertas disminuye después del día 1 probablemente debido a la depuración celular. Las células CCD-19Lu no se vieron afectadas por el fármaco. Una ventaja adicional de esta plataforma es la generación de datos cuantitativos. Por ejemplo, en nuestro experimento de co-cultivo, se encontró que una subpoblación inicial de 1.118 (75.8%) células H3255 era 2.817 (87.9%) o 396 (57.2%) después de tres días sin o con erlotinaIb, respectivamente ( Figura 4 ). Debido a que podemos generar recuentos de células reales en lugar de porcentaje relativo (como con los métodos de citometría de flujo), concluimos que el cambio en la composición durante el tratamiento con fármacos se debe a una disminución de las células H3255 y no a un aumento de CCD-19Lu. No vale la pena que las tasas de mortalidad se pueden subestimar debido a la depuración celular, que es difícil de evaluar experimentalmente y probablemente difiere entre los tipos de células.
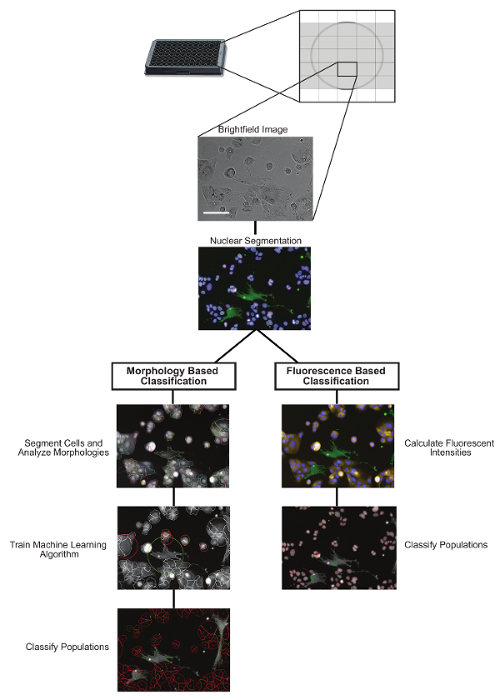
Figura 1: Visión general del Protocolo de análisis de imágenes. Dos posibles tuberías de análisis de imagen aguas abajo para clasificar las poblaciones heterocelulares utilizando una clasificación basada en morfología o basada en fluorescencia. Barras de escala = 100 μm. Por favor haga clic en ellaE para ver una versión más grande de esta figura.

Figura 2: Concordancia entre la Morfología y la Clasificación basada en la Fluorescencia. ( A ) Gráfico de concordancia que muestra la superposición de los dos protocolos de clasificación. Las mismas células se clasificaron como H3255 utilizando tanto la morfología y la fluorescencia basada en la clasificación. Los dos protocolos estuvieron de acuerdo, con clasificación de 97,4% (n = 1403) de células no tratadas y 92,5% (n = 916) de células tratadas con erlotinib (Nota: el área blanca es demasiado pequeña para visualizarse). ( B ) imágenes 10X que representan ejemplos de buena y mala concordancia entre la clasificación basada en la fluorescencia y la morfología basada. Las flechas blancas apuntan a las celdas que fueron clasificadas inconsistentemente entre las plataformas. Imagen de entrada: núcleos azules (Hoechst); Verde - CCD19Lu (GFP). ClassiImágenes de marcado: Rojo - H3255; Verde - CCD-19Lu. Barra de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
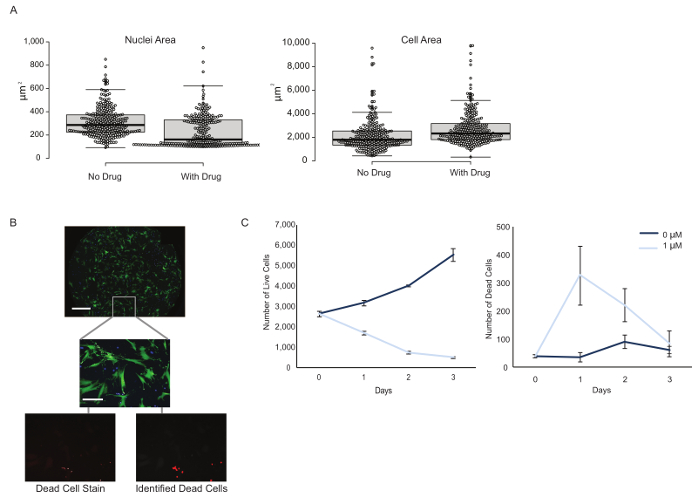
Figura 3: Mediciones fenotípicas multiplexadas de una sola configuración experimental. ( A ) Se calcularon características morfológicas, tales como núcleos y área celular, en el nivel de célula única en presencia y ausencia de fármaco. Nota: Las áreas celulares que miden menos de 100 μm 2 se consideraron escombros y se excluyeron de los análisis. La gráfica de la caja representa la mediana con los rangos del primer y tercer cuartil y las barras de error del intervalo de confianza del 95%. ( B ) Se co-cultivaron células H3255 (azul) y CCD-19Lu (verde) y se identificaron células muertas basándose en la intensidad de las células propídicasIum iodide mancha (rojo) y imágenes utilizando un objetivo 10X. Barra de escala = 1 mm (panel superior); 100 μm (imágenes del fondo). ( C ) Se calculó el número total de células vivas y muertas durante tres días con o sin tratamiento con fármaco, con una disminución obvia en el número de células vivas y aumento de células muertas con la adición de erlotinib. Las barras de error representan el error estándar de la media basada en tres repeticiones. Haga clic aquí para ver una versión más grande de esta figura.
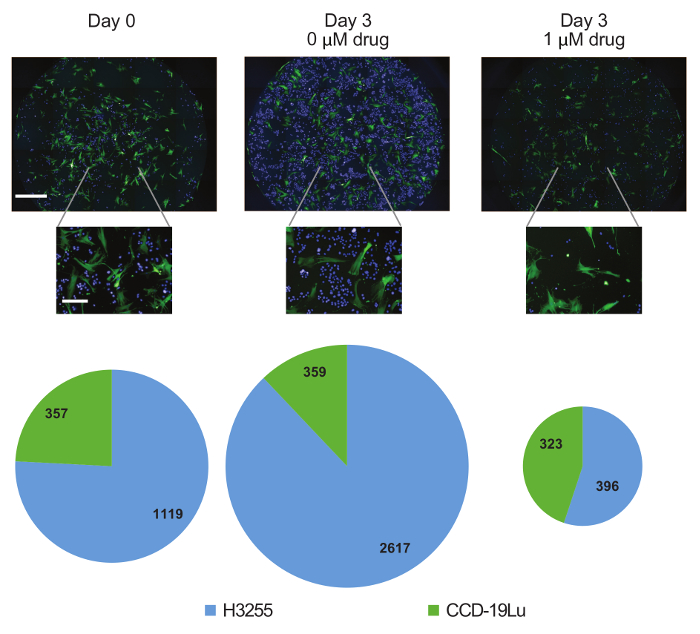
Figura 4: Dinámica de la Subpoblación en el Tiempo. Imágenes representativas 10X de pocillos que contienen H3255 (azul) y CCD-19Lu (verde) el día 0 o el día 3 con y sin fármaco. Se contaron las células pertenecientes a cada subpoblación y los gráficos circulares proporcionales muestran la aCambios en la composición de la población en las muestras. Barras de escala = 1 mm (paneles centrales, shpwn en el panel más alto), 1 mm (imagen superior), 100 μm (imágenes del fondo). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
The protocol described above improves upon current cell biology assays by providing more comprehensive insights into phenotypic dynamics of multiple cell types in response to environmental perturbations while using reduced reagents and time. A major advantage of this experimental design is the ability to analyze multiple phenotypes with a single setup and generate quantitative data characterizing these phenotypes on a single cell level. One technical advantage to this platform is the ease of initial troubleshooting compared to other assays. Because this method is image-based, one is able to visualize the wells for apparent over/under seeding. It is advisable to have cells in the exponential growth phase for the duration of the experiment and not be limited by nutrient or spatial constraints or confounded by senescence due to scarce seeding. Otherwise, birth and death rates may not be reproducible between experiments. For reference, CellPD is a publicly available program for computation of birth and death rates14. Additionally, one can visualize whether adequate concentrations of dyes were added to each well. Pipetting issues in an individual well could result in missegmentation and skewed data, but can easily be detected with the aforementioned protocol.
Unfortunately, not all cell types may be amenable to this application. It is important to be able to accurately segment the nuclei and cells, therefore analysis of cells that organize in more sphere-like or clumped structures may not be suitable. For some cells, it also may be advantageous to use a cell strainer prior to seeding to ensure initial seeding of single cells. In addition, the linear classifier technique is only applicable to cell types that can be readily distinguished based on morphology features.
For the success of the protocol, it is important to first optimize the imaging conditions, as the validity of downstream analyses is dependent on the quality of the images. While here we performed experiments using a high-content screening platform, image acquisition can also be performed using any fluorescent microscope (although an automated imaging platform is ideal for high-throughput approaches). Test images should be taken prior to each imaging time point to ensure that there are no problems with the microscope or protocol. The signal to noise ratio should be high, especially for the channels that will be used for segmentation (i.e. nuclei, cell stains). Additionally, it is important to image in the optimal plane of focus. If the images are out of focus, segmentation becomes much more difficult and the calculated morphological features will likely be inaccurate. Illumination differences between fields can cause problems with image segmentation as well. Large differences in brightness make the automated selection of threshold values difficult. Additionally, if there are heterogeneous fluorescence intensities between cells, a single threshold value may not sufficiently segment all the cells in an image. In this analysis, these problems were overcome by creating masks around brighter and dimmer cell populations and segmenting each population separately.
While the phenotypes under investigation in this protocol are limited to live, dead, and morphological characterization, they can easily be expanded to investigate other features. For example, functional genetic studies can be added with RNAi, overexpression, or other chemical perturbations.
In this paper, the capabilities of the protocol to measure the response of non-small cell lung cancer cells to erlotinib in the presence and absence of CAFs was demonstrated. However, this is merely one example of the many cell types and microenvironmental parameters that can be tested. We have extended this protocol to be used with other cell types and drug studies, including primary cells isolated from patient tumors13,15.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por el Instituto Nacional del Cáncer (NCI) Subvenciones U54CA143798 y U54CA143907 para establecer Ciencias Físicas-Oncología Centros (PS-OC) en el Dana-Farber Cancer Institute y la Universidad del Sur de California, respectivamente. SM Mumenthaler recibió un premio de transnetwork de PS-OC que apoyó parte de este trabajo.
Quisiéramos expresar nuestra más profunda gratitud a nuestros partidarios filantrópicos, en particular a la familia Stephenson, Emmet, Toni y Tessa, por su donación de la plataforma Operetta HCS. También queremos agradecer a J. Foo por orientación, y al Centro de Medicina Aplicada miembros del equipo de Medicina: D. Agus para la orientación clínica y mentoría, K. Patsch para las discusiones significativas con el diseño experimental, R. Rawat para la ayuda en los protocolos de análisis de imágenes , J. Katz para la asistencia técnica con la Operetta, y P. Macklin y D. Ruderman para las discusiones provechosas y la regeneración.
Materiales
| Name | Company | Catalog Number | Comments |
| RPMI-1640 | Corning | 10-040-CV | cell culture medium |
| Fetal Bovine Serum (FBS) | Gemini | 100-106 | medium supplement |
| Penicillin/streptomycin | Gibco | 15-140-122 | medium supplement |
| TC20 | Biorad | 1450102 | cell counter |
| TC20 slides with trypan blue | Biorad | 1450003 | cell counter slides |
| 96-well plates | Corning | 3904 | clear bottom black plates |
| Erlotinib | LC Laboratories | E-4007 | |
| DMSO | VWR | 317275-100ML | solution to resuspend drug |
| Hoechst | Invitrogen | H21491 | nuclear dye |
| Propidium Iodide (PI) | Invitrogen | P1304MP | dead cell stain |
| Cell Tracker Orange CMRA | Life Technologies | C34551 | whole cell stain |
| Operetta high content imaging system | Perkin Elmer | ||
| CellProfiler | Broad Institue | version 2.2.0 (rev 9969f42) | http://cellprofiler.org/releases/ |
| Cell culture incubator | Any cell culture incubator will be suitable - cells were cultured under 37 ºC at 5% CO2. | ||
| 15 mL Falcon conical tubes | Falcon | 14-959-53A | |
| 10 cm2 cell culture plates | TPP | 93040 |
Referencias
- Kanemura, Y., et al. Evaluation of in vitro proliferative activity of human fetal neural stem/progenitor cells using indirect measurements of viable cells based on cellular metabolic activity. J Neurosci Res. 69 (6), 869-879 (2002).
- Plumb, J. A., Milroy, R., Kaye, S. B. Effects of the pH dependence of 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl-tetrazolium bromide-formazan absorption on chemosensitivity determined by a novel tetrazolium-based assay. Cancer Res. 49 (16), 4435-4440 (1989).
- Hsu, S., et al. Green tea polyphenols induce differentiation and proliferation in epidermal keratinocytes. J Pharmacol Exp Ther. 306 (1), 29-34 (2003).
- Caicedo, J. C., Singh, S., Carpenter, A. E. Applications in image-based profiling of perturbations. Curr Opin Biotechnol. 39, 134-142 (2016).
- Sero, J. E., et al. Cell shape and the microenvironment regulate nuclear translocation of NF-kappaB in breast epithelial and tumor cells. Mol Syst Biol. 11 (3), 790 (2015).
- Chen, J. F., et al. Subclassification of prostate cancer circulating tumor cells by nuclear size reveals very small nuclear circulating tumor cells in patients with visceral metastases. Cancer. 121 (18), 3240-3251 (2015).
- Ohlund, D., Elyada, E., Tuveson, D. Fibroblast heterogeneity in the cancer wound. J Exp Med. 211 (8), 1503-1523 (2014).
- Paraiso, K. H., Smalley, K. S. Fibroblast-mediated drug resistance in cancer. Biochem Pharmacol. 85 (8), 1033-1041 (2013).
- Kalluri, R. The biology and function of fibroblasts in cancer. Nat Rev Cancer. 16 (9), 582-598 (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), R100 (2006).
- Jones, T. R., et al. CellProfiler Analyst: data exploration and analysis software for complex image-based screens. BMC Bioinformatics. 9, 482 (2008).
- Dao, D., et al. CellProfiler Analyst: interactive data exploration, analysis and classification of large biological image sets. Bioinformatics. 32 (20), 3210-3212 (2016).
- Garvey, C. M., et al. A high-content image-based method for quantitatively studying context-dependent cell population dynamics. Sci Rep. 6, 29752 (2016).
- Juarez, E. F., et al. Quantifying differences in cell line population dynamics using CellPD. BMC Syst Biol. 10 (1), 92 (2016).
- Mumenthaler, S. M., et al. The Impact of Microenvironmental Heterogeneity on the Evolution of Drug Resistance in Cancer Cells. Cancer Inform. 14, 19-31 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados