Method Article
Reconocimiento de secuencia específica y selectivo de RNAs de doble cadena sobre ARN monocatenario de modifica químicamente los ácidos nucleicos péptidos
En este artículo
Resumen
Divulgamos los protocolos para la síntesis y purificación de oligómeros del péptido de ácido nucleico (PNA) incorporando modificado residuos. Se describen los métodos bioquímicos y biofísicos para la caracterización del reconocimiento de dúplex RNA por las PNAs modificados.
Resumen
RNAs están emergiendo como importantes biomarcadores y dianas terapéuticas. Así, hay gran potencial en el desarrollo de sondas químicas y terapéuticas ligandos para el reconocimiento de la estructura y secuencia de ARN. Modificar químicamente péptidos ácidos nucleicos (PNA) oligómeros se han desarrollado recientemente que puede reconocer dúplex RNA de una manera específica de secuencia. PNAs son químicamente estables con un esqueleto de péptidos como neutral. PNAs se pueden sintetizar relativamente fácilmente por el método de síntesis de péptidos de fase sólida manual de Boc-química. PNAs son purificadas por HPLC de fase inversa, seguido de la caracterización del peso molecular por láser asistida por matriz de desorción/ionización-tiempo de vuelo (MALDI-TOF). Técnica de electroforesis (PAGE) de gel de poliacrilamida no desnaturalizantes facilita la proyección de imagen de la formación de triplex, porque cuidadosamente diseñado gratis RNA duplex construye y PNA obligado triplex a menudo Mostrar tasas de migración diferentes. Página no desnaturalizar con bromuro de etidio post coloración es a menudo una técnica fácil e informativa para la caracterización de las afinidades de Unión y especificidades de oligómeros PNA. Por lo general, múltiples horquillas de RNA o dúplex con mutaciones de pares solo puede utilizarse para caracterizar propiedades de enlace de PNA, como vinculante las afinidades y especificidades. 2-aminopurina es un isómero de la adenina (6-aminopurina); la intensidad de fluorescencia de la 2-aminopurina es sensible a los cambios del entorno estructural y es conveniente para la supervisión de formación de triplex con el residuo de la 2-aminopurina incorporado cerca el sitio de unión de la Prefectura Naval Argentina. 2-aminopurina valoración de fluorescencia puede utilizarse también para confirmar la selectividad de unión de PNAs modificados hacia objetivos RNAs de doble hebra (dsARN) en ARN monocatenario (ssRNAs). Experimentos de fusión termal detección de absorbancia UV permiten la medición de la estabilidad térmica de PNA-ARN dúplex y PNA· Tríplex de2 RNA. Aquí, describimos la síntesis y purificación de oligómeros PNA incorporando modifica residuos y describir los métodos bioquímicos y biofísicos para la caracterización del reconocimiento de dúplex RNA por las PNAs modificados.
Introducción
RNAs están emergiendo como importantes biomarcadores y dianas terapéuticas, debido a los recientes avances en los descubrimientos de los roles de los RNAs en la regulación y la catálisis de procesos biológicos diversos1,2,3. Tradicionalmente, se han utilizado hilos antisentidos para enlazar a ssRNAs a través de Watson-Crick formación duplex3,4,5,6,7,8, 9 , 10 , 11 , 12 , 13 , 14 , 15 , 16 , 17 , 18 , 19 , 20 , 21 , 22 , 23 , 24 , 25 , 26 , 27. recientemente, triplex formando péptidos ácidos nucleicos (TFPNAs) han sido diseñados para enlazar a dsRNAs vía hidrógeno Hoogsteen vinculación (figura 1)3,28,29. dsRNA regiones están presentes en la mayoría de los ARNs antisentido dirigidos tradicionales, incluyendo pre-mRNAs y mRNAs, pre- o pri-miRNAs3y muchos otros no-codificación RNAs1,26,27. DsARNs a través de la formación de la triple hélice con TFPNAs puede ser ventajosa debido a la especificidad de su estructura y es de gran potencial para el uso en la restauración de las funciones normales de las RNAs, que son normales en las enfermedades, por ejemplo.
El recientemente publicado trabajo de Rozners et al.y nosotros3,28,29,30,31,32,33, 34,35,36,37,38,39,40,41, informó de los esfuerzos en la mejora de la Unión selectiva de TFPNAs modificados a dsRNAs con mayor afinidad. Hemos desarrollado métodos de síntesis de monómeros PNA racionalmente diseñados (figura 2) incluyendo thio-pseudoisocytosine (L) monómero30 y guanidina-modificado 5-metil citosina (Q) monómero31. A través de varios métodos de caracterización bioquímica y biofísica, hemos demostrado que PNAs relativamente corto (6-10 residuos) incorporación de residuos de L y Q muestren mejor reconocimiento de pares de bases Watson-Crick G-C y C-G, respectivamente, en dsRNAs. Por otra parte, comparado con PNAs sin modificar, PNAs que contiene residuos de L y Q mostraron enlace más selectiva hacia dsARN ssRNA y dsDNA. La funcionalidad de guanidina42 en la base de Q permite PNAs entrar en las células de HeLa31.
En nuestro laboratorio, sintetizamos PNAs por la Boc-química manual (Boc o t- Boc es tert-butyloxycarbony (ver figura 2) de método de síntesis de péptidos de fase sólida4. La síntesis del monómero PNA con Boc como la amina protección de grupo es conveniente que el grupo Boc es sterically menos voluminoso en comparación con la amina fluorenylmethyloxycarbonyl (Fmoc) protección de grupo, que puede ser beneficioso durante el monómero PNA de acoplamiento la soporte sólido. El grupo Boc es ácido lábil y se puede quitar fácilmente en el soporte sólido por 20-50% de ácido trifluoroacético (TFA) en diclorometano (DCM) durante la síntesis de PNA. Un sintetizador de péptidos automatizada se puede emplear para sintetizar oligómeros PNA; sin embargo, 3-5-fold exceso de monómero PNA es necesario para un sintetizador de péptidos automatizada. Síntesis manual requiere significativamente menos monómero ANP (2-3-fold exceso), con cada acoplamiento fácilmente monitoreado por el Kaiser prueba43. Además, muchos sintetizadores automatizados no son compatibles con la síntesis de la estrategia de Boc debido al uso de TFA corrosivo durante el paso de eliminación de Boc.
Los oligómeros PNA pueden ser purificados por fase reversa cromatografía líquida alto rendimiento (RP-HPLC) seguida de la caracterización del peso molecular por MALDI-TOF (figuras 3 y 4)30, 31. empleamos página no desnaturalizantes para monitorear la triple formación, debido a que construye duplex RNA libre y PNA obligado triplex a menudo muestran diferente migración tasas (figura 5)30,31 . No etiquetado es necesario si la coloración eficiente puede lograrse para ambos el RNA duplex y PNA· Bandas triples de RNA2 . Una relativamente pequeña cantidad de muestra se necesita para no desnaturalizar página experimentos. Sin embargo, los almacenadores intermediarios del cargamento (incubación) y los buffers de corrientes (pH 8,3) pueden no ser el mismo, dando por resultado las medidas se limita a la conexión cinéticamente estable, debido a un pH relativamente alto de 8.3 significativamente puede desestabilizar un triplex.
2-aminopurina es un isómero de la adenina (6-aminopurina); la intensidad de fluorescencia de la 2-aminopurina (con un pico de emisión en alrededor de 370 nm) es sensible a los cambios del entorno estructural y es conveniente para el seguimiento de la formación de triplex con el residuo de la 2-aminopurina incorporados cerca de la Prefectura Naval Argentina (sitio de Unión Figura 6) 31. a diferencia de muchos otros tintes que muestran la emisión de fluorescencia en la gama visible, RNA 2-aminopurina-labeled puede estar expuesto a luz de la habitación sin blanquear de la foto. A diferencia del experimento de la página en la que con frecuencia es necesaria un corriente buffer de pH 8.3, 2-aminopurina basado en titulación de fluorescencia permite la medición de la Unión en una sola solución a un pH determinado y así puede permitir la medición y detección de relativamente débil y enlace cinéticamente inestable equilibrio.
Experimentos de fusión termal detección de absorbancia UV permiten la medición de la estabilidad térmica de dúplex (figura 7)31 y tricomplejos30,32,44,45. Dependiendo de la composición de longitud y secuencia, la fusión de conexión puede o no puede mostrar una transición clara. Parámetros termodinámicos pueden obtenerse si se superponen las curvas de calentamiento y enfriamiento. Parámetros termodinámicos exactos se pueden obtener por titulación isotérmica calorimetría (ITC)32; sin embargo, cantidades relativamente grandes de muestras son generalmente necesarios para la ITC.
Protocolo
1. manual Solid-phase péptido síntesis de PNAs usando Boc química
Nota: para el éxito y la facilidad de la deseada síntesis de oligómero de Prefectura Naval Argentina, todos los disolventes y reactivos deben ser anhidros. Añadir los tamices moleculares apropiados (4A, bolitas de 1-2 mm de diámetro) y de vez en cuando purga de nitrógeno seco en botellas. Para la síntesis de monómeros PNA modificados, pueden utilizar los protocolos reportados en referencias respectivas 30 , 31. Monómeros PNA pueden adquirirse de fuentes comerciales. En cada uno de los pasos de lavado, la cantidad apropiada de solvente se agrega a la resina, formando una mezcla, antes de se drena apagado
Resina de poliestireno- carga del primer monómero y tapado el exceso aminas primarias libres en la resina
- pesar 30 mg de clorhidrato de 4-methylbenzhydrylamine (MBHA·HCl) (disponible; cargar valor 0.7-1.4 mmol/g; 100-200 mallas), 5 mL y recipiente de la reacción de fase sólida péptido provisto de un tapón de vidrio y llave de paso.
- Remojo la resina en una cantidad apropiada de DCM durante 1 hora, permitiendo a la resina a hincharse y a exponer las aminas (sal del ácido clorhídrico).
Nota: Resina debe siempre estar totalmente sumergida en solventes a través de la síntesis. - Drenaje del DCM, aplicando un suave flujo de gas de nitrógeno seco en la parte superior del matraz. Añadir 1 mL de 50% (v/v) N, N-diisopropylethylamine (DIPEA) en DCM y dejarla por 15 minutos. Esto neutraliza el ácido clorhídrico sal a las aminas libres en la resina.
- Repetir paso 1.1.3. Mientras tanto, pesan 6 μmol de monómero y 6 μmol de hexafluorofosfato (benzotriazol-1-yl-oxy) tripyrrolidinophosphonium (PyBOP) y transferir a un tubo de 1,5 mL. Añadir 200 μL de dimetilformamida (DMF) y 12 μmol de DIPEA. Vórtice de la solución de acoplamiento para 3-5 min
Nota: El valor de la carga deseada utilizado es 0,2 mmol/g. El primer monómero añadido es el monómero en el C-terminal de la secuencia deseada de la Prefectura Naval Argentina. El valor de la carga del primer aminoácido se puede calcular por el método de ácido pícrico 46.
Solución de - drenaje de DIPEA del DCM. Lavar la resina con DCM (x 3), seguida de DMF (x 3) y cierre la llave de paso. Añadir la solución de acoplamiento dispuesto a la resina y agitar suavemente. Introduzca la resina a lo largo de las paredes internas del vaso la solución de acoplamiento con el uso de una espátula de acero inoxidable limpio. Sujete el tapón de cristal y asegure el vaso en un agitador incubador durante 3 h a 40 ° C.
Nota: Alternativamente, el recipiente de la reacción puede ser mantenido constante a temperatura ambiente durante 6-8 h permitir la finalización de la reacción de acoplamiento de péptido. - Preparar la solución que capsula mezclando 240 μmol de anhídrido acético y 360 μmol de DIPEA en 200 μL de DCM. Elimine la solución de acoplamiento y lavar la resina con DMF (x 3) y DCM (x 3). Añadir en la solución de tapado y dejar el recipiente por 30 min, agitando de vez en cuando el vaso suavemente. Recubrimiento enmascara los grupos de amina primaria gratis exceso de las resinas por acetilación.
- Repetir paso 1.1.6. La solución que capsula de drenar y lavar la resina con DCM (x 3).
- Eliminar una pequeña alícuota de granos de la resina mediante un tubo capilar fino y colocarlos en un frasco de vidrio pequeño de 1,5 mL. Realizar la prueba de Kaiser del 43. Añadir 15 μl de cada uno de lo Kaiser soluciones de ensayo en el vaso del frasco y calientan mediante una pistola de calor. Observar el color de los granos después del calentamiento. El color de los granos debe permanecer sin cambios, lo que indica la falta de grupos de la amina libre en la resina.
Nota: El kit de la prueba de Kaiser disponible comercialmente o puede ser preparado según el protocolo reportado. Solución A: ninhidrina en etanol; solución B: fenol en etanol; solución C: cianuro de potasio (KCN) en piridina.
PRECAUCIÓN: KCN es altamente tóxico; se debe usar ropa protectora adecuada y calefacción debe realizarse en una campana bien ventilada, en ausencia de disolventes inflamables o reactivos. - Repetir paso 1.1.6 Si los granos de resina color azul tenue o azul la pantalla.
- Retiro de grupo protector de amino N-terminal
- escurrir los solventes del recipiente de la reacción y añadir una solución de 50% (v/v) de ácido trifluoroacético (TFA) en DCM, asegurando que las resinas están completamente sumergidas. Deje el recipiente durante 15 minutos, sacudiendo de vez en cuando para facilitar la desprotección de los grupos de la amina. Repetir 2 ciclos más.
PRECAUCIÓN: TFA es altamente corrosivo. Se debe usar ropa protectora adecuada cuando manejo. - Lavar la resina con DCM (x 3), DMF (x 3) y DCM (x 3). Agregar una solución de 5% DIPEA en DCM. Deje el recipiente durante 15 minutos. Este paso activa el aminas libres mediante la neutralización de los aniones de contador TFA. Repetir una vez más.
- Lave DIPEA de la solución de DCM. Lavar la resina con DCM (x 3). Realizar la prueba de Kaiser (paso de 1.1.8).
Nota: Éxito desprotección de los grupos de Amina dará coloración azulada de los granos. Una vez que la desprotección es exitoso, acoplamiento posterior de monómeros por acoplamiento de péptidos puede llevarse a cabo.
- escurrir los solventes del recipiente de la reacción y añadir una solución de 50% (v/v) de ácido trifluoroacético (TFA) en DCM, asegurando que las resinas están completamente sumergidas. Deje el recipiente durante 15 minutos, sacudiendo de vez en cuando para facilitar la desprotección de los grupos de la amina. Repetir 2 ciclos más.
- Acoplamiento de monómeros posterior
- pesa 18 μmol de deseado monómero (para el monómero de Boc-PNA-Q-OH, 13,2 mg de monómero se pesa) y 18 μmol de PyBOP en un tubo de 1,5 mL. Añadir 200 μL de DMF y 36 μmol de DIPEA en el tubo de 1,5 mL. Vortex hasta que se disuelvan todos los compuestos sólidos.
- Lavar la resina con DMF (x 3). Añadir la solución de acoplamiento en el recipiente de la reacción y agite suavemente. Introduzca la resina a lo largo de las paredes internas del vaso la solución de acoplamiento con el uso de una espátula de acero inoxidable limpio. Sujete el tapón de cristal y asegure el vaso en un agitador incubador durante 3 h a 40 ° C.
- Drenaje de la solución de acoplamiento y lava la resina con DMF (x 3) y DCM (x 3). Realizar paso 1.1.8. Si el color de los granos sigue siendo 1.2.1-1.3.3 sin cambios, repetir los pasos hasta completa la secuencia deseada de la Prefectura Naval Argentina. Si se observa decoloración azul de granos después de la Unión de un monómero, repita los pasos 1.3.1-1.3.3. Si la decoloración persiste, realizar la nivelación de nuevo (paso 1.1.6).
Nota: Se recomiendan extendido de acoplamiento tiempo (3-12 h) y/o el uso del equivalente exceso de monómero y acoplamiento reactivos si surgen problemas con el acoplador. - Una vez completada la secuencia deseada de la PNA, lavar la resina con DMF (x 3) y DCM (x 3). Secar completamente la resina mediante la aplicación de un flujo continuo de gas de nitrógeno seco durante 15 minutos. Esta resina seca puede ser dividida y utilizada para fijar la lisina o una etiqueta fluorescente como Cianina 3 (Cy3) y carboxyfluorescein en N-terminal de la ANP.
- Accesorio de la etiqueta de lisina o fluorescentes (Cy3, Cy5 o carboxyfluorescein) a N-terminal de ANP
- pesan 10 mg de resina, cargada con la secuencia deseada de la Prefectura Naval Argentina. Transferir a un recipiente de reacción de 5 mL. Reposar la resina en DCM 1 h.
- Para la fijación de la lisina, realizar pasos 1.2.1-1.3.3, reemplazando el monómero con cualquiera de los dos OH Boc-Lys (Z) - OH o Fmoc-Lys (Boc)-.
- Para el accesorio de carboxyfluorescein, realizar pasos 1.2.1-1.3.3, con 10 veces exceso de 5 6-carboxyfluorescein como monómero, y N, N '-diisopropilcarbodiimida (DIPC) y hydroxybenzotriazole (TOHB) como los reactivos de acoplamiento en DMF. Dejar durante la noche la reacción de acoplamiento en la coctelera de la incubadora a los 40 ° C.
Nota: Como carboxyfluorescein es sensible a la luz, el recipiente de la reacción debe cubrirse con papel de aluminio. - Para la fijación del tinte Cy3 o Cy5, etiqueta por el método de química haga clic en realizar pasos 1.2.1-1.3.3, reemplazando el monómero con N-Boc-2-propargyl-L-glycine. Esto functionalizes el N-terminal de la ANP con un grupo de alquino. Realizar la reacción de cobre-catalizada del tecleo para sujetar el Cy3 que contiene azida o Cy5 fluorescente tinte 12 , 47 , 48 , 49
- Escote PNA de soporte sólido, purificación y caracterización
Nota: Si cualquier grupo de la amina está protegido con el Fmoc protección de grupo, primero desprotejera el grupo Amina por tratamiento con piperdine de 20% en DMF solución durante 15 min (2 ciclos). Lavar la resina con DMF (x 3) seguido por DCM (x 3). Seque completamente la resina mediante la aplicación de un flujo continuo de gas de nitrógeno seco durante 15 minutos.- Transferencia de 5 mg de resina seca en un frasco pequeño. Añada 10 μl de tioanisol y 4 μL de 1, 2-Etanoditiol, asegurando que la resina se sumerge en los reactivos. Dejar el tubo a temperatura ambiente durante 5 minutos
Nota: Estos reactivos actúan como carroñeros, que atrapar especies catiónicas reactivas que se forman durante la extracción de la protección de grupos en la Prefectura Naval Argentina. Adsorbente adecuado puede elegir basado en la cadena lateral de protección de los grupos. - Agregar 100 μl de TFA en el tubo que contiene la resina y carroñeros. Suavemente el vórtice la mezcla y sujeto a centrifugado breve. Dejar el tubo a temperatura ambiente durante 10 minutos
PRECAUCIÓN: TFA es altamente corrosivo. Se debe usar ropa protectora adecuada cuando manejo.
Cuidadosamente, - añada 20 μl de Ácido trifluorometanosulfónico (TFMSA) al tubo. Frote suavemente la mezcla de reacción antes de someter a centrifugación breve a temperatura ambiente. Dejar el tubo constante a temperatura ambiente durante 2 h.
PRECAUCIÓN: TFMSA es altamente corrosivo. Se debe usar ropa protectora adecuada cuando manejo. - Filtro del escote cocktail en un matraz de fondo redondo 5 mL (FBR) con el uso de un vidrio Pasteur pipeta provista de algodón. Use una pequeña cantidad de TFA para lavar la resina.
- Purgar el gas de nitrógeno seco para el filtrado recogido hasta que todos los disolventes volátiles se evaporan. Añadir 1 mL de éter dietílico frío en el RBF.
Nota: El éter dietílico causará la PNA a precipitar.- Enjuague el RBF con el dietil éter varias veces antes de transferir la solución turbia en un tubo de 1,5 mL. Sujeto el tubo de centrifugación para permitir que el precipitado PNA resolver. Decantar los solventes y añadir 300-500 μl de agua esterilizada para el precipitado. Vortex para disolver la ANP.
- Purificar la cruda muestra de ANP mediante RP-HPLC utilizando water-acetonitrile-0.1% TFA como fase móvil. Recoger las fracciones correspondientes, se evaporan los disolventes utilizando un concentrador de vacío antes de volver a disolver la ANP purificada en agua esterilizada.
- Caracterizan la ANP purificada mediante análisis de MALDI-TOF con el uso de ácido α-ciano-4-hidroxicinámico (CHCA) como la matriz de cristalización muestra.
- Medir la absorbancia de UV (260 nm) de la Prefectura Naval Argentina a 65 ° C. calcular la concentración de la Autoridad Nacional Palestina con la ecuación:

Nota: aquí c es la concentración, es la lectura de absorbancia obtenida, ε es el coeficiente de extinción de la secuencia de ARN, y l es la longitud del camino óptico de la cubeta (1 cm). el coeficiente de extinción de la secuencia de la PNA es la suma del coeficiente de extinción de monómeros individuales 50. Los coeficientes de extinción de adenina, citosina, guanina y timina son 15.4, 7.3, 11.7 y 8.8 mL/µmol·cm, respectivamente. El coeficiente de extinción de los monomers L y Q utilizadas se supone que es igual a la de la base citosina (C).
- Transferencia de 5 mg de resina seca en un frasco pequeño. Añada 10 μl de tioanisol y 4 μL de 1, 2-Etanoditiol, asegurando que la resina se sumerge en los reactivos. Dejar el tubo a temperatura ambiente durante 5 minutos
2. Página de desnaturalización no
tampón de incubación preparación de- preparación de soluciones tampón necesaria
- (10 mL) usando 116,88 mg NaCl (200 mM), 50 μl de ácido de 100 mM etilendiaminotetracético (EDTA) (0,5 mM), 200 μL de stock de 1 M HEPES (20 mM), 9,75 m H 2 O; ajustar el buffer a pH 7.5.
- Prepare 1 x Tris-borato-EDTA (TBE) corriente almacenador intermediario (1 L) con 100 mL 10 x Tris-borato-EDTA a pH 8.3 y 900 mL de H 2 O.
- Preparar solución de persulfato (APS) de amonio de 10% (300 μL) con persulfato de amonio de 30 mg y 300 μL de H 2 O.
- Preparación del gel de poliacrilamida 12%
- limpiar el peine bien formando placas de fundición de vidrio y separadores con etanol, el peine bien conformado y espaciadores son 1 mm de espesor. Establecer la Asamblea de gel y selle con cinta de sellado de gel.
- Para un gel de poliacrilamida de 22 cm x 16.5 cm x 1 dimensión mm, 50 mL de la solución en gel es adecuado. Pesa 5,7 g de acrilamida y 0,3 g de N, N '-Metilenbisacrilamida (19:1) y transferencia en una centrífuga de 50 mL tubo.
PRECAUCIÓN: La acrilamida es carcinogénico. Evite respirar polvo vapores de acrilamida y asegurarse que la ropa protectora apropiada es usada cuando manejo. - Disolver compuestos sólidos en 50 mL 1 X TBE corriente de tampón colocando el tubo de centrífuga en un baño de agua de 50 ° C durante 15 minutos o hasta que se disuelvan todos los compuestos sólidos. Llevar a cabo la centrifugación (3.000 rpm, 5 min, 25 ° C) para eliminar todas las burbujas de aire dentro de la solución. Deje que la solución se enfríe a temperatura ambiente.
- Añadir 250 μl de solución APS 10% y 50 μl de tetramethylethylenediamine (TEMED) en la solución de gel y mezclar suavemente con una espátula. Inmediatamente verter la solución entre las placas de vidrio, asegurándose de no introducir burbujas de aire. Inserte el peine bien conformado y dejar la configuración del gel a temperatura ambiente durante al menos 60 min permitir la polimerización que se produzca, antes de guardar a 4 ° C hasta que esté listo para usar.
- Preparación de muestras
- Retire cantidad requerida de horquilla de RNA (1 μm) de la acción principal en un tubo limpio de 1.5 mL. La solución de RNA utilizando un concentrador de vacío en seco.
Nota: Cada muestra contiene 1 μm del ARN en 20 μl de tampón de incubación. Por lo general, 13 muestras de ARN están preparadas para experimentar una sola página. El RNA para todas las muestras se puede preparar juntos en un solo tubo. - Quitar los volúmenes requeridos de ANP específica (con la concentración final de hasta 50 μm) de la acción principal y transferencia en tubos separados 1,5 mL. Evaporar el agua de las soluciones PNA utilizando un concentrador de vacío.
- Añadir 260 μl de tampón de incubación en 1,5 mL de tubo que contiene secado ARN y mezcla bien para asegurar que todos los RNA es dissolved. Tema del RNA para rápido enfriamiento: lugar el tubo en un bloque de calor (precalentado a 95 ° C) durante 5 minutos inmediatamente transferir a un baño de hielo y dejar durante 10 minutos Tubos de
- Añadir 20 μl del ARN en cada uno de 1.5 mL con habían secado PNA y mezcla bien. Realizar el recocido: Coloque el tubo que contiene mezcla de ARN y PNA en un bloque de calor (precalentado a 65 ° C) durante 10 minutos a su vez la energía del bloque de calor y deje enfríen las muestras lentamente a temperatura ambiente. Incubar las muestras a 4 ° C durante la noche.
- Retire cantidad requerida de horquilla de RNA (1 μm) de la acción principal en un tubo limpio de 1.5 mL. La solución de RNA utilizando un concentrador de vacío en seco.
- Ejecución y procesamiento de gel
- Retire la cinta de sellado de gel en la parte inferior de la placa de vidrio. Monte y fije la placa de vidrio sobre el gel vertical soporte con pinzas de plástico.
Nota: El gel de ejecución se lleva a cabo en una cámara fría a aproximadamente 4 º C. Todos los equipos, las muestras y tampones se enfrían a 4 º C antes de ejecutar el gel de. - Llene el depósito de buffer inferior con 1 x TBE funcionamiento tampón hasta que la placa quede sumergida en aproximadamente 1-2 cm del almacenador intermediario de funcionamiento. Llenar el depósito tampón superior con corriente buffer hasta que el nivel de búfer supera la parte superior del gel por 1-2 cm. lentamente y retire suavemente el peine bien formando, permitiendo que el buffer corriente llenar los pozos de.
- Conecte el soporte del gel a una fuente de alimentación. Funcionamiento previo el gel para por lo menos 30 min con una constante tensión de 250 V, que está optimizado para una 22 cm x 16,5 cm x 1 gel mm.
- Mientras tanto, añadir 4 μL (20% del volumen de la muestra) de 35% glicerol solución a cada una de las muestras y mezclar suavemente. Una vez terminado el plazo anterior, cargar 20 μl de cada muestra (incluyendo una muestra solo RNA más muestras que contienen mezcla de ARN y PNA) cuidadosamente en el fondo del pozo utilizando una micropipeta y gel tips, asegurándose de no introducir burbujas de aire de carga. Correr el gel a una constante tensión de 250 V, similar a la carrera previa, de 5 h.
- Detener la fuente de alimentación después de 5 h y retire la placa del soporte. Retire la cinta de sellado de gel restante y desmontar la placa de vidrio. Suavemente retirar y sumergir el gel en un recipiente llenado con 350 mL de agua desionizada. Cuidadosamente añadir 35 μl de bromuro de etidio (10 mg/mL) y coloque el recipiente sobre un agitador de plataforma (baja velocidad) para 30 minutos
PRECAUCIÓN: El bromuro de etidio es un mutágeno. Se debe usar ropa protectora adecuada cuando manejo. - Deseche la solución de bromuro de etidio en un cesto de basura designado. Enjuagar el gel con 1.5-2 L de agua destilada. Escanear el gel usando un sensor (véase Tabla de materiales) con un láser verde de 532 nm y la emisión filtran ajustado a 610 nm.
- Retire la cinta de sellado de gel en la parte inferior de la placa de vidrio. Monte y fije la placa de vidrio sobre el gel vertical soporte con pinzas de plástico.
- Análisis del gel
- cuantificar las intensidades de la banda de gel usando un software libre, GelQuant.NET (http://www.biochemlabsolutions.com/GelQuantNET.html). Normalizar las intensidades de la banda según:

Nota: aquí me max duplex es la intensidad de la banda de la horquilla de RNA sin la adición de PNA y me max triple es la intensidad de triple banda con la mayor concentración de ANP ha añadido.- Calcular la fracción de triple formación según:

- Calcular la fracción de triple formación según:
- parcela la fracción de la formación de triplex (Y) contra la concentración de ANP agregado () ΜM). Ajustar los datos a la ecuación para obtener la constante de disociación (K d):
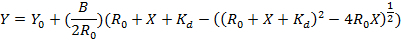
Nota: aquí R 0 es la concentración de la horquilla de RNA (1 μm). Aquí Y 0 y B están la inicial y el cambio máximo de la fracción triple, respectivamente. Y es la fracción de triplex en variada concentración de ANP. X es la concentración total de PNA y K d es la constante de disociación.
- cuantificar las intensidades de la banda de gel usando un software libre, GelQuant.NET (http://www.biochemlabsolutions.com/GelQuantNET.html). Normalizar las intensidades de la banda según:
3. 2-aminopurina fluorescencia enlace ensayo
- preparación de las muestras (contiene dsARN)
- Retire la cantidad necesaria de la 2-aminopurina (2AP) etiquetada dsRNA (1 μm de cada uno del filamento) de la acción principal en un tubo limpio de 1.5 mL. Evaporar agua en las soluciones de RNA utilizando un concentrador de vacío.
Nota: Cada muestra contiene 1 μm del ARN en 75 μl de tampón de incubación. Se preparan normalmente, 13 muestras de ARN. RNA de las muestras se puede preparar juntos en un solo tubo. - Quitar los volúmenes requeridos de la ANP específica para diferentes concentraciones de la acción principal y transferencia en tubos separados 1,5 mL. Evaporar el agua de las soluciones PNA utilizando un concentrador de vacío.
- Añadir 975 μl de tampón de incubación en 1,5 mL de tubo que contiene secado RNA y mezcla bien para que RNA todo se disuelve. Centrifugar brevemente la solución de RNA y sometidos a recocido: Coloque el tubo en un bloque de calor (precalentado a 95 ° C) durante 10 minutos a su vez la energía del bloque de calor y deje enfríen las muestras lentamente a temperatura ambiente. Tubos de
- añadir 75 μl de RNA en cada uno de 1.5 mL con habían secado PNA y mezcla bien. Dejar las muestras a temperatura ambiente durante por lo menos 1 h. Incubar las muestras a 4 ° C durante la noche.
- Retire la cantidad necesaria de la 2-aminopurina (2AP) etiquetada dsRNA (1 μm de cada uno del filamento) de la acción principal en un tubo limpio de 1.5 mL. Evaporar agua en las soluciones de RNA utilizando un concentrador de vacío.
- Preparación de las muestras (contiene ssRNA)
- quitar cantidad necesaria de la etiqueta 2AP ssRNA (1 μm) de las acciones principales en tubos de 1,5 mL limpio. Extraer los volúmenes requeridos de ANP específica para diferentes concentraciones de la acción principal y transferencia en los tubos de 1.5ml respectivos que contengan el ssRNA. La mezcla de ARN y PNA utilizando un concentrador de vacío en seco.
- Añadir 75 μl de incubación buffer en cada uno de los tubos de 1.5 mL y homogeneizar. Someter la mezcla a recocido: Coloque el tubo en un bloque de calor (precalentado a 95 ° C) durante 10 minutos a su vez de la energía y deje que las muestras se enfríen lentamente a temperatura ambiente. Incubar las muestras a 4 ° C durante la noche.
- Medición y análisis
- utilizar un espectrofotómetro de fluorescencia para medir las emisiones en un rango de longitud de onda de 330-550 nm. Utilizar una longitud de onda de excitación de 303 nm.
Nota: Las muestras se miden a temperatura ambiente y cada muestra se midió 3 veces, en el que se toma la media. - Transferencia de 70 μl de incubación buffer en la cubeta cuadrada de 1 cm. Iniciar la medición.
- Quitar el tampón de la cubeta. Enjuagar la cubeta con agua destilada y purgar con gas nitrógeno para secar. Repita para todas las muestras. Restar las medidas del almacenador intermediario de todas las muestras de.
- Trama de la intensidad de fluorescencia (a.u.) contra longitud de onda (nm). Registrar la intensidad de fluorescencia a 370 nm para todas las muestras. Parcela la intensidad de fluorescencia a 370 nm (a.u.) contra las concentraciones correspondientes de la ANP añadió (μm).
- Ajustar los datos a la ecuación del paso 2.5.2 para obtener la constante de disociación (K d): Y = Y 0 + (B / (2R 0)) (X, R 0 + K d-((R 0 + X + K d) 2 -4R 0 X) 1/2), donde R 0 es la concentración de dsARN etiquetado 2AP (1 μm).
Nota: Aquí Y 0 y B están el cambio inicial y máximo de intensidad de fluorescencia a 370 nm, respectivamente. Y es la intensidad de fluorescencia a 370 nm a variada concentración de ANP. X es la concentración total de PNA y K d es la constante de disociación.
- utilizar un espectrofotómetro de fluorescencia para medir las emisiones en un rango de longitud de onda de 330-550 nm. Utilizar una longitud de onda de excitación de 303 nm.
4. Experimentos de detección de absorbancia UV de fusión térmica
- preparación de las muestras
Nota: Mida la absorbancia de UV (260 nm) del ARN a 95 ° C (para asegurarse de que las estructuras secundarias del RNA se interrumpen). Calcular la concentración del RNA con la ecuación:
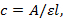
donde c es la concentración, es la lectura de absorbancia obtenida, ε es el coeficiente de extinción de la secuencia de RNA, y l es la longitud del camino óptico de la cubeta (1 cm). El coeficiente de extinción del ARN se calcula basándose en un modelo de vecino más cercano con MeltWin 51 , 52. El paquete de programa puede suministrarse bajo pedido. Tubos- Retire la cantidad necesaria de ssRNA (5 μm) de las acciones principales en limpio 1,5 mL. Quitar los volúmenes requeridos de ANP específica (5 μm) de la acción principal y transferencia en los tubos de 1.5ml respectivos que contengan el ssRNA. La mezcla de ARN y PNA utilizando un concentrador de vacío en seco.
- Añadir 130 μl de incubación buffer en cada uno de los tubos de 1.5 mL y homogeneizar. Someter la mezcla a recocido: Coloque el tubo en un bloque de calor (precalentado a 95 ° C) durante 10 minutos a su vez de la energía y deje que las muestras se enfríen lentamente a temperatura ambiente. Incubar las muestras a 4 ° C durante la noche.
- Medición y análisis
- utilice un espectrofotómetro UV-Vis para medir la absorbancia a 260 nm con una cubeta de microcell 8 con una longitud de ruta de acceso de 1 cm. medir las muestras ' absorbancia en el aumento de temperatura de 15 a 95 ° C, seguido por la disminución de temperatura de 95 a 15 ° C a una tasa de rampa de 0,5 ° C/min
- Transferencia de 130 μl de muestra en cada uno de lo bien, asegurándose de que uno también contiene el tampón de incubación. Iniciar la medición. Repita según sea necesario.
- Normalizar los valores de absorbancia con la alta temperatura de normalizado a la unidad y represente la absorbancia normalizada lectura contra temperatura (° C). Trama de la primera derivada de las curvas. Obtener la temperatura de fusión encajando las primera curvas derivadas de una función Gaussiana.
Resultados
HPLC de fase inversa permite la purificación de oligómeros PNA. Podemos obtener oligómeros PNA puros con dos rondas de la purificación de HPLC (figura 3). La identidad de los PNAs puede confirmarse por análisis de MALDI-TOF (figura 4).
Página de desnaturalización no es una técnica fácil e informativa para la caracterización de las afinidades de Unión y especificidades de oligómeros PNA. Por lo general utilizamos múltiples horquillas de RNA o dúplex con mutaciones de base par caracterizar propiedades de enlace (figura 5). Los datos de la página de desnaturalización no que se muestra en la figura 5 indican claramente que el Q - y L-modificado PNA puede reconocer una región de dsRNA con un par de C-G (figura 5B, panel inferior) pero no uno sin un par de C-G (figura 5B, superior panel). Este reconocimiento específico y mayor es el T· A-U, L· G-C y Q· C-G PNA· RNA2 base triple (figura 1A, C, D) la formación. Varios PNAs con una o varias mutaciones pueden usarse para demostrar las propiedades de enlace mejorado de un ANP modificado. Hemos demostrado que añadir 2 mM Mg2 + en el tampón de incubación no afecta el atascamiento perceptiblemente31.
Hemos demostrado por titulación de la fluorescencia de la 2-aminopurina que una PNA Q y L modificado se une a una región de dsRNA dirigido (figura 6A, 6 C, 6 D) pero no ssRNA (figura 6B, 6E, 6F). P3 de la Prefectura Naval Argentina se une al dsRNA 2-aminopurina-etiquetados con un valor ded Kde 0,8 ± 0,1 μm. La intensidad de fluorescencia a 370 nm para la 2-aminopurina-labeled ssRNA permanece relativamente constante con la variada concentración de P3, indicando la falta de unión de PNA P3 a la ssRNA.
PNAs con show de residuos (P2 y P3) Q no termo fusión de transiciones (figura 7), no sugiriendo vinculante a la ssRNA. Esto es debido al choque estérico presente en Watson-Crick como par Q-G. En comparación con P1 PNA sin modificar, PNAs P4 y P5 que contiene modificaron L residuos pero sin residuos Q, mostrar disminución de temperatura de fusión para la dúplex RNA-PNA correspondiente debido al choque estérico presente en Watson-Crick como par L-G. Los datos de fusión termal detección de absorbancia UV son consistentes con los datos de valoración de fluorescencia 2-aminopurina, que también muestran que una PNA que contiene residuos de Q y L no se une a ssRNA apreciable (figura 6B, 6E, 6F). Incorpora una base Q es más desestabilizadora que una base de L, como una base Q tiene un choque estérico más importante en la formación de un par de Watson-Crick-como Q-G (figura 1F) comparado con un par de Watson-Crick-como L-G (figura 1E ).
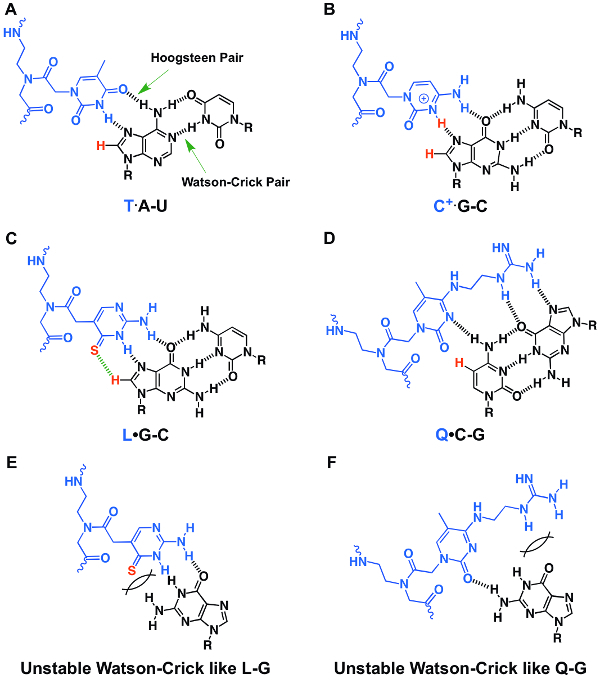
Figura 1 : Estructuras químicas de estructuras estable base triple e inestable par. (A, D) PNA· mayor-surco RNA2 base triples de T· A-U (A), C+· G-C (B), L· G-C (C) y Q· C-G (D). (E, F) Watson-Crick inestable como pares bajos PNA-RNA L-G (E) y Q-G (F). La letra R representa lo azúcar-fosfato de ARN. Enlaces de hidrógeno están indicados por líneas discontinuas negras. Se reproduce la figura de referencia31. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2 : Estructuras químicas de los monómeros PNA de. Se muestran cuatro monómeros PNA (T, C, L y Q). Haga clic aquí para ver una versión más grande de esta figura.
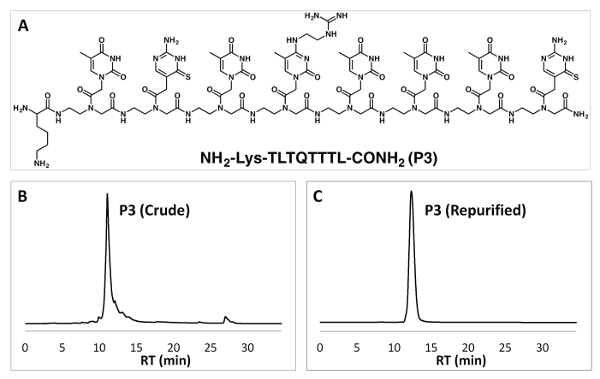
Figura 3 : Estructura química de un oligómero PNA y de purificación por RP-HPLC. (A) estructura química de la secuencia PNA P3. (B, C) Datos de RP-HPLC de crudo PNA P3 (B) y volver a PNA P3 (C). Haga clic aquí para ver una versión más grande de esta figura.
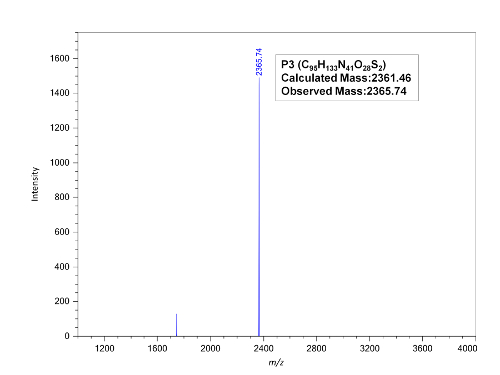
Figura 4 : Espectro MALDI-TOF de purificada PNA P3. Haga clic aquí para ver una versión más grande de esta figura.
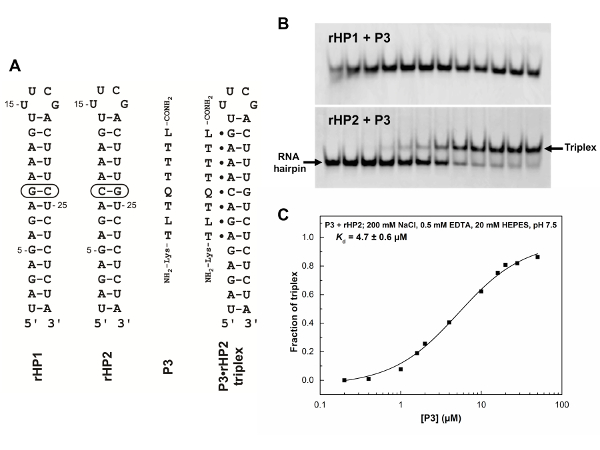
Figura 5 : Horquilla de RNA y las secuencias PNA y caracterización vinculante por página no desnaturalizantes. Horquillas para el pelo (A) RNA (rHP1 y rHP2), PNA P3 y un PNA· RNA2 triplex formado entre PNA P3 y rHP2. Página de desnaturalización no (B) (12%) resultados para rHP1 y atascamiento de rHP2 a PNA P3. El tampón de incubación es 200 mM NaCl, 0.5 mM EDTA 20 mM HEPES, pH 7,5. Las horquillas cargadas de RNA (rHP1 y rHP2) están a 1 μm de 20 μl. Las concentraciones de la Prefectura Naval Argentina en los carriles de izquierda a derecha son 0, 0.2, 0.4, 1, 1.6, 2, 4, 10, 16, 20, 28, y 50 μm. PNA P3 no enlazar a rHP1 (panel superior) pero se une a rHP2 (panel inferior). (C) Kd determinación P3 Unión rHP2. La fracción de la formación de triplex (Y) se enfrenta a la concentración de ANP. La figura es adaptada de la referencia31. Haga clic aquí para ver una versión más grande de esta figura.
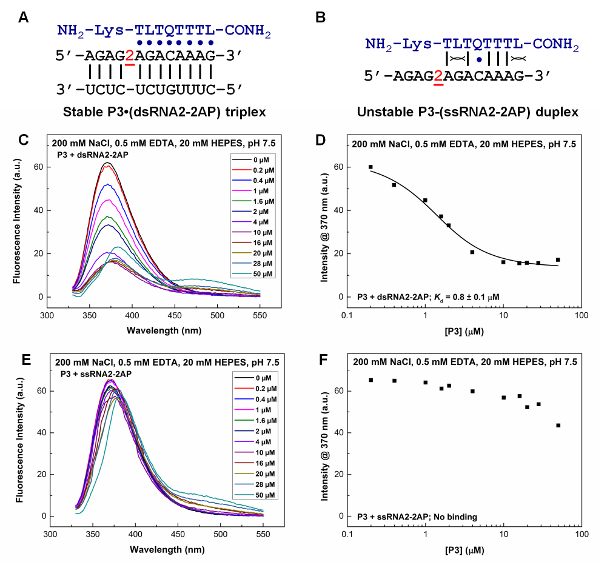
Figura 6 : Estudio de valoración de fluorescencia de enlace PNA P3a la 2-aminopurina-labeled RNAs. El residuo de la 2-aminopurina es señalado como '2' en la secuencia de RNA. El tampón de incubación es 200 mM NaCl, 0.5 mM EDTA 20 mM HEPES, pH 7,5. (A) un PNA· RNA2 triplex formado entre P3 y un dsRNA etiquetado 2-aminopurina (dsRNA2-2AP). (B) un duplex de PNA-RNA hipotético formado entre P3 y un 2-aminopurina-labeled ssRNA (ssRNA2-2AP). (C, E) Espectros de emisión de fluorescencia para la 2-aminopurina-labeled RNA duplex (1 μm) y ssRNA (1 μm), respectivamente, con variar P3 concentración a pH 7,5. El pico en alrededor de 475 nm es debido a la emisión de fluorescencia débil de la base de la L en la Prefectura Naval Argentina. (D, F) K d determinación basada en las parcelas de la intensidad de fluorescencia de la 2-aminopurina (a 370 nm) de la RNA duplex y ssRNA, respectivamente, frente a concentración de ANP P3. La figura es adaptada de la referencia31. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7 : Termo fusión resultados para dúplex RNA-Prefectura Naval Argentina. El tampón de incubación es 200 mM NaCl, 0.5 mM EDTA 20 mM NaH2PO4, pH 7,5. Todas las muestras contienen 5 μm de RNA monocatenario (ssRNA1) y PNA en 130 μl. (A) RNA monocatenario (ssRNA1), PNAs (P1, P2, P3, P4 y P5) y un dúplex de PNA-RNA hipotético formado entre PNA P3 y ssRNA1 en una orientación paralela. El choque estérico está indicado para el Crick de Watson como pares L G Q G. (B) fusión de curvas para diferentes PNAs a ssRNA1. La temperatura de fusión se muestra las curvas con transiciones de fusión. La figura es adaptada de la referencia31.
Discusión
Oligómeros PNA RNA duplex vinculante (por ejemplo, 10-mers) son moléculas de tamaño mediano y así puede mostrar cambio de movilidad electroforética a enlace de RNAs con un tamaño comparable o ligeramente mayor (por ejemplo, 50-mer o menor). Si un ARN es significativamente mayor que la Prefectura Naval Argentina, valoración de PNA en el RNA puede no funcionar debido a un cambio de movilidad limitada gel. Así, el RNA grande puede truncarse por los análisis de la página no desnaturalizantes. Valoración de un RNA grande en una PNA fluoróforo permite el seguimiento de la formación del triple por un gel de agarosa desnaturalizantes no con la muestra cargada en el gel de40.
Para un experimento de valoración por página no desnaturalizar con una concentración total constante de ARN, por lo general usamos una concentración de RNA sin etiqueta de 1 μm para eficiente post coloración del RNA libre y bandas triples por bromuro de etidio. Una concentración de RNA tan baja como 0.2 μm también puede ser suficiente según el RNA construir31. La concentración del RNA sin etiqueta (0,2 μm) determina que los valores ded de Kque pueden medirse con precisión deben ser de 0,2 μm o más grandes. Otros colorantes de tinción pueden utilizarse para mejorar la eficacia de la coloración. Por otra parte, nuestros datos no publicados sugieren que Cy3 RNAs de marcado tinte puede utilizarse en condiciones no desnaturalizantes página experimentos para medir los eventos atascamiento apretado.
Debido a que sólo es moderadamente fluorescente 2-aminopurina, 2-aminopurina valoración de fluorescencia también es limitada a la medida del enlace con valores ded de Kcerca o por encima de 0,2 μm31. El ARN o la ANP puede etiquetarse con un tinte relativamente brillante para cuantificar un atascamiento relativamente estrecha en la solución a través de la valoración de la fluorescencia, si la Unión resulta en cambios en la fluorescencia señales53,54, 55.
La estrategia de focalización estructuras de ARN por Unión dsRNA PNAs ha sido probada para un número limitado de RNAs. Es probable que enlace propiedades puede variar por dsRNAs con diferentes composiciones de secuencias y pares. Uno siempre puede elegir la hebra rica en purina de un dúplex para el diseño de TFPNAs. Es fundamental entender cómo consecutivos Q· C-G triples pueden afectar la estabilidad de un triplex. Estudios más extensos de la secuencia-dependientes son necesarias para comprender las propiedades de enlace dependiente de la secuencia de TFPNAs.
La afinidad de TFPNAs puede mejorarse aumentando la longitud y/o modificar aún más las bases y redes troncales56,57 TFPNAs. Sin embargo, una región continua de dos caras puede a menudo consiste de más de 10 pares de bases consecutivos sin la interrupción por estructuras de Watson-Crick. Uno puede conjugar TFPNAs con pequeñas moléculas para el reconocimiento de Watson-Crick estructuras adyacentes a las regiones de dsRNA. En principio, se espera que una conjugación de la molécula de TFPNA pequeñas han mejorado la afinidad y especificidad en comparación con un TFPNA o solamente pequeñas moléculas. Sin embargo, las propiedades químicas y físicas de la linker para la conjugación58,59,60,61,62,63,64 debe ser optimizado.
El hecho de que TFPNAs puede atar selectivamente a dsRNAs ssRNAs y dsDNAs sugiere que es posible desarrollar TFPNAs sondas químicas muy útiles como ligandos potenciales terapéuticos a través de la regulación de la dinámica estructural del RNA y las interacciones con proteínas y metabolitos. La absorción celular de TFPNAs puede facilitarse a través de la conjugación con moléculas de penetrar en la célula como moléculas pequeñas, péptidos y nanopartículas, o formación de complejos con estructuras supramoleculares como liposomas5,6 ,12,17,25,31,41,65,66. Funcionalización de TFPNAs con las etiquetas de Bioimagen como fluoróforos y radioisótopos puede facilitar aún más la detección, la proyección de imagen y el señalamiento de estructuras de RNA funcionales en los organismos vivos.
Divulgaciones
Que se haya presentado una solicitud de patente (PAT/179/14/15/PCT) basada en el trabajo reportado aquí.
Agradecimientos
Este trabajo fue apoyado por el Singapur Ministerio de Educación (MOE) nivel 1 (RGT3/13 y RG42/15 a G.C.) y MOE nivel 2 (MOE2013-T2-2-024 y MOE2015-T2-1-028 a G.C.).
Materiales
| Name | Company | Catalog Number | Comments |
| Molecular sieves, 4A, 1-2 mm diameter pellets | Alfa Aesar | 87956 | |
| 4-Methylbenzhydrylamine hydrochloride (MBHAŸHCl) | Sigma-Aldrich | 532444 | |
| N,N-diisopropylethylamine (DIPEA) | Alfa Aesar | A11801 | |
| (Benzotriazol-1-yl-oxy)tripyrrolidinophosphonium hexafluorophosphate (PyBOP) | Alfa Aesar | B25251 | |
| Acetic anhyride | Sigma-Aldrich | 320102 | |
| Kaiser Test Kit | Sigma-Aldrich | 60017 | |
| Trifluoroacetic acid (TFA) | Alfa Aesar | L06374 | |
| Unmodified PNA monomers | ASM Research Chemicals GmbH | 5004007, 5004008, 5004009, 5004010 | |
| Boc-Lys(Z)-OH / Fmoc-Lys(Boc)-OH | Sigma-Aldrich | B8389 / 47624 | |
| Thioanisole | Alfa Aesar | A14846 | |
| 1,2-Ethanedithiol | Alfa Aesar | L12865 | |
| Trifluoromethanesulfonic acid | Alfa Aesar | A10173 | |
| LiChrosper® 100 RP-18 endcapped (5 µm) LiChroCART® 250-4 | Merck Millipore | 150838 | |
| RNA Oligos | Sigma-Aldrich | Customized | |
| α-cyano-4-hydroxycinnamic acid (CHCA) | Sigma-Aldrich | 39468 | |
| Ethylenediaminetetraacetic acid (EDTA) | Alfa Aesar | J15694 | |
| HEPES | Lonza | 17-737E | |
| Acrylamide | Sigma-Aldrich | A8887 | |
| N.N'-methylenebisacrylamide | Sigma-Aldrich | 146072 | |
| Ammonium persulfate (APS) | Bio-rad | 161-0700 | |
| Tetramethylethylenediamine (TEMED) | Bio-rad | 161-0800 | |
| 10X Tris-Borate-EDTA (TBE) Buffer, pH 8.3 | 1st Base | BUF-3010-10X1L | |
| Glycerol | Promega | H5433 | |
| Ethidium bromide (10 mg/mL) | Bio-rad | 161-0433 | |
| High Precision Cell (Quartz Suprasil, 200-2500 nm) | Hellma Analytics | 105.250-QS |
Referencias
- Cech, T. R., Steitz, J. A. The noncoding RNA revolution-trashing old rules to forge new ones. Cell. 157 (1), 77-94 (2014).
- Velagapudi, S. P., Gallo, S. M., Disney, M. D. Sequence-based design of bioactive small molecules that target precursor microRNAs. Nat Chem Biol. 10, 291-297 (2014).
- Patil, K. M., Chen, G., Jurga, S., Erdmann, V. A., Barciszewski, J. . Modified Nucleic Acids in Biology and Medicine. , 299-317 (2016).
- Hyrup, B., Nielsen, P. E. Peptide nucleic acids (PNA): synthesis, properties and potential applications. Bioorg Med Chem. 4 (1), 5-23 (1996).
- Shiraishi, T., Nielsen, P. E. Improved cellular uptake of antisense peptide nucleic acids by conjugation to a cell-penetrating peptide and a lipid domain. Methods Mol Biol. 751, 209-221 (2011).
- Shiraishi, T., Nielsen, P. E. Nanomolar cellular antisense activity of peptide nucleic acid (PNA) cholic acid ("umbrella") and cholesterol conjugates delivered by cationic lipids. Bioconjugate Chem. 23 (2), 196-202 (2012).
- Khoo, B., Roca, X., Chew, S. L., Krainer, A. R. Antisense oligonucleotide-induced alternative splicing of the APOB mRNA generates a novel isoform of APOB. BMC Mol Biol. 8, 3 (2007).
- Stein, C. A., et al. Efficient gene silencing by delivery of locked nucleic acid antisense oligonucleotides, unassisted by transfection reagents. Nucleic Acids Res. 38 (1), 3 (2010).
- Kole, R., Krainer, A. R., Altman, S. RNA therapeutics: beyond RNA interference and antisense oligonucleotides. Nat Rev Drug Discov. 11 (2), 125-140 (2012).
- Peacey, E., Rodriguez, L., Liu, Y., Wolfe, M. S. Targeting a pre-mRNA structure with bipartite antisense molecules modulates tau alternative splicing. Nucleic Acids Res. 40 (19), 9836-9849 (2012).
- Stenvang, J., Petri, A., Lindow, M., Obad, S., Kauppinen, S. Inhibition of microRNA function by antimiR oligonucleotides. Silence. 3 (1), 1 (2012).
- Ma, X., et al. Intracellular delivery of antisense peptide nucleic acid by fluorescent mesoporous silica nanoparticles. Bioconjugate Chem. 25 (8), 1412-1420 (2014).
- Wojtkowiak-Szlachcic, A., et al. Short antisense-locked nucleic acids (all-LNAs) correct alternative splicing abnormalities in myotonic dystrophy. Nucleic Acids Res. 43 (6), 3318-3331 (2015).
- Lenartowicz, E., et al. Antisense Oligonucleotides Targeting Influenza A Segment 8 Genomic RNA Inhibit Viral Replication. Nucleic Acid Ther. 26 (5), 277-285 (2016).
- Avitabile, C., et al. Targeting pre-miRNA by peptide nucleic acids: a new strategy to interfere in the miRNA maturation. Artif DNA PNA XNA. 3 (2), 88-96 (2012).
- Barczak, A. K., et al. RNA signatures allow rapid identification of pathogens and antibiotic susceptibilities. Proc Natl Acad Sci USA. 109 (16), 6217-6222 (2012).
- Das, I., et al. A peptide nucleic acid-aminosugar conjugate targeting transactivation response element of HIV-1 RNA genome shows a high bioavailability in human cells and strongly inhibits tat-mediated transactivation of HIV-1 transcription. J Med Chem. 55 (13), 6021-6032 (2012).
- Fabani, M. M., et al. Efficient inhibition of miR-155 function in vivo by peptide nucleic acids. Nucleic Acids Res. 38 (13), 4466-4475 (2010).
- Riguet, E., et al. A peptide nucleic acid-neamine conjugate that targets and cleaves HIV-1 TAR RNA inhibits viral replication. J Med Chem. 47 (20), 4806-4809 (2004).
- Torres, A. G., et al. Chemical structure requirements and cellular targeting of microRNA-122 by peptide nucleic acids anti-miRs. Nucleic Acids Res. 40 (5), 2152-2167 (2012).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Mol Cell Proteomics. 12 (6), 1539-1552 (2013).
- Wesolowski, D., et al. Basic peptide-morpholino oligomer conjugate that is very effective in killing bacteria by gene-specific and nonspecific modes. Proc Natl Acad Sci USA. 108 (40), 16582-16587 (2011).
- Armitage, B. A. The impact of nucleic acid secondary structure on PNA hybridization. Drug Discov Today. 8 (5), 222-228 (2003).
- Thomas, S. M., et al. Antitumor effects of EGFR antisense guanidine-based peptide nucleic acids in cancer models. ACS Chem Biol. 8 (2), 345-352 (2013).
- Bahal, R., McNeer, N. A., Ly, D. H., Saltzman, W. M., Glazer, P. M. Nanoparticle for delivery of antisense gammaPNA oligomers targeting CCR5. Artif DNA PNA XNA. 4 (2), 49-57 (2013).
- Adams, B. D., Parsons, C., Walker, L., Zhang, W. C., Slack, F. J. Targeting noncoding RNAs in disease. J. Clin. Invest. 127 (3), 761-771 (2017).
- Matsui, M., Corey, D. R. Non-coding RNAs as drug targets. Nat Rev Drug Discov. 16 (3), 167-179 (2017).
- Devi, G., Zhou, Y., Zhong, Z., Toh, D. -. F. K., Chen, G. RNA triplexes: from structural principles to biological and biotech applications. Wiley Interdiscip Rev RNA. 6 (1), 111-128 (2015).
- Rozners, E. Recent Advances in Chemical Modification of Peptide Nucleic Acids. J Nucleic Acids. 2012, 8 (2012).
- Devi, G., Yuan, Z., Lu, Y., Zhao, Y., Chen, G. Incorporation of thio-pseudoisocytosine into triplex-forming peptide nucleic acids for enhanced recognition of RNA duplexes. Nucleic Acids Res. 42 (6), 4008-4018 (2014).
- Toh, D. K., et al. Incorporating a guanidine-modified cytosine base into triplex-forming PNAs for the recognition of a C-G pyrimidine-purine inversion site of an RNA duplex. Nucleic Acids Res. 44 (19), 9071-9082 (2016).
- Li, M., Zengeya, T., Rozners, E. Short peptide nucleic acids bind strongly to homopurine tract of double helical RNA at pH 5.5. J Am Chem Soc. 132 (25), 8676-8681 (2010).
- Gupta, P., Zengeya, T., Rozners, E. Triple helical recognition of pyrimidine inversions in polypurine tracts of RNA by nucleobase-modified PNA. Chem Commun. 47 (39), 11125-11127 (2011).
- Zengeya, T., Li, M., Rozners, E. PNA containing isocytidine nucleobase: synthesis and recognition of double helical RNA. Bioorg Med Chem Lett. 21 (7), 2121-2124 (2011).
- Gupta, P., Muse, O., Rozners, E. Recognition of double-stranded RNA by guanidine-modified peptide nucleic acids. Biochemistry. 51 (1), 63-73 (2012).
- Zengeya, T., Gupta, P., Rozners, E. Triple-helical recognition of RNA using 2-aminopyridine-modified PNA at physiologically relevant conditions. Angew Chem Int Ed. 51 (50), 12593-12596 (2012).
- Muse, O., et al. Sequence selective recognition of double-stranded RNA at physiologically relevant conditions using PNA-peptide conjugates. ACS Chem Biol. 8 (8), 1683-1686 (2013).
- Hnedzko, D., Cheruiyot, S. K., Rozners, E. Using Triple Helix Forming Peptide Nucleic Acids for Sequence-selective Recognition of Double-stranded RNA. Curr Protoc Nucleic Acid Chem. 58, 61-64 (2014).
- Zengeya, T., Gupta, P., Rozners, E. Sequence selective recognition of double-stranded RNA using triple helix-forming peptide nucleic acids. Methods Mol Biol. 1050, 83-94 (2014).
- Endoh, T., Hnedzko, D., Rozners, E., Sugimoto, N. Nucleobase-Modified PNA Suppresses Translation by Forming a Triple Helix with a Hairpin Structure in mRNA In Vitro and in Cells. Angew Chem Int Ed. 55 (3), 899-903 (2016).
- Hnedzko, D., McGee, D. W., Karamitas, Y. A., Rozners, E. Sequence-selective recognition of double-stranded RNA and enhanced cellular uptake of cationic nucleobase and backbone-modified peptide nucleic acids. RNA. 23 (1), 58-69 (2017).
- Wexselblatt, E., Esko, J. D., Tor, Y. On guanidinium and cellular uptake. J Org Chem. 79 (15), 6766-6774 (2014).
- Kaiser, E., Colescott, R. L., Bossinger, C. D., Cook, P. I. Color test for detection of free terminal amino groups in the solid-phase synthesis of peptides. Anal. Biochem. 34 (2), 595-598 (1970).
- Roberts, R. W., Crothers, D. M. Stability and properties of double and triple helices: dramatic effects of RNA or DNA backbone composition. Science. 258 (5087), 1463-1466 (1992).
- Zhou, Y., et al. Recognition of RNA duplexes by chemically modified triplex-forming oligonucleotides. Nucleic Acids Res. 41 (13), 6664-6673 (2013).
- Gisin, B. F. The monitoring of reactions in solid-phase peptide synthesis with picric acid. Anal. Chim. Acta. 58 (1), 248-249 (1972).
- Gogoi, K., Mane, M. V., Kunte, S. S., Kumar, V. A. A versatile method for the preparation of conjugates of peptides with DNA/PNA/analog by employing chemo-selective click reaction in water. Nucleic Acids Res. 35 (21), 139 (2007).
- Shabanpoor, F., Gait, M. J. Development of a general methodology for labelling peptide-morpholino oligonucleotide conjugates using alkyne-azide click chemistry. Chem Commun. 49 (87), 10260-10262 (2013).
- Rostovtsev, V. V., Green, L. G., Fokin, V. V., Sharpless, K. B. A Stepwise Huisgen Cycloaddition Process: Copper(I)-Catalyzed Regioselective "Ligation" of Azides and Terminal Alkynes. Angew Chem Int Ed. 41 (14), 2596-2599 (2002).
- Haaima, G., Hansen, F. H., Christensen, L., Dahl, O., Nielsen, P. E. Increased DNA binding and sequence discrimination of PNA oligomers containing 2,6-diaminopurine. Nucleic Acids Res. 25 (22), 4639-4643 (1997).
- Schroeder, S. J., Turner, D. H. . Methods Enzymol. 468, 371-387 (2009).
- McDowell, J. A., Turner, D. H. Investigation of the Structural Basis for Thermodynamic Stabilities of Tandem GU Mismatches: Solution Structure of (rGAGGUCUC)2 by Two-Dimensional NMR and Simulated Annealing. Biochemistry. 35 (45), 14077-14089 (1996).
- Sato, T., Sato, Y., Nishizawa, S. Triplex-Forming Peptide Nucleic Acid Probe Having Thiazole Orange as a Base Surrogate for Fluorescence Sensing of Double-stranded RNA. J Am Chem Soc. 138 (30), 9397-9400 (2016).
- Cheruiyot, S. K., Rozners, E. Fluorescent 2-Aminopyridine Nucleobases for Triplex-Forming Peptide Nucleic Acids. ChemBioChem. 17 (16), 1558-1562 (2016).
- Sato, T., Sato, Y., Nishizawa, S. Optimization of the Alkyl Linker of TO Base Surrogate in Triplex-Forming PNA for Enhanced Binding to Double-Stranded RNA. Chem Eur J. 23 (17), 4079-4088 (2017).
- Virta, P. M., Tahtinen, V., Granqvist, L., Murtola, M., Stromberg, R. 19F NMR spectroscopic analysis of the binding modes in triple helical PNA/microRNA-complexes. Chem Eur J. , (2017).
- Zengeya, T., Gindin, A., Rozners, E. Improvement of sequence selectivity in triple helical recognition of RNA by phenylalanine-derived PNA. Artif DNA PNA XNA. 4 (3), 69-76 (2013).
- Moses, A. C., Huang, S. W., Schepartz, A. Inhibition of Rev.RRE complexation by triplex tethered oligonucleotide probes. Bioorg Med Chem. 5 (6), 1123-1129 (1997).
- Ben Gaied, N., Zhao, Z., Gerrard, S. R., Fox, K. R., Brown, T. Potent triple helix stabilization by 5',3'-modified triplex-forming oligonucleotides. ChemBioChem. 10 (11), 1839-1851 (2009).
- Grimm, G. N., Boutorine, A. S., Lincoln, P., Norden, B., Helene, C. Formation of DNA triple helices by an oligonucleotide conjugated to a fluorescent ruthenium complex. ChemBioChem. 3 (4), 324-331 (2002).
- Tran, T., et al. Targeting the r(CGG) Repeats That Cause FXTAS with Modularly Assembled Small Molecules and Oligonucleotides. ACS Chem Biol. , (2014).
- Gianolio, D. A., Segismundo, J. M., McLaughlin, L. W. Tethered naphthalene diimide-based intercalators for DNA triplex stabilization. Nucleic Acids Res. 28 (10), 2128-2134 (2000).
- Rumney, S., Kool, E. T. Structural Optimization of Non-Nucleotide Loop Replacements for Duplex and Triplex DNAs. J Am Chem Soc. 117, 5635-5646 (1995).
- Stafford, R. L., Dervan, P. B. The reach of linear protein-DNA dimerizers. J Am Chem Soc. 129 (45), 14026-14033 (2007).
- Gupta, A., Bahal, R., Gupta, M., Glazer, P. M., Saltzman, W. M. Nanotechnology for delivery of peptide nucleic acids (PNAs). J Control Release. 240, 302-311 (2016).
- Avitabile, C., et al. Incorporation of Naked Peptide Nucleic Acids into Liposomes Leads to Fast and Efficient Delivery. Bioconjugate Chem. 26 (8), 1533-1541 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados