Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Alto rendimiento de secuenciación paralelo a la idoneidad de la medida de Leptospira interrogans Transposon inserción mutantes durante oro sirios Hamster infección
En este artículo
Resumen
Aquí describimos una técnica que combina la mutagénesis de transposon con secuenciación de alto rendimiento para identificar y cuantificar a mutantes de transposon leptoespiral en tejidos después de un desafío de hámsters. Este protocolo puede utilizarse para mutantes de pantalla para la supervivencia y difusión en los animales y también se puede aplicar a estudios en vitro .
Resumen
En este manuscrito, describimos un transposón (Tn-Seq) técnica para identificar y cuantificar a Leptospira interrogans mutantes alterados en gimnasio durante la infección de hámsters sirios dorados de secuenciación. TN-Seq combina mutagénesis de transposon al azar con el poder de la tecnología de secuenciación de alto rendimiento. Los animales se enfrentan con un grupo de mutantes de transposon (entrada piscina), seguido por la recolección de la sangre y los tejidos unos días más tarde para identificar y cuantificar el número de mutantes en cada órgano (piscinas de salida). Las piscinas de salida se comparan con la piscina entrada para evaluar la aptitud en vivo de cada mutante. Este enfoque permite la proyección de un gran número de mutantes en un número limitado de animales. Con pequeñas modificaciones, este protocolo se puede realizar con cualquier modelo animal de la leptospirosis, embalse host modelos como las ratas y la infección aguda como hámsters, así como estudios en vitro . TN-Seq proporciona una poderosa herramienta para pantalla para mutantes con defectos de fitness en vivo y en vitro .
Introducción
Identificación de genes de virulencia de algunas bacterias como Leptospira spp., es difícil debido al número limitado de herramientas genéticas disponibles. Un método comúnmente utilizado es la creación de una colección de mutantes por mutagénesis de transposon al azar seguido de la identificación del sitio de inserción en cada mutante y virulencia pruebas de mutantes de transposon individual en un modelo animal. Este enfoque es lento, costoso y requiere un gran número de animales.
Cuando mutagénesis al azar primero fue desarrollada para el patógeno Leptospira interrogans, genes implicados en virulencia fueron identificados por pruebas individuales mutantes en un modelo animal1. Mutantes se seleccionaron con base en criterios tales como su papel potencial en la señalización o motilidad o su membrana externa prevista o ubicación superficial. Como la mayoría de leptoespiral genes codifican proteínas hipotéticas de función desconocida2, selección de mutantes basan en estos límites de criterio la capacidad para descubrir genes de virulencia leptoespiral novela.
Más recientemente, revisaron las piscinas de mutantes de L. interrogans transposon de infectividad en los modelos de ratón y hámster3. Cada animal fue desafiada con una piscina de hasta 10 mutantes. Infectividad de un mutante lo marcó como positivo si fue detectado por PCR de cultivos obtenidos de sangre y los riñones. Pruebas de PCR fue laborioso, porque requiere una reacción de PCR individual para cada mutante en la piscina. Porque no se cuantificó la frecuencia de cada mutante en las culturas, el enfoque fue parcial hacia la identificación de mutantes altamente atenuadas.
Describimos un transposon secuencia técnica (Tn-Seq), como una estrategia para más eficientemente detectar genes de virulencia. TN-seq consiste en la creación de una biblioteca de mutantes por mutagénesis de transposon seguida de secuenciación masiva en paralelo4,5,6. Brevemente, mutantes de transposon se agruparon inoculados en animales y más tarde recuperados de diferentes órganos (piscinas de salida). El ADN de las piscinas de salida es extraído y digerido con enzimas de restricción o esquilado por sonicación. Se realizan dos rondas de PCR a las uniones de los sitios de inserción de transposones. Este paso permite la adición de los adaptadores necesarios para la secuenciación. Los productos PCR obtenidos son analizados por secuenciación de alto rendimiento para identificar el sitio de inserción del transposón de cada mutante de la piscina junto con su abundancia relativa, que es comparado con la composición inicial de la piscina del mutante.
La principal ventaja de este enfoque es la capacidad de la pantalla al mismo tiempo un gran número de mutantes con un pequeño número de animales. TN-Seq no requiere el conocimiento previo de los sitios de inserción de transposones que aumenta las posibilidades de descubrir nuevas Leptospira-genes específicos implicados en virulencia con menos tiempo y mayor eficiencia. Porque carga leptoespiral en tejidos es relativamente alta en roedores modelos susceptibles a la letal infección (normalmente 104 108 bacterias/g de tejido)7,8,9 , así como anfitriones del depósito 10,11, los tejidos pueden ser analizados directamente sin necesidad de cultivo, reduciendo sesgos debido al crecimiento en vitro .
En los estudios de Tn-Seq con mayoría patógenos bacterianos descritos hasta la fecha, la alta frecuencia de mutagénesis de insertional permite la infección con grandes piscinas que contengan a mutantes colectivamente con inserciones de transposones espaciamiento múltiples dentro de cada gen4 ,12,13,14. TN-Seq también ha sido desarrollado para una bacteria para que la frecuencia de mutagénesis es mucho inferior6. Con Leptospira, una biblioteca de mutantes de transposon puede generarse introduciendo el transposon en un plásmido movilizable por la conjugación de Slamti et al15. Sin embargo, la frecuencia de mutagénesis de transposon de L. interrogans es baja. Cuando el transposon Himar1 fue introducido en un plásmido conjugativos, la frecuencia de transconjugant se informó que solamente 8.5 x 10-8 por receptor de la célula con la cepa Lai de L. interrogans16 y es probable que del mismo modo pobre con más otras cepas de L. interrogans. El protocolo descrito aquí está en parte basado en que se convirtió para Borrelia burgdorferi, en el que la frecuencia de mutagénesis de insertional transposón es también baja6.
Para nuestro experimento piloto con el protocolo17, se realizó mutagénesis de transposon con L. interrogans serovar cepa Manilae L495 debido al éxito de otros grupos en el aislamiento de mutantes de inserción de transposones en la cepa junto con su bajo LD50 (dosis letal) de virulencia1. Se había defendido a 42 mutantes por Tn-Seq y se identificaron a varios candidatos mutantes defectuosos en virulencia, dos de ellos con inserciones en un gen de la adenilato ciclasa de candidato. Prueba individual de los dos mutantes en hámsteres confirmó que eran deficientes en virulencia17.
Protocolo
PRECAUCIÓN: Las cepas patógenas de Leptospira spp deben manipularse bajo procedimientos de contención de bioseguridad nivel 2 (BSL-2). Debe llevarse equipo de protección personal (EPP). Un gabinete de bioseguridad clase II debe ser utilizado para todas las manipulaciones de patógena Leptospira spp.
1. creación de los transposones mutante biblioteca15
-
Transferencia de los transposones en Leptospira spp. por conjugación (figura 1)
- Inocular un volumen de la fase exponencial cultura de spp de Leptospira patógena correspondiente a 107 células en 10 mL de Ellinghausen-McCullough-Johnson-Harris (EMJH) medio18,19. Incubar a 30 ° C con 150 rpm agitación hasta que la densidad llega a 2-8 x 108 células/mL.
Nota: El tiempo de duplicación de leptospiras patógenas es 12 a 24 h, dependiendo de la cepa. - Inoculan 50 μl del donante Escherichia coli cepa β216320 llevar el transposon movilizable plasmid (pCjTKS2)16 en 5 mL de caldo Luria (LB) suplementado con 0.3 mM de 2, 6-diaminopimelicacid (DAP), 50 μg/mL de kanamicina (Km) y 50 μg/mL de espectinomicina (Spc) y lugar durante la noche en una incubadora de 37 ° C a 255 rpm.
- Inocular 60 μL de células de e. coli en 3 mL de EMJH suplementado con 0.3m m de DAP (EMJH + DAP). Incubar a 37 ° C en agitación 255 rpm durante 3-4 h hasta un OD600 nm≈ 0.3.
- Para montar la unidad de filtración (figura 1), coloque la base en un brazo-lateral 125 mL matraz Erlenmeyer, colocar un filtro de acetato-celulosa (tamaño de poro 0,1 mm, diámetro 25 mm) en la base y el embudo en la base. Conecte la unidad de filtración a un sistema de vacío.
- Añadir 5 mL de Leptospira spp. cultura y 0,5 mL de cultivo de e. coli en el embudo. Aspire el líquido a través del filtro.
- Transferir el filtro con la superficie de las bacterias hacia arriba en un EMJH + DAPplate. Incubar a 30 ° C durante la noche con el filtro hacia arriba.
- Coloque el filtro en un tubo de 15 mL que contiene 1 mL de EMJH y de vortex por 10 s para liberar las bacterias en los medios de comunicación. Extensión 200 μL de la suspensión en 5 EMJH platescontaining 50 μg/mL de Km con 10-15 perlas de vidrio estéril de 1 mm o un separador desechable estéril. Envuelva las placas con parafilm e incúbelos invertida a 30 ° C durante 3 a 4 semanas hasta que las colonias sean visibles.
- Transferencia de colonias individualmente en 3 mL de EMJH 50 μg/ml de Km (EMJH + Km) a 30° C bajo agitación de 150 rpm durante 7 a 10 días hasta que la cultura alcance una densidad de ≈ 108/ml.
Nota: Las culturas pueden almacenarse a-80 ° C o en nitrógeno líquido (con 4% de glicerol).
- Inocular un volumen de la fase exponencial cultura de spp de Leptospira patógena correspondiente a 107 células en 10 mL de Ellinghausen-McCullough-Johnson-Harris (EMJH) medio18,19. Incubar a 30 ° C con 150 rpm agitación hasta que la densidad llega a 2-8 x 108 células/mL.
-
Identificación del sitio de inserción de transposones por PCR anidada (figura 2)
- Lisan 50 μl de cada mutante de transposon en tubos de PCR por la incubación a 95 ° C durante 15 minutos.
Nota: ADN puede purificarse en su lugar, utilizando un kit de extracción de ADN. - Preparar la mezcla PCR con iniciadores Deg1 y Tnk1 (tabla 3) según tabla 1. Transferencia de 23,7 μl de la mezcla a cada tubo PCR y agregar 1,3 μl de células sometidas a lisis. Ejecute el programa: 95° C por 5 min; 40 ciclos: 95 ° C por 15 s, 40 ° C por 1 min, 72 ° C por 2 min; 72 ° C durante 10 minutos.
- Hacer la mezcla PCR con iniciadores de etiqueta y TnkN1 (tabla 3) según tabla 2. Transferencia 24.2 μl de la mezcla a cada tubo PCR y añadir 0,8 μl de reacción de PCR #1. Ejecute el programa: 95 ° C por 5 min; 35 ciclos de: 95 ° C por 15 s, 55 ° C por 30 s, 72 ° C por 2 min; 72 ° C durante 10 minutos.
- Ejecutar 3 μl de los productos PCR en un gel de agarosa 1% con 1 Tampón X Tris-acetato-EDTA (TAE) en 10-15 V/cm (figura 2B).
- Purificar productos PCR de muestras positivas con el equipo de purificación PCR. Eluir el ADN con el volumen más bajo permitido por el kit para maximizar la concentración de ADN recuperado.
- Envíe productos PCR purificados para Sanger secuenciación utilizando la cartilla de TnkN1 (tabla 3).
- Identificar los sitios de inserción comparando la secuencia resultante con la secuencia del genoma de la cepa parental por análisis BLASTN (http://blast.ncbi.nlm.nih.gov/) o usando la base de datos (http://www.genoscope.cns.fr/agc/mage) SpiroScope del21.
- Confirmar el sitio de inserción del transposón por PCR usando las cartillas recocido a las secuencias que flanquean de host.
Nota: El transposón aumenta el tamaño de la secuencia wild type ≈ 2 kb.
- Lisan 50 μl de cada mutante de transposon en tubos de PCR por la incubación a 95 ° C durante 15 minutos.
2. animal experimento (figura 3)
-
Cultivo de Leptospira mutantes
- Crecer a individualmente cada mutante del transposon seleccionada en 10 mL de EMJH + Km a 30 ° C en agitación de 150 rpm a una densidad de7-10 108 leptospiras/mL.
- Cuenta las leptospiras por microscopia de campo oscuro con un contador de Petroff-Hausser o descrito por Miller23.
- Diluir cada cultura en EMJH para la misma densidad, por ejemplo 106 células/mL.
- Para montar la piscina entrada, mezclar las culturas diluidas en volúmenes iguales.
Nota: Incluir controles en la entrada piscina añadiendo mutantes con defectos de aptitud conocida como loa2217,24 y mutantes con aptitud inalterado como ligB17,25, respectivamente. Piscinas de mutantes pueden almacenarse con 4% de glicerol a-80 ° C o en nitrógeno líquido.
-
Desafío
Nota: Los métodos descritos aquí fueron aprobados por los veteranos asuntos mayor Los Ángeles institucional Animal cuidado y uso (protocolo #09018-14).- Inyectar por vía intraperitoneal de 1 mL de la piscina entrada a cada animal con una jeringa de insulina U-100 con aguja 26 x ½".
Nota: En el experimento piloto, 8 animales fueron desafiados con 1 mL de la piscina de entrada, es decir, 106 bacterias total17. La infección fue permitida proceder durante 4 días antes de la eutanasia. - Recoger 10 mL de la piscina entrada y vuelta por 20 min a x 3.220 g. eliminar cuidadosamente el sobrenadante sin perturbar el pellet. Guarde el precipitado de células en - 80˚C hasta su uso (paso 3.1.3).
- Supervisar diariamente los animales hasta que se terminan en el punto final determinado. Pesan los hámsteres diariamente y buscar criterios de punto final: pérdida de apetito, andar o respirar dificultad, postración, con volantes de piel, o la pérdida de > 10% del peso máximo alcanzado.
- Inyectar por vía intraperitoneal de 1 mL de la piscina entrada a cada animal con una jeringa de insulina U-100 con aguja 26 x ½".
-
Experimento in vitro
- En el día del desafío, inocular 3 matraces de 25 mL de EMJH + Km con 5 mL de la piscina entrada. Crecen cultivos a 30 ° C bajo agitación de 150 rpm.
- Cuenta las leptospiras diariamente mediante el uso de uno de los métodos de la sección 2.1.2. Cuando la densidad alcanza ≈ 1 x 108/ml, girar por cada suspensión por 20 min a 3.220 x g.
- Almacenar los pellets de células a-80 ° C hasta su uso.
-
Cosecha y almacenamiento de los tejidos
- Eutanasia a los animales por inhalación isoflurano seguida de toracotomía bilateral26.
- Recoja inmediatamente 1-2 mL de sangre por punción cardiaca con una jeringa de 3 mL y una aguja de 25 x 5/8". Transferencia de la sangre en tubo con EDTA. Mezclar por inversión, 5 o 6 veces.
- Recoger un riñón y ⅓ a 1/2 del lóbulo medio ventral del hígado en criotubos.
- Almacenar los tejidos a-80 ° C hasta su uso.
3. construcción de librerías genómicas de secuenciación de alto rendimiento (figura 4)
-
Extracción de ADN
- Extracción de ADN de la sangre.
- Transferir 100 μl de sangre desde el tubo de EDTA para un tubo de microcentrífuga.
- Purificar el ADN utilizando un kit de extracción de ADN. Siga las instrucciones del fabricante.
- Extracción de ADN de los tejidos.
- Utilizando bisturís y tijeras, dados entre 50 a 80 mg de cada órgano en trozos pequeños (1 mm x 1 mm) y transferir a un tubo seco estéril de tapón de rosca. Medir el peso del tejido con una balanza de precisión.
- Añadir 500 μl de PBS estéril en el tubo.
- Homogeneizar las muestras utilizando el disruptor durante 1 min a 5 movimientos por segundo.
- Calcular el volumen correspondiente a 25 mg de tejido utilizando la siguiente ecuación:
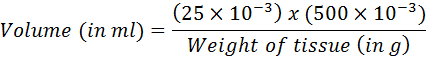
- Transferir el volumen calculado en el kit de extracción de ADN. Proceder a la purificación de ADN siguiendo las instrucciones del fabricante.
- Extracción de ADN de culturas entradas piscina y en vitro .
- Descongele la pelotillas bacteriano a temperatura ambiente durante 5-10 minutos.
- Proceder a la extracción de ADN siguiendo las instrucciones que acompañan el kit.
- ADN tienda a-80 ° C hasta su uso.
- Extracción de ADN de la sangre.
-
Corte del ADN (figura 5)
- Transferir 50 μl del ADN extraído en un tubo de microcentrífuga de 1,5 mL.
- Colocar los tubos en el rack del sonicador Copa cuerno llenado de agua fría (4 ° C).
- Hacer funcionar el sonicador durante 3 minutos en el 80% de intensidad con 10 s de 5 y pulso s de pulso.
PRECAUCIÓN: Usar orejeras o tapones para los oídos para proteger la audición. - 2.5 μl de la esquilada DNA en gel de agarosa al 2% para confirmar que la mayoría de la DNA es < 600 bp en tamaño.
-
Además de la C de cola
- Medir la concentración de ADN con un espectrofotómetro de pequeño volumen.
- Calcular el volumen correspondiente a 500 ng de ADN utilizando la siguiente ecuación:
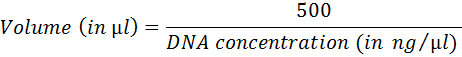
- Preparar la reacción de seguimiento (tabla 4). Incubar 1 h a 37 ° C; inactivar a 75 ° C por 20 min.
Nota: Para las muestras que requieren ajuste a un volumen superior a 14,5 μl, aumentar el volumen final de reacción a 40 μl y ampliar en consecuencia los componentes restantes. - Limpiar las muestras con un kit de potabilización de la polimerización en cadena. Eluir el ADN con 12 μl de tampón de elución.
-
PCR anidada
- POLIMERIZACIÓN EN CADENA #1
- Preparar mezclas PCR según las tablas 3 y 5. Transferencia 22 μL de la mezcla de la biblioteca a cada tubo PCR y agregar 3 μl de DNA purificado. Proceder igualmente con la mezcla de control.
Nota: Primer TnkN317 es específico para el transposón y primer olj3766 es específico a la C de cola. La mezcla de control carece de la cartilla de la TnkN3, que dirige específicamente el transposon. - Ejecutar el siguiente programa: 95 ° C por 2 min; 24 ciclos de: 95 ° C por 30 s, 60 ° C durante 30 s, 72 ° C por 2 min; 72 ° C por 2 min.
- Preparar mezclas PCR según las tablas 3 y 5. Transferencia 22 μL de la mezcla de la biblioteca a cada tubo PCR y agregar 3 μl de DNA purificado. Proceder igualmente con la mezcla de control.
- POLIMERIZACIÓN EN CADENA #2.
- Preparar las mezclas PCR segundo según las tablas 3 y 6. Transferencia 49 μl de la mezcla de la biblioteca a cada tubo PCR y añadir 1 μl de reacción de PCR #1 biblioteca. 24.5 μl de la mezcla de control la transferencia a cada tubo PCR y añadir 0,5 μl de reacción de PCR #1 control.
Nota: Primer pMargent2 es específico para el transposón y el IP cartillas6 contienen base-pares seis barras y secuencias específicas, reconocidas por la plataforma de secuenciación de próxima generación. - Ejecutar el siguiente programa: 95 ° C por 2 min; 18 ciclos de: 95 ° C por 30 s, 60 ° C durante 30 s, 72 ° C por 2 min; 72 ° C por 2 min.
- Preparar las mezclas PCR segundo según las tablas 3 y 6. Transferencia 49 μl de la mezcla de la biblioteca a cada tubo PCR y añadir 1 μl de reacción de PCR #1 biblioteca. 24.5 μl de la mezcla de control la transferencia a cada tubo PCR y añadir 0,5 μl de reacción de PCR #1 control.
- Ejecutar 3 μL en un gel de agarosa al 2%. La biblioteca debe mostrar un frotis con la mayoría de la señal entre 200 a 600 bp (figura 6) y la no amplificación por la reacción de control.
- POLIMERIZACIÓN EN CADENA #1
-
Purificación de productos PCR.
Nota: Limpie las bibliotecas genómicas con un kit de purificación PCR siguiendo las instrucciones del fabricante. Eluir el ADN con 30 μl de tampón de elución. - Medir la concentración de ADN con un fluorómetro. Promedio de 2 a 3 lecturas.
- Calcular el volumen de cada biblioteca equivalente a 15 o 20 ng utilizando la siguiente ecuación:
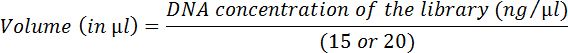
-
Mezclar todas las bibliotecas juntos según los anteriores cálculos. Determinar la concentración molar de la DNA con la ecuación de la siguiente página de Web:
http://www.molbiol.edu.ru/ENG/scripts/01_07.html
Atención: Requisitos de concentración y el volumen de ADN dependen de la plataforma de secuenciación.
4. alto rendimiento secuenciación y análisis de datos
- Secuencia
- Bibliotecas de secuencia como 64 Lee solo-fin de bp mediante la secuenciación personalizada cartilla pMargent3 y la cartilla de la secuencia comercial estándar. La plataforma de secuenciación le proporcionará FastQ archivos con todas las lecturas de secuenciación.
- Análisis con software Galaxy
- Descargar el archivo de secuencia de genoma
- En la página de base de datos de SpiroScope (ver paso 1.2.7 enlace), seleccione el organismo utilizado para el experimento y haga clic en la opción de "Carga en BROWSER del genoma".
- En la barra de herramientas (en la parte superior de la Página principal), seleccione "búsqueda y exportación > descargar datos", en la línea de "Secuencia (fasta)", haga clic en "Genoma" para descargar la secuencia.
- Abra el archivo con bloc de notas (PC) o TextEdit (Mac) y cambiar el nombre de cromosoma (por ejemplo "mente"). Mantener el formato de fasta.
- Siga los mismos pasos para descargar la secuencia del cromosoma II. Combinar ambas secuencias del cromosoma en un archivo .txt solo copiar y pegar.
Nota: Secuencias del cromosoma pueden ser descargadas desde el sitio web NCBI (https://www.ncbi.nlm.nih.gov) y combinar en un archivo .txt solo.
- Subir archivos en el servidor de la galaxia
Nota: El Galaxy es de código abierto, plataforma web para la gestión de datos bioinformática intensos flujos de trabajo27,28,29,30 y puede consultarse en https://usegalaxy.org/.- En el menú herramientas, seleccione "obtener datos > subir archivo desde su computadora". Arrastrar y soltar los archivos de .fastq generados por la plataforma de secuenciación en la ventana y haga clic en "Inicio".
- Siga los mismos pasos para cargar el archivo de .txt de secuencia de genoma de Leptospira .
- Novio Lee
- Seleccione "NGS: manipulación y control de calidad > FASTQ Groomer" en el menú herramientas.
- Junto al archivo para novio, seleccione las bibliotecas cargado en el paso 4.2.2. En entrada FASTQ calidad partituras tipo, seleccione el sistema apropiado de la secuencia. Para opciones avanzadas, deje ocultar avanzada oficina seleccionadas.
- Haga clic en "Ejecutar".
- Eliminar artefactos de secuenciación
- Seleccione "NGS: manipulación y control de calidad > eliminar artefactos de la secuencia". Junto a la biblioteca para filtrar, seleccione los archivos arreglados generados en el paso 4.2.3. Haga clic en "Ejecutar".
- Quitar secuencias C-tail
Nota: Repita estos pasos una vez o dos veces para todos C-colas se quitan.- Seleccione "NGS: manipulación y control de calidad > Clip secuencias de adaptador".
- Seleccione o escriba lo siguiente:
Clip de la biblioteca: seleccione los archivos generados en el paso 4.2.4.
Longitud mínima de la secuencia: 15
Fuente: introducir secuencia personalizada
Introduzca la secuencia de recorte personalizado: CCCCCCC
Introduzca valor distinto de cero para mantener el adaptador secuencias y bases x que le siguen: 0
Deseche las secuencias con desconocido (N) bases: sí
Opciones de salida: salida de secuencias recortadas y sin recortar - Haga clic en "Ejecutar".
- Quitar secuencias de adaptador
- Seleccione "NGS: manipulación y control de calidad > Clip secuencias de adaptador".
- Seleccione o escriba lo siguiente:
- Clip de la biblioteca: seleccione los archivos generados en el paso 4.2.5.
- Longitud mínima de la secuencia: 15
- Fuente: introducir secuencia personalizada
- Introduzca la secuencia de recorte personalizado: CGTATGCCGTCTTCTGCTTG
- Introduzca valor distinto de cero para mantener el adaptador secuencias y bases x que le siguen: 0
- Deseche las secuencias con desconocido (N) bases: sí
- Opciones de salida: salida de secuencias recortadas y sin recortar
- Haga clic en "Ejecutar".
- Filtro lee en base a su calidad
- Seleccione "NGS: manipulación y control de calidad > filtro de calidad".
Nota: Esta herramienta selecciona lecturas basadas en los resultados de calidad. - Seleccione los siguientes:
Biblioteca de filtro: seleccione los archivos generados en el paso 4.2.6.
Valor de corte de calidad: 20
Por ciento de las bases en la secuencia que debe tener calidad igual a o más alta que el valor de corte: 95 - Haga clic en "Ejecutar".
Nota: Con estas opciones, se descartan lecturas más cortas de 20 nucleótidos o con una puntuación de calidad de 20 o menos para el 95% de los ciclos. Adaptar los ajustes de rigor para su experimento.
- Seleccione "NGS: manipulación y control de calidad > filtro de calidad".
- Mapa Lee32
- Seleccione "NGS: cartografía > Bowtie2"32.
- Seleccione lo siguiente en los campos en la ventana principal:
Es esta biblioteca unitarios o emparejada: solo-fin
Fichero FASTQ: seleccione la biblioteca de filtrado para la calidad del paso 4.2.7.
Escribir no alineados (en formato fastq) lecturas para separar archivos: no
Escribir alineadas (en formato fastq) lecturas para separar archivos: no
¿Seleccione un genoma de referencia de su historia o utilizar un índice incorporado?: utilizar un genoma de la historia y construir el índice
Seleccione el genoma de referencia: seleccione el archivo de genome.txt de Leptospira cargado en el paso 4.2.2.
Conjunto de Lee la información de los grupos?: no
Modo de análisis Select: 1: por defecto configuración sólo
¿Desea usar presets?: No, sólo tiene que utilizar por defecto
Guardar las estadísticas de asignación de bowtie2 en la historia: No
Parámetros de recursos de empleo: utilizar parámetros de recursos de trabajo por defecto - Haga clic en "Ejecutar" para alinear Lee al genoma.
- Convertir archivos
- Seleccione "NGS: SAMtools > BAM a SAM convertir BAM a SAM".
- Seleccione los siguientes:
Archivo de BAM para convertir: seleccione asignado biblioteca de paso 4.2.8.
Opciones de encabezado: incluir encabezado en salida de SAM (-h) - Haga clic en "Ejecutar".
- Convertir archivos
- Seleccione "NGS: SAMtools > convertir SAM intervalo".
- Seleccione los siguientes:
Seleccione conjunto de datos a convertir: seleccione el archivo de biblioteca asignadas SAM generado en el paso 4.2.9.
Imprimir todo?: sí - Haga clic en "Ejecutar".
- Suerte Lee
- Seleccione "filtrar y ordenar > ordenar los datos en orden ascendente o descendente".
- Seleccione los siguientes:
Conjunto de datos de tipo: seleccione el archivo de intervalo generado en el paso 4.2.10.
en la columna: 2
con sabor: orden numérico
todo en: orden ascendente - Haga clic en "Ejecutar".
- Seleccione lecturas que cromosoma
- Seleccione "filtrar y ordenar > seleccionar las líneas que coincidan con una expresión".
- Seleccione los siguientes:
Seleccionar líneas de: seleccione Archivo clasificado lecturas generada en el paso 4.2.11.
que: coincidencia
el patrón: Introduzca el nombre de cromosoma determinado en el paso 4.2.1.3 (p. ej., "mente"). - Haga clic en "Ejecutar".
- Seleccione Lee correspondiente cromosoma II
Proceder siguiendo paso 4.2.12. - Grupo Lee según los sitios de inserción en el cromosoma I
- Seleccionar "unir, sustraer y grupo > agrupar datos por una columna y realizar operación agregada en otras columnas".
- Seleccione los siguientes:
Seleccionar datos: seleccionar archivo resultante del paso 4.2.12.
Grupo de columnas: 2
Ignorar el caso mientras la agrupación?: No
Ignorar las líneas a partir de estos personajes: Ø
Operación > + Insertar operación
Tipo: cuenta
En la columna: 2
Ronda el resultado al entero más cercano: NO - Haga clic en "Ejecutar".
- Grupo Lee según los sitios de inserción en el cromosoma II
Proceder siguiendo paso 4.2.14. - Sitios de inserción de tipo en el cromosoma I
- Seleccione "filtrar y ordenar > ordenar los datos en orden ascendente o descendente".
- Seleccione los siguientes:
Conjunto de datos de tipo: Seleccione el archivo del paso 4.2.14.
en la columna: 1
con sabor: orden numérico
todo en: orden ascendente - Haga clic en "Ejecutar".
- Ordenar los sitios de inserción en el cromosoma II
Proceder siguiendo paso 4.2.16.
- Descargar el archivo de secuencia de genoma
- Análisis estadístico
- Transferir los datos de la galaxia a archivos de hoja de cálculo por copiar y pegar las dos columnas de pasos 4.2.16. y 4.2.17. en un archivo de Excel.
Nota: La primera columna es la coordenada del nucleótido del sitio de inserción de transposones, y la segunda columna es el número de lecturas en cada sitio de inserción. - Identifican el gen que el transposon usando la coordenada de nucleótido en la tabla.
Nota: por ejemplo, un transposón insertado en el nucleótido #30718 del cromosoma es situado en el gen LIC10024 , que se extiende por nucleótidos cromosoma en 29263-31539. - Calcular frecuencias relativas (F) para cada mutante en cada tejido y en la piscina entrada siguiendo la siguiente ecuación:
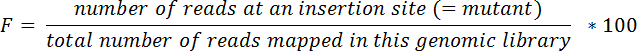
- Calcular relaciones de entrada y salida (R) para cada mutante y tejido usando la siguiente ecuación:
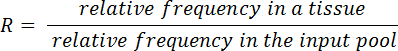
- Wilcoxon firmar-alinea la prueba
- Normalizar todos relaciones de entrada y salida mediante el establecimiento de la relación media de cada tejido en cada animal a 1.0. Una proporción de 1.0 es neutral, > 1.0 es desventajosa, y < 1.0 es ventajoso33.
- Comparar relaciones de entrada y salida a 1.0 (aptitud neutral) usando la prueba de Wilcoxon rank con valores de P < 0,05 considerado como estadísticamente significativo.
- Transferir los datos de la galaxia a archivos de hoja de cálculo por copiar y pegar las dos columnas de pasos 4.2.16. y 4.2.17. en un archivo de Excel.
Resultados
Creación de una biblioteca de mutantes de transposon en L. interrogans por verbal requiere una unidad de filtración, como se muestra en la figura 1. Hemos recuperado 100-200 transconjugants de cada apareamiento.
El sitio de inserción del transposón se identifica en cada mutante por secuenciación del producto PCR generado por PCR semi-random que se dirige al final de los transposones y ...
Discusión
Aunque se presentan los resultados de nuestro experimento piloto para hámster desafiado por vía intraperitoneal con mutantes de L. interrogans 4217, esperamos que piscinas más grandes de mutantes pueden ser defendidos por Tn-SS. Porque la frecuencia de transconjugants es baja (100-200 transconjugants/apareamiento), varios apareamientos son necesarias para generar un número suficiente de mutantes para los grandes experimentos de Tn-Seq. Mantener un gran número de mutantes en cultivos ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por un premio de mérito de asuntos de veteranos (para D.A.H.) y un Instituto Nacional de salud concede R01 AI 034431 (a D.A.H.).
Materiales
| Name | Company | Catalog Number | Comments |
| Kanamycin sulfate from Streptomyces kanamyceticus | Sigma-Aldrich | K4000 | |
| 2,6-diaminopimelic acid | Sigma-Aldrich | D1377 | |
| Spectinomycin dihydrochloride pentahydrate | Sigma-Aldrich | S4014 | |
| Axio Lab A1 microscope with a darkfieldcondenser | Zeiss | 490950-001-000 | |
| DNeasy blood and tissue kit | Qiagen | 69504/69506 | |
| MinElute PCR Purification | Qiagen | 28004/28006 | |
| QIAquick PCR purification kit | Qiagen | 28104/28106 | |
| Model 505 Sonic Dismembrator | Fisher Scientific | FB-505 | |
| 2.5" Cup horn | Fisher Scientific | FB-4625 | |
| Bead Ruptor 24 | Omni International | 19-010 | Step 3.1.2.4 |
| Terminal deoxynucleotidyl transferase | Promega | M828C | |
| Master mix Phusion | Thermo Scientific | F531 | Preparation of genomic libraries, step 3.4. |
| DreamTaq Master Mix | Thermo Scientific | K9011/K9012 | Identification of the transposon insertion site, step 1.2. |
| dCTP | Thermo Scientific | R0151 | |
| ddCTP | Affymetrix/ USBProducts | 77112 | |
| T100 Thermal cycler | BioRad | 1861096 | |
| Qubit 2.0 fluorometer | Invitrogen | Q32866 | step 3.6. |
| Qubit dsDNA HS assay kit | Invitrogen | Q32851/Q32854 | step 3.6. |
| Qubit assay tubes | life technologies | Q32856 | step 3.6. |
| PBS pH 7.2 (1X) | Gibco | 20012-027 20012-050 | |
| Disposable scalpel No10 | Feather | 2975#10 | |
| Plastic K2 EDTA 2 ml tubes | BD vacutainer | 367841 | |
| syringe U-100 with 26G x ½” needle | BD vacutainer | 329652 | IP challenge, step 2.2.1. |
| 3 mL Luer-Lok tip syringe | BD vacutainer | 309657 | Cardiac puncture, step 2.4.2. |
| 25G X 5/8” needle | BD vacutainer | 305901 | Cardiac puncture, step 2.4.2. |
| 25 mm fritted glass base with stopper | EMD Millipore | XX1002502 | Filtration unit system, step 1.1.7. |
| 25 mm aluminum spring clamp | EMD Millipore | XX1002503 | Filtration unit system, step 1.1.7. |
| 15 ml borosilcate glass funnel | EMD Millipore | XX1002514 | Filtration unit system, step 1.1.7. |
| 125 ml side-arm Erlenmeyer flask | EMD Millipore | XX1002505 | Filtration unit system, step 1.1.7. |
| Acetate-cellulose filter VVPP (pore size 0.1 mm; diameter 25 mm) | EMD Millipore | VVLP02500 |
Referencias
- Murray, G. L., et al. Genome-wide transposon mutagenesis in pathogenic Leptospira species. Infect Immun. 77 (2), 810-816 (2009).
- Adler, B., Lo, M., Seemann, T., Murray, G. L. Pathogenesis of leptospirosis: the influence of genomics. Vet Microbiol. 153 (1-2), 73-81 (2011).
- Marcsisin, R. A., et al. Use of a high-throughput screen to identify Leptospira mutants unable to colonize the carrier host or cause disease in the acute model of infection. J Med Microbiol. 62, 1601-1608 (2013).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat Methods. 6 (10), 767-772 (2009).
- van Opijnen, T., Lazinski, D. W., Camilli, A. Genome-wide fitness and genetic interactions determined by Tn-seq, a high-throughput massively parallel sequencing method for microorganisms. Curr Protoc Microbiol. 36, 1E.3.1-1E.3.24 (2015).
- Troy, E. B., et al. Understanding barriers to Borrelia burgdorferi dissemination during infection using massively parallel sequencing. Infect Immun. 81 (7), 2347-2357 (2013).
- Wunder, E. A., et al. Real-time PCR reveals rapid dissemination of Leptospira interrogans after intraperitoneal and conjunctival inoculation of hamsters. Infect Immun. 84 (7), 2105-2115 (2016).
- Lourdault, K., Aviat, F., Picardeau, M. Use of quantitative real-time PCR for studying the dissemination of Leptospira interrogans in the guinea pig infection model of leptospirosis. J Med Microbiol. 58, 648-655 (2009).
- Coutinho, M. L., et al. Kinetics of Leptospira interrogans infection in hamsters after intradermal and subcutaneous challenge. PLoS Negl Trop Dis. 8 (11), e3307 (2014).
- Athanazio, D. A., et al. Rattus norvegicus as a model for persistent renal colonization by pathogenic Leptospira interrogans. Acta Trop. 105 (2), 176-180 (2008).
- Ratet, G., et al. Live imaging of bioluminescent Leptospira interrogans in mice reveals renal colonization as a stealth escape from the blood defenses and antibiotics. PLoS Negl Trop Dis. 8 (12), e3359 (2014).
- Gutierrez, M. G., Yoder-Himes, D. R., Warawa, J. M. Comprehensive identification of virulence factors required for respiratory melioidosis using Tn-seq mutagenesis. Front Cell Infect Microbiol. 5 (78), (2015).
- Gawronski, J. D., Wong, S. M., Giannoukos, G., Ward, D. V., Akerley, B. J. Tracking insertion mutants within libraries by deep sequencing and a genome-wide screen for Haemophilus genes required in the lung. Proc Natl Acad Sci U S A. 106 (38), 16422-16427 (2009).
- Gallagher, L. A., Shendure, J., Manoil, C. Genome-scale identification of resistance functions in Pseudomonas aeruginosa using Tn-seq. MBio. 2 (1), (2011).
- Slamti, L., Picardeau, M. Construction of a library of random mutants in the spirochete Leptospira biflexa using a mariner transposon. Methods Mol Biol. 859, (2012).
- Picardeau, M. Conjugative transfer between Escherichia coli and Leptospira spp. as a new genetic tool. Appl Environ Microbiol. 74 (1), 319-322 (2008).
- Lourdault, K., Matsunaga, J., Haake, D. A. High-throughput parallel sequencing to measure fitness of Leptospira interrogans transposon insertion mutants during acute infection. PLoS Negl Trop Dis. 10 (11), e0005117 (2016).
- Ellinghausen, H. C., McCullough, W. G. Nutrition of Leptospirapomona and growth of 13 other serotypes: Fractionation of oleic albumin complex and a medium of bovine albumin and polysorbate 80. Am J Vet Res. 26, 45-51 (1965).
- Johnson, R. C., Harris, V. G. Differentiation of pathogenic and saprophytic letospires. I. Growth at low temperatures. J Bacteriol. 94 (1), 27-31 (1967).
- Demarre, G., et al. A new family of mobilizable suicide plasmids based on broad host range R388 plasmid (IncW) and RP4 plasmid (IncPa) conjugative machineries and their cognate Escherichia coli host strains. Res Microbiol. 156 (2), 245-255 (2005).
- Vallenet, D., et al. MicroScope: a platform for microbial genome annotation and comparative genomics. Database (Oxford). , bap021 (2009).
- Goodman, A. L., Wu, M., Gordon, J. I. Identifying microbial fitness determinants by insertion sequencing using genome-wide transposon mutant libraries. Nat. Protocols. 6 (12), 1969-1980 (2011).
- Miller, J. . Spirochetes in body fluids and tissues manual of investigative methods. , 22-23 (1971).
- Ristow, P., et al. The OmpA-like protein Loa22 is essential for leptospiral virulence. PLoS Pathog. 3 (7), (2007).
- Croda, J., et al. Targeted mutagenesis in pathogenic Leptospira species: disruption of the LigB gene does not affect virulence in animal models of leptospirosis. Infect Immun. 76 (12), 5826-5833 (2008).
- Haake, D. A. Hamster model of leptospirosis. Curr Protoc Microbiol. 12, 2 (2006).
- Giardine, B., et al. Galaxy: a platform for interactive large-scale genome analysis. Genome Res. 15 (10), 1451-1455 (2005).
- Goecks, J., Nekrutenko, A., Taylor, J., Galaxy, T. Galaxy: a comprehensive approach for supporting accessible, reproducible, and transparent computational research in the life sciences. Genome Biol. 11 (8), R86 (2010).
- Blankenberg, D., et al. Galaxy: a web-based genome analysis tool for experimentalists. Curr Protoc Mol Biol. (Chapter 19), 11-21 (2010).
- Afgan, E., et al. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Res. 8 (44), (2016).
- McCoy, K. M., Antonio, M. L., van Opijnen, T. MAGenTA; a Galaxy implemented tool for complete Tn-Seq analysis and data visualization. Bioinformatics. , 1367 (2017).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biol. 10 (3), R25 (2009).
- Kamp, H. D., Patimalla-Dipali, B., Lazinski, D. W., Wallace-Gadsden, F., Camilli, A. Gene fitness landscapes of Vibrio cholerae at important stages of its life cycle. PLoS Pathog. 9 (12), e1003800 (2013).
- Poggi, D., Oliveira de Giuseppe, P., Picardeau, M. Antibiotic resistance markers for genetic manipulations of Leptospira spp. Appl Environ Microbiol. 76 (14), 4882-4885 (2010).
- van Opijnen, T., Camilli, A. Transposon insertion sequencing: a new tool for systems-level analysis of microorganisms. Nat Rev Microbiol. 11 (7), 435-442 (2013).
- Pappas, C. J., Picardeau, M. Control of gene expression in Leptospira spp. by Transcription activator-like effectors demonstrates a potential role for LigA and LigB in Leptospira interrogans virulence. Appl Environ Microbiol. 81 (22), 7888-7892 (2015).
- Koskiniemi, S., Sun, S., Berg, O. G., Andersson, D. I. Selection-driven gene loss in bacteria. PLoS Genet. 8 (6), e1002787 (2012).
- Troy, E. B., et al. Global Tn-seq analysis of carbohydrate utilization and vertebrate infectivity of Borrelia burgdorferi. Mol Microbiol. 101 (6), 1003-1023 (2016).
- Ramsey, M. E., et al. A high-throughput genetic screen identifies previously uncharacterized Borrelia burgdorferi genes important for resistance against reactive oxygen and nitrogen species. PLoS Negl Trop Dis. 13 (2), e1006225 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados