Method Article
Protocolo de diferenciación cerebelosa 3D optimizada con modificación 2D opcional
En este artículo
Resumen
Describimos un medio de protocolo para hPSCs, utilizando la diferenciación 3D simplificada y reducida de factores de crecimiento, capaces de generar agregados de células con estructuras neuroepiteliales temprano y el positivos para cerebelosa asociada a marcadores, así como un opcional Modificación 2D para diferenciar las células como una monocapa para generar neuronas funcionales.
Resumen
Reducir la complejidad y el costo de los protocolos de diferenciación es importante para los investigadores. Este interés se ajusta con las preocupaciones acerca de posibles efectos no deseados que factores extrínsecos patrones podrían introducir en la célula de vástago de pluripotent humanas (hPSC) los modelos de desarrollo del cerebro y Fisiopatología, como enmascarar el fenotipo de la enfermedad. Aquí, presentamos dos protocolos de diferenciación cerebelosa hPSCs, diseñado con el método de inicio más simple, menos factores de diseño y menos requisitos materiales que protocolos anteriores. Recientemente, hemos desarrollado procedimientos de cultivo, que generan (flotante 3 dimensiones 3D) los productos compatibles con otros protocolos de "organoide" del cerebro, incluyendo morfología rómbica y relevante a modelado el desarrollo del cerebro como zona ventricular sub- labio-como las estructuras. La segunda utiliza un procedimiento de monocapa adherente, 2D a la completa diferenciación, que se muestra capaz de generar neuronas funcionales cerebelosas, como productos son positivos para los marcadores cerebelosa asociada y exhiben flujos de calcio similar a la neurona. Juntos, estos protocolos ofrecen a los científicos de opciones adecuada para fines de investigación, así como un modelo básico para probar otros tipos de diferenciación neuronal optimizada.
Introducción
Protocolos in vitro para diferenciar hPSCs hacia linajes cerebelosas inicialmente funcionan en el principio de la mímica en vivo el desarrollo cerebeloso1,2,3,4. Como tal, requiere una sucesión de factores introducidos en momentos específicos para impulsar la maduración y patrones pro-cerebelosa. Entre éstos estaban WNT, ósea proteínas morfogenéticas (BMPs) y factores de crecimiento fibroblástico (FGFs) con papeles conocidos en cerebelo medio desarrollo y formación de la ístmica organizador5,6,7. Por supuesto, cada paso adicional y factor significa un incremento en manipulaciones que requieren mucho trabajo y gasto mayor para el investigador, y así el desarrollo de protocolos más simples capaces de lograr resultados iguales es de interés. Este problema práctico encaja muy bien con la hipotética pregunta, si las células requieren tal apretado control externo sobre su desarrollo in vitro.
Diferenciación cerebelosa, un protocolo publicado en 2015 aborda la necesidad de utilizar una gran cantidad de factores de crecimiento utilizando sólo FGF2 FGF19 y factor derivado de células estromal 1 (SDF1) patrones objetivos8. Este estudio también diferenció de anteriores protocolos cerebelosos, mediante un sistema de flotación libre cultura 3D. Además de producir las células positivas para marcadores cerebelosos, el cerebro "organoides" generadas por su técnica mostraron exhiben morfología pertinente, disponible en culturas tradicionales monocapa 2D, como estructuras labio rómbicas. Aunque menos compleja y costosa con respecto a los factores de crecimiento, otras características tales como la formación de cuerpos de embryoid uniforme (EBs) y la cultura en placas de 96 pocillos (96WPs), hecho procesalmente compleja durante los pasos iniciales. Otro protocolo 3D había publicado el mismo año, informó la diferenciación exitosa linajes neuronales usando celular común y barato cultura técnicas9. Aunque este grupo estaba investigando en lugar de diferenciación cerebelosa cortical, aplicación de su concepto de diferenciación cerebelosa no podría ser descontado.
Nos informó recientemente un protocolo de diferenciación cerebelosa 3D utilizando un número reducido de factores de diseño (es decir, FGF2, 4 y 8), así como una configuración simplificada manteniendo las células en placas de 6 pozos (6WPs) en todas partes para reducir al mínimo necesidades a medio10. Para ayudar a la producción de células del gránulo, agonista pulida (SAG) fue utilizado durante la etapa de maduración final. SAG es una alternativa menos costosa química a sonic hedgehog (SHH), que había sido utilizado en anteriores protocolos cerebelosos, debido a su papel en promover el crecimiento del gránulo de la célula precursores (GCP) en vivo1,2, 11,12,13. Diferenciación productos fueron consistentes con los de otros protocolos 3D, incluyendo la presencia de marcadores cerebeloso asociado en estructuras morfológicamente relevantes8,9. Estos resultados refuerzan el mensaje anterior que detallan la mímica en vivo ambiente puede no ser necesario para el complejo 3D en vitro protocolos de diferenciación.
Además del protocolo 3D, este informe describe un protocolo 2D, diseñado con la misma configuración rápida, materiales básicos y reducido número de factores de crecimiento. Es capaz de producir las células de células madre embrionarias humanas (hESCs) o inducida de células madre pluripotentes (hiPSCs), positivo para los marcadores asociados a principios neuronales, cerebelosa e identidades de la célula del gránulo. Además, la proyección de imagen de calcio indica la presencia de neuronas funcionales humanas. La posibilidad de elegir entre protocolos, agrega un nivel de flexibilidad para los investigadores, para los interesados ya sea: (1) generar células específicas tipos, desarrollo del cerebro humano (2) modelado y asociadas estructuras, (3) análisis optimizado en monocapa ajustes (por ejemplo, grabaciones de patch-clamp), o (4) interacciones de la célula-célula en cultivos neuronales mixtos. Su naturaleza simple y de bajo costo los hace accesibles para los investigadores que son nuevos en el campo de hPSC, o necesitan de procedimientos hPSC base para explorar otras opciones de diferenciación.
Protocolo
1. preparaciones
Nota: Para todas las medidas consulte Tabla de materiales para elementos específicos.
-
Preparar medio de cultivo de 500 mL definido hPSC hPSC cultura
Nota: Use medio de pasos 2.1-2.6.- Descongelar hPSC medio suplemento durante la noche (o/n) a 4 ° C. Quitar 12,5 mL de medio base hPSC de media botella, luego añadir 10 mL de suplemento y 2,5 mL (100 U/L por lo que) penicilina/estreptomicina (Pen/Strep) a la botella.
- Almacenar a 4 ° C y usar dentro de 2 semanas.
Nota: Se puede utilizar el medio de hPSC congelar abajo las células mediante la adición de 10 μm ROCK inhibidor (RI) y 10% DMSO.
-
Preparar medio de mantenimiento neuronal 1 L (NMM) para la cultura de la diferenciación
Nota: Use medio de pasos 3.1-4.4.- Glutamina mezcla fortificada DMEM/F12 y medio de base neural (proporción 1:1) en 1 L botella, entonces suplemento con suplemento de N2 (1 x), (1 x) suplemento B27, 5 μg/mL insulina, 1,5 mM L-glutamina, 100 μm aminoácidos no esenciales (AANE), 100 U/L pluma/Strep y 10 μm beta-mercaptoetanol.
- Almacenar el medio a 4 ° C y usar dentro de 3 semanas.
Nota: Antes de agregar suplementos a mixto medio basal, quitar el volumen adecuado para ajustar el volumen requerido de agregado componentes basados en concentraciones de población.
-
Preparar 500 mL de solución de trabajo de 0,5 mM EDTA pases hPSCs
Nota: Use medio de pasos 2.4 y 2.5.- Bajo una campana de flujo transferencia 49 mL de fosfato tampón salino (PBS) de una botella de 500 mL de PBS estéril en un tubo de 50 mL. Agregar 0,5 mL de EDTA de 0,5 M y 0,9 g de NaCl en el tubo de 50 mL. Mezcla suavemente para disolver.
- Filtro esterilizar la solución con un filtro de 0,22 μm y la transferencia a la botella 500 mL de PBS estéril. Almacenar a temperatura ambiente (RT).
-
Preparar hPSC placas hPSC cultura
Nota: Use las placas para pasos 2.1-2.6.- Hacer 50 x solución de trabajo de capa adherente adecuado hPSC (PAAC): descongelar un vial de PAAC o/n a 4 ° C. Diluir el PAAC en proporción 1:1 con DMEM/F12 y transferencia como 400 alícuotas μL en tubos de 1,5 mL. Solución de trabajo 50 x PAAC a-80 ° C.
Nota: PAAC (importante) se solidifica rápidamente a temperatura ambiente, por lo tanto es necesario que todos los componentes (DMEM, tubos, etc.) se mantienen en hielo (o a 4 ° C). - Descongelar el tubo de 50 x solución de trabajo de PAAC a 4 ° C, luego diluir 50 x en frío DMEM/F12. Añadir 750 μL/pocillo de PAAC diluido a un 6WP. Incubar la placa durante al menos 1 h a 37 ° C.
Nota: Placas PAAC pueden almacenarse durante 1 semana a 4 ° C, envolviendo la placa después de período de incubación de 1 h. Placa caliente a 37 ° C antes de usar.
- Hacer 50 x solución de trabajo de capa adherente adecuado hPSC (PAAC): descongelar un vial de PAAC o/n a 4 ° C. Diluir el PAAC en proporción 1:1 con DMEM/F12 y transferencia como 400 alícuotas μL en tubos de 1,5 mL. Solución de trabajo 50 x PAAC a-80 ° C.
-
Preparar platos para antiadherente (AA) para la cultura de la diferenciación
Nota: Use las placas para pasos 3.1-4.1.- Hacer 5 mg/mL poli (metacrilato de 2-hidroxietilo) solución (poly-HEMA) en 95% etanol. Agite o/n a 37 ° C hasta obtener una solución clara. Tienda a TA.
Nota: Filtración con filtro 0,22 μm puede quitar sin disolver poly-HEMA. - Añadir poly-HEMA a la placa de cultivo para que cubra el fondo de cada pozo. Incubar la placa a 37 ° C durante 2 días e inspeccione la placa para asegurar la completa evaporación de la capa uniforme de líquido de los pozos. Placas de AA pueden ser envuelto y almacenadas a TA.
- Hacer 5 mg/mL poli (metacrilato de 2-hidroxietilo) solución (poly-HEMA) en 95% etanol. Agite o/n a 37 ° C hasta obtener una solución clara. Tienda a TA.
-
Preparar las placas de Poly-L-ornitina/laminina (PLO/LAM) cultura de diferenciación
Nota: Use las placas para pasos 4.2-4.4.- Capa de la superficie de los pozos, usando 20 PLO μg/mL disuelta en PBS estéril. Incubar la placa o/n a 37 ° C. PLO de aspirar y lavar 3 veces con PBS.
Nota: (Opcional) incubada la placa con PLO puede envolverse y almacenada a 4 ° C hasta que se necesite. - Recubrir las superficies de los pocillos recubiertos de PLO, utilizando 10 μg/mL LAM disuelta en PBS estéril. Incubar durante al menos 2 h a 37 ° C o o/n a 4 ° C. Eliminar fuga y lavado pozos 2-3 x con PBS, luego inmediatamente agregar medio apropiado o células.
Nota: Solución quitar LAM (opcional) puede ser almacenado a 4 ° C y reutilizado hasta 2 veces. (Importante) No permita que superficies recubiertas de LAM a secar; para evitar esto inmediatamente añadir PBS o medio apropiado.
- Capa de la superficie de los pozos, usando 20 PLO μg/mL disuelta en PBS estéril. Incubar la placa o/n a 37 ° C. PLO de aspirar y lavar 3 veces con PBS.
2. Protocolo n ° 1: Libre de alimentador hPSC cultura
Nota: hESCs se obtuvieron de una organización no comercial (línea H01, véase Tabla de materiales). Tres líneas de control hiPSC (hvs51, 60 y 88) fueron generadas por la reprogramación de fibroblastos de tres pacientes humanos sanos (fibroblastos fueron derivadas de donantes anónimos, no identificable y por lo tanto exentos de aprobación IRB)10, 17.
- Mantener hPSCs en la cultura libre de alimentador
- Después de descongelar y galjanoplastia hPSCs sobre placas PAAC en medio hPSC (vea el paso 2.2), mantener hPSCs a 37 ° C con 5% CO2. Actualizar hPSC medio diario (ver paso 2.3), excepto el día después de la descongelación o pases y examina las células bajo el microscopio (objetivos: 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1) para observar el crecimiento de la tarifa e identificar posibles áreas de diferenciación (figura 3 , parte superior del panel izquierdo muestra el ejemplo de la diferenciación).
- Paso hPSCs cada 3-4 días, o cuando la cultura llega a > 80% de confluencia. Si menos del 5% de las células exhiben diferenciación, uso normal hPSC pases método (véase paso 2.4), de lo contrario usar el método (ver paso 2.5). Cuando ya no sea necesario en la cultura, hPSCs puede ser congelado para su almacenamiento a largo plazo (ver paso 2.6).
- HPSCs de deshielo en medio hPSC
- Transferir el volumen requerido de hPSC medio a tubos estériles para el proceso de descongelación (9 tubos criogénicos o mL) y a la placa PAAC preparada para recibir las células descongeladas. Suplementar el medio de ambos tubos con 10 μm RI.
- Recuperar el criotubo de LN2 almacenamiento y lugar directamente en un baño de agua (37 ° C). Cuando solamente un pequeño cristal restos de hielo, retire del baño María y transferir el contenido del tubo criogénico al tubo para el proceso de descongelación (volumen total de 10 mL). Centrifugar el tubo a 290 x g durante 5 min a TA.
- Utilice una pipeta serológica para quitar el PAAC solución de los pozos de la placa del PAAC (ver paso 1.4) destinado a recibir las células y añadir medio hPSC con 10 μm RI.
Nota: (importante) no no aspirada solución PAAC con una aguja de succión, o se puede solidificar y obstruir las líneas de la bomba de vacío. - Quite el sobrenadante del tubo y resuspender las células en medio hPSC con 10 μm RI. Distribuyen las células a la placa de destino en proporción de 1 tubo criogénico/pozo de 6WP. Incubar a 37 ° C con 5% CO2y no actualizar medio para 1 día.
Nota: A partir de células en el 5% O2 puede aumentar la supervivencia de la célula.
- Actualizar hPSC medio
- Caliente el volumen necesario de hPSC medio en un tubo estéril a temperatura ambiente o en baño de agua; se sugiere 2 mL/pozo de 6WP.
Nota: (Opcional): mediante la adición de una cantidad extra de medio hPSC, hPSCs puede permanecer un día más sin actualizar; sin embargo, no permita que esto más de una vez por semana. - Aspire el medio de los pozos que contienen hPSCs y añadir medio fresco hPSC.
- La cultura hPSCs en una incubadora a 37 ° C y 5% CO2.
- Caliente el volumen necesario de hPSC medio en un tubo estéril a temperatura ambiente o en baño de agua; se sugiere 2 mL/pozo de 6WP.
- HPSCs de paso en medio de hPSC
- Transferir el volumen requerido de hPSC medio a tubos estériles, para proceso de pases y para la preparación de la placa PAAC recibir las células de los. Suplementar el medio de la placa de destino con 10 μm RI. Caliente los tubos medio a temperatura ambiente o en baño de agua.
Nota: La preparación y manejo serán diferente si utilizando un material de revestimiento alternativo que aparece en la Tabla de materiales. - Utilice una pipeta serológica para quitar el PAAC solución de los pozos de la placa del PAAC (ver paso 1.4) destinado a recibir las células y añadir medio hPSC con 10 μm RI.
Nota: (importante) no no Aspire la solución PAAC con una aspiración de la aguja, ya que puede solidificar y obstruir las líneas de la bomba de vacío. - Aspirar el medio de los pozos con hPSCs para ser pasados, lavan las células dos veces con 0,5 mM de EDTA, añada 0,5 mM EDTA e incubar durante 2-5 min a 37 ° C.
Nota: 1 mL/pozo de 6WP es un volumen de EDTA suficiente para lavado e incubación. - Compruebe los pozos bajo el microscopio (objetivos: 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1). Si las células están empezando a separar, aspirar solución de EDTA y libre por medio de hPSC ras células.
Nota: (importante) tenga cuidado de no quitar todo hPSC colonias cuando aspirando EDTA (no espere a colonias enteras se separación). No lave las células de más de 5 veces ya que pueden dañar hPSCs y afectan la pluripotencia. También, no dejar que las células soporte en medio hPSC con RI antes de enjuagar las células de los pozos, como pueden volver a adherirse a la placa. - Basado en la determinación empírica (generalmente se relaciona con la confluencia, tamaño de las colonias y la tasa de crecimiento), transferencia de hPSCs a los pocillos de la placa de destino mediante una relación de división de 1:4-1:16(es decir, 1 pozo de la placa original a 4 pocillos del destino placa). Incubar a 37 ° C con 5% CO2y no actualizar medio para 1 día.
Nota: Partir de proporciones tan altas como sea posible evitar el hacinamiento y mejorar el aspecto de las colonias 1:16-1:20.
- Transferir el volumen requerido de hPSC medio a tubos estériles, para proceso de pases y para la preparación de la placa PAAC recibir las células de los. Suplementar el medio de la placa de destino con 10 μm RI. Caliente los tubos medio a temperatura ambiente o en baño de agua.
- Paso hPSCs con el suave-método (G)
- Transferir el volumen requerido de hPSC medio a tubos estériles, para el proceso de passaging y para la preparación de la placa de autoridad pública recibir las células de los. Suplementar el medio de placa de destino con 10 μm RI. Tubos de medios de calientes a temperatura ambiente o en baño María a 37 ° C.
- Pipeta serológica de uso para quitar PAAC solución de pozos de la placa PAAC (ver paso 1.4) destinado a recibir las células y añadir medio hPSC con 10 μm RI.
Nota: (importante) no no PAAC de aspirado con una aguja de succión, o se puede solidificar y obstruir las líneas de la bomba de vacío. - Aspirar el medio de los pozos con hPSCs a ser pasados y lavan las células dos veces con 0,5 mM de EDTA. En el segundo lavado, esperar 30 s antes de aspiración EDTA, luego añada 1 mL de PBS e incubar 4-9 min a 37 ° C. Mientras espera, prepare un tubo con 4 mL de PBS estéril.
Nota: 1 mL/pozo de 6WP es suficiente volumen de EDTA para el lavado. - Compruebe los pozos bajo el microscopio (objetivos: 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1). Si las células están empezando a soltar, golpee cuidadosamente los lados de la placa para ayudar a las colonias libres. Cuando > 50% de las colonias están libre flotantes, utilizar una pipeta serológica de 5 mL para transferir las colonias en 1 mL de PBS para el tubo que contiene 4 mL de PBS (no triturate).
Nota: (importante) ya que el propósito es limpiar la cultura hPSC, verifique para determinar si se distinguen las células siguen siendo conectada a la placa. Además, no es necesario todas las colonias en un bien mediante este proceso, para que colonias que permanecen adjuntas pueden quedar atrás del paso. - Esperar 5-10 min a temperatura ambiente para que las células se instalan en el tubo (no centrifugar). Aspire el PBS del tubo, teniendo cuidado de no para quitar hPSCs colocado. Con cuidado resuspender las células en medio hPSC (triturate no) y las células se transfieren a la placa de destino mediante una relación de división de 1:4-1:16. Incubar a 37 ° C con 5% CO2y no actualizar medio para 1 día.
- Congelación de hPSCs
- Dependiendo de la confluencia, utilice 1 bien de hPSCs, en 2-3 días (máximos) en la cultura, para llenar frascos de criopreservación de 1-2 para el almacenamiento de información en LN2.
- Al final de pases (paso de 2.5), utilizar 500 μl o 1 mL de medio de hPSC para transferir células de 1 bien de 6WP a 1 o 2 frascos de criopreservación, respectivamente (500 μl/vial). A cada tubo, agregar 500 μl de 2 x de congelación medio que contiene medio de hPSC, 20 μm RI y 20% DMSO.
Nota: (Opcional) las células pueden ser transferidas directamente en 1 x medio de a 1 mL/vial de congelación. - Colocar los tubos criogénicos en un contenedor criogénico (contiene isopropanol) previamente enfriados a 4 ° C y almacenar inmediatamente a-80 ° C.
- Al día siguiente, transferir los tubos criogénicos a un tanque de2 LN para almacenamiento a largo plazo.
3. Protocolo 2: 3D "organoide" diferenciación
-
Configuración de la diferenciación con G-método modificado de pases de hPSCs
- Transferir el volumen requerido de NMM para el número de pozos de destino a un tubo estéril. Suplemento con 4 ng/mL FGF2 y 10 μm RI. Caliente el tubo de medio a temperatura ambiente o en baño María a 37 ° C.
Nota: Dependiendo de la confluencia de los pozos de origen hPSCs se concentran durante la distribución a la placa de destino en un 2:1 o 3:1 ratio (es decir, 2 pozos de la placa original a la 1 de la placa de destino), con un volumen final de 2,5 mL/pocillo de 6WP. - Aspire el medio de los pozos que contienen hPSCs ser distinguido y lavan las células dos veces con 0,5 mM de EDTA. En el segundo lavado, esperar 30 s antes de aspiración EDTA, luego añada 1 mL de PBS e incubar 4-9 min a 37 ° C. Mientras espera, prepare un tubo con 4 mL de PBS estéril.
Nota: 1 mL/pozo de 6WP es suficiente volumen de EDTA para el lavado. (Importante) Es preferible usar hPSCs que eran no más de 3 días en cultivo tras el último paso y al menos 1-2 pasajes después de descongelar. - Compruebe los pozos bajo el microscopio (objetivos: 2.5x/0.06, 5 x / 0.12 Ph0, 10 x / 0.25 Ph1). Si las células están empezando a separar las células golpeando suavemente los lados de la placa. Transferir las células a un tubo que contiene 4 mL de PBS con una pipeta de 5 mL.
Nota: (importante) luz lavado y trituración permite romper las colonias y recoger células sueltas, pero ignorar las células que permanecen adheridas a la placa. - Deje el tubo por 10 min a temperatura ambiente, para la separación por gravedad. Opcionalmente, centrifugar las células ligeramente (no más de 200 g x a TA, 5 minutos) si es necesario.
- Aspirado el PBS del tubo, teniendo cuidado de no para quitar hPSCs colocado, resuspender las células en NMM con 4 ng/mL FGF2 y 10 μm RI y luego distribuir a la placa revestida de AA en una proporción 2:1 o 3:1. Incubar a 37 ° C con 5% CO2y no actualiza el medio durante 3 días, a menos que requerido (ver paso 3.2.1).
Nota: (Opcional) para la conveniencia de la transferencia de las células, agregar una porción de medio de cultivo a los pocillos de la placa de destino antes de su distribución y resuspender las células en un volumen más pequeño. Además, hPSCs puede manchado y contado definir la densidad exacta de la célula partida. Sin embargo, el volumen final en la placa de destino debe ser 2,5 mL/pozo de 6WP.
- Transferir el volumen requerido de NMM para el número de pozos de destino a un tubo estéril. Suplemento con 4 ng/mL FGF2 y 10 μm RI. Caliente el tubo de medio a temperatura ambiente o en baño María a 37 ° C.
-
Mantener la cultura de diferenciación flotante a 37 ° C (5% CO2)
- Verifique las placas cada día cambios en el color medio, acumulación de células muertas, aglutinación y adherencia a fondos.
- Opcional: Sin importar el medio cambio horario, actualizar (incluyendo los 3 primeros días no refrescante) el medio si ha vuelto amarillo y siga las instrucciones para el cambio/actualización de media (paso 3.3). Si la mayoría de las células aparece muerta, siga las instrucciones para el cambio/refresco mediano con separación por gravedad (paso 3.4).
Nota: Se espera formación de EBs y crecimiento en agregados de células grandes, pero las células y agregados de células pueden agruparse en grandes masas no se debe a crecimiento individual/proliferación. Si esto se observa, trituración ligera para romper las masas está permitido. Si las células comienzan a adherirse a la superficie de la placa AA, el resto flotante contenido de pozos puede ser transferido directamente a pozos nuevos, o transferido durante el proceso de actualización de cambio de medio. No trate de transferir las células que se han adherido a la placa.
- Opcional: Sin importar el medio cambio horario, actualizar (incluyendo los 3 primeros días no refrescante) el medio si ha vuelto amarillo y siga las instrucciones para el cambio/actualización de media (paso 3.3). Si la mayoría de las células aparece muerta, siga las instrucciones para el cambio/refresco mediano con separación por gravedad (paso 3.4).
- El día 3, cambie el medio a NMM con 4 ng/mL FGF2. Actualizar el medio día.
- En el día 7, cambiar el medio a NMM con 1 μm ácido retinoico (RA), FGF8B de 100 ng/mL y 4 ng/mL FGF2. Actualizar el medio día.
Nota: RA (importante) es sensible a la luz. Proteger las muestras de cultura RA de la luz. - El día 14, cambiar el medio a NMM con FGF8B de 100 ng/mL y 100 ng/mL FGF4 FGF2 de 20 ng/mL. Actualizar el medio día.
- Día 17, cambia el medio al NMM con 100 ng/mL FGF8B. Actualizar el medio día.
- El día 21, cambiar el medio a NMM con cerebro deriva Neurotrophic Factor (BDNF) de 100 ng/mL y 10 ng/mL de Factor neurotrófico derivado Glial (GDNF). Actualizar el medio día.
- En el día 28, cambiar el medio a NMM con BDNF de 100 ng/mL y 10 ng/mL GDNF, SAG, 100 ng/mL Factor neurotrófico 3 (NT3) y 25 mM KCl. actualizar el medio día de 3 ng/mL.
- En el día 35, recoger las organitas 3D para el análisis.
- Verifique las placas cada día cambios en el color medio, acumulación de células muertas, aglutinación y adherencia a fondos.
-
Cambio/actualización medio de diferenciación de la cultura 3D
- Transferir el volumen necesario de NMM con los componentes adecuados (ver pasos 3.2.2-3.2.7 para programación de componente) a un tubo estéril. Caliente en un baño de agua a 37 ° C.
- Punta de la placa y agítelo suavemente hasta que las células se colocan a los bordes inferiores de los pozos. Con cuidado retirar 2 mL del medio viejo con una pipeta serológica, evitando la eliminación de las células, luego añada 2 mL de medio fresco. Incubar a 37 ° C con 5% CO2.
Nota: Volumen (importante) al final debe ser 2,5 mL/pozo de 6WP. Si se produce la evaporación, no retire 2 mL de medio como este además de secar a los cultivos celulares; en su lugar, añada medio extra.
-
Cambio/actualización medio de diferenciación con la separación de gravedad
- Transferir el volumen necesario de NMM con los componentes apropiados a un tubo estéril. Caliente en un baño de agua a 37 ° C.
- Transferir el contenido de los pocillos a un tubo estéril y permitir que el tubo por 10 min a temperatura ambiente, para la separación por gravedad.
- Utilice una pipeta para quitar viejos medio del tubo, teniendo cuidado de no quitar colocado células, resuspender en NMM con componentes adecuados y luego distribuir a los nuevos pozos recubiertos de AA. Incubar a 37 ° C con 5% CO2.
Nota: (Opcional) para mayor comodidad, una porción de medio de cultivo se puede Agregar al destino los pozos antes de la distribución, diferenciando las células suspendidas en un volumen menor. El volumen final debe ser 2,5 mL/pozo de 6WP.
4. Protocolo 3: Diferenciación 2D alternativa cultura
-
Iniciar y mantener la cultura siguiendo los pasos según la sección 3 para el protocolo 3D hasta el día 12
- Seguir pasos 3.1 3.2.3 y cambio/actualización medio según pasos 3.3 y 3.4.
-
Cambiar a y mantener la cultura monocapa 2D
- El día 13 de diferenciación, siga las instrucciones para el cambio/actualización medio con separación por gravedad (paso 3.4), sólo distribuimos los células/agregados a PLO/LAM cubrió la placa (ver paso 1.6) con un volumen final de 2,5 mL/pozo de 6WP.
Nota: El medio puede complementarse con 10 μm RI durante la chapa inicial para ayudar a la adherencia y la supervivencia de las células. (Importante) Es deseable difundir uniformemente las células en los pozos, para evitar la baja densidad o hacinamiento en las placas y paso (ver paso 4.4) según sea necesario. El tamaño preferido de placas PLO/LAM cubierto (es decir, 6WP, 12WP, etc.) debe ser empíricamente determinado, basada en la tasa de proliferación de la línea celular y el propósito para el producto. Instrucciones darán volúmenes de 6WP y pueden ser convertidas por reducir a la mitad por cada duplicación de la cantidad de bien (es decir, 2 mL/pozo de 6WP, 1 mL/pozo de 12WP, etc.) - El día 14, cambiar el medio a NMM con FGF8B de 100 ng/mL y 100 ng/mL FGF4 FGF2 de 20 ng/mL. Actualizar el medio día como se describe en el paso 4.3.
- Día 17, cambia el medio al NMM con 100 ng/mL FGF8B. Actualizar el medio día como se describe en el paso 4.3.
- El día 21, cambiar el medio a NMM con BDNF de 100 ng/mL y 10 ng/mL GDNF. Actualizar el medio día como se describe en el paso 4.3.
- En el día 28, cambiar el medio a NMM con 100 ng/mL BDNF y 10 ng/mL GDNF, hundimiento de 3 ng/mL, 100 ng/mL NT3 y 25 mM KCl. actualizar el medio día como se describe en el paso 4.3.
- En el día 35, recolectar células para el análisis, o mantener en el mismo medio como paso 4.2.5 extendida cultura (límite potencial no probado).
- El día 13 de diferenciación, siga las instrucciones para el cambio/actualización medio con separación por gravedad (paso 3.4), sólo distribuimos los células/agregados a PLO/LAM cubrió la placa (ver paso 1.6) con un volumen final de 2,5 mL/pozo de 6WP.
-
Cambio/actualización medio de diferenciación de la cultura 2D
- Transferir el volumen necesario de NMM con componentes adecuados (ver pasos 4.2.2-4.2.5 para programación de componente) a un tubo estéril. Caliente en un baño de agua a 37 ° C.
- Aspirar el medio de los pozos, luego añada 2 mL medio nuevo. Incubar a 37 ° C con 5% CO2.
Nota: Volumen (opcional) al final puede conservarse en 2,5 mL/pozo de 6WP, usando una pipeta para extraer 2 mL del medio viejo y añadir 2 mL de medio fresco. Reservar una porción de medio en los pozos y las células prevención de contacto con el aire, puede reducir golpes a las células durante los pasos de cambio.
-
Cultura de diferenciación 2D paso
- Transferir el volumen necesario de NMM con los componentes adecuados (ver pasos 4.2.2-4.2.5 para programación de componente) a tubos estériles, para el proceso de pases y, por separado preparar PLO/LAM cubierta placa para recibir los células. Suplementar el medio de la placa de destino con 10 μm RI. Caliente los tubos medio a temperatura ambiente o en baño de agua a 37 ° C. Para guardar en componentes, es posible utilizar NMM solamente para el lavado de las células durante el proceso de passaging.
Nota: (importante) si se usan productos en proyección de imagen de calcio experimentos, asegurar a las células de pasaje entre 2-6 días antes del final de la diferenciación. - Aspire el medio de los pozos a ser pasados. Añadir 300 μL/pocillo de agente de disociación base de tripsina (véase Tabla de materiales), placa de remolino para cubrir pozos, a continuación, retire inmediatamente el agente de disociación.
- Deje que la plancha por 2 minutos a temperatura ambiente, luego aflojar las células dando golpecitos en los lados de la placa. Añadir 600 μL/pocillo definido inhibidor (DTI) y transferencia de las células en DTI a un tubo estéril con 5 mL NMM.
- Centrifugar el tubo a 290 x g durante 15 min a TA. aspirado del medio y añadir otros 5 mL NMM al tubo.
- Centrifugar el tubo a 290 x g durante 15 min a TA. aspirado del medio y resuspender las células en el medio apropiado con RI.
- Distribuir las celdas de la placa de PLO/LAM con una relación de división de 1:1-1:12, dependiendo de la confluencia inicial, tasa de proliferación y tamaño diferencial en origen a la placa de destino y se mantiene a 37 ° C con 5% CO2.
- Transferir el volumen necesario de NMM con los componentes adecuados (ver pasos 4.2.2-4.2.5 para programación de componente) a tubos estériles, para el proceso de pases y, por separado preparar PLO/LAM cubierta placa para recibir los células. Suplementar el medio de la placa de destino con 10 μm RI. Caliente los tubos medio a temperatura ambiente o en baño de agua a 37 ° C. Para guardar en componentes, es posible utilizar NMM solamente para el lavado de las células durante el proceso de passaging.
Resultados
Visión general de reducción del crecimiento Factor protocolos de diferenciación cerebelosa 2D y 3D
La figura 1 muestra la cronología general de los protocolos de diferenciación cerebelosa 2D y 3D, identificación de factores extrínsecos y el tiempo de la galjanoplastia. Los avances típicos para hPSCs que experimentan diferenciación cerebelosa 3D se muestra en la figura 2: con la línea de hESCs H01 a partir como colonias de cultura libre de alimentador en el día 0 (parte superior izquierda de la figura); sometidos a formación de EB por día 2 (medio superior); crecimiento en agregados de células más grandes con lumen aparente después de la inducción neural con RA y FGF8 en el día 14 (superior derecha); formación de agregados de diferente tamaño y forma al día 28 (abajo a la izquierda); desarrollo en complejidad con diferentes estructuras de un solo agregado indicado al día 28 (media baja); y con cambios morfológicos en las mismas estructuras en el día 35 (abajo a la derecha). Los avances típicos para hPSCs que experimentan diferenciación cerebelosa 2D es representado en la figura 3: con células línea H01 como colonias de cultura libre de alimentador en el día 0 (arriba a la izquierda de la figura, con el círculo que indica zona de células diferenciadas entre las colonias de células) ; sometidos a formación de EB por día 2 (medio superior); crecimiento en agregados de células más grandes con lumen aparente después de la inducción neural con RA y FGF8 en el día 13 (superior derecha); proliferar como células adherentes después de galjanoplastia del día 14 (abajo a la izquierda); y luego como una monocapa de células con morfología más complejo/madurar en el día 35 bajo alta magnificación (abajo a la derecha) y baja (inferior centro).
Marcadores de exposición de productos en 3D y las estructuras de neuroepitelio temprano
Figura 2 (imagen inferior izquierda) y figura 4 muestran la heterogeneidad de la morfología total 3D visto a través de cultivo, debido a la variación de crecimiento o tasas de maduración, así como la fusión estocástico o romper aparte de los agregados. A pesar de la heterogeneidad, cada diferenciación produce agregados exhibiendo marcadores neuronales y nervios tempranos, como marcador de gránulo cerebeloso ZIC1, indicado por inmunocitoquímica (ICC) la coloración en la figura 5. Más importante aún, figura 5 y figura 6 sugieren que una simple cultura 3D, con factores de crecimiento reducidos, es capaz de generar agregados con estructuras complejas relacionadas con el desarrollo del cerebro como el neuroepitelio temprano y labio rómbico.
Productos 2D exhiben marcadores cerebelosos y actividad Neuronal funcional
Aunque las culturas 2D no pueden reproducir estructuras 3D complejas, son capaces de generar células exponiendo principios marcadores neuronales y neuronales, como marcador de la célula del gránulo cerebeloso ZIC1, indicó manchando de ICC en la figura 7. Análisis de expresión génica por RT-PCR, como se ve en la figura 8, es compatible con resultados de tinción de ICC, aunque la presencia de un marcador temprano de la célula del gránulo ATOH1 es variable entre líneas y experimentos. Proyección de imagen de calcio es más fácilmente manejado en la cultura 2D. Como se ve en la figura 9, suplementario Video 1y Video suplementario 2, células eléctricamente estimuladas muestran flujos de calcio que son típicas de los patrones de disparo neuronal, lo que sugiere la generación de neuronas funcionales.
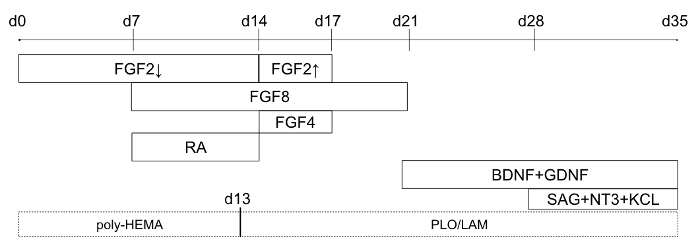
Figura 1: línea de tiempo del Protocolo de diferenciación (a partir de día 0 de la diferenciación). Cajas de línea, indicar factores específicos se agregan al medio de cultivo y cajas de la línea de puntos indican placa de recubrimiento para modificación 2D opcional. Para FGF2, la flecha hacia abajo se refiere a concentraciones más bajas (4 ng/mL), y la flecha hacia arriba se refiere a concentraciones más altas (20 ng/mL). Esta figura ha sido modificada de Holmes y Heine10. Haga clic aquí para ver una versión más grande de esta figura.
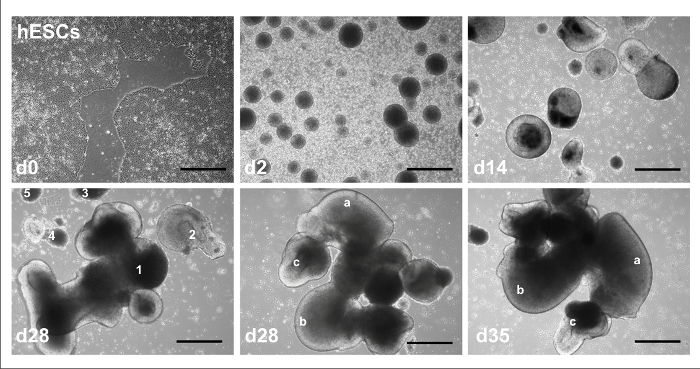
Figura 2: imágenes de brightfield representante de protocolo 3D. colonias de células en el d0, EBs en d2, agregados después de la inducción en d14, agregados de distinto tamaño y morfología (numerados 1-5) en el d28, un agregado único con características únicas, identificables (indicado por letras una–c) en el d28, y cambios visibles en las mismas características en d35. Barra de escala = 100 μm. Esta figura ha sido modificada de Holmes y Heine10. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: imágenes de brightfield representante de protocolo 2D. colonias de células un d0, EBs en d2, agregados después de la inducción en d13, después galjanoplastia agregados en d14, después de la maduración en d35 como se ve en 5 aumentos y 20 aumentos. El círculo blanco en el panel superior de la izquierda muestra el área de las células diferenciadas entre las colonias de células. Barra de escala = 50 μm. haga clic aquí para ver una versión más grande de esta figura.
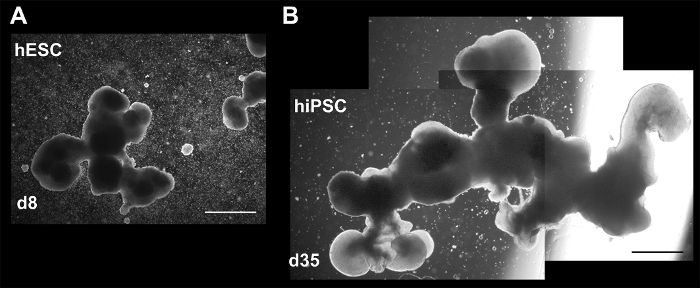
Figura 4: imágenes de Brightfield con diferente tamaño y complejidad en 3D cultura. Agregados en (A) día 8 (hESCs) y (B) d35 (hiPSCs). La imagen de este último se compone de tres imágenes separadas para mostrar todo el agregado. Ambos agregados puedan haber sido afectados por la fusión de pequeños agregados o pérdida (interrumpiendo) de estructuras. Barra de escala = 200 μm. Esta figura es republicada de Holmes y Heine10. Haga clic aquí para ver una versión más grande de esta figura.
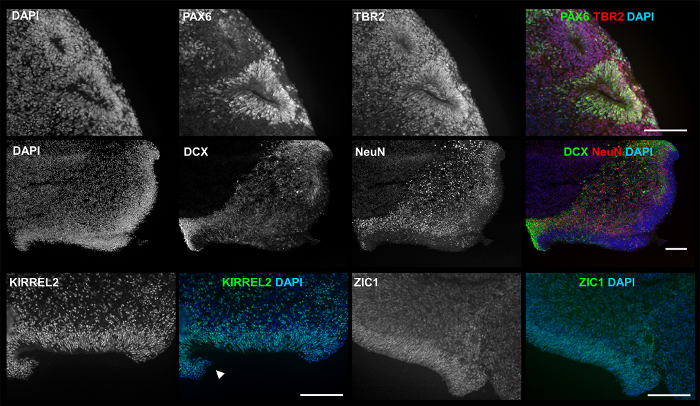
Figura 5: ICC imágenes de productos 3D muestran estructuras y marcadores relevantes. En cultura d35, exhiben productos 3D: PAX6 (verde) y TBR2 (rojo) por lumen de nervios formación de roseta (primera fila); DCX (verde) y NeuN (rojo) que se extiende desde la zona ventricular del borde exterior (VZ)-como la estructura (segunda fila); KIRREL2, un marcador asociado con neuroepitelio cerebelosa (tercera fila, izquierda); y ZIC1 un marcador asociado con el gránulo cerebeloso células (tercera fila, derecho). El experimento se realizó varias veces usando cuatro hPSC diferentes líneas: línea de hESCs H01 (n = 5) y hvs88 de líneas de iPSC (n = 4), hvs60 (n = 3) y hvs51 (n = 1). Las flechas apuntan hacia el labio rómbico (RL)-como la estructura. Barra de escala = 100 μm. Esta figura es republicada de Holmes y Heine10. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: gran escala ventricular zona-como la estructura en 3D producto. En cultura d35, un agregado de células derivadas es positivo para PAX6 (verde) y TBR2 (rojo) comúnmente asociados con el tempranas neuronas encontradas zonas ventriculares (VZs) y zonas subventricular (SVZs), en vivo. (Top) Un asterisco (*) marca el lado apical de una región de VZ-como correr por el borde del agregado, entre paréntesis indicando profundidad/División de VZ / SVZs. (medio) combinada señales muestran secciones dispersas de PAX6 + TBR2-células aumentando de tamaño hacia el extremo derecho superior de la VZ. (Parte inferior) Imagen de mayor ampliación de la sección indicada por un rectángulo en el panel central. Barra de escala = 100 μm. Esta figura ha sido modificada de Holmes y Heine10. Haga clic aquí para ver una versión más grande de esta figura.
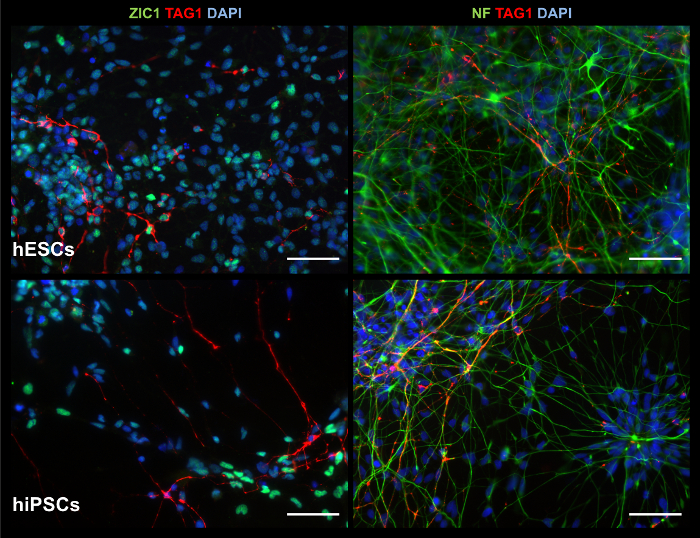
Figura 7: imágenes de ICC de productos 2D muestran marcadores relevantes. En cultura d35, exposición de productos 2D, de hESCs (fila superior) y hiPSCs (fila inferior), las células positivas para: gránulo de la célula marcador ZIC1 y migratorias neurona cerebelosa marcador TAG1 (columna de la izquierda); y del neurofilament marcador neuronal (NF) y TAG1 (columna derecha). Barra de escala = 50 μm. haga clic aquí para ver una versión más grande de esta figura.
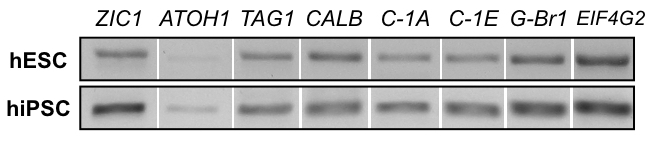
Figura 8: RT-PCR de productos 2D. Análisis de expresión de mRNA de hESCs línea H01 (fila superior) y hiPSC línea hvs60 (fila inferior) en el extremo del 2D protocolo muestra productos con electroforesis en gel de: marcador de células de gránulo ZIC1, gránulo de la célula marcador ATOH1, neurona cerebelosa migratorias marcador TAG1, Purkinje marcador de células Calbindin (CALB), canales de calcio dependientes de voltaje CACNA1A (C-1A), CACNA1E (C-1E), receptor del ácido gamma - aminobutírico (GABA) B 1 (G-Br1) y limpieza gene EIF4G2).
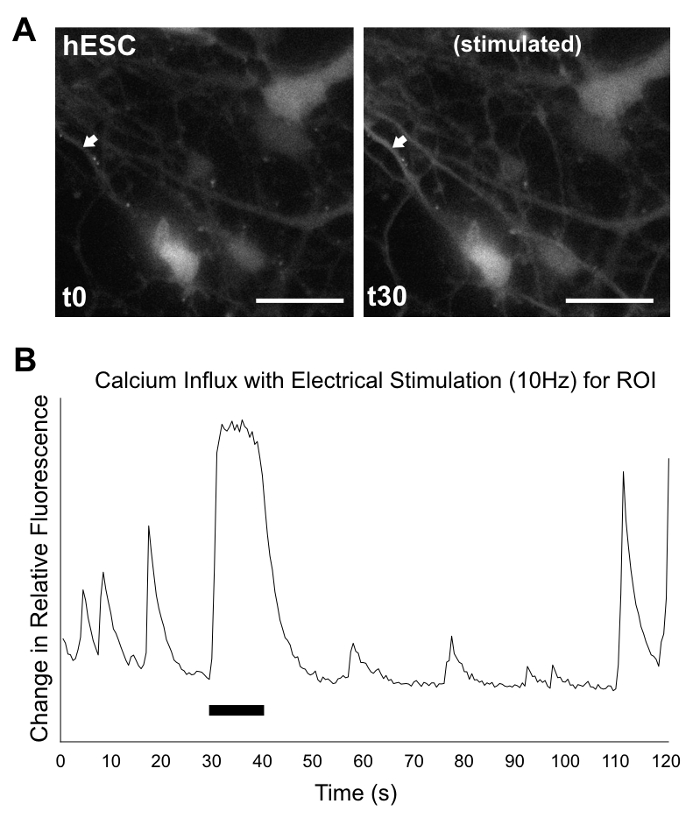
Figura 9: proyección de imagen de calcio/análisis de productos 2D. En cultura d35, productos de diferenciación hPSC registraron durante 2 min bajo el microscopio, después de la incubación con tinte fluor5. En 30 s, las células fueron estimuladas eléctricamente durante 10 s a 10 Hz. (A) imágenes fijas Mostrar hESCs en 0 s (izquierda) y después del comienzo de la estimulación eléctrica a 30 s (derecha). Las flechas indican la región de interés (ROI) para el análisis de la afluencia del calcio. Muestra de análisis gráfico (B) cambio en la fluorescencia relativa frente al tiempo de retorno de la inversión (cambio en fluorescencia = (F-F0) / f0, donde F0 = (∑F1-n) / n), con picos de neurona-como ocurriendo antes, durante y después de estimulación. Barra negra indica la longitud de la estimulación eléctrica. Escala (blanco) de la barra = 50 μm. completo grabación hESCs (como se ve aquí) y una línea de hiPSC (no mostrado) están disponibles en 1 Video complementario y suplementario Video 2, respectivamente. Las grabaciones son en formato AVI, a velocidad 4 x. Haga clic aquí para ver una versión más grande de esta figura.
1 Video suplementario: calcio imágenes vídeo de producto 2D de hESCs línea H01. En cultura d35, productos de la diferenciación de células línea H01 registraron durante 2 min bajo el microscopio, después de la incubación con fluor5 del tinte. En 30 s, las células fueron estimuladas eléctricamente durante 10 s a 10 Hz. grabaciones fueron hechas a 2 fotogramas/s y transformado en vídeo AVI a ~ 7 fotogramas/s, produciendo un video que dura 30 s, a ~ 4 x. Haga clic aquí para descargar este archivo.
2 Video suplementario: calcio imágenes vídeo de producto 2D hiPSC línea hvs51. En cultura d35, productos diferenciación de hiPSC línea hvs51 se registraron por 2 min bajo el microscopio, después de la incubación con tinte fluor5. En 30 s, las células fueron estimuladas eléctricamente durante 10 s a 10 Hz. grabaciones fueron hechas a 2 fotogramas/s y transformado en vídeo AVI a ~ 7 fotogramas/s, produciendo un video que dura 30 s, a ~ 4 x. Haga clic aquí para descargar este archivo.
Discusión
Complejidad y los costos son factores relevantes para los investigadores de células madre antes de seleccionar o desarrollar protocolos de diferenciación. Esto es especialmente cierto ya que es una cuestión abierta cuánto control externo es requerido para generar los tipos celulares deseados, o — para plantear diferentemente, como hPSCs competentes en la producción de su propio entorno de desarrollo, si se deja a sí mismos con suficiente nutrientes. Introducción de factores extrínsecos en vitro puede producir muy bien productos de célula deseada, pero también podrían interferir con las capacidades intrínsecas del desarrollo las células habrían expuesto en vivo. Estas consideraciones son importantes, particularmente si el objetivo es el uso de iPSCs derivados del paciente para el modelado de la enfermedad. Uso extenso de patrones o factores de crecimiento podría enmascarar los fenotipos de la enfermedad. Los protocolos detallados en este informe siguen la tendencia de los estudios anteriores para reducir la complejidad, costo y uso de patrones extrínsecos factores8,9.
Base en los resultados reportados por Muguruma et al. y nuestro reciente estudio, parece que es posible lograr diferenciación hacia destinos cerebelosas sin esfuerzos para reproducir en vivo las condiciones, como estudios anteriores han hecho 1 , 2 , 3 , 4 , 8 , 10. la parte intrigante es que los dos estudios utilizaron diferentes conjuntos de factores de crecimiento, sugiriendo que ni conjunto era necesaria, aunque ambos utilizan FGF2. Corrimos pruebas adicionales, donde FGFs fueron selectivamente excluidos del protocolo y mostró que las células eran capaces de generar los mismos productos sin extrínseca FGFs10. Diferencias entre nuestros estudios fueron calificadas por el hecho de que utiliza hPSC diferentes líneas y métodos de cultivo, indujo la diferenciación de los nervios con RA e incluye componentes para apoyar la supervivencia de la célula del gránulo y maduración (BDNF, GDNF, SAG y el KCL)11 –14. Además, se empleó un método menos complejo de inicio, comparado con Opie et al.. Su protocolo comenzó generando uniforme EBs en 96WPs, que aislado físicamente y químicamente entre sí. El protocolo aquí tenía todos los PSCs relativamente lleno juntos en 6WPs durante la formación del EB, que les permitieron interactuar libremente. Cómo esto puede haber diferencialmente afectado el ambiente físico y químico de EBs y organitas posterior (incluida la producción intrínseca de los compuestos de señalización) es desconocido y podría ser explorado. Además, mientras nos muestra la expresión de genes asociados con — y tan sugestivos de — origen cerebeloso, situado dentro de estructuras morfológicamente similares a los reportados por Muguruma et al., no podemos excluir generación neuronal-como las estructuras que son de identidad no cerebeloso. Los estudios futuros, utilizando un amplio panel de anticuerpos como los reportados por Muguruma et al. (es decir, ATOH1, CALB, etc.) haría que tales asignaciones y comparación entre los productos de ambos protocolos, más contundentes.
En el protocolo 3D, es importante para empezar y mantener un número suficiente de células en cultivo para asegurar un número suficiente de los productos finales para el análisis. Teniendo en cuenta decrecimiento significativo en el protocolo, le recomendamos comenzar con más de 500 EBs/bien durante los primeros 3 días en cultivo (figura 1). Esto no debe ser difícil de lograr dado Colonia tamaños para hPSCs en la cultura libre de alimentación, pero podría no ser tan fácil para aquellos que todavía utilizan métodos dependientes de alimentador. Dado el gran número de células, es importante ver el cambio de color en medio (lo que indica cambios en el pH) y acumulación de células muertas. Ambos deben corregirse para evitar el colapso de la cultura. También puede haber aglutinación de las células y agregados en estructuras masivas. Aunque todavía puede resultar en agregados que pueden analizarse, cantidad de producto será grandemente reducido, para romper en pequeños agregados de trituración suave puede ser útil. Sin embargo, evitar agregados normales inquietantes, que se pueden alcanzar grandes tamaños (figura 4). Si queda demasiado escasos agregados, se recomienda combinar pozos para que los agregados no están completamente aislados. Variabilidad del producto (en número, tamaño y morfología) es un problema bien conocido en la cultura celular 3D, incluso para los protocolos a partir de aislados, uniforme EB formación pasos, lo que sugiere que un procedimiento menos complejo de inicio (por ejemplo, el protocolo descrito aquí ) puede ser más práctico8,15. Mientras que esta heterogeneidad es algo que los investigadores deben tener en cuenta, particularmente durante el análisis, el protocolo divulgado genera productos consistentes con los encontrados en otros protocolos 3D8,9,15. Basado en tamaño y morfología, caen dentro de la gama de roseta neural al organoide cerebral, como se describe en un informe reciente por Kelava y Lancaster15, con el máximo ajuste a la clasificación de esferoide. Particularmente notables, son sugestivos de neuronales rosetas con lumen, (sub) zonas ventriculares y rombal labio como características la presencia de estructuras 3D (figura 5 y figura 6) identificado por otros grupos8 , 15 , 16 , 17. puesto que todos los experimentos produjeron por lo menos uno agregado con supuesta VZ/SVZs y cerebelosa asociada a marcadores (ZIC1, KIRREL2), son criterios útiles para determinar el éxito de una diferenciación 3D usando nuestro protocolo con RL-como características de apoyo adicional. Extender la longitud de la cultura pasados 35 días no fue probado, pero podría aplicarse para determinar los límites del crecimiento, complejidad y madurez permitido por esta técnica.
El protocolo 2D utiliza la misma formación de EB no adherente y proceso de inducción neural como el protocolo 3D y así que los comentarios anteriores también se aplican. Una vez plateado, se debe considerar un conjunto de consideraciones diferentes. El EBs debe adherirse rápidamente para que las células proliferan hacia fuera en el plato. Si hay problemas con la adherencia, además de RI (si no utilizado), reducción del volumen del medio, o pueden aplicarse cambios experimentales en la concentración de PLO/LAM. Es importante que las células crezcan demasiado denso o RALO (preferentemente cultivados entre confluencia 20-80%) en los pozos; seguimiento diario y pases oportuna es importante, para evitar que las células sobre-confluencia o flotantes. A diferencia del protocolo 3D, no debe haber decrecimiento significativo en cultura, aunque puede haber áreas de crecimiento pobre, o una disminución de las tasas de proliferación. Pases afectan el estado de maduración de las células (por ejemplo, eliminación de procesos celulares y redes desarrolladas entre las células) y deben tenerse en cuenta al acercarse a los puntos donde las células se recogen o analizadas de alguna manera. Por ejemplo, calcio lo proyección de imagen es muy importante a las células de pasaje entre 2-6 días antes del análisis. Pases demasiado análisis pueden significar las células no han tenido tiempo para conectar o maduro y demasiado puede causar células hacinamiento, dificulta la proyección de imagen. Aunque puede existir variabilidad entre experimentos, los resultados son constantes con ésos divulgados en protocolos cerebelosa 2D inicial1,2. ICC la coloración y gene expresión análisis corroboran la presencia de células positivas para el marcador de la célula del gránulo ZIC1, mientras también identificación de marcadores asociados con otras identidades neurales y cerebelosas (figura 7 y figura 8). Proyección de imagen de calcio, que consiste en la estimulación eléctrica de las células se incubaron con el tinte de fluor5, indica actividad neuronal funcional (figura 9, suplementario Figura 1y figura 2 complementaria), aunque no está confirmado si estos eran células del gránulo. Es discutible que dando células más tiempo para madurar ampliando la longitud de la cultura últimos 35 días, debe aumentar la cantidad de actividad neuronal funcional. Este potencial podría ser explorado en el futuro.
Además de las líneas de investigación sugeridas anteriormente, sería de interés para determinar diferencias en la identidad del producto (cantidad y calidad) entre los protocolos de 2D y 3D. La importancia de la FGFs extrínsecas no fue probada en el protocolo de 2D, y sería útil saber si carecen de estructura 3D después de placas y así las vías de señalización asociadas, que culturas 2D más o menos dependiente de los patrones temprano compuestos. Más despojados protocolos (por ejemplo, no RA, BDNF, SAG) son igualmente plausibles líneas para análisis adicionales. Por último, los estudios futuros podrían beneficiarse de nuevas herramientas de investigación para mejor caracterizar (y evaluar la eficacia de la generación de) cerebelosos subtipos neuronales humanos específicos.
Con el dados advertencias en mente, ambos registrados deben utilizarse protocolos para diferenciaciones cerebelosas, con productos adecuados a diferentes propósitos. Pueden servir como puntos de partida prácticos para los investigadores realizar estudios piloto, pruebas de viabilidad de líneas celulares para tales diferenciaciones, o como un modelo básico para otros tipos de diferenciación específica de los nervios.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Gerbren Jacobs y Jurjen Broeke su asistencia técnica experta, a Prisca Leferink para contribuir a la generación y caracterización de dos líneas de iPSC de control y a Lisa Gasparotto para demostrar nuestros procedimientos.
Materiales
| Name | Company | Catalog Number | Comments |
| DMEM/F12+Glutamax | Gibco | 31331-028 | glutamine fortified DMEM/F12 |
| Neurobasal medium | Gibco | 21103-049 | neural basic medium |
| N2 supplement | Gibco | 17502-048 | |
| B27 supplement | Gibco | 17504001 | |
| Insulin | Imgen | PT468-B | |
| L-glutamine | Gibco | 25030-024 | |
| Non-essential Amino Acids (NEAA) | Gibco | 11140-035 | |
| beta-mercaptoethanol | Gibco | 21985-023 | |
| Poly-L-Ornithine | Sigma | P3655 | |
| Poly (2-hydroxyethyl methacrylate) | Sigma | P3932 | aka Poly-Hema |
| Laminin | Sigma | L2020 | |
| E8 medium and supplement | Gibco | A1517001 | hPSC medium and supplement |
| Penicillin/Streptomycin | Gibco | 15140-122 | |
| Sodium Chloride | Sigma | S-5886 | |
| y-27632 (ROCK inhibitor) | SelleckChem | S1049-10mg | |
| DMSO | Sigma | D-2650 | |
| Geltrex | Gibco | A1413302 | hPSC-appropriate adherent coating (PAAC) |
| 0,5M EDTA | Gibco | 15575-020 | |
| 0.2 um filter | VWR | 28145-77 | |
| 1.5 mL Eppendorf tube | VWR | 525-0130 | |
| DMEM/F12 | Gibco | 21331-020 | |
| Ethanol | VWR | 83804360 | |
| Parafilm | Sigma | PM996 | wrap for culture plates |
| cryotubes | ThermoFisher | 368632 | |
| TrypLE | Gibco | 12563-029 | trypsin-based dissociation agent |
| Defined Trypsin Inhibitor (DTI) | Gibco | R-007-100 | |
| FGF-2 | Peprotech | 100-18B | |
| FGF-4 | R&D Systems | 100-31 | |
| FGF-8B | Peprotech | 100-25 | |
| Retinoic Acid | Sigma | R2625 | |
| Brain Derived Neurotrophic Factor | Peprotech | 450-02 | |
| Glial Derived Neurotrophic Factor | Peprotech | 450-10 | |
| Potassium Chloride | Sigma | P5405 | |
| Neurotrophic Factor 3 | Peprotech | 450-03 | |
| Smoothened Agonist (SAG) | Cayman | 11914 | CAS 912545-86-9 |
| Axiovert 40C microscope | Zeiss | Brightfield imaging microscope | |
| Axiocam | Zeiss | Brightfield imaging - image aquisition | |
| Eppendorf Centrifuge 5810 | Eppendorf | 521-0996 | centrifuge for cell culture |
| PBS (gebufferde natrium oplossing) | Braun Medical | 3623140 | |
| 5 ml Serological pipets | VWR | 612-4950 | |
| 10 ml Serological pipets | VWR | 612-4951 | |
| 6-wells culture plates | VWR | 734-2323 | |
| 12-wells culture plates | VWR | 734-2324 | |
| hESCs | WiCELL | line H01 |
Referencias
- Erceg, S., et al. Efficient differentiation of human embryonic stem cells into functional cerebellar-like cells. Stem Cells Dev. 19, 1745-1756 (2010).
- Erceg, S., Lukovic, D., Moreno-Manzano, V., Stojkovic, M., Bhattacharya, S. S. Derivation of cerebellar neurons from human pluripotent stem cells. Curr Protoc Stem Cell Biol. , (2012).
- Su, H. L., Muguruma, K., Matsuo-Takasaki, M., Kengaku, M., Watanabe, K., Sasai, Y. Generation of cerebellar neuron precursors from embryonic stem cells. Dev Biol. 290, 287-296 (2006).
- Salero, E., Hatten, M. E. Differentiation of ES cells into cerebellar neurons. Proc Natl Acad Sci U S A. 104, 2997-3002 (2007).
- Joyner, A. L. Engrailed, Wnt and Pax genes regulate midbrain--hindbrain development. Trends Genet. 12, 15-20 (1996).
- Joyner, A. L., Liu, A., Millet, S. Otx2, Gbx2 and Fgf8 interact to position and maintain a mid-hindbrain organizer. Curr Opin Cell Biol. 12, 736-741 (2000).
- Tam, E. W. Y., Benders, M. J. N. L., Heine, V. M. Cerebellar Development-The Impact of Preterm Birth and Comorbidities. Fetal and Neonatal Physiology. , (2017).
- Muguruma, K., Nishiyama, A., Kawakami, H., Hashimoto, K., Sasai, Y. Self-organization of polarized cerebellar tissue in 3D culture of human pluripotent stem cells. Cell Rep. 10, 537-550 (2015).
- Pasca, A. M., et al. Functional cortical neurons and astrocytes from human pluripotent stem cells in 3D culture. Nat Methods. 12, 671-678 (2015).
- Holmes, D. B., Heine, V. M. Simplified 3D protocol capable of generating early cortical neuroepithelium. Biology Open. 6, 402-406 (2017).
- Chen, J. K., Taipale, J., Cooper, M. K., Beachy, P. A. Inhibition of Hedgehog signaling by direct binding of cyclopamine to Smoothened. Genes Dev. 16, 2743-2748 (2002).
- Chen, J. K., Taipale, J., Young, K. E., Maiti, T., Beachy, P. A. Small molecule modulation of Smoothened activity. Proc Natl Acad Sci U S A. 99, 14071-14076 (2002).
- Dahmane, N., Ruiz-i-Altaba, A. Sonic hedgehog regulates the growth and patterning of the cerebellum. Development. 126, 3089-3100 (1999).
- Borghesani, P. R., et al. BDNF stimulates migration of cerebellar granule cells. Development. 129, 1435-1442 (2002).
- Kelava, I., Lancaster, M. A. Stem Cell Models of Human Brain Development. Cell Stem Cell. 18, 736-748 (2016).
- Kadoshima, T., et al. Self-organization of axial polarity, inside-out layer pattern, and species-specific progenitor dynamics in human ES cell-derived neocortex. Proc Natl Acad Sci U S A. 110, 20284-20289 (2013).
- Englund, C., et al. Pax6, Tbr2, and Tbr1 are expressed sequentially by radial glia, intermediate progenitor cells, and postmitotic neurons in developing neocortex. J Neurosci. 25, 247-251 (2005).
- Warlich, E., et al. Lentiviral vector design and imaging approaches to visualize the early stages of cellular reprogramming. Mol Ther. 19, 782-789 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados