Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Microsondeo electrónico capilar electroforesis de espectrometría de masas para unicelular metabolómica en embriones de Rana viva (Xenopus laevis)
En este artículo
Resumen
Describimos los pasos que permiten muestreo rápido in situ de una pequeña porción de una célula individual de alta precisión y mínima invasión, mediante micro-muestras basadas en el tubo capilar, para facilitar la caracterización química de una instantánea de la actividad metabólica en embriones vivos utilizando una plataforma de espectrometría de masas y electroforesis capilar célula hecha a la medida.
Resumen
La cuantificación de moléculas pequeñas en las células provoca nuevos potenciales para la mejor comprensión de los procesos básicos que subyacen el desarrollo embrionario. A activar sola célula investigaciones directamente en embriones vivos, nuevos enfoques analíticos son necesarios, especialmente aquellos que son sensibles, selectivos, cuantitativa, robusto y escalable para tamaños diferentes de la célula. Aquí, presentamos un protocolo que permite el análisis en situ del metabolismo en las células en el libre desarrollo de embriones de la rana con garras sudafricana (Xenopus laevis), un modelo potente en biología celular y del desarrollo. Este enfoque utiliza una microsonda capilar para aspirar una porción definida de células identificadas en el embrión, dejando las células vecinas intactas para posterior análisis. El contenido celular recogido es analizado por una interfaz de microescala electroforesis capilar electrospray ionización (CE-ESI) acoplada a un espectrómetro de masas tándem de alta resolución. Este método es escalable a diferentes tamaños de célula y compatible con la compleja estructura tridimensional del embrión en desarrollo. Por ejemplo, demostramos eso microprobe que unicelular CE-ESI-MS permite la aclaración de la heterogeneidad metabólica de la célula que se desarrolla como una célula progenitora da origen a descendientes durante el desarrollo del embrión. Además de la célula y biología de desarrollo, los protocolos de análisis unicelular aquí descritos son susceptibles a otros tamaños de célula, tipos celulares o modelos animales.
Introducción
Una comprensión integral del desarrollo embrionario requiere caracterización de todos los cambios moleculares que se despliegan en cada célula del organismo en desarrollo. Secuenciación de próxima generación con amplificación molecular permite medición profunda de transcriptomas unicelular1 en desarrollo sistemas2,3, mucho menos se sabe sobre el conjunto de moléculas más pequeñas producido en células embrionarias, incluyendo las proteínas y, especialmente, metabolitos (masa molecular < Da ~ 1.500). Con una respuesta rápida y dinámica a los eventos intrínsecos y extrínsecos, el metaboloma sirve como un poderoso descriptor del estado molecular de la célula. El metaboloma unicelular, por lo tanto, aumenta el potencial para seguir el desarrollo espacial y temporal de la heterogeneidad celular en el embrión y para identificar nuevas moléculas para estudios funcionales. Sin embargo, sin amplificación molecular disponible para estas moléculas, detección del metaboloma exige una sensibilidad excepcional utilizando espectrometría de masas (MS), que es la tecnología de elección para el análisis de metabolitos.
Sola célula MS es una colección de tecnologías con suficiente sensibilidad para medir metabolitos en las células (ver comentarios 4,5,6,7,8,9 ,10,11,12,13,14,15). Muestreo reproducible de las células y eficiente extracción de los metabolitos son esenciales para la acertada detección de metabolitos en las células. Disección de célula entera de identificadas células de embriones de Xenopus ha permitido la caracterización de pequeñas moléculas y péptidos16. Otros enfoques utilizan Micropipetas para células vivas individuales seguidas de detección mediante la ionización por electrospray (ESI) MS de la muestra. Por ejemplo, los metabilitos fueron medidos en planta o en células de mamífero por unicelular video MS17, presión sonda18, sonda solo19y fuerza fluídica microscopia20, entre otras técnicas21, 22,23,24. Además, incorporación de separación química antes de la ionización en el flujo de trabajo de MS sola célula eficiente simplifica el metaboloma, aliviando así posibles interferencias durante la generación de iones antes de la detección. Lo importante, separación también proporciona información específica del compuesto para ayudar en la identificación molecular. Electroforesis capilar (CE) se ha utilizado para detectar metabolitos en disecado solo25,26 o microsampled27las neuronas, captando moléculas pequeñas diferencias entre fenotipos de neurona. Recientemente adaptamos CE al tándem ESI MS para permitir la detección de nivel de seguimiento de cientos de metabolitos en las células individuales que fueron disectadas desde tempranos embriones de Xenopus laevis16,28. Estos estudios revelaron sorprendentes diferencias metabólicas entre las células embrionarias en una fase temprana de desarrollo y condujeron al descubrimiento de metabolitos con el previamente desconocido impactos del desarrollo16.
Aquí proporcionamos un protocolo que permitió la detección de metabolitos en las células en un embrión vertebrado vivo utilizando microsonda unicelular CE-ESI-MS29,30. El organismo modelo elegido es la 8-a-32-célula X. laevis embrión, aunque el enfoque es también aplicable a etapas posteriores de desarrollo y otros tipos de organismos modelo. Este protocolo utiliza capilares afilados con varios eje control traduccional bajo dirección por un sistema de proyección de imagen de alta resolución para aspirar una porción de nL ~ 10 de células identificados en situ en el embrión en desarrollo morfológicamente complejo. Esta microsonda es escalable a células más pequeñas y opera dentro de los segundos, que es lo suficientemente rápido para seguir linajes celulares en el embrión. Después de extracción polar o apolar de moléculas pequeñas, como metabolitos y péptidos, de la muestra recogida en ~ 4-5 solución de extracción μl, un ~ 10 nL del extracto resultante se analiza en una plataforma a la medida de CE separe a un espectrómetro de masas ESI. Construcción y operación de la plataforma de CE-ESI-MS se basa en protocolos descritos en otras partes. 31 , 32 la interfaz coaxial de CE-ESI es construida como se describe en otra parte. 31 esta plataforma se mantiene en el régimen cono-chorro rociado lograr sensibilidad de seguimiento-nivel capacidad de cuantificación sobre un rango dinámico del orden de registro de 4-5 (relativa28,29,30 o absoluta16). La plataforma de CE-ESI-MS ofrece un 60-amol límite inferior de detección con 8% de desviación estándar relativa (RSD) en la cuantificación en un rango probado de 10 nM y 1 μm para moléculas pequeñas16, que son suficientes para caracterizar metabolitos endógenos en X. Laevis de las células. Microprobed células continúan dividir a medida que avanza el embrión a través de desarrollo30, lo que permite análisis temporal y espacialmente resueltos de metabolismo celular. De hecho, unicelular CE-ESI-MS puede usarse para encontrar las diferencias metabólicas entre las células que ocupan el dorsal ventral16,29, animal-vegetal16y ejes del desarrollo de izquierda a derecha28 así como células forman el linaje predestinado dorsal de tejido neural de una célula progenitora común en X. laevis30. Además de consultar diferencias metabólicas entre las células embrionarias individuales en diferentes etapas de desarrollo del embrión X. laevis 30, Anticipamos que los protocolos descritos aquí son aplicables a una amplia gama de biomoléculas y microsampled de las células de diferentes etapas de desarrollo embrionario, así como otros tipos de células y organismos modelo. Además, la microsonda podría utilizarse para microsampling, mientras que otra plataforma compatible con minúsculas muestras podría ser utilizada para la separación y caracterización de biomoléculas.
Protocolo
Todos los protocolos relacionados con el mantenimiento y manejo de Xenopus laevis fueron aprobado por el institucional Animal cuidado y uso de la Universidad George Washington (IACUC no. A311).
1. preparación de muestras de instrumentos, medios, solventes y platos de muestreo
- Preparar solución de Steinberg (SS) 1 x mediante la disolución de las sales siguientes en agua ultrapura (~18.2 MΩ.cm a 25 ° C) en el siguiente orden y en las concentraciones indicadas siguiendo un estándar de protocolo33: cloruro de sodio (58,2 mM), cloruro de potasio ( 0,67 mM), nitrato de calcio (0,34 mM), sulfato de magnesio (0,83 mM), Tris-clorhidrato (4,19 mM) y Tris base (0,66 mM). Hacer 0,5 x SS por dos veces y 0.1 x SS por dilución diez veces, de la 1 x SS con agua ultrapura.
- Preparar platos de muestreo por primera agarosa 2% haciendo en 1 x SS. Autoclave a 120 ° C durante 20 minutos para disolver. Mientras que todavía líquido, cubrir el fondo de los platos de Petri de 60 mm con la solución. Una vez que el gel de agarosa ha enfriado y solidificado, llama al final de una pipeta Pasteur de 6 pulgadas hasta que se forme una bola y toque ligeramente el extremo calentado para imprimir pocillos de 5-10, ~ 1 mm de profundidad, en la agarosa.
Nota: Estos pozos se utilizan para inmovilizar los embriones durante el muestreo. - Preparar el disolvente de extracción de metabolitos. Adaptar las propiedades fisicoquímicas del solvente (por ejemplo, la polaridad y el pH) a las clases de moléculas que son de interés en el estudio.
Nota: por ejemplo, utilizamos 40% de metanol y 40% acetonitrilo en LC-MS-grade del agua como un enfoque de descubrimiento metabolitos principalmente polares y apolar algunos péptidos y metabolitos28. - Hacer lazos de pelo con cabello limpio y una pipeta de Pasteur como se describe en otra parte33 para mover suavemente los embriones en las cajas Petri con mínima perturbación.
- Fabrican Micropipetas de punta cónica como se muestra en la Figura 1a.
- En primer lugar, tire de capilares de borosilicato (1.000/500 μm diámetro de exterior/interior) en un extractor capilar tipo llamas-marrón con los siguientes ajustes: calor = 355; Tire = 65, velocidad = 80; tiempo = 150.
- Luego, rompa-apagado la punta de la micropipeta tirada con un par de pinzas afiladas finas para obtener una punta capilar de diámetro externo de 20 μm. realizar este paso bajo un estereomicroscopio para facilitar la precisión y reproducibilidad.
Nota: Tubos capilares con un pequeño Consejo son propensos a la obstrucción durante la aspiración del citoplasma viscoso. Mientras que tubos capilares con una punta más grande sin duda ayudan a evitar la obstrucción y aspirar a más del contenido celular, capilares de gran diámetro pueden plantean desafíos durante el muestreo de células más pequeñas y posiblemente dañar la celda de muestreo posterior. Ajuste cuidadoso de la presión y el tiempo de aspiración parcialmente pueden aliviar estos problemas. Nos encontramos con Micropipetas con ~ 20 μm diámetro exterior ideal para el trabajo presentado aquí.
2. Microsampling solo las células y la extracción de metabolitos
- Obtener embriones (óvulos fecundados) mediante monta natural inducida por la gonadotropina de adultos Xenopus laevis o mediante fertilización in vitro como se describe en otra parte protocolos33,34.
Nota: Monta Natural asegura que las etapas del desarrollo embrionarias se tambaleó mientras que los embriones obtenidos por fertilización in vitro son más confiables en el suministro. Sin embargo, la fertilización in vitro requiere sacrificar la rana macho adulto. - Recién 2% cisteína dejellying solución disolviendo 4 g de cisteína en 200 mL de agua ultrapura y ajustar gota a gota la solución a pH 8 con solución de hidróxido de sodio 10 N.
- Eliminar las capas de gelatina que rodea los embriones cuando empiezan a cleave en la etapa de células 2 como sigue: dejar embriones descansan en la solución de dejellying por 2 min, luego agitar suavemente por un minuto 2 adicional impedir que embriones adhiriéndose a la superficie de la colección plato.
- Con cuidado, vierta el contenido del plato en un vaso limpio y decantar rápidamente la solución dejellying de la taza. Inmediatamente cubrir los huevos con 0.1 x SS para enjuagar el resto dejellying solución, agitar suavemente y luego decantar la solución. Repita este paso cuatro veces para lavar completamente los embriones.
Nota: Limitar la exposición de los embriones a la solución dejellying a 4 minutos para asegurar la viabilidad. Protocolos integrales para eliminar las capas de gelatina están disponibles en otra parte33. - Transferencia de embriones dejellied en 1 x SS en una placa Petri. Para minimizar la aglomeración dentro de las placas, colocar ~ 100 embriones por 100 mm plato33.
Nota: Los platos que contienen embriones pueden almacenarse entre 14-18 ° C para frenar desarrollo y obtener embriones en etapas de desarrollo escalonadas de los mismos padres. Se publican nuevas directrices en dependencia de la temperatura de crecimiento y desarrollo en Xenbase y en otros lugares33,35,36,37. - Tipo hendiendo los embriones en la fase de 2 células en un plato separado en que la pigmentación estereotipada con confianza marca el eje de la dorsal-ventral, con referencia a células establecidas sino mapas de38,39,40.
- Identificar correctamente Hendedoras embriones garantizando que el primer surco de escote, que demarks el plano de corte sagital medio, divide el oscuro (ventral) y ligeramente pigmentado (dorsal) polo animal tal que las dos mitades son imágenes espejo41.
- Montar una micropipeta fabricada en un micromanipulador multi-eje (manual o accionado). Conecte la micropipeta a un microinyector.
- Utilizar una pipeta de plástico para aspirar ~ 5 de los embriones de 8 células y transferirlos en el recipiente de muestreo que contengan 0.5 x SS.
Nota: Identificar las células basadas en pigmentación y posición en el embrión, con referencia a la celda destino mapas38,39,40.
Nota: por ejemplo, rutinariamente aspirar ~ 10-15 volumen de nL de la célula mediante la aplicación de ~ 3 impulsos de psi-30 para el tubo capilar. Este paso toda dura ~ 5 s para la aspiración30.
- Cierre firmemente el frasco para evitar la evaporación y coloque el frasco en el cubo de hielo de 4 ° C hasta que el muestreo es completado. Deseche la micropipeta utilizada en un contenedor de objetos punzantes para evitar el riesgo de punción.
Nota: Para determinar el volumen del contenido celular aspirado, inyecte el aspirado en aceite mineral, donde obtiene una forma esférica. El diámetro de esta esfera puede medirse utilizando un microscopio. Calcular el volumen aspirado: V = 4/3 π r3, donde V es el volumen, y r es el radio de la esfera.
3. CE-ESI-MS medida
- Preparación de estándares y soluciones para CE-ESI-MS
- Preparar el electrolito de fondo (BGE) compuesto por 1% ácido fórmico en agua de calidad LC-MS.
- Preparar la solución de vaina que contiene 50% metanol en LC-MS grado agua y 0.1% de ácido fórmico.
- Preparar solución de acetilcolina de 50 nM en la solución de la vaina para la evaluación diaria del desempeño del sistema CE-ESI-MS.
- Preparar solución de cloruro de sodio de 150 mM como estándar de calibración de masa para la gama baja de m/z en el modo de ion positivo. Una precisión de masa (m/z) de < 10 ppm se recomienda. Siga las instrucciones del proveedor de espectrómetro de masas para llevar a cabo este paso.
Nota: Como alternativa, otros estándares con valores conocidos de m/z pueden utilizarse para masa-calibrar el espectrómetro de masas.
- Construcción de la plataforma de CE-ESI
- Construir una plataforma de inyección de CE capaz de rápida traducción vertical de una etapa con el frasco BGE y la muestra carga microvial. Para la construcción y operación de la plataforma, consulte los detalles en referencia31.
- Montar la interfaz CE-ESI (figura 1C) como sigue. Monte el emisor metal electrospray (130/260 μm interno/externo diámetro y ~ 35 mm de longitud) en un puerto de 3 T-Unión. Alimentación del tubo capilar de separación CE (40/105 μm interno/externo diámetro y ~ 100 cm de longitud) por el emisor de electrospray permitiendo que sobresalga ~ 40-100 μm más allá de la punta del emisor. Trabajar bajo un estereomicroscopio para facilitar la precisión.
- Conecte el tubo capilar la solución de la vaina (75/360 μm interno/externo diámetro y ~ 100 cm de longitud) al puerto restante para proveer la solución de electrospray. Utilizar fundas adecuadas y apriete las conexiones para operación libre de fugas de la interfaz de CE-ESI. Referirse a anteriores protocolos31,32 para obtener información sobre la Asamblea y resolución de problemas de esta interfaz.
Nota: Dimensiones del tubo capilar afectan la relación de señal a ruido (S/N) y la duración de la separación. Por ejemplo, tubos capilares de diámetro estrecho y corto facilitan separaciones rápidas con mayor separación voltajes42,43. Además, dependiendo de los tipos de moléculas que son de interés en un estudio, capilares de separación pueden ser cubiertos para minimizar o evitar los no deseados pared capilar molécula interacciones44.
- Conecte el tubo capilar la solución de la vaina (75/360 μm interno/externo diámetro y ~ 100 cm de longitud) al puerto restante para proveer la solución de electrospray. Utilizar fundas adecuadas y apriete las conexiones para operación libre de fugas de la interfaz de CE-ESI. Referirse a anteriores protocolos31,32 para obtener información sobre la Asamblea y resolución de problemas de esta interfaz.
- Usando un sostenedor de la placa, instale la interfaz CE-ESI en una etapa de la traducción de tres ejes y posicione la punta del emisor de electrospray ~ 2 mm desde el orificio del espectrómetro de masas (figura 1C).
- Para limpiarlos componentes de la interfaz, enjuague la solución de la vaina de electrospray mediante electrospray emisor de 1 μl/min y el BGE a través de la separación de CE capilar. Utilizar bombas de la jeringuilla para alimentar los solventes a un ritmo constante.
- Lave el capilar de separación CE antes de cada medición mediante la conexión de una jeringa en el extremo de entrada capilar. Usar jeringas suficientemente grandes para minimizar el relleno y cebado de líneas de alimentación de solvente.
Nota: Experimentos normalmente utilizan jeringas gas tight de 1 mL para el solvente de la vaina de electrospray y una jeringa de 500 μL para purgar el capilar de separación de la fuente.
- Validación de plataforma de CE-ESI-MS y la medición de metabolitos
Nota: El objetivo de este paso es confirmar la sensibilidad analítica del instrumento CE-ESI-MS día antes analizando extractos de unicelulares.
- Después de enjuagar la separación capilar por ~ 5 min, transferir su entrada en la solución BGE en un frasco de acero inoxidable.
- Colocar la punta del emisor de electrospray ~ 2 mm del orificio de espectrómetro de masas y ajuste fino de esta distancia usando una etapa de la traducción para generar electrospray en el régimen cono-chorro estable mientras el aerosol con un estereomicroscopio (ver referencias 31,45). Monitorear la estabilidad de la actual total del ion (TIC) ~ 30-45 min para asegurar una operación estable.
- Aplicar 20 kV al frasco de BGE por gradualmente incrementando el potencial de más de ~ 15 s, típicamente generando ~7.5 mA actual a través del tubo capilar de separación utilizando 1% de ácido fórmico como el BGE. Antes de cada medición, asegurar la estabilidad del sistema mediante el control del perfil TIC ~ 5-10 min y luego baje step-wise el potencial de separación (CE) a 0 V (tierra).
Nota: Para semi-automatizar este proceso, utilizamos un software de medida para controlar remotamente el CE alto voltaje potencia fuente31. Si la plataforma de CE-ESI-MS es inestable, cuidadosamente evaluar la fuente de inestabilidad probando la plataforma de CE-ESI-MS primero en modo de sólo ESI y luego en el modo de funcionamiento del CE-ESI como recomienda en otra parte31. Brevemente, para probar la plataforma en el modo sólo ESI, apague la alta tensión de la CE y monitorear las TIC ~ 30 min en el régimen de pulverización de cono-chorro. Si es necesario, proceder a errores de dirección: () Inspeccione las conexiones por fugas; (ii) limpiar el emisor electrospray con agua, isopropanol, agua y metanol; (iii) de gases solventes; (iv) descarga el emisor ~ 25 min antes de probar otra vez. Si la plataforma de CE-ESI se encuentra estable en modo de sólo ESI pero llega a ser inestable durante la separación de la CE, inspeccione el sistema para el calentamiento de Joule o electrólisis: () eliminar el capilar de separación con BGE ~ 25 min y repetir el experimento; (ii) utilizar potenciales de separación inferiores para mantener una respuesta resistiva lineal (es decir, lineal CE actual vs curva de tensión de separación); (iii) inspeccionar el capilar de CE por posibles daños, tales como grietas y vuelva a colocar el tubo capilar si es necesario. - Analizar ~ 6 nL de la muestra como sigue:
- Pipetear 1 μl de la solución estándar de acetilcolina en el frasco de la inyección.
- Transferir el capilar de separación del frasco de BGE en el frasco de la inyección.
- Elevación a la etapa de inyección del CE 15 cm en 1s.
- Mantenga la etapa elevada de 60 s para inyectar hidrodinámico ~ 6 nL de la muestra en el capilar de separación.
- Posteriormente, traducir la etapa a partir de niveles (en consonancia con la salida del capilar).
- Mueva suavemente el extremo de entrada capilar en el BGE.
- Inmediatamente después, la rampa a la tensión de la CE para iniciar la separación electroforética.
- Adquisición de datos de MS de inicio.
Nota: El rendimiento del sistema puede caracterizarse con cualquier estándar químico. El entrega de CE-ESI-MS sola célula inferior límites de detección de ~ 10 nM (~ 60 amol) para la acetilcolina, metionina e histidina16. El volumen inyectado (Vinj), en el tubo capilar en nL, depende de la diferencia de altura (H, cm) durante la inyección, densidad (ρ, g cm-3) y viscosidad (η, kg m-1s-1) de BGE, longitud (L, m) y radio (r, μm) de la CE capilar y la duración de inyección (tinj, s). Esta relación se expresa mediante la siguiente fórmula, donde g (m s-2) es la aceleración de la gravedad:
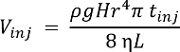
- Una vez que la norma ha sido detectada, dejar de adquisición de datos, bajar la tensión de separación gradual a 0 V (tierra) y luego recuperar el emisor a 2 cm del orificio. Ras la separación capilar durante 5 minutos antes de analizar la célula extrae.
- Medida 10 nL de la célula de solo extraer repitiendo los pasos 3.3.1-3.3.5 con 90 s hidrodinámico inyectar la muestra.
Nota: El objetivo del tratamiento es identificar y cuantificar compuestos entre las células. El protocolo de CE-ESI-MS unicelular genera picos de electropherographic estrechas con anchos de base típico de unos pocos segundos. Mediante la realización de análisis de datos semi-manualmente, es posible encontrar características moleculares (valores únicos m/z con tiempos de migración única) siguiendo estos pasos. Representante de separación se muestra para seleccionarlos metabolitos identificados en la Figura 2a.
- Masa-calibrar los archivos de datos raw post adquisición de datos.
Nota: Utilizamos señales de sodio formiato de los racimos que se genera durante la separación de iones de sodio abundante de la muestra, que forma nativa presentes en las células o extraídos de los medios de cultivo de embrión. El objetivo de este paso es mejorar la identificación de metabolitos en pasos posteriores garantizando una precisión alta masa (m/z), preferiblemente < 5 mDa, o < 10 ppm, entre m/z 50-1, 000. Aquí, post calibración de adquisición de datos permite rutinariamente obtener exactitudes masa < 1 mDa, o < 2 ppm de m/z 50-500. - Utilizando un script de procesamiento, búsqueda de características moleculares a través del rango total detectado. Promedio de espectros de masas a través de cada pico para determinar la masa exacta y anote sus correspondientes épocas de migración. Para la identificación de metabolitos de baja masa en el rango de m/z 50-500, utilice una ventana de paso de 500 mDa para supervisar características moleculares con S/N > 3.
- Integrar el pico zona-bajo-la-curva para cada característica molecular manualmente o automáticamente. Los valores de área obtenidos se utilizan como una medida de abundancia de metabolitos.
- Identificación de las características moleculares de interés con alta confianza como sigue (ver Fig.
- En primer lugar, compare la masa exacta de las características moleculares contra una base de datos de metabolitos (por ejemplo, Metlin46 y HMDB47) con una precisión de 10 ppm para obtener una lista de supuestos partidos de masa.
- A continuación, evaluar estos partidos de masas comparando su espectro de masas tándem de la célula extractos con datos disponibles en las bases de datos de metabolitos o el espectro de masas tándem miden para el correspondiente producto químico estándar.
- Por último, validar estas asignaciones comparando el tiempo de la migración molecular características registrados en los extractos de célula con estándares químicos relacionados por el mismo instrumento de CE-ESI-MS.
Nota: Para mejorar el rendimiento experimental, típicamente identificar características moleculares que son estadística y significativamente diferentes entre las condiciones experimentales o tipos de células. Identificaciones representativas se muestran en la figura 2. Para identificar metabolitos de alta precisión total, recomendamos externamente calibrar el espectrómetro de masas diariamente, realizar nueva calibración en tiempo real durante cada medición utilizando un patrón interno o externamente masa calibrando cada medición se recomienda el archivo posterior de adquisición de datos (por ejemplo, para clusters de formiato de sodio aquí).
Nota: En nuestros experimentos, software en línea plataformas47 fueron utilizados para llevar a cabo todos los pasos posteriores del análisis de datos, incluyendo los siguientes pasos: i) filtrado de características moleculares con ocurrencia en por lo menos el 50% de cada conjunto de muestra (por ejemplo, la célula tipo); II) normalización de los datos; III) estadística (por ejemplo, t-test) y análisis de datos multivariados como el análisis de componentes principales (PCA) y análisis de conglomerados jerárquico (HCA). Utilizamos p < 0.05 (de Student t-test) para marcar la significación estadística y cambio de doblez ≥ 1.5 tener en cuenta la significación biológica.
Resultados
Recientemente se empleó microprobe unicelular CE-ESI-MS para caracterizar metabolitos en las células individuales identificadas en el libre desarrollo de embriones de Xenopus laevis 29,30. La microsonda permite rápido (~ 5 seg/celular), en situ la aspiración de ~ 10 nL de una célula individual, aspiraciones múltiples de la misma célula o varias células diferentes dentro de las misma o posteriores etapas d...
Discusión
MicroProbe CE-ESI-MS permite la caracterización directa de los metabolitos en las células en vivo, libremente desarrollar embriones. En el corazón de la estrategia son dos subcomponentes técnicos, es decir en situ capilar microsampling y alta sensibilidad CE-ESI-MS. Comparado con celulares disección, microsampling capilar tiene la ventaja de un funcionamiento rápido (algunos segundos y 5 minutos / celular por disección), compatibilidad con la morfología tridimensional complejo de embriones y escalabilida...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por los institutos nacionales de salud subvenciones GM114854 (a p) y CA211635 (a p), Arnold y Mabel Beckman Fundación Beckman joven investigador conceden (a p), el premio DuPont joven profesor (a p), la sociedad americana para la masa Premio de investigación de espectrometría (a p) y becas de la Fundación del Club COSMOS (a R.M.O. y E.P.P.). Las opiniones y conclusiones expresadas en esta publicación son únicamente las de los autores y no representan necesariamente las opiniones oficiales de las fuentes de financiación.
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents for Embryo Culture Media | |||
| Potasium chloride | Fisher Scientific | BP 366-1 | |
| Magnesium sulfate | Fisher Scientific | M 65-3 | |
| Calcium nitrate | Sigma Aldrich | C1396 | |
| Cysteine | MP Biomedicals | 101444 | |
| Trizma hydrochloride | Sigma Aldrich | T3253 | |
| Trizma base | Sigma Aldrich | T1503 | |
| Sodium chloride | Fisher Scientific | 5641-212 | |
| Name | Company | Catalog Number | Comments |
| Metabolite Extraction Solvents | |||
| Acetonitrile (LC-MS-grade) | Fisher Scientific | A955 | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Name | Company | Catalog Number | Comments |
| Solvents and Standards for CE-ESI-MS | |||
| Formic acid (LC-MS-grade) | Fisher Scientific | A11710X1-AMP | |
| Methanol (LC-MS-grade) | Fisher Scientific | A456-4 | |
| Water (LC-MS-grade) | Fisher Scientific | W6 | |
| Sodium chloride | Fisher Scientific | 5641-212 | |
| Acetylcholine chloride | Acros Organics | 159170050 | |
| Name | Company | Catalog Number | Comments |
| Microprobe Fabrication Setup | |||
| Micropippette puller | Sutter Instrument Co. | P-1000 | |
| Borosilicate capillaries | Sutter Instrument Co. | B100-50-10 | |
| Fine sharp forceps: Dumont #5, Biologie/Dumoxel | Fine Science Tools (USA) Inc | 11252-30 | Corrosion resitant and autoclavable. |
| Name | Company | Catalog Number | Comments |
| Microprobe Sampling Setup | |||
| Micromanipulator | Eppendorf, Hauppauge, NY | TransferMan 4r | |
| Stereomicroscope | Nikon | SMZ18 | Should be vibrationally isolated. |
| Illuminator e.g. Goosenecks | Nikon | C-FLED2 | |
| Microinjector | Warner Instrument, Handem, CT | PLI-100A | |
| Transfer pipettes (Plastic, disposable) | Fisher Scientific | 13-711-7M | |
| Petri dish 60 mm and 80 mm | Fisher Scientific | S08184 | |
| Glass Pasteur Pipets ( Borosilicate, disposable) | Fisher Scientific | 13-678-20A | |
| Centrifuge | Thermo Scientific | Sorvall Legend X1R | |
| Name | Company | Catalog Number | Comments |
| CE-ESI-MS Setup | |||
| High voltage power supply | Spellman | CZE1000R | The HVPS may be controlled remotely using a low-voltage program generated by a personal computer. Caution: High voltage presents electrical shock hazard; all connective parts must be grounded or carefully shielded to prevent users from accidental exposure. |
| Syringe pumps (2) | Harvard Apparatus | 704506 | |
| Stereomicroscope | Amscope | SM-3BZZ | Stereomicroscope capable of 4.5× magnification, equipped with an illuminator to monitor the spraying mode of the CE-ESI interface. |
| XYZ translation stage | Thorlabs | PT3 | |
| XYZ translation stage | Custom-built | This platform is capable of loading nanoliter-amounts of sample into the separation capillary via hydrodynamic injection and supplying the BGE for CE. Both interfaces described in this work were able to inject 6–10 nL of sample within 1 min into a 1 m separation capillary | |
| Stainless steel sample vials | Custom-built | ||
| Stainless steel BGE vial | Custom-built | ||
| Fused silica capillary (40 µm/105 µm ID/OD; 100 cm) | Polymicro technologies | TSP040105 | |
| Fused silica capillary (75 µm/360 µm ID/OD; 100 cm) | Polymicro technologies | TSP075375 | |
| Stainless steel emitter with blunt tips (130/260 µm ID/OD) | Hamilton Co. | 21031A | For better performance, laser-cleave and fine-polish the emitter tip. |
| Syringes (gas-tight): 500 - 1000 µL | Hamilton Co. | 1750TTL | |
| Digital multimeter | Fluke | Fluke 117 | |
| High-resolution Mass Spectrometer | Bruker Daltonics | Maxis Impact HD | High-resolution tandem mass spectrometer equipped with an atmospheric-pressure interface configured for ESI |
| Tunning mixture for mass spectrometer calibration | Agilent technologies | ESI-L G1969-85000 | |
| Data Analysis ver. 4.3 software | Bruker Daltonics | ||
| Name | Company | Catalog Number | Comments |
| Ancillary Equipment | |||
| Vacuum concentrator capable of operation at 4–10°C | Labconco | 7310022 | |
| Analytical microbalance (XSE105DU) | Fisher Scientific | 01911005 | |
| Freezer (-20 °C) | Fisher Scientific | 97-926-1 | |
| Freezer (-80 °C) | Fisher Scientific | 88300ASP | |
| Refrigerated Incubator | Fisher Scientific | 11475126 | |
| Vortex-mixer | Benchmark | BS-VM-1000 |
Referencias
- Tang, F. C., Lao, K. Q., Surani, M. A. Development and applications of single-cell transcriptome analysis. Nat. Methods. 8 (4), S6-S11 (2011).
- Veselovska, L., et al. Deep sequencing and de novo assembly of the mouse oocyte transcriptome define the contribution of transcription to the DNA methylation landscape. Genome Biol. 16 (209), (2015).
- Tran, D. A., Bai, A. Y., Singh, P., Wu, X. W., Szabo, P. E. Characterization of the imprinting signature of mouse embryo fibroblasts by RNA deep sequencing. Nucleic Acids Res. 42 (3), 1772-1783 (2014).
- Wang, D. J., Bodovitz, S. Single cell analysis: the new frontier in 'omics'. Trends Biotechnol. 28 (6), 281-290 (2010).
- Svatos, A. Single-cell metabolomics comes of age: new developments in mass spectrometry profiling and imaging. Anal. Chem. 83 (13), 5037-5044 (2011).
- Rubakhin, S. S., Romanova, E. V., Nemes, P., Sweedler, J. V. Profiling metabolites and peptides in single cells. Nat. Methods. 8 (4), S20-S29 (2011).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat. Biotechnol. 30 (9), 858-889 (2012).
- Rubakhin, S. S., Lanni, E. J., Sweedler, J. V. Progress toward single cell metabolomics. Curr. Opin. Biotechnol. 24 (1), 95-104 (2013).
- Kleparnik, K., Foret, F. Recent advances in the development of single cell analysis: A review. Anal. Chim. Acta. 800, 12-21 (2013).
- Zenobi, R. Single-cell metabolomics: Analytical and biological perspectives. Science. 342 (6163), 1243259 (2013).
- Gholipour, Y., Erra-Balsells, R., Nonami, H. In situ pressure probe sampling and UV-MALDI MS for profiling metabolites in living single cells. Mass Spectrom (Tokyo). 1 (1), A0003 (2012).
- Comi, T. J., Do, T. D., Rubakhin, S. S., Sweedler, J. V. Categorizing cells on the basis of their chemical profiles: progress in single-cell mass spectrometry. J. Am. Chem. Soc. 139 (11), 3920-3929 (2017).
- Lombard-Banek, C., Portero, E. P., Onjiko, R. M., Nemes, P. New-generation mass spectrometry expands the toolbox of cell and developmental biology. Genesis. 55, e23012 (2017).
- Yang, Y. Y., et al. Single-cell analysis by ambient mass spectrometry. Trac-Trends Anal. Chem. 90, 14-26 (2017).
- Lanni, E. J., Rubakhin, S. S., Sweedler, J. V. Mass spectrometry imaging and profiling of single cells. J. Proteomics. 75 (16), 5036-5051 (2012).
- Onjiko, R. M., Moody, S. A., Nemes, P. Single-cell mass spectrometry reveals small molecules that affect cell fates in the 16-cell embryo. Proc. Natl. Acad. Sci. U. S. A. 112 (21), 6545-6550 (2015).
- Mizuno, H., Tsuyama, N., Harada, T., Masujima, T. Live single-cell video-mass spectrometry for cellular and subcellular molecular detection and cell classification. J. Mass Spectrom. 43 (12), 1692-1700 (2008).
- Nakashima, T., et al. Single-cell metabolite profiling of stalk and glandular cells of intact trichomes with internal electrode capillary pressure probe electrospray ionization mass spectrometry. Anal. Chem. 88 (6), 3049-3057 (2016).
- Pan, N., et al. The single-probe: A miniaturized multifunctional device for single cell mass spectrometry analysis. Anal. Chem. 86 (19), 9376-9380 (2014).
- Guillaume-Gentil, O., et al. Single-cell mass spectrometry of metabolites extracted from live cells by fluidic force microscopy. Anal. Chem. 89 (9), 5017-5023 (2017).
- Saha-Shah, A., Green, C. M., Abraham, D. H., Baker, L. A. Segmented flow sampling with push-pull theta pipettes. Analyst. 141 (6), 1958-1965 (2016).
- Hu, J., et al. Synchronized polarization induced electrospray: Comprehensively profiling biomolecules in single cells by combining both positive-ion and negative-ion mass spectra. Anal. Chem. 88 (14), 7245-7251 (2016).
- Zhang, L. W., Vertes, A. Energy charge, redox state, and metabolite turnover in single human hepatocytes revealed by capillary microsampling mass spectrometry. Anal. Chem. 87 (20), 10397-10405 (2015).
- Zhang, L. W., et al. In Situ metabolic analysis of single plant cells by capillary microsampling and electrospray ionization mass spectrometry with ion mobility separation. Analyst. 139 (20), 5079-5085 (2014).
- Lapainis, T., Rubakhin, S. S., Sweedler, J. V. Capillary electrophoresis with electrospray ionization mass spectrometric detection for single-cell metabolomics. Anal. Chem. 81 (14), 5858-5864 (2009).
- Nemes, P., Knolhoff, A. M., Rubakhin, S. S., Sweedler, J. V. Metabolic differentiation of neuronal phenotypes by single-cell capillary electrophoresis electrospray ionization mass spectrometry. Anal. Chem. 83 (17), 6810-6817 (2011).
- Aerts, J. T., et al. Patch clamp electrophysiology and capillary electrophoresis mass spectrometry metabolomics for single cell characterization. Anal. Chem. 86 (6), 3203-3208 (2014).
- Onjiko, R. M., Morris, S. E., Moody, S. A., Nemes, P. Single-cell mass spectrometry with multi-solvent extraction identifies metabolic differences between left and right blastomeres in the 8-cell frog (Xenopus) embryo. Analyst. 141 (12), 3648-3656 (2016).
- Onjiko, R. M., Plotnick, D. O., Moody, S. A., Nemes, P. Metabolic comparison of dorsal versus ventral cells directly in the live 8-cell frog embryo by microprobe single-cell CE-ESI-MS. Anal. Methods. , (2017).
- Onjiko, R. M., Portero, E. P., Moody, S. A., Nemes, P. In situ microprobe single-cell capillary electrophoresis mass spectrometry: Metabolic reorganization in single differentiating cells in the live vertebrate (Xenopus laevis) embryo. Anal. Chem. 89, 7069-7076 (2017).
- Nemes, P., Rubakhin, S. S., Aerts, J. T., Sweedler, J. V. Qualitative and quantitative metabolomic investigation of single neurons by capillary electrophoresis electrospray ionization mass spectrometry. Nat. Protoc. 8 (4), 783-799 (2013).
- Knolhoff, A. M., Nemes, P., Rubakhin, S. S., Sweedler, J. V., Wevers, R., Lutz, N., Sweedler, J. V. . Methodologies for Metabolomics. , 119-139 (2013).
- Sive, H. L., Grainger, R. M., Harland, R. M. . Early development of Xenopus laevis: a laboratory manual. , (2000).
- Moody, S. A. Cell lineage analysis in Xenopus embryos. Methods Mol Biol. 135, 331-347 (2000).
- Bowes, J. B., et al. Xenbase: a Xenopus biology and genomics resource. Nucleic Acids Res. 36, D761-D767 (2008).
- Karpinka, J. B., et al. Xenbase, the Xenopus model organism database; new virtualized system, data types and genomes. Nucleic Acids Res. 43 (D1), D756-D763 (2015).
- James-Zorn, C., et al. Xenbase: expansion and updates of the Xenopus model organism database. Nucleic Acids Res. 41 (D1), D865-D870 (2013).
- Moody, S. A. Fates of the blastomeres of the 16-cell stage Xenopus embryo. Dev. Biol. 119 (2), 560-578 (1987).
- Moody, S. A. Fates of the blastomeres of the 32-cell-stage Xenopus embryo. Dev. Biol. 122 (2), 300-319 (1987).
- Dale, L., Slack, J. M. W. Fate map for the 32-cell stage of Xenopus laevis. Development. 99 (4), 527-551 (1987).
- Klein, S. L. The first cleavage furrow demarcates the dorsal-ventral axis in Xenopus embryos. Dev. Biol. 120 (1), 299-304 (1987).
- Rollman, C. M., Moini, M. Ultrafast capillary electrophoresis/mass spectrometry of controlled substances with optical isomer separation in about a minute. Rapid Commun. Mass Spectrom. 30 (18), 2070-2076 (2016).
- Moini, M., Martinez, B. Ultrafast capillary electrophoresis/mass spectrometry with adjustable porous tip for a rapid analysis of protein digest in about a minute. Rapid Commun. Mass Spectrom. 28 (3), 305-310 (2014).
- Huhn, C., Ramautar, R., Wuhrer, M., Somsen, G. W. Relevance and use of capillary coatings in capillary electrophoresis-mass spectrometry. Anal. Bioanal. Chem. 396 (1), 297-314 (2010).
- Nemes, P., Marginean, I., Vertes, A. Spraying mode effect on droplet formation and ion chemistry in electrosprays. Anal. Chem. 79 (8), 3105-3116 (2007).
- Zhu, Z. J., et al. Liquid chromatography quadrupole time-of-flight mass spectrometry characterization of metabolites guided by the METLIN database. Nat. Protoc. 8 (3), 451-460 (2013).
- Wishart, D. S., et al. HMDB 3.0 The Human Metabolome Database in 2013. Nucleic Acids Res. 41 (D1), D801-D807 (2013).
- Liu, J. X., Aerts, J. T., Rubakhin, S. S., Zhang, X. X., Sweedler, J. V. Analysis of endogenous nucleotides by single cell capillary electrophoresis-mass spectrometry. Analyst. 139 (22), 5835-5842 (2014).
- Hubrecht, L., Nieuwkoop, P. D., Faber, J. . Normal table of Xenopus laevis (Daudin). A systematical and chronological survey of the development from the fertilized egg till the end of metamorphosis. , (1967).
- Grant, P. A., Herold, M. B., Moody, S. A. Blastomere explants to test for cell fate commitment during embryonic development. J. Vis. Exp. (71), (2013).
- Sellick, C. A., Hansen, R., Stephens, G. M., Goodacre, R., Dickson, A. J. Metabolite extraction from suspension-cultured mammalian cells for global metabolite profiling. Nat. Protoc. 6 (8), 1241-1249 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados