Method Article
Una plataforma de ultra rendimiento microfluidos para la secuenciación del genoma de unicelular
En este artículo
Resumen
Sola célula secuencia revela heterogeneidad genotípica en los sistemas biológicos, pero las tecnologías actuales carecen de la capacidad necesaria para el perfilado profundo de la función y composición de la comunidad. Aquí, describimos un flujo de trabajo de microfluidos para la secuencia > 50.000 genomas unicelulares de diversas poblaciones de la célula.
Resumen
Tecnologías de secuenciación han experimentado un cambio de paradigma de bulto a la resolución sola célula en respuesta a una comprensión de la evolución del papel de la heterogeneidad celular en los sistemas biológicos. Sin embargo, unicelular secuenciación de grandes poblaciones se ha visto obstaculizado por limitaciones en el procesamiento de genomas para la secuencia. En este papel, describimos un método de secuenciación del genoma de unicelular (SiC-seq) que utiliza gotas de microfluídica para aislar, amplificar y código de barras los genomas de las células. Encapsulación de la célula en microgels permite la purificación compartimentado y tagmentation de la DNA, mientras que una fusión de microfluidos pares eficientemente cada genoma con un código de barras único oligonucleótido unicelular que permite > 50.000 células individuales a ser ordenados por ejecutar. Los datos de secuenciación se demultiplexan por código de barras, generación de grupos de Lee origina de las células. Como método de alto rendimiento y bajo sesgo de secuencia unicelular, SiC-seq permitirá una gama más amplia de estudios genómicos dirigidos a poblaciones de células diferentes.
Introducción
El genoma sirve como un modelo de identidad celular y función, que contiene la totalidad de un organismo del potencial de codificación. Una comprensión de la biología celular a nivel de genoma puede explicar la diversidad fenotípica observada en poblaciones celulares heterogéneas. Esta heterogeneidad es evidente en los sistemas biológicos y tiene amplias implicaciones para la salud y la enfermedad. Por ejemplo, las variaciones número de la copia del gene entre las células del tumor están ligadas a la evolución y propagación de cáncer1,2. Infecciones bacterianas, Islas de patogenicidad presentes en una pequeña fracción de los genomas pueden ser transferidas horizontalmente y conducen a la proliferación de bacterias antibiotic-resistant3,4. Un desafío principal en el estudio de genomas a nivel unicelular es la baja cantidad de ADN disponible, así como la necesidad de analizar miles de células a la muestra la completa diversidad de genotipos. Por estas razones, limitaciones en el rendimiento experimental han obstaculizado la efectividad de los estudios de unicelulares, sesgar los resultados hacia las células más abundantes. Técnicas de aislamiento de células individuales como flujo clasificación5,6, pinza óptica7, empotramiento en a granel geles8y microfluídica9 son capaces de procesar cientos de células para secuenciación; sin embargo, esto representa sólo una pequeña fracción de la mayoría de las muestras. Un método de secuenciación del genoma de unicelular con rendimiento de procesamiento substancialmente más alta permitiría más profunda y más completa de perfiles de poblaciones celulares, así dilucidar el papel de la diversidad genotípica dentro de estas comunidades.
Microfluídica de gotas permite la manipulación de alto rendimiento de las células y reactivos biológicos dentro de millones de reactores picolitros-escala. Hasta la fecha, microdroplet tecnologías se han utilizado para estudiar patrones de expresión diferencial entre las células de los tejidos heterogéneos10,11,12, profundamente la secuencia de moléculas largas13,14 ,15y análisis de secuencia (ChiP-seq) de inmunoprecipitación conducta cromatina de las células16. De hecho, microgotas son capaces de operaciones de alto rendimiento, compartimentadas, haciéndolos susceptibles a aplicaciones en genómica unicelular. El desarrollo de esta tecnología presenta sus propios retos tecnológicos, sin embargo. Células deben lisis, purificadas y amplificadas con sesgo mínimo, uniformemente las poblaciones celulares de muestra. Además, a diferencia de cofia transcripciones del mRNA en células de mamíferos, no existe motivo molecular comparable en el genoma para facilitar la captura del ácido nucleico Diana. Por estas razones, secuenciación del genoma de la célula de solo ha sido difícil de implementar en plataformas microdroplet.
En este trabajo, proporcionamos un protocolo detallado de nuestro enfoque previamente divulgados microfluídicos unicelular capaz de secuenciar los genomas de decenas de miles de células en un solo experimento17. Con esta tecnología, llamada SiC-seq, las células bacterianas son encapsuladas en microescala hidrogeles individualmente lisis, tagmented y se fusionó con un microdroplet que contiene un código de barras único oligonucleótido, que se empalma en el ADN genómico de la célula a través de un superposición única extensión reacción en cadena de polimerasa (PCR). Los hidrogeles sirven como contenedores aislados en los que DNA genómico de alto peso molecular sterically se encajona, permitiendo que las moléculas más pequeñas tales como detergentes y enzimas líticas para acceder y purificar el ADN antes de código de barras18. Este protocolo de procesos > 50.000 células en cuestión de horas, lo que resulta en una biblioteca de código de barras para la secuencia. Tras la secuencia, las lecturas son demultiplexed según su secuencia de barras unicelular, resultando en un conjunto de datos conformada por millones de lecturas, cada una con un índice de celular.
Protocolo
1. fabricación de dispositivo de microfluidos
- Preparar la microfluídica diseños máscara utilizando el software de diseño asistido por ordenador (CAD) (siempre como. DWG; Ver Archivos complementarios). Con estos diseños de impresos por el proveedor con una resolución de 10 μm en una película de placa de circuito.
Nota: Para los dispositivos microfluídicos varias capas, las máscaras correspondientes contienen marcas de alineación. -
Para cada dispositivo, como sigue a fabricar el molde maestro SU-8 (figura 1A).
- Preparar una oblea de silicio de 3 in de diámetro por verter aproximadamente 1 mL de photoresist 3025 SU-8 en el centro de la oblea. Asegure la oblea en el recubridor spin chuck aplicando succión.
- Vea la tabla 1 para obtener un listado de los espesores de capa velocidades y exprimido para cada dispositivo. Para todos los dispositivos, iniciar el recubrimiento centrifugado con 30 s a 500 rpm, seguido de 30 s a la velocidad indicada.
- Sacar la oblea de silicio-8-SU del recubridor spin y suave cuece al horno sobre una placa para 135 ° C durante 30 minutos permite la oblea se enfríe a temperatura ambiente después de la cocción.
- Exponer la placa de SU-8-silicio con la máscara de microfluidos adecuada bajo un LED UV colimado 190 mW, 365 nm por 3 min.
- Después de la exposición, duro hornear la oblea en la placa caliente a 135 ° C durante 1 minuto. Después de este paso horno, permiten que la oblea se enfríe a temperatura ambiente.
- Para un dispositivo de microfluidos sola capa, vaya al paso 1.2.7. Para un dispositivo de microfluidos multicapa, repita los pasos 1.2.1 - 1.2.5 para la segunda capa de photoresist (figura 1B).
- Tras la primera duro cuece al horno para un dispositivo de una sola capa (o el segundo horneado duro para un dispositivo multicapa), desarrollar la oblea sumergiéndolo en un baño de acetato de Propilenglicol Monometil Éter (PGMEA) durante 30 minutos.
- Después del desarrollo de la oblea, utilice una botella de squirt que contiene PGMEA para enjuagar la oblea. Luego enjuague la oblea con una botella de squirt que contienen isopropanol antes de colocar sobre una placa de 135 ° C durante 1 minuto secar.
- Colocar la oblea (en adelante referida como el maestro) en una placa de Petri para el casting con polidimetilsiloxano (PDMS).
-
Con el maestro, preparado en el paso 1.2, proceder a realizar la fabricación del dispositivo con un casting de PDMS.
- Preparar el PDMS combinando una base de silicona con un agente endurecedor en una proporción 11:1 en masa. Mezclar a mano la base de silicona y agente de curado con una varilla de mezclar.
- Desgasificar el PDMS colocando en una cámara de desgasificación y aplicar vacío. Permitir el PDMS desgasificación hasta que las burbujas de aire ya no son visibles (típicamente 30 minutos).
- Vierta con cuidado el PDMS desgasificada sobre el amo, a un espesor final de PDMS-capa de aproximadamente 5 mm. Degas el PDMS para asegurar la eliminación de burbujas de aire.
- Después de la desgasificación, cuece al horno la PDMS y el amo a 80 ° C durante 80 minutos.
- Cuidadosamente suprimir la losa PDMS curada del maestro al horno con una hoja de afeitar. Asegurar que todos los cortes están encima de la oblea de silicio.
Nota: Cualquier corte hacen de la oblea de silicio puede resultar en un labio evitando una adhesión uniforme. - Perforar las entradas y salidas con un punzón de biopsia de 0,75 mm. Retirar polvo o PDMS callejeros usando una cinta adhesiva en el lado de la característica del dispositivo.
- Antes de tratar el dispositivo de plasma, limpiar un portaobjetos de vidrio de 50 mm x 75 mm por enjuague con isopropanol y secarla.
- Para el tratamiento de plasma, coloque el portaobjetos de losa y vidrio PDMS en la bonder de plasma con las características hacia arriba. Realizar el tratamiento de plasma utilizando 1 mbar O2 plasma para 1min Bond el dispositivo el vidrio diapositiva trayendo los expuestos o boca arriba, los lados juntos.
- Tras el tratamiento de plasma, hornee el dispositivo a 80 ° C durante 40 minutos.
- Finalmente, inyectar fluido de tratamiento superficial de vidrio en una de las entradas para representar los canales de microfluidos hidrofóbico. Asegúrese de que todos los canales son totalmente inundados con la solución y repetición la inyección de cada dropmaker. Hornear el dispositivo tratado a 80 ° C durante 10 minutos evaporar el disolvente sobrante.
2. encapsulación de células en agarosa Microgels
Nota: Vea la figura 2A.
- Preparar 1 mL de agarosa de temperatura de bajo punto de fusión 3% w/v de 1 x de buffer Tris-EDTA (TE). Mantener la solución de agarosa en un bloque de 90 ° C de calor hasta que inmediatamente antes de la jeringa de carga.
-
Preparar la suspensión de células.
Nota: Este protocolo y sus dispositivos microfluídicos asociados han sido validados para trabajar con las células bacterianas, de un stock congelado o fresco preparación. Las células mamíferas, dependiendo del tipo de célula, pueden requerir un ajuste de las dimensiones de canal de microfluidos para acomodar los tamaños de célula más grandes.- Resuspender las células en 1 mL de tampón fosfato salino (PBS).
- Contar las células en un hemocitómetro o por clasificación de flujo. Para microgels de 25 μm con una tasa de encapsulación celular objetivo de 1 en 10, prepare 1 mL de la suspensión celular a una concentración final de 2.4 x 107 células/mL.
- Desactivación de las células a 3.000 x g durante 3 min aspirar el sobrenadante y resuspender el precipitado de células en 1 mL de 17% v/v densidad gradiente medio (véase Tabla de materiales) en PBS. No se en hielo hasta la carga de la jeringa.
- Cargar una jeringa de 3 mL con aceite fluorado (HFE) que contiene un agente tensioactivo de 2% w/w de perfluoropoliéter polietileno glicol (PEG de PFPE), ajustar con una aguja de 27 G y lo coloca en una bomba de jeringa.
Nota: Ajuste los pasos todas las jeringas con aguja de 27 G de la microfluídica en este protocolo. Para reducir el riesgo de pinchazo accidental, mantenga tapas en todas las agujas hasta que comience la operación de la bomba. - Cargar la suspensión de la célula y la agarosa fundida en jeringas de 1 mL, coloque ambos con agujas de calibre 27 y coloque en bombas de la jeringuilla.
- Mantener la jeringa de la agarosa y la bomba caliente con un pequeño calefactor para evitar que la agarosa de gelificación en la jeringa y el tubo de entrada. Ajuste el espacio a alta y posición para que la superficie de calefacción es de unos 10 cm de la jeringa. Asegurar que la temperatura medida en la jeringa es aproximadamente de 80 ° C.
Nota: Se informa a los usuarios para mantener el calentador a la distancia recomendada desde el aparato de bombeo para reducir el riesgo de daño al equipo, incluyendo la fusión de la tubería. -
Generar 25 gotas de microgel μm utilizando el dispositivo de dropmaking de flujo de trabajo.
Nota: Ver Figura 3A para un dispositivo esquemático indicando la ubicación de la salida y entradas de reactivo.- Conectar las agujas de jeringa a las entradas de dispositivo de microfluidos con trozos de tubería de polietileno (PE). Antes de insertar los tubos en el dispositivo, cebar los bombeadores para quitar el aire de la línea.
- Conecte un trozo de tubería a la salida y coloque el extremo libre en un tubo de colección de 15 mL.
- Utilice las siguientes tasas de flujo (recomendado) para dropmaking: 800 μl/h para el HFE 2% w/w PFPE-PEG; 200 μL/h para la suspensión de células en PBS; y 200 μL/h para el 3% w/v agarosa.
- Después de la dropmaking, coloque el tubo de la colección a 4 ° C durante 30 minutos para la completa gelificación de la agarosa.
3. rompiendo y lavar el Microgels de la agarosa
- Quitar la capa más baja del aceite del tubo de colección utilizando una jeringa de 3 mL con una aguja de 20 G, teniendo cuidado de no para perturbar la capa superior de las gotas de agarosa.
-
Rotura de las emulsiones con perfluorooctanol (PFO).
- Añadir 1 mL de 10% v/v PFO en HFE a las gotas de agarosa. Pipeta de esta solución hacia arriba y abajo durante 1 minuto cubrir completamente las emulsiones.
Nota: Para el microgel lavado pasos, pipetear soluciones con una punta de 1.000 μl. La suspensión de microgel debe aparecer homogéneo y libre de grumos después de su uso lo. - Girar el tubo cónico a 2.000 x g durante 1 min a recoger la microgels de la agarosa. Retire el sobrenadante de la FOP/HFE por aspiración; las microgels libre de la capa de surfactante y aparece claras.
- Añadir 1 mL de 10% v/v PFO en HFE a las gotas de agarosa. Pipeta de esta solución hacia arriba y abajo durante 1 minuto cubrir completamente las emulsiones.
-
Lavar la microgels con un agente tensioactivo en hexano.
PRECAUCIÓN: El hexano es un solvente orgánico volátil, y el lavado en el paso 3.3 debe llevarse a cabo en una campana de humos.- Añadir 2 mL de 1% v/v del sorbitán monooleato agente tensioactivo no iónico (véase Tabla de materiales) en hexano a la microgels de la agarosa. Pipeta de arriba y abajo 10 veces para mezclar, asegurando la completa desintegración de la microgel de pellets.
- Girar el tubo a 1.000 x g durante 1 min a recoger la microgels. Aspirar el sobrenadante para quitar la solución de surfactante/hexano.
- Repita el lavado del surfactante/hexano.
-
Lavar la microgels en un tampón acuoso para eliminar cualquier solvente orgánico residual.
- Añadir 5 mL de tampón de TET [0.1% v/v Octilfenol etoxilado no iónicos detergente (véase Tabla de materiales) en TE 1 x] en el tubo cónico. Pipeta de arriba y abajo 10 veces para mezclar.
- Girar el tubo cónico a 2.000 x g durante 2 min a recoger la microgels. Aspirar el sobrenadante para quitar el tampón de TTE.
- Repita la TTE lavar x 2.
- Añadir 5 mL de buffer de TE de x 1 en el tubo cónico. Pipeta de arriba y abajo 10 veces para mezclar.
- Girar el tubo cónico a 2.000 x g durante 2 min a recoger la microgels. Aspirar el sobrenadante para quitar el tampón TE.
- Repita el lavado TE.
- Verificar la encapsulación de la célula en el microgels bajo el microscopio en un aumento de X 400 por una alícuota de 10 μl de geles con la coloración de la mancha 1 x de ácido nucleico (véase Tabla de materiales).
Nota: Microgels aparece clara en el canal transparente mientras celda de ADN que es fluorescente bajo el canal GFP (497/520 nm excitación/emisión de onda). Un recubrimiento de canales transparentes y fluorescentes mostrará las células en el microgels como se muestra en la Figura 4A.
4. lisis de las células en agarosa mediante enzimas líticas
- Preparar 1 mL de una enzima lítica cóctel con 800 μl de buffer TE (1 x), 2 μl de enzima lítica de levadura (5 U/μl de valores, 10 U/mL final), 30 μl de Ditiotreitol (stock de 1 M, final 30 mM), 60 mg de lisozima (polvo liofilizado), 15 μl de EDTA (0.5 M stock final 7,5 mM), 2 μl de mutanolysin (bolsa de 100 U/μl, 200 U/mL final), 2 μl de lysostaphin (bolsa de 10 U/μl, 20 U/mL final) y 30 μl de NaCl (1 M stock, 30 mM final).
- Añadir TE adicional para llevar el volumen a 1 mL. Mezclar la solución con un vórtex.
- Añadir el todo 1 mL de la solución de enzima lítica a no más de 1 mL de microgels lavado. Mezclar por pipeteo 10 x. Incubar la mezcla a 37 º C durante > 2 h en un agitador (la máxima es de incubación durante la noche).
5. base de detergente Microgel tratamiento
-
Después de la lisis mediante enzimas líticas (paso 4), lavar las microgels.
- Desactivación de la microgels a 2.000 x g durante 2 min y aspirar el sobrenadante. Las gotitas aparecerá blanco opaco debido a restos de células dispersados en el precipitado.
- Resuspender el microgels en 5 mL de tampón de Tris-HCl 10 mM y la pipeta hacia arriba y abajo 10 veces para mezclar.
- Desactivación la mezcla a 2.000 x g durante 2 min., luego retirar el sobrenadante por aspiración.
-
Realizar un tratamiento de detergente en el microgels.
- Resuspender los geles en un tampón de lisis de litio dodecil sulfato (tapas) (0.5% w/v tapas en 20 mM Tris-HCl) y 60 μL de EDTA de 0,5 M hasta un volumen final de 3 mL.
- Añadir 5 μl de enzima proteinasa K (stock de 800 U/mL). Pipeta de arriba y abajo 10 veces para mezclar, luego incubar la mezcla a 42 ° C en un bloque de calor durante 1 hora solubilizar las membranas celulares y digerir las proteínas.
-
Tras el tratamiento detergente, lavar las microgels.
- Girar el tubo cónico con la microgels por a 2.000 x g durante 2 minutos retirar el sobrenadante por aspiración.
- Lave el microgels con 10 mL de polisorbato de v/v 2% 20 en agua. Pipeta de arriba y abajo 10 veces para mezclar.
- Girar el tubo cónico hacia abajo a 2.000 x g durante 2 min., luego retirar el sobrenadante por aspiración.
- Lave el microgels con 10 mL de etanol al 100% para inactivar cualquier enzima residual. Pipeta de arriba y abajo 10 veces para mezclar.
- Girar el tubo cónico hacia abajo a 2.000 x g durante 2 min., luego retirar el sobrenadante por aspiración.
- Lave el microgels con 10 mL de polisorbato de 0.02% v/v 20 en agua. Pipeta de arriba y abajo 10 veces para mezclar.
- Girar el tubo cónico hacia abajo a 2.000 x g durante 2 min., luego retirar el sobrenadante por aspiración.
- Repetir el polisorbato 20 lavar 3 x. Pasar la solución por un colador de celda de 100 μm antes del último lavado para eliminar cualquier grumos grandes.
- Resuspender el microgels en 5 mL de tampón de Tris-HCl 10 mM para evitar la degradación del ADN. Los microgels pueden ser almacenadas a 4 ° C hasta 1 semana antes de la tagmentation (paso 7).
6. generar gotitas de código de barras por PCR Digital
- Preparar un stock de 500 pM de barra cartilla (tabla 2) en un 1 x de buffer de TE en un tubo de vínculo de baja. Antes de cada uso, diluir la cartilla a una bolsa de trabajo de 13:00 y calentar a 70 ° C por 1 min en un bloque de calor.
- Preparar una mezcla de reacción de PCR 150 μL con 75 μl de la master de arranque en caliente alta fidelidad de la mezcla (véase Tabla de materiales) (2 x), 42 μl de agua grado PCR, 3 μl de primer DNA_BAR (10 stock μm, 0,2 μm de final), 3 μl de primer P7_BAR (stock 10 μm Final 0.2 μm), 6 μl de dilución de código de barras (stock de 13:00, final 40 fM), 6 μl de polisorbato 20 (50% v/v stock, 2% final) y 15 μl de PEG 6 k (50% w/v stock, 5% final). Mezclar mediante pipeteo arriba y abajo de 10 x.
- Preparar una jeringa con respaldo de HFE dibujando 200 μL de HFE en aceite en una jeringa e introducirlo con una aguja. Coloque una sección de tubería de PE en la aguja y cebe la línea a mano. Inserte el extremo de la tubería de PE en la solución de destino y dibujar cuidadosamente los 150 μL de la mezcla PCR en la tubería de PE y la jeringa. Cargue la jeringa en una bomba de jeringa.
- Cargar una jeringa de 1 mL con aceite fluorado (HFE) que contiene un agente tensioactivo de 2% w/w de perfluoropoliéter polietileno glicol (PEG de PFPE), ajuste con una aguja y lo coloca en una bomba de jeringa.
-
Generar 25 μm gotas de código de barras utilizando el dispositivo de dropmaking de flujo de trabajo.
Nota: Ver Figura 3A para un dispositivo esquemático indicando la ubicación de la salida y entradas de reactivo.- Conecte la entrada de las células de un dispositivo de flujo Co dropmaker con un pequeño trozo de soldadura de plomo.
- Conectar las jeringas cargadas con HFE y PCR mezcla a las entradas de dispositivo de microfluidos con trozos de tubería de PE, con la entrada de la Agarosa fundida para el código de barras PCR. Antes de insertar los tubos en el dispositivo, cebar los bombeadores para quitar el aire de la línea.
- Conecte un trozo de tubería a la salida y coloque el extremo libre en un tubo PCR de 0,2 mL. Utilice las siguientes tasas de flujo (recomendado) para dropmaking: 600 μl/h para el HFE 2% w/w PFPE-PEG y 200 μL/h para la mezcla PCR. Recoger las gotas a los tubos de PCR con aproximadamente 50 μl de gotas en cada tubo.
- Después de la dropmaking, retire con cuidado la capa más baja de aceite HFE de las emulsiones utilizando puntas de pipeta de carga de gel y sustituirla por FC-40 aceite fluorado que contiene un surfactante de PFPE PEG 5% p/p. Ciclo térmico con el siguiente protocolo: 98 ° C por 3 min, 40 x (98 ° C por 10 s, 62 ° C por 20 s, 72 ° C por 20 s), 72 ° C por 5 min y luego espera a 12 ° C.
Nota: Las gotitas de un ciclo térmico pueden ser almacenadas a 4 ° C hasta por 1 día. -
Verificar el tipo de amplificación y encapsulación de código de barras.
- Preparar un ácido nucleico de 1 x de la mancha (véase Tabla de materiales) en HFE con un surfactante de PFPE-PEG 2% w/w; la mancha es ligeramente miscible en HFE y se unirá al ADN en las gotitas.
- Añadir 1 μl de código de barras térmica ciclo emulsión a 10 μl de aceite tinción. Incúbelos durante 5 minutos a temperatura ambiente.
- Gotitas de imagen por microscopía fluorescente (GFP canal, longitudes de excitación/emisión de 497/520 nm) en una magnificación de 200 X y cuenta el tipo de encapsulación de código de barras. En cuenta que la señal es discreta: las gotitas que contienen códigos de barras amplificado se fluorescencia brillante, mientras que las gotas vacías aparecerá oscuras (Figura 4B).
7. Tagmentation de la DNA Genomic en gotas
Nota: Vea la figura 2B.
- Preparar 500 μl de solución de tagmentation utilizando los reactivos de una preparación de biblioteca de secuenciación de próxima generación kit (véase Tabla de materiales). Usar 7 μl de enzima de tagmentation y 250 μl de tampón de tagmentation 243 μl de agua grado PCR. Mezclar con un vórtex y girar la mezcla por recoger. Cargar esta solución en una jeringa de 1 mL de aceite-con el respaldo de HFE y montar con una aguja.
-
Preparar el microgels para la reinyección.
- Girar hacia abajo el tubo de microgel por 2 min a 2.000 x g y aspire el sobrenadante. Transfiera 200 μL de geles a la parte superior de una jeringa de HFE-respaldado con una punta de la pipeta de carga de gel y sellar la boquilla con un trozo pequeño de cinta.
- Usando un adaptador 3D impreso centrífuga (ver suplemento de archivos de 1 y 2), girar la jeringa microgel por 3 min a 3.000 x g.
- Quite el sobrenadante líquido de la jeringa con una punta de la pipeta de carga de gel. Empuje la microgel capa a la base de la boquilla de la jeringa. Coloque la jeringa con una aguja.
- Cargar una jeringa de 3 mL con aceite fluorado (HFE) que contiene un agente tensioactivo de 2% w/w de perfluoropoliéter polietileno glicol (PEG de PFPE), ajustar con una aguja y lo coloca en una bomba de jeringa.
- Volver a encapsular las microgels en gotitas que contienen reactivos de tagmentation.
Nota: Ver figura 3B para un dispositivo esquemático indicando la ubicación de la salida y entradas de reactivo.- Conectar las jeringas que contienen HFE, mezcla de tagmentation y microgels a las entradas de dispositivo de microfluidos con trozos de tubería de PE. Antes de insertar los tubos en el dispositivo, cebar los bombeadores para quitar el aire de la línea.
- Conecte un trozo de tubería a la salida y coloque el extremo libre en una jeringuilla de 1 mL vacía con el émbolo a la línea de 1 mL.
- Utilice las siguientes tasas de flujo (recomendado) para dropmaking: 2.000 μl/h para el HFE 2% w/w PFPE-PEG, 200 μL/h para la microgels y 500 μl/h para la mezcla de tagmentation.
- Verificar el tipo de encapsulación de microgel un microscopio óptico con un aumento de X 400. Aproximadamente el 80-90% de las gotas debe contener una microgel, como se muestra en la figura 4.
- Colocar la jeringa que contiene las emulsiones de tagmentation con una aguja e incubar verticalmente en un bloque de calor o en el horno durante 1 hora a 55 º C para fragmentar el ADN genómico.
8. el código de barras sola célula por fusión de microfluidos doble
Nota: Ver figura 2.
- Preparar las gotas de código de barras para la fusión mediante la sustitución de la fracción de aceite FC-40 con HFE 2% w/w PFPE-PEG. Cuidadosamente transfiera estas gotas a una jeringa de 1 mL, ajustar con una aguja y lo coloca en una bomba de jeringa.
- Cargar la jeringa de la gotita de microgel incubada y tagmented en una bomba de jeringa.
- Preparar 500 μl de la mezcla PCR. Añadir los reactivos en el siguiente orden para impedir la formación de precipitados: 140 μl de agua grado PCR, 10 μl de la cartilla de P5_DNA (10 stock μm, 0,2 μm de final), 10 μl de la cartilla de P7_BAR (10 μm acción, final 0.2 μm), 50 μl de PEG 6k (stock 50% w/v 5% final), 50 μl de polisorbato 20 (50% v/v stock, 5% final), 250 μl de Taq Master Mix (2 x) (véase Tabla de materiales), 10 μl de polimerasa isotermo (véase Tabla de materiales) y 10 μl de tampón de neutralización (véase Tabla de materiales). Mezclar mediante pipeteo arriba y abajo de 10 x y exprimido la mezcla hacia abajo para recogerlo. Cargar esta solución en una jeringa de 1 mL con respaldo de HFE, ajuste con una aguja y lo coloca en una bomba de jeringa.
- Carga tres mL 3 jeringas con aceite fluorado (HFE) que contiene surfactante 2% w/w de perfluoropoliéter polietileno glicol (PEG de PFPE), ajuste cada uno con una aguja y colocarlos en las bombas de jeringa.
- Carga tres jeringas de 1 mL con 2 M de NaCl, ajuste cada uno con una aguja y dejar de lado.
-
Combinar las gotitas de código de barras, tagmented gotitas de genoma, y mezcla de PCR utilizando el dispositivo de doble fusión.
Nota: Consulte la figura 5 para un dispositivo esquemático indicando la ubicación de la salida y entradas de reactivo y el electrodo.- Conectar las 3 jeringas de NaCl a las 2 entradas de electrodo y foso solo inletusing pedazos de tubería de PE. Para los electrodos, aplique presión a las jeringas manualmente hasta que los electrodos están completamente llenos con solución salina. Después de llenar los electrodos, aplicar presión manual en la jeringa del foso hasta el foso se llena. Conecte el extremo libre del foso con un pequeño pedazo de plomo de la soldadura.
- Conectar las 3 jeringas HFE en bombas a las 2 entradas de aceite separador y dropmaking aceite inletusing pedazos de tubería de PE. Antes de insertar los tubos en el dispositivo, cebar los bombeadores para quitar el aire de la línea.
- Conecte la jeringa de mezcla PCR, microgel gotas jeringa, código de barras los drops y jeringa en sus respectivas entradas utilizar tubería de PE. Dispara a todas tuberías de reinyección de gota con una pistola antiestática antes de conectar a las agujas de la jeringa; el tratamiento antiestático reduce el riesgo de fusión de gota inducida por cargas estáticas en la tubería de PE.
- Conecte un pedazo de tubo PE a la salida y coloque el extremo libre en un tubo PCR de 0,2 mL.
- Conecte la aguja de la jeringa de electrodo a un inversor fluorescente de cátodo frío, utilizando una pinza de cocodrilo. La posición DC fuente de alimentación del inversor 2 V.
- El equipo puede utilizar doble fusión con los caudales recomendados: 300 μL/h para las tagmented microgel gotas, 100 μl/h para las gotas de código de barras, 1500 μl/h para el HFE 2% w/w PFPE-PEG (dropmaking aceite), 600 μl/h para la amplificación de la mezcla, 200 μL/h para el HFE 2% w/w PFPE-PEG ( código de barras separadores de aceite) y 700 μl/h para el HFE 2% w/w PFPE-PEG (microgel de separadores de aceite). Recoger las gotas en los tubos de PCR con aproximadamente 50 μl de la emulsión en cada tubo.
- Antes de los ciclos térmicos, retire con cuidado la capa más baja de aceite HFE de las emulsiones utilizando puntas de pipeta de carga de gel y reemplazarlos con FC-40 aceite fluorado que contiene un surfactante de PFPE PEG 5% p/p. Ciclo térmico con el siguiente protocolo: 65 ° C durante 5 min, 95 ° C por 2 min, x 30 (95 ° C por 15 s, 60 ° C por 1 min, 72 ° C por 1 min), 72 ° C por 5 min y luego espera a 12 ° C.
-
Recuperar el código de barras de ADN de las gotitas de un ciclo térmico.
- Piscina las gotitas en un tubo de microcentrífuga y romper las emulsiones con 20 μl de FOP. Vortex por 10 s para mezclar.
- Girar el tubo a 10.000 x g durante 1 min a fraccionar la mezcla acuosa (superior) y las fases del aceite (inferior). Con cuidado quite la capa acuosa superior del tubo utilizando una pipeta y transferir a un tubo nuevo de microcentrífuga. Descartar la fase oleosa.
- Purificar el producto PCR con código de barras en una columna de giro según protocolo del fabricante y eluir en 20 μl de tampón de TE de x 1.
- Proceder a la secuenciación realizada y análisis según los pasos 9 y 10.
9. Biblioteca preparación y secuenciación
- Preparar la biblioteca sola célula para la secuenciación siguiendo los protocolos del fabricante para la selección del tamaño del fragmento y cuantificación.
- Secuencia la biblioteca final junto con química por defecto para lectura 1 y lectura 2. Usar el primer Índice 1 personalizado I7_READ (tabla 2) para un índice de 15 puntos leer, correspondiente a la sola célula de código de barras.
10. Análisis de datos unicelular
Nota: Los scripts de Python personalizado para el control de calidad y análisis preliminar de datos del SiC-seq se pueden descargar de https://www.github.com/AbateLab/SiC-seq.
- Ejecute el script "barcodeCleanup.py" para realizar control de calidad en el código de barras Lee y exportar los datos de unicelular a una base de datos SQLite. Para un experimento de control, use este script con el "-Alinee" conjunto para alinear la lee a genomas de referencia conocido de la bandera.
- Analizar la pureza de los grupos de código de barras (para un experimento de control) usando el script "purity.py" y confirmar los valores de alta pureza consistentes con la Figura 6B.
Resultados
El flujo de trabajo experimental de SiC-seq contiene 3 dispositivos de microfluidos PDMS fabricados mediante un procedimiento de litografía blanda (figura 1). Un dropmaker de flujo Co (Figura 3A) genera 25 μm de código de barras digital gotitas para el etiquetado de ADN genómico con un identificador único de unicelulares. Los oligonucleótidos de código de barras consisten en una secuencia degenerada de 15 bp flanqueado por las manijas de la PCR para la amplificación (tabla 2, BAR la cartilla). Los códigos de barras son diluidas a una concentración de femtomolar para lograr el encapsulamiento de una sola molécula, y todas las gotas reciben fragmentos de código de barras ya sea 0 o 1. Se amplifican las gotitas que contienen un código de barras, produciendo muchas copias de amplicones de código de barras de doble hebra. Una mancha de ácido nucleico se utiliza para comprobar la amplificación exitosa y cuantificar la tasa de encapsulación de los fragmentos de código de barras (Figura 4B). Los microgels son generados por fluir conjuntamente una suspensión bacteriana y un gel de agarosa fundida en caudales iguales (figura 2A). La agarosa está dispuesta en dos veces la concentración final deseada, como el proceso de dropmaking de flujo Co diluye efectivamente las soluciones acuosas por un factor de 2. Como la agarosa se enfría, se solidifica en un microgel de 25 μm de diámetro ocupando el volumen esférico de la gota.
Una serie de pasos de lavado y lisis purifica el DNA genómico de alto peso molecular en el microgels (figura 2B). Después de romper las emulsiones, se realizan lavados acuosos en grandes volúmenes para diluir un rastro disolventes orgánicos que pueden inhibir los posteriores tratamientos enzimáticos. El lavado microgels se observan bajo un microscopio para verificar la tasa de encapsulación de la célula (Figura 4A). Un cóctel de enzimas con actividad lítica amplia se añade a la suspensión de microgel para digerir las paredes celulares de las bacterias y microbios eucarióticos19. Un segundo tratamiento con proteinasa K y detergente degrada las proteínas y solubiliza restos celulares.
Tagmentation de la DNA purificada se lleva a cabo en gotas para evitar la contaminación cruzada potencial resultante de la difusión de fragmentos de ADN de pequeñas tagmented entre los microgels18. Un dispositivo de encapsulado de la gota (figura 3B) compartmentalizes cada microgel con una enzima tampón y tagmentation, que al mismo tiempo fragmentos de ADN de doble hebra mientras también "etiquetado" con un oligonucleótido precargado20. El microgels se cargan en las gotitas como partículas cerrar-embalado, lograr tasas de encapsulación acercarse 1 microgel para cada gota con pocos dobletes21 (figura 4).
En la etapa final del flujo de trabajo microfluídicos (figura 2), un dispositivo realiza una operación de doble fusión combinando 1 gota de código de barras, 1 gota que contiene el microgel y la mezcla de amplificación en un proceso de dos pasos controlados. En primer lugar, una gota de reactivo PCR está emparejada y se fusionó con una gota de código de barras en la región que se muestra en amarillo (figura 5). Los electrodos agua salados en el canal microfluídico producen un gradiente de alto campo eléctrico que provoca la fusión de gotas. De manera similar, la primera gota se fusionó es apareada con una gotita de microgel y combinó una segunda vez en la región que se muestra en rojo. Las gotitas son recogidas y off-chip en una extensión de solo-solapamiento (SOE) PCR ciclo térmico. Los extremos complementarios superpuestos del código de barras y el ADN genómico de tagmented permiten la fusión y la amplificación exponencial de construcciones de código de barras sólo correctamente.
Los datos de la secuencia es primero filtrados por una calidad de lectura y luego analizados agrupando las lecturas según su secuencia de 15-bp unicelular código de barras. Para que un grupo de código de barras ser considerado válido, debe contener un mínimo de lecturas; este umbral limita el análisis a las células con una cantidad útil de datos de secuenciación y elimina el código de barras de PCR-transformado "huérfanos" del conjunto de datos. En este ejemplo se ejecute, el mínimo se establece en 7,5 kbps por grupo (50 lecturas de 150 bp cada uno). Un histograma de las cuentas de código de barras versus el tamaño del grupo muestra que una porción significativa de los grupos de código de barras válido es justo por encima del tamaño de umbral (figura 6A).
En un experimento de control donde se conoce la composición de la comunidad microbiana, la pureza y abundancia relativa métricas se utilizan para evaluar la calidad de un SiC-seq ejecutar. Aquí, una comunidad de 10 células sintética de 3 bacterias Gram-negativas, 5 bacterias Gram positivas y 2 levaduras se analiza. La pureza de un grupo de código de barras determinado se define como el número de lecturas con el genoma más común en el grupo dividido por el número total de lecturas en el grupo. La gran mayoría de los grupos de código de barras tiene purezas superiores a 0.95 (Figura 6B). Abundancia relativa de los tipos de células se calcula contando el raw Lee y contando los grupos de código de barras, donde los grupos se asignan a un tipo de célula correspondiente al consenso de sus lecturas de miembro (figura 6). La abundancia de grupos de código de barras y lee la pista en proporciones aproximadamente iguales, lo que indica que las poblaciones celulares son ser muestreadas que ciertas especies no figuran en el código de barras desproporcionadamente pequeños o grandes grupos. Trazar la cobertura total de todos los grupos de código de barras de una sola especie indica una alta cobertura a través de todo el genoma, con pocos o ningún regiones de deserción (figura 7). La uniformidad de la cobertura puede ser verificada con una distribución de frecuencia de valores normalizados de la cobertura, con mayoría centrados alrededor de la media de los valores (figura 7, de la inserción).
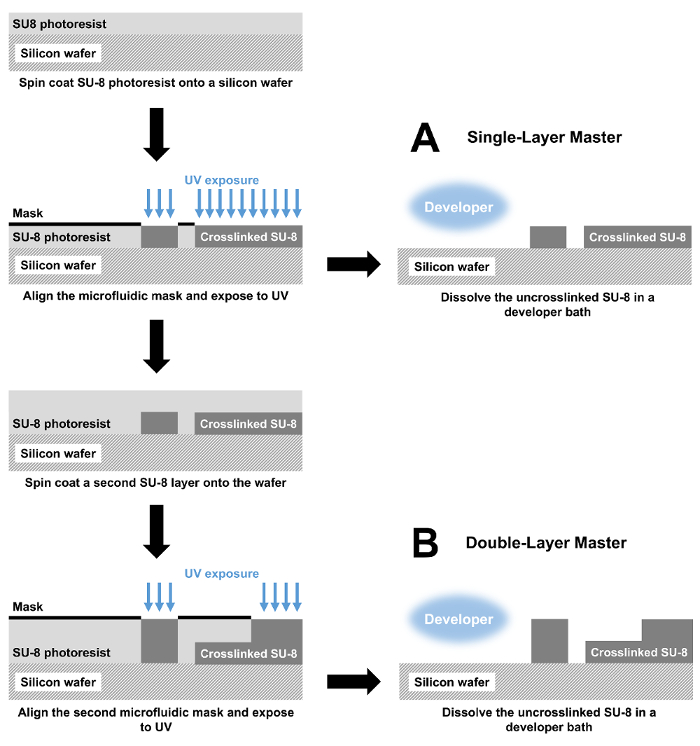
Figura 1 : Fabricación de dispositivos microfluídicos por Fotolitografía. Maestro (A) moldes con una altura característica solo se fabrican por una capa de photoresist SU-8 sobre una oblea de silicio de la capa de la vuelta. La fotoresistencia es entonces modelado con una máscara photolithographic y luz UV, reticulando el expuesto SU-8. Por último, todo SU-8 se disuelve en un baño de desarrollador. El molde resultante se utiliza para la fundición de PDMS que se enlaza a un portaobjetos de vidrio para producir el dispositivo de microfluidos completa. ()B) para un dispositivo de doble capa, la fabricación del mismo modo comienza con pasos de capa y la exposición de vuelta. Luego se repiten estos pasos para crear un dispositivo de dos capas. Haga clic aquí para ver una versión más grande de esta figura.
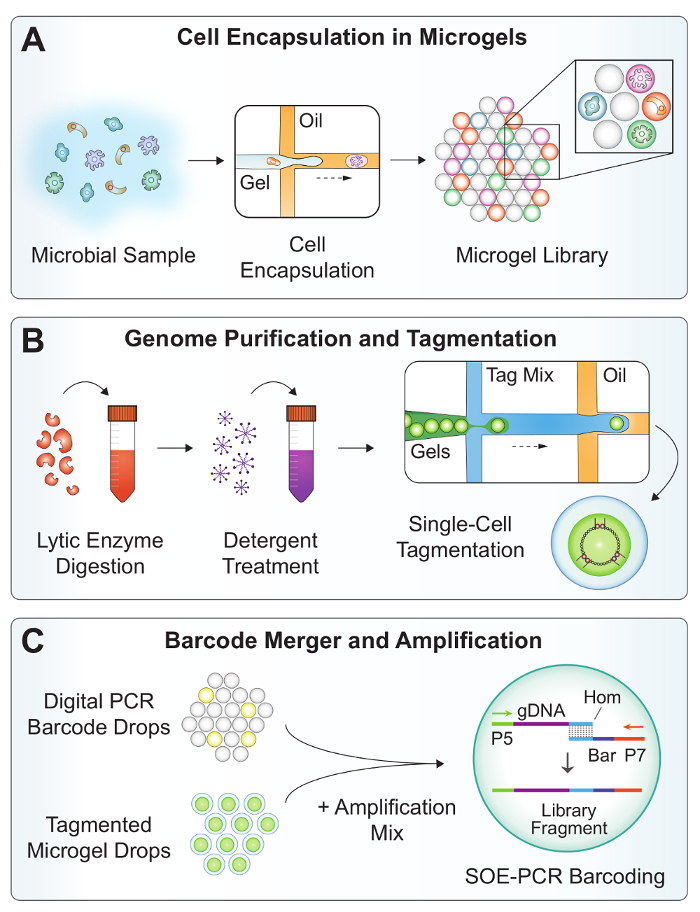
Figura 2: Resumen del flujo de trabajo SiC-seq. Suspensión microbiana (A) A es co fluyó con agarosa fundida en un dispositivo de dropmaker para encapsular las células en microgels. (B) los microgels son sometidos a una serie de lavados para purificar el ADN genómico bacteriano. Enzimas líticas digestión las paredes celulares de bacterias Gram positivas y levaduras, y detergente solubiliza los desechos celulares. Los microgels son re encapsulados en gotas para la tagmentation reducir la contaminación cruzada. (C) la fusión de microfluidos combina un código de barras digital de PCR, un genoma de microgel tagmented y una mezcla de amplificación a una velocidad > 1 kHz. Off-chip SOE-PCR empalma un código de barras único unicelular en el genoma de tagmented y amplifica selectivamente plenamente construcciones de código de barras. Haga clic aquí para ver una versión más grande de esta figura.
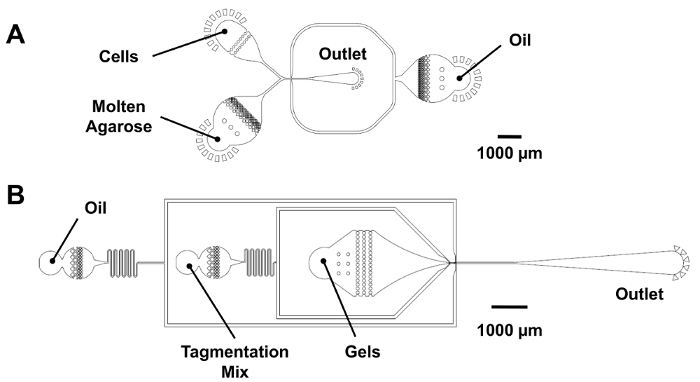
Figura 3: Dispositivos de microfluidos para la encapsulación de la dropmaking y microgel. (A) este panel muestra un dropmaker de flujo Co (25 μm de altura característica). Células y agarosa fundida se introducen el dispositivo en caudales iguales para producir 25 gotitas de μm en un cruce de μm μm x 25 25. Para la dropmaking de código de barras digital, está conectada la entrada de la célula, y una mezcla PCR se introduce en la entrada de la agarosa. (B) este panel muestra un dispositivo de re-encapsulación microgel (25 μm de altura característica). Las microgels de flujo en un orificio de entrada en forma de embudo para mantener su cerrar-embalado el pedido y recibir un volumen de la mezcla tagmentation antes de la encapsulación en un cruce de μm 25 μm x 30. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4 : Microfotografías de las gotas y microgels. (A) este panel muestra lavado 25 μm microgels antes de la lisis enzimática. Las bacterias se tiñen fluorescente para la cuantificación de la tasa de encapsulación. Estadísticas de carga de Poisson determinan que las células deben ser encapsulado en una tasa de 1 en 10 gotas o menos para reducir al mínimo la frecuencia de encapsulamiento de múltiples eventos. (B) este panel muestra una imagen de microscopía de fluorescencia de 25 μm gotas de código de barras digital tratada con una tinción de ácido nucleico. Las gotitas que contienen fragmentos de código de barras amplificado producen una señal de fluorescencia fuerte. (C) este panel muestra microgels volver a encapsularse en 50 μm gotas. El cerrar-embalaje de la microgels permite tarifas de encapsulación acercarse a 1 gel por cada gota con pocos dobletes. Haga clic aquí para ver una versión más grande de esta figura.
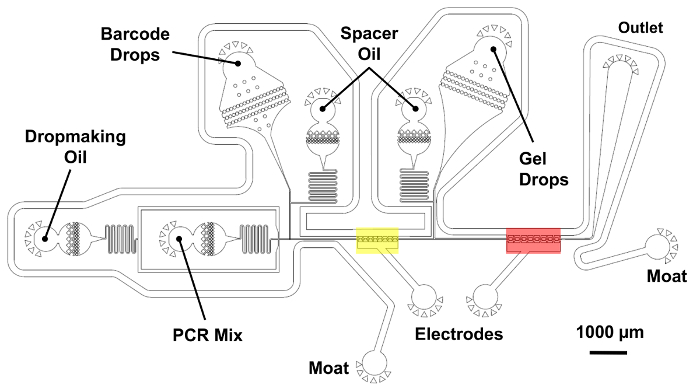
Figura 5 : Dispositivo de doble fusión de microfluidos para código de barras sola célula genoma. Una operación de fusión de dos etapas pares de gotas de código de barras con genomas de tagmented con un alto rendimiento. Una gota de la mezcla PCR es primero generada y se fusionó con una gotita de código de barras en la región que se muestra en amarillo utilizando electrodos agua salados. A continuación, una gota que contiene un microgel se introduce y se fusionaron una segunda vez en la región que se muestra en rojo. Entradas de aceite permiten un control preciso de la distancia entre las gotas reinjected. La cámara de reinyección de código de barras y sus separadores de aceite se colocan en la capa más corta de 25 μm, sombreada en azul. Todas las demás características dispositivo pertenecen a la capa más gruesa con 45 μm de altura total. Haga clic aquí para ver una versión más grande de esta figura.
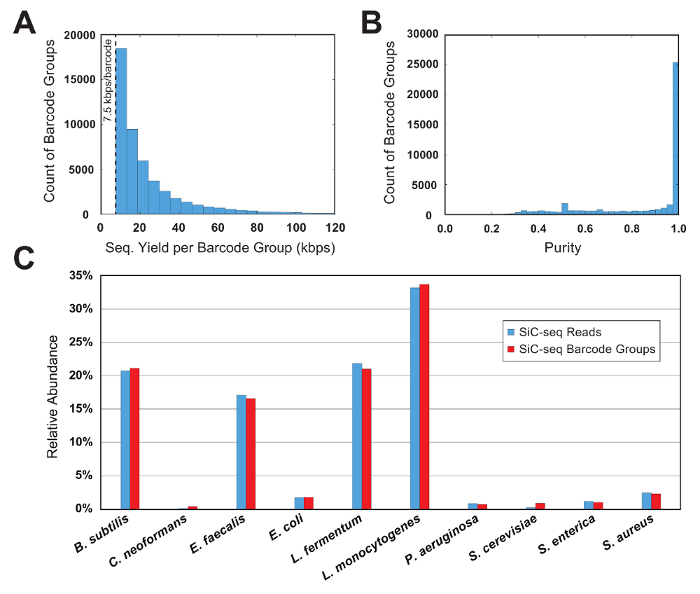
Figura 6 : Métricas de grupo de código de barras para una comunidad microbiana sintético 10 celular. (A) este panel muestra la distribución del código de barras tamaños de grupo. El número de grupos de un tamaño determinado disminuye exponencialmente como el aumento de tamaño de grupo. Un umbral mínimo de 7,5 kbps por grupo limita el análisis a los grupos con una cantidad suficiente de información y elimina la secuencia mutada de PCR "huérfanos." (B) este panel muestra la distribución de código de barras purezas de grupo. La gran mayoría (> 90%) de los grupos son de muy alta pureza (> 95%). (C) este panel muestra la abundancia relativa de 10 especies calculado a nivel de grupo de lectura y código de barras. 2 contar métodos producen resultados similares, indicando que los tamaños de grupo de código de barras son compatibles entre especies. Haga clic aquí para ver una versión más grande de esta figura.
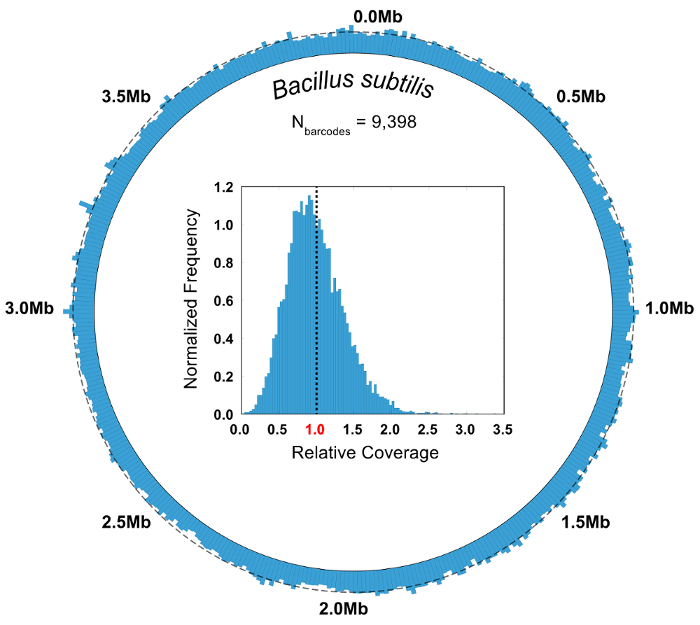
Figura 7 : Agregado cobertura genómica de Bacillus subtilis grupos de código de barras. Las lecturas de todos los grupos de código de barras con la bacteria B. subtilis (N = 9.398) son agrupados y analizados en conjunto. Mapa cobertura circular ilustra la uniformidad de la cobertura de la SiC-seq Lee, con las regiones de no deserción observables. Una línea discontinua alrededor de la circunferencia indica la cobertura promedio (5,55 x). El histograma de la inserción de las frecuencias de cobertura relativa muestra que una mayor parte de las bases están cubiertas en una profundidad cerca de la media de todo el genoma, representado por la línea punteada. Haga clic aquí para ver una versión más grande de esta figura.
| Dispositivo de | 1 altura de la capa (μm) | 1 º capa Spin Speed (rpm) | 2 º altura de la capa (μm) | 2 º capa Spin Speed (rpm) |
| Dropmaker de flujo de Co | 25 | 4000 | N / A | N / A |
| Re-encapsulador gel | 25 | 4000 | N / A | N / A |
| Doble fusión | 25 | 4000 | 20 | 5000 |
Tabla 1: parámetros de fabricación de dispositivos microfluídicos. Esta tabla muestra un listado de los dispositivos de microfluidos en el flujo de trabajo del SiC-seq con sus velocidades requiere de vuelta photoresist capa (basado en las especificaciones del fabricante para SU-8 3025).
| Etiqueta | Secuencia (5' > 3') | ||||
| BAR | GCAGCTGGCGTAATAGCGAGTACAATCTGCTCTGATGCCGCATAGNNNNNNNNNNNNNNNTAAGCCAGCCCCGACACT | ||||
| DNA_BAR | CTGTCTCTTATACACATCTCCGAGCCCACGAGACGTGTCGGGGCTGGCTTA | ||||
| P7_BAR | CAAGCAGAAGACGGCATACGAGATCAGCTGGCGTAATAGCG | ||||
| P5_DNA | AATGATACGGCGACCACCGAGATCTACACTCGTCGGCAGCGTC | ||||
| I7_READ | GCCCACGAGACGTGTCGGGGCTGGCTTA | ||||
Tabla 2: Secuencias de la cartilla.
Archivo adicional 1: Haga clic aquí para descargar este archivo.
Archivo adicional 2: Haga clic aquí para descargar este archivo.
Discusión
El flujo de trabajo de SiC-seq microfluídicos produce sola célula genoma secuencia datos de miles de células bacterianas. Códigos de barras digitales empalmados en los genomas de las células encapsuladas de microgel permiten la deconvolución en silico de datos NGS en grupos de código de barras Lee originarios de la misma célula. Un experimento de control con una comunidad microbiana de composición conocida es necesario para evaluar la pureza de los grupos de código de barras. Una gran parte de los grupos de baja pureza indica que la tasa de encapsulación de la célula es demasiado alto o que hay gota significativa la contaminación cruzada que ocurre durante las etapas de procesamiento de microfluidos. Según las estadísticas de Poisson, las células y códigos de barras deben ser encapsuladas en una proporción de blanco de 1 partícula por 10 gotas limitar la tasa de múltiples eventos de encapsulación a menos del 5% de todas las gotas no vacío. Una mayor tasa de encapsulación aumenta las tasas de Dobletes exponencialmente, por lo que la verificación de la relación de encapsulación durante el proceso de dropmaking es de importancia crítica. Los usuarios deben ser particularmente cautelosos de la encapsulación de varias celdas en una sola microgel ya lee de diversas células que comparten la misma secuencia de código de barras no puede ser bioinformatically separado. En el caso de 1 célula recibe 2 códigos de barras diferentes, la pureza del grupo de código de barras no se ve afectada aunque los parámetros de la abundancia están sesgados cuando cuenta la secuencia de código de barras.
Gota la contaminación cruzada también puede presentarse debido a las condiciones de fusión subóptima. Durante una operación de éxito, el dispositivo de fusión de microfluidos (figura 5) puede controllably par 1 gotita de código de barras con 1 microgel y un volumen de reactivo PCR. Las tasas de flujo no ideal se traducirá en una gota de emparejamiento en proporciones incorrectas: 1 código de barras puede combinarse con 2 microgels, por ejemplo. Todas las tarifas de flujo enumeradas en el protocolo pretenden ser las estimaciones y pueden necesitar ser ajustado según pequeñas variaciones en los tamaños de geometría y gotita de dispositivo. Los usuarios con acceso a cámaras con capacidad de grabación de alta velocidad (> 10.000 frames/s) deben verificar la fusión correcta gota al principio y en el transcurso de la operación de microfluidos. Los usuarios sin acceso a una cámara de alta velocidad pueden recoger un pequeño volumen de la salida fusionada y medir manualmente los tamaños de gota bajo el microscopio. El tamaño de las gotas debe ser uniforme: un exceso de códigos de barras sin combinar o microgel gotas indica que las tarifas de reinyección deben reducirse en consecuencia.
Varios general Precauciones al manejar microgels y microgotas para preservar su integridad. Microgels, aunque mecánicamente robusto, debe ser suficientemente refrigerado por antes de la ruptura y lavado pasos para asegurar la completa gelificación. Microgels no-esféricas son una indicación que la agarosa no se da suficiente tiempo para solidificar. Lavado microgels, desactivación las suspensiones a la velocidad requerida para evitar una pérdida de producto. Hidrogel de agarosa tiene un índice de refracción cerca que la del agua y puede ser difícil de ver en un tubo22, así que los usuarios deben identificar cuidadosamente el límite de gel líquido antes de la aspiración. Gotas de agua en aceite son susceptibles a la fusión por la acumulación de fuerzas estáticas23 laboratorio guantes y tubería. Por esta razón, se recomiendan cargar las jeringas de reinyección de gotitas con las manos desnudas y el tratamiento de todas las líneas de reinyección con una pistola antiestática antes de cebado de la bomba. Gotas grandes coaligadas pueden eliminarse lentamente girando las emulsiones en una jeringa y aspirar manualmente las gotas más grandes, que se acumulan en la parte superior debido a su mayor fuerza boyante.
SiC-seq es la primera tecnología para demostrar la secuenciación del genoma de unicelular de > 50.000 células bacterianas. Esta plataforma ofrece ventajas significativas en el rendimiento sobre los enfoques existentes y permite un muestreo más profundo de las comunidades microbianas heterogéneas. Hasta la fecha, tecnologías de microfluidos para la secuenciación del genoma de la célula solo han empleado microchambers micropocillos y9 24 para aislamiento de células y la amplificación, pero con rendimientos en el rango de sólo decenas a cientos de células. La clasificación del flujo de las células en wellplates5,6 no requiere ninguna instrumentación especializada microfluídicos pero posee un rendimiento igualmente bajo. Dado que las muestras de suelo y agua del ambiente comúnmente tienen las diversidades alfa de > 1.000 en las especies de nivel25,26, SiC-seq es altamente ventajosa en virtud de su capacidad para un número mucho mayor de organismos de la muestra. El flujo de trabajo de SiC-seq es adaptable a las entradas de celda de laboratorio cultura, el medio ambiente natural o un anfitrión vivo. Una muestra de células hay que estar en una suspensión acuosa y libre de partículas grandes (> 10 μm) adecuada para la encapsulación de microfluidos. Por ejemplo, el método se ha aplicado previamente a una muestra de agua de mar utilizando una serie de lavado y filtrado de pasos para el procesamiento previo de las células antes de la encapsulación17.
El protocolo de SiC-seq genera una cantidad relativamente escasa de los datos de la secuencia de cada célula individual y puede no ser adecuado para todas las aplicaciones. Algunos algoritmos de Bioinformática como Asamblea de genoma de novo o variante de un solo nucleótido (SNV) que requieren profundidades de cobertura más alto para trabajar con eficacia. En cambio, grupos de código de barras pueden ser agrupado en silico por binning taxonomía de métodos27 para que los algoritmos pueden ser aplicados en grandes conjuntos de lecturas. La relativamente baja eficiencia de código de barras general del flujo de trabajo SiC-seq también puede presentar retos en casos donde la disponibilidad de la muestra de entrada es baja. SiC-seq se basa en un paso de la encapsulación de código de barras de distribución de Poisson, por lo tanto, aproximadamente el 10% de las células recibe un código de barras molecular y se amplifican durante la etapa de preparación final de la biblioteca. Mientras que esto es comparable a otros planes microdroplet basado en código de barras del10, los usuarios que trabajan con muestras de células preciosas pueden tener dificultad para lograr el rendimiento adecuado de la biblioteca para la secuencia y pueden necesitar aumentar el número de ciclos PCR en la final etapa de AMPLIFICACION. Otra posible solución para usuarios con conocimientos de microfluidos es ordenar gotas de código de barras positivas después del paso PCR digital, así que la eficacia total de código de barras > 85%28.
Una potencial futura dirección para la tecnología SiC-seq es adaptar el flujo de trabajo para el uso con las células mamíferas, allanando el camino para nuevos estudios clínicos de unicelulares. Por ejemplo, un análisis de la variación del número de copia entre cáncer solo células mayo mejorar nuestra comprensión del papel de la heterogeneidad en la patología de cáncer2. Por otra parte, integrar SiC-seq con métodos existentes para probar y enriquecer secuencias de ADN de interés29 permitiría la secuenciación específica de unicelular de subpoblaciones o raras cepas de células. Con muestras ambientales, genes de dentro de un camino metabólico conocido podrían dirigidos y analizados contextualmente junto a vecinos de genes para identificar novela islas genómicas. Desde dentro de un ambiente del anfitrión humano, muestras de bacterias patógenas bajo título podrían ser aisladas y secuenciaron en el nivel unicelular para examinar más de cerca sus orígenes genotípicos de virulencia.
Divulgaciones
Patentes relativas a este flujo de trabajo pueden ser licenciado en la Bio de la misión, de que Adam R. Abate es un accionista.
Agradecimientos
Este trabajo fue financiado por la National Science Foundation a través de un premio de carrera (número DBI-1253293); los institutos nacionales de salud (NIH) (grant números HG007233-01, 01-EB019453-R01, 1R21HG007233, DP2-AR068129-01, R01-HG008978); y la defensa avanzada investigación proyectos Agencia fundiciones programa de vida (número de contrato HR0011-12-C-0065, N66001-12-C-4211, HR0011-12-C-0066).
Materiales
| Name | Company | Catalog Number | Comments |
| 3" silicon wafers, P type, virgin test grade | University Wafers | 447 | |
| SU-8 3025 photoresist | Microchem | 17030192 | |
| Spin coater | Specialty Coating Systems | G3P-8 | |
| Photomasks | CadArt Servcies | (custom) | See Supplemental Files for mask designs |
| PGMEA developer | Sigma-Aldrich | 484431 | |
| Isopropanol | Sigma-Aldrich | 109827 | |
| Sylgard 184 silicone elastomer kit | Krayden | 4019862 | |
| Degassing chamber | Bel-Art | 42025 | |
| 0.75 mm biopsy punch | World Precision Instruments | 504529 | |
| Glass microscope slides (75 mm x 50 mm) | Corning | 294775X50 | |
| Aquapel (hydrophobic glass treatment) | Pittsburgh Glass Works | 47100 | |
| PE-2 polyethylene tubing | Scientific Commodities | B31695-PE/2 | |
| 1 mL syringes | BD | 309628 | |
| 27 gauge needles | BD | 305109 | |
| Syringe pump | New Era Pump Systems | NE-501 | |
| Novec HFE-7500 fluorinated oil (HFE) | 3M | 98-0212-2928-5 | |
| FC-40 fluorinated oil | Sigma-Aldrich | F9755 | |
| PEG-PFPE surfactant | Ran Biotechnologies | 008-FluoroSurfactant | |
| Space heater | Lasko | CD09250 | |
| Agarose, low gelling temperature | Sigma-Aldrich | a9414 | |
| TE (10X) | Rockland | mb-007 | |
| PBS 1X, pH 7.4 | E&K Scientific Products | EK-65083 | |
| OptiPrep (density gradient medium) | Sigma-Aldrich | d1556 | |
| 1H,1H,2H-Perfluoro-1-Octanol (PFO) | Sigma-Aldrich | 370533 | |
| Span 80 (sorbitane monooleate) | Sigma-Aldrich | s6760 | |
| Hexane | Sigma-Aldrich | 139386 | |
| Tween 20 (polysorbate 20) | Sigma-Aldrich | p2287 | |
| Lysozyme Type IV | MP Biomedicals | 195303 | |
| Mutanolysin | Sigma-Aldrich | M9901 | |
| Zymolyase (yeast lytic enzyme) | Zymo Research | e1004 | |
| Lysostaphin | Sigma-Aldrich | L7386 | |
| Sodium chloride | Sigma-Aldrich | S9888 | |
| EDTA | Sigma-Aldrich | E6758 | |
| Tris-HCl, pH 7.5, 1M | Invitrogen | 15567-027 | |
| Dithiothreitol (DTT) | Teknova | d9750 | |
| Lithium dodecyl sulfate | Sigma-Aldrich | L9781 | |
| Proteinase K | New England Biosciences | P8107S | |
| Ethanol, 200 Proof (100%) | Koptec | V1001 | |
| SYBR Green I (nucleic acid stain) | Invitrogen | S7563 | |
| PEG 6k | Sigma-Aldrich | 81260 | |
| Triton X-100 (octylphenol ethoxylate) | Sigma-Aldrich | t8787 | |
| Nextera DNA Library Prep Kit | Illumina | FC-121-1030 | |
| Phusion Hot Start Flex Master Mix (High-Fidelity Hot Start Master Mix) | New England Biosciences | m05365 | |
| Platinum Multiplex PCR Master Mix (Taq Master Mix) | Applied Biosystems | 4464263 | |
| Warmstart 2.0 Bst Polymerase (isothermal polymerase) | New England Biosciences | m0538m | |
| NT buffer from Nextera XT kit (neutralization buffer) | Illumina | FC-131-1024 | |
| Cold cathode fluorescent inverter | (custom) | (custom) | |
| DC power supply | Mastech | HY1503D | |
| Zerostat 3 anti-static gun | Milty | 5036694022153 | |
| 3D-printed centrifuge syringe holder | (custom) | (custom) | See Supplemental Files for 3D print file |
| Zymo DNA Clean & Concentrator-5 | Zymo Research | D4003 |
Referencias
- Navin, N., et al. Tumour evolution inferred by single-cell sequencing. Nature. 472 (7341), 90-94 (2011).
- Ni, X., et al. Reproducible copy number variation patterns among single circulating tumor cells of lung cancer patients. Proceedings of the National Academy of Sciences of the United States of America. 110 (52), 21083-21088 (2013).
- Schmidt, H., Hensel, M. Pathogenicity islands in bacterial pathogenesis. Clinical Microbiology Reviews. 17, 14-56 (2004).
- Martínez, J. L., Baquero, F. Interactions among strategies associated with bacterial infection: pathogenicity epidemicity, and antibiotic resistance. Clinical Microbiology Reviews. 15 (4), 647-679 (2002).
- Rinke, C., et al. Obtaining genomes from uncultivated environmental microorganisms using FACS-based single-cell genomics. Nature Protocols. 9 (5), 1038-1048 (2014).
- Rinke, C., et al. Insights into the phylogeny and coding potential of microbial dark matter. Nature. 499 (7459), 431-437 (2013).
- Zhang, H., Liu, K. K. Optical tweezers for single cells. Journal of the Royal Society Interface. 5 (24), 671-690 (2008).
- Xu, L., Brito, I. L., Alm, E. J., Blainey, P. C. Virtual microfluidics for digital quantification and single-cell sequencing. Nature Methods. 13 (9), 759-762 (2016).
- Gawad, C., Koh, W., Quake, S. R. Dissecting the clonal origins of childhood acute lymphoblastic leukemia by single-cell genomics. Proceedings of the National Academy of Sciences of the United States of America. 111 (50), 17947-17952 (2014).
- Macosko, E. Z., et al. Highly parallel genome-wide expression profiling of individual cells using nanoliter droplets. Cell. 161 (5), 1202-1214 (2015).
- Klein, A. M., et al. Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell. 161 (5), 1187-1201 (2015).
- Rotem, A., et al. High-throughput single-cell labeling (Hi-SCL) for RNA-Seq using drop-based microfluidics. PLoS One. 10 (5), 1-14 (2015).
- Amini, S., et al. Haplotype-resolved whole-genome sequencing by contiguity-preserving transposition and combinatorial indexing. Nature Reviews Genetics. 46 (12), 1343-1349 (2014).
- Zheng, G. X. Y., et al. Haplotyping germline and cancer genomes with high-throughput linked-read sequencing. Nature Biotechnology. 34 (3), 303-311 (2016).
- Lan, F., Haliburton, J. R., Yuan, A., Abate, A. R. Droplet barcoding for massively parallel single-molecule deep sequencing. Nature Communications. 7, 11784(2016).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Lan, F., Demaree, B., Ahmed, N., Abate, A. R. Single-cell genome sequencing at ultra-high-throughput with microfluidic droplet barcoding. Nature Biotechnology. 35 (7), 640-646 (2017).
- Novak, R., et al. Single-cell multiplex gene detection and sequencing with microfluidically generated agarose emulsions. Angewandte Chemie Internation Edition. 50 (2), 390-395 (2011).
- Gill, C., Van De Wijgert, J. H. H. M., Blow, F., Darby, A. C. Evaluation of lysis methods for the extraction of bacterial DNA for analysis of the vaginal microbiota. PLoS One. 11 (9), 1-16 (2016).
- Picelli, S., et al. Tn5 transposase and tagmentation procedures for massively scaled sequencing projects. Genome Research. 24 (12), 2033-2040 (2014).
- Abate, A. R., Chen, C. H., Agresti, J. J., Weitz, D. A. Beating Poisson encapsulation statistics using close-packed ordering. Lab on a Chip. 9 (18), 2628(2009).
- Jain, A., Yang, A. H. J., Erickson, D. Gel-based optical waveguides with live cell encapsulation and integrated microfluidics. Optic Letters. 37 (9), 1472(2012).
- Karbaschi, M., Shahi, P., Abate, A. R. Rapid chemical-free breaking of microfluidic emulsions with a hand-held antistatic gun. Biomicrofluidics. 11 (4), 1-6 (2017).
- Gole, J., et al. Massively parallel polymerase cloning and genome sequencing of single cells using nanoliter microwells. Nature Biotechnology. 31 (12), 1126-1132 (2013).
- Chao, Y., et al. Metagenomic analysis reveals significant changes of microbial compositions and protective functions during drinking water treatment. Scientific Reports. 3 (1), 3550(2013).
- Fierer, N., et al. Cross-biome metagenomic analyses of soil microbial communities and their functional attributes. Proceedings of the National Academy of Sciences of the United States of America. 109 (52), 21390-21395 (2012).
- Mande, S. S., Mohammed, M. H., Ghosh, T. S. Classification of metagenomic sequences: methods and challenges. Briefings in Bioinformatics. 13 (6), 669-681 (2012).
- Eastburn, D. J., et al. Microfluidic droplet enrichment for targeted sequencing. Nucleic Acids Research. 43 (13), e86(2015).
- Clark, I. C., Abate, A. R. Finding a helix in a haystack: nucleic acid cytometry with droplet microfluidics. Lab on a Chip. 17 (12), 2032-2045 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados