Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Selección de varios subconjuntos de biomarcadores con semejantemente eficaz clasificación binaria actuaciones
En este artículo
Resumen
Algoritmos existentes generan una solución para un conjunto de datos de detección de biomarcadores. Este protocolo demuestra la existencia de varias soluciones igualmente eficaces y presenta un software fácil de usar para ayudar a los investigadores biomédicos a investigar sus conjuntos de datos para el reto propuesto. Científicos de la computación también pueden proporcionar esta característica en sus biomarcadores algoritmos de detección.
Resumen
Detección de biomarcadores es una de las cuestiones biomédicas más importantes para investigadores de alto rendimiento "ómicas", y casi todos los algoritmos de detección de biomarcadores generan un subconjunto de biomarcadores con la medición del desempeño optimizado para un determinado conjunto de datos . Sin embargo, un reciente estudio demostró la existencia de varios subconjuntos de biomarcadores con actuaciones de clasificación igualmente efectivos o incluso idénticos. Este protocolo presenta una metodología simple y directa para la detección de subconjuntos del biomarcador con las actuaciones de clasificación binario, mejores que un corte definido por el usuario. El protocolo consiste en preparación de datos y carga, Resumen de información de línea de base, parámetro ajuste, detección de biomarcadores, visualización resultado interpretación, anotaciones de genes biomarcadores y exportación de resultados y visualización en calidad de la publicación. El biomarcador propuesto evaluación estrategia es intuitivo y muestra una regla general para el desarrollo de algoritmos de detección de biomarcadores. Una interfaz de usuario gráfica fácil de usar (GUI) fue desarrollada usando el lenguaje de programación Python, permitiendo a los investigadores biomédicos tienen acceso directo a sus resultados. Pueden descargarse el código fuente y manual de kSolutionVis de http://www.healthinformaticslab.org/supp/resources.php.
Introducción
Clasificación binaria, uno de los más comúnmente investigada y datos difíciles problemas en el área biomédico, de la explotación minera se utiliza para construir un modelo de clasificación en dos grupos de muestras con la más precisa discriminación poder1, 2 , 3 , 4 , 5 , 6 , 7. sin embargo, los grandes datos generados en el ámbito biomédico tienen la inherente "p pequeño n grande" paradigma, con el número de características generalmente mucho mayores que el número de muestras6,8,9. Por lo tanto, los investigadores biomédicos tienen que reducir la dimensión de función antes de utilizar los algoritmos de clasificación para evitar el problema overfitting8,9. Biomarcadores de diagnóstico se definen como un subconjunto de características detectados separar a pacientes de una determinada enfermedad de control sano muestras10,11. Los pacientes generalmente se definen como las muestras positivas, y los controles sanos se definen como muestras negativas12.
Estudios recientes han sugerido que existe más de una solución con las actuaciones de clasificación idéntico o igualmente efectiva para un conjunto de datos biomédica5. Casi todos los algoritmos de selección de función son algoritmos deterministas, produciendo una única solución para el mismo conjunto de datos. Algoritmos genéticos simultáneamente pueden generar múltiples soluciones con actuaciones similares, pero aún así tratar de seleccionar una solución con la mejor función de la aptitud como la salida para un conjunto dado de datos13,14.
Algoritmos de selección de función se pueden agrupar áspero como filtros o envolturas12. Un algoritmo de filtro elige el top -k características por su importante asociación individual con las etiquetas de clase binaria basada en la suposición de que dispone son independientes de uno a15,16,17 . Aunque esta suposición no tiene verdadera para casi todos datos del mundo real, la regla de filtro heurístico realiza bien en muchos casos, por ejemplo, mRMR (redundancia mínima y máxima relevancia) algoritmo, el Wilcoxon test basado en función filtrado (WRank) algoritmo de filtrado (ROCRank) basado en algoritmo y el diagrama ROC (característica operativa del receptor). mRMR, es un algoritmo de filtro eficiente porque aproxima el problema de combinatoria de estimación con una serie de problemas mucho más pequeños, en comparación con el algoritmo de selección de función de máxima dependencia, cada uno de los cuales sólo involucra dos variables, y por lo tanto utiliza pares probabilidades conjuntas que son más robustas de18,19. Sin embargo, mRMR puede subestimar la utilidad de algunas de las características que no mide las interacciones entre las características que pueden aumentar la relevancia y así pierde algunas combinaciones de funciones que sirven individualmente pero que son útiles sólo cuando se combinan. El algoritmo WRank calcula una puntuación no paramétrica de forma discriminativa una característica entre dos clases de muestras y es conocida por su robustez para afloramientos20,21. Además, el algoritmo de ROCRank evalúa cómo importante es el área bajo ROC la curva (AUC) de una función concreta para la clasificación binaria investigados rendimiento22,23.
Por otro lado, un contenedor evalúa el rendimiento del clasificador previamente definida de un subconjunto de la característica dada, generado iterativamente una regla heurística y crea el subconjunto de la característica con el mejor rendimiento medida24. Un contenedor generalmente supera a un filtro en el rendimiento de la clasificación pero corre lento25. Por ejemplo, el algoritmo de27 26,de bosque al azar regularizado (RRF) utiliza una regla codiciosa, evaluando las características en un subconjunto de los datos del entrenamiento en cada nodo del bosque al azar, y los puntos función importancia son evaluados por el índice de Gini . La elección de una nueva característica se penalizará si no mejora la ganancia de información de las características solicitadas. Además, el análisis de predicción de Microarrays (PAM)28,29 algoritmo, también un algoritmo de envoltura, calcula un centroide para cada una de las etiquetas de clase y luego selecciona características para reducir el tamaño los centroides gen hacia el general centroide de la clase. PAM es robusto para las características.
Soluciones múltiples con el rendimiento de la clasificación superior pueden ser necesarias para cualquier conjunto dado de datos. En primer lugar, el objetivo de la optimización de un algoritmo determinista es definido por una fórmula matemática, por ejemplo, tasa de error mínimo30, que no es necesariamente ideal para muestras biológicas. En segundo lugar, un conjunto de datos puede tener soluciones significativamente diferentes, múltiples, con actuaciones similares de efectivas o incluso idénticos. Casi todos los algoritmos de selección existentes de la característica de estas soluciones seleccionará al azar la salida31.
Este estudio presenta un protocolo analítico de informática para la generación de múltiples soluciones de selección de función con actuaciones similares para cualquier conjunto de datos de clasificación binario dado. Teniendo en cuenta que investigadores biomédicos más no están familiarizados con las técnicas informáticas o codificación de la computadora, una interfaz de usuario gráfica fácil de usar (GUI) fue desarrollada para facilitar el rápido análisis de datos biomédicos clasificación binaria. El protocolo analítico consiste en carga de datos y resumir, parámetro tuning, ejecución de ductos e interpretaciones del resultado. Con un simple clic, el investigador es capaz de generar el subconjuntos de biomarcadores y de la parcelas de visualización de calidad de publicación. El protocolo ha sido probado utilizando los transcriptomas de dos conjuntos de clasificación binaria de la leucemia linfoblástica aguda (ALL), es decir, ALL1 y ALL212. Los conjuntos de datos de ALL1 y ALL2 se descargaron desde el amplio Instituto genoma análisis centro de datos, disponible en http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi. ALL1 contiene 128 muestras con 12.625 características. De estas muestras, 95 son células B todo y 33 son células T todos. ALL2 incluye 100 muestras con 12.625 características así. De estas muestras, hay 65 pacientes que sufrieron recaída y 35 pacientes que no lo hizo. ALL1 era un conjunto de datos de fácil clasificación binaria, con una precisión mínima de cuatro filtros y cuatro envolturas que 96.7% y 6 de los algoritmos de selección de 8 función logro 100%12. ALL2 fue un conjunto de datos más difícil, con los algoritmos de selección 8 característica anterior logrando no es mejor que el 83,7% exactitud12. Esta mayor precisión se logró con 56 características detectadas por el algoritmo de la envoltura, selección basada en la correlación de función (SFC).
Protocolo
Nota: El siguiente protocolo describe los detalles del procedimiento analítico informática y seudo códigos de los módulos principales. El sistema de análisis automático se desarrolló usando Python versión 3.6.0 y los pandas de módulos Python, abc, numpy, scipy, sklearn, sys, PyQt5, sys, mRMR, matemáticas y matplotlib. Los materiales utilizados en este estudio se enumeran en la Tabla de materiales.
1. preparar la matriz de datos y etiquetas de la clase
- Preparar el archivo de la matriz de datos como un archivo delimitado por TABULACIONES o comas de la matriz, como se ilustra en la figura 1A.
Nota: Cada fila tiene todos los valores de una función, y el primer elemento es el nombre de función. Una función es un identificador de probeset del conjunto de datos basados en microarrays transcriptoma o puede ser otro valor ID como un residuo de cisteína con su valor de metilación en un conjunto de datos de methylomic. Cada columna da los valores de característica de una muestra, con el primer elemento es el nombre de la muestra. Una fila se separa en columnas por una ficha (figura 1B) o una coma (figura 1). Un archivo delimitado por TABULADORES de la matriz es reconocido por el archivo extensión .tsv, y un archivo delimitado por comas matriz tiene la extensión .csv. Este archivo puede ser generado por salvar una matriz como el formato .tsv o .csv de software como Microsoft Excel. La matriz de datos también puede ser generada por computadora codificación. - Preparar el archivo de la etiqueta de clase como un delimitado por TABULACIONES o por comas matriz archivo (figura 1), similar al archivo de la matriz de datos.
Nota: La primera columna da el nombre de la muestra, y la etiqueta de clase de cada muestra se da en la columna titulada clase. Compatibilidad máxima se considera en el proceso de codificación, por lo que se pueden agregar columnas adicionales. El archivo de la etiqueta de clase puede ser formateado como un archivo .tsv o CSV. Los nombres en la columna de clase pueden ser cualquiera de los términos, y puede haber más de dos clases de muestras. El usuario puede elegir dos de las clases para el siguiente análisis.
2. cargar la matriz de datos y etiquetas de la clase
- Cargar las etiquetas de clase y matriz de datos en el software. Haga clic en el botón de matriz de datos de carga para seleccionar el archivo de la matriz de datos especificado por el usuario. Haga clic en el botón etiquetas de clase de carga para seleccionar el archivo de la etiqueta de clase correspondiente.
Nota: Después de que ambos archivos se cargan, kSolutionVis a cabo una rutina pantalla de compatibilidad entre los dos archivos. - Resumir las características y las muestras del archivo de la matriz de datos. Estimar el tamaño del matriz del archivo de datos.
- Resumir las clases en el archivo de la etiqueta de clase y las muestras. Estimar el tamaño del archivo de la etiqueta de clase.
- Comprobar si cada muestra de la matriz de datos tiene una etiqueta de clase. Resumen de los números de las muestras con las etiquetas de clase.
3. resume y muestra las estadísticas de la base del conjunto de datos
- Clic en resumir, sin cualquier palabra clave especificado de entrada, y el software mostrará 20 características indexadas y los nombres de características correspondiente.
Nota: Los usuarios deben especificar el nombre de la función que desea encontrar para ver sus estadísticas base y la correspondiente distribución de valor entre todas las muestras de entrada. - Proporcionar una palabra clave, por ejemplo, "1000_at", en el cuadro de texto función para encontrar una característica específica que se resumirá. Clic en resumir para obtener las estadísticas de base para esta función dada.
Nota: La palabra clave puede aparecer en cualquier lugar los nombres de función objetivo, facilitando el proceso de búsqueda para los usuarios. - Clic en resumir para encontrar más de una función con la palabra clave determinada y luego especifique el ID de característica única para proceder con el paso anterior de resumir una característica particular.
4. determinar las etiquetas de clase y el número de mejores características
- Elegir los nombres de clases negativa ("N (95)") y positivo ("P (33)") en los cuadros de lista desplegable Clase positivo y Negativo de clase, como se muestra en la figura 2 (medio).
Nota: Se sugiere elegir que una clasificación binario equilibrado conjunto de datos, es decir, la diferencia entre el número de muestras positivas y negativas es mínima. El número de muestras también se da entre paréntesis después del nombre de cada etiqueta de clase en los dos cuadros de lista desplegable. - Elegir 10 como el número de mejores características (parámetro pTopX) en el menú desplegable Top_X (?) para una pantalla completa de la característica-subconjunto.
Nota: El software automáticamente alinea todas las características de la P-valor calculado de una prueba de t de cada característica comparando las clases positivas y negativas. Una característica una menor P-valor tiene un poder discriminar mejor entre las dos clases de muestras. El módulo de proyección global es computacionalmente intensivo. El parámetro pTopX es de 10 por defecto. Los usuarios pueden cambiar este parámetro en el rango de 10 a 50, hasta que encuentren satisfacción tienen subconjuntos con las actuaciones de buena clasificación.
5. ajustar parámetros del sistema para diferentes espectáculos
- Elegir la medición del desempeño (pMeasurement) exactitud (Acc) en el menú desplegable Acc/bAcc (?) para el clasificador seleccionado máquina de aprendizaje extrema (ELM). Otra opción de este parámetro es la medida de precisión de equilibrado (bAcc).
Nota: TP, FN, TN, y FP el número de verdaderos positivos, falsos negativos, verdaderos negativos y falsos positivos, respectivamente. La medición Acc se define como (TP+TN)/(TP+FN+TN+FP), que funciona mejor en un conjunto equilibrado de datos6. Pero un clasificador optimizado para Acc tiende a asignar todas las muestras a la clase negativa si el número de muestras negativas es mucho mayor que la de los positivos. El bAcc se define como (Sn + Sp) / 2, donde Sn = TP/(TP+FN) y Sp = TN/(TN+FP) son las tarifas correctamente predichas para el positivo y negativo de las muestras, respectivamente. Por lo tanto, bAcc normaliza las actuaciones de predicción sobre las dos clases y puede conducir a un funcionamiento equilibrado de la predicción sobre dos clases no balanceadas. ACC es la opción por defecto de pMeasurement. El software utiliza el clasificador de olmo por defecto para calcular los rendimientos de la clasificación. El usuario también puede elegir un clasificador de SVM (máquina de vectores soporte), KNN (k vecino más cercano), árbol de decisión o Naïve Bayes. - Elija el valor de corte 0.70 (parámetro pCutoff) para la medición de rendimiento especificado en el cuadro de pCutoff:.
Nota: Acc y bAcc oscilan entre 0 y 1, tanto el usuario puede especificar un valor pCutoff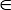 [0, 1] como el atajo para mostrar las soluciones combinadas. El software lleva a cabo un amplio subconjunto de característica proyección, y una adecuada selección de pCutoff hará la visualización 3D más intuitivo y explícito. El valor predeterminado para pCutoff es 0.70.
[0, 1] como el atajo para mostrar las soluciones combinadas. El software lleva a cabo un amplio subconjunto de característica proyección, y una adecuada selección de pCutoff hará la visualización 3D más intuitivo y explícito. El valor predeterminado para pCutoff es 0.70.
6. Haga funcionar la tubería y los resultados visualizados interactivo
- Haga clic en el botón analizar para correr la tubería y generar las parcelas de visualización, como se muestra en la figura 2 (parte inferior).
Nota: La tabla izquierda da todos los subconjuntos de la función y sus pMeasurement calculado por la estrategia de validación cruzada 10 veces del clasificador olmo, como se describió anteriormente5. Para el procedimiento de proyección característica subconjunto con los ajustes actuales se generan dos diagramas de dispersión 3D y parcelas de dos líneas. - Elegir 0.70 como el valor predeterminado de la pMeasurement corte (parámetro piCutoff, cuadro de entrada de valor) y 10 como el valor predeterminado del número de mejores subconjuntos de la función (parámetro piFSNum).
Nota: La tubería se realiza mediante los parámetros pTopX, pMeasurement y pCutoff. La función detectada subconjuntos pueden ser más evaluados usando el corte piCutoff, sin embargo piCutoff no puede ser menor que pCutoff. Por lo tanto, piCutoff se inicializa como pCutoff y a visualizar sólo los subconjuntos de la característica con la medición de desempeño ≥ piCutoff . El valor predeterminado de piCutoff es pCutoff. A veces kSolutionVis detecta muchas soluciones y sólo el mejor piFSNum (predeterminado: 10) serán visualizar subconjuntos de la función. Si el número de subconjuntos de característica detectada por el software es menor que piFSNum, todos los subconjuntos de la función se visualiza. - Recoger e interpretar las características detectadas por el software, como se muestra en la figura 3.
Nota: La tabla en el cuadro de la izquierda muestra los subconjuntos característica detectada y sus mediciones de desempeño. Los nombres de las tres primeras columnas son "F1" "F2" y "F3". Las tres características de cada subconjunto de la característica se dan en su orden de clasificación en una fila (F1 < F2 < F3). La última columna da la medición del desempeño (Acc o bAcc) de cada subconjunto de la característica, y su nombre de columna (Acc o bAcc) es el valor de pMeasurement.
7. interpretar la dispersión 3D parcelas-visualizar e interpretar los subconjuntos de la función con las actuaciones de clasificación binario semejantemente eficaz mediante diagramas de dispersión 3D
- Haga clic en el botón analizar para generar la trama de dispersión 3D de los subconjuntos de la función superior de 10 con las mejores actuaciones de clasificación (Acc o bAcc) detectada por el software, como se muestra en la figura 3 (caja media). Ordenar las tres características en un subconjunto de la característica en ascendente de sus filas y utilizar las filas de las tres características como los ejes F1/F2/F3, es decir, F1 < F2 < F3.
Nota: El color de un punto representa el rendimiento de la clasificación binaria del correspondiente subconjunto de la característica. Un conjunto de datos puede tener múltiples subconjuntos de la función con semejantemente las mediciones de desempeño eficaz. Por lo tanto, es necesario un diagrama de dispersión interactivo y simplificada. - Cambie el valor 0,70 en la caja pCutoff: y haga clic en el botón analizar para generar el diagrama de dispersión 3D de los subconjuntos de la característica con la medición de desempeño ≥ piCutoff, como se ve en la figura 3 (cuadro derecho). Haga clic en el botón 3D tuning para abrir una nueva ventana para sintonizar manualmente los ángulos de visión de la trama de dispersión 3D.
Nota: Cada subconjunto de la característica está representada por un punto en la misma forma que anteriormente. El diagrama de dispersión 3D se generó en el ángulo por defecto. Para facilitar la visualización en 3D y sintonía, una ventana separada se abrirá haciendo clic en el botón 3D tuning. - Haga clic en el botón reducir para reducir la redundancia de los subconjuntos de la función detectado.
Nota: Si desean que los usuarios seleccionar a los tríos de la función y minimizar la redundancia de los subconjuntos de la función, el software también proporciona esta función mediante el algoritmo de selección de función de mRMR. Después de hacer clic en el botón reducir , kSolutionVis quitará esas características redundantes en los tríos de característica y regenerar la tabla y los dos scatter diagramas antes mencionados. Las características quitadas de los tríos de característica se reemplazará por la palabra clave en la tabla. Los valores de ninguno en el eje de F1/F2/F3 serán denotados como el valor de piFSNum (el rango del valor normal de F1/F2/F3 es [1, top_x]). Por lo tanto, los puntos que incluyen un valor ninguno parece ser parcelas puntos "outlier" en 3D. Las parcelas 3D manualmente ajustables pueden encontrarse en "Sintonización Manual de los diagramas de punto 3D" en el material complementario.
8. encontrar anotaciones de genes y sus asociaciones con enfermedades humanas
Nota: Pasos 8 a 10 muestran cómo anotar un gen desde el nivel de secuencia de DNA y proteínas. En primer lugar, el símbolo del gen de cada identificación de biomarcadores de los pasos anteriores se recuperará de la base de datos de DAVID32, y luego dos servidores web representativa se utilizará para analizar este símbolo del gene de los niveles de DNA y proteínas, respectivamente. El servidor GeneCard proporciona una amplia anotación funcional de un símbolo determinado gen, y la herencia mendeliana en línea en base de datos de hombre (OMIM) ofrece la más completa conservación de asociaciones del gen de la enfermedad. El servidor UniProtKB es una de la más completa base de datos de la proteína, y el servidor de sistema de predicción basado en grupo (GPS) predice la señalización fosforilación una lista muy grande de las quinasas.
- Copiar y pegar el enlace de la base de datos de DAVID en un navegador web y abrir la página web de esta base de datos. La función de IDs 38319_at/38147_at/33238_at del primer biomarcador subconjunto del conjunto de datos ALL1 (Figura 4B) de entrada y haga clic en el enlace Gene ID conversión vista en la Figura 4A . Haga clic en el enlace Lista de Gene y haga clic en Presentar lista como se muestra en la Figura 4B. Recuperar las anotaciones de interés y haga clic en Mostrar lista de gen (figura 4). Obtener la lista de los símbolos del gene (figura 4).
Nota: Los símbolos del gene obtenidos aquí se utilizará para más anotaciones funcionales en los siguientes pasos. - Copiar y pegar el enlace de la base de datos de tarjetas de Gene en un navegador web y abrir la página web de esta base de datos. Buscar nombre de un gen CD3D en el cuadro de entrada de consulta de base de datos y encontrar las anotaciones de este gene del Gene tarjetas33,34, como se muestra en la tabla 1 y figura 5A.
Nota: Tarjetas de Gene es una base de conocimientos gene integral, proporcionando nomenclatura, genómica, proteómica, localización subcelular y vías involucradas y otros módulos funcionales. También proporciona enlaces externos a varias otras bases de datos biomédicas como PDB/PDB_REDO35, Entrez Gene36, OMIM37y38de UniProtKB. Si el nombre de función no es un símbolo del gen estándar, utilizar la base de datos de ENSEMBL para convertirlo en39. CD3D es el nombre del gene de la célula de T del Receptor T3 Delta cadena. - Copiar y pegar el enlace de la base de datos OMIM en un navegador web y abrir la página web de esta base de datos. Buscar nombre de un gen CD3D y encontrar las anotaciones de este gen de la base de datos OMIM37, como se muestra en la tabla 1 y la figura 5B.
Nota: OMIM sirve ahora como una de las fuentes más completa y autorizadas de las conexiones del gene humano con enfermedades hereditarias. OMIM fue iniciado por el Dr. Victor A. McKusick catalogar las mutaciones genéticas asociadas a enfermedad40. OMIM cubre ahora más de 15.000 genes humanos y más de 8.500 fenotipos, a partir de diciembre 1st 2017.
9. anotar las proteínas codificadas y las modificaciones post-traduccionales
- Copiar y pegar el enlace de la base de datos UniProtKB en un navegador web y abrir la página web de esta base de datos. Buscar nombre de un gen CD3D en el cuadro de consulta de UniProtKB y encontrar las anotaciones de este gen de la base de datos38, como se muestra en la tabla 1 y figura 5.
Nota: UniProtKB recoge una rica fuente de anotaciones para las proteínas, incluyendo la nomenclatura y la información funcional. Esta base de datos también proporciona enlaces externos a otras bases de datos ampliamente utilizados, incluyendo PDB/PDB_REDO35, OMIM37y41de Pfam. - Copiar y pegar el enlace del servidor web GPS en un navegador web y abrir la página web de este servidor web. Recuperar la secuencia de la proteína codificada por el gen CD3D del biomarcador de la base de datos de UniProtKB38 y predecir residuos de modificación poste-de translación (PTM) de la proteína usando la herramienta de GPS, como se muestra en la tabla 1 y figura 5.
Nota: Un sistema biológico es dinámico y complicado, y las bases de datos recogen sólo información. Por lo tanto, herramientas en línea de predicción biomédica así como programas sin conexión pueden proporcionar la evidencia útil para complementar un mecanismo hipotético. GPS ha sido desarrollado y mejorado para más de 12 años7,42 y puede utilizarse para predecir residuos PTM de la proteína en un determinado péptido secuencia43,44. Herramientas también están disponibles para diversos temas de investigación, incluyendo la predicción de la localización subcelular45 y transcripción factor vinculante motivos 46 entre otros de una proteína.
10. anotar las interacciones proteína-proteína y sus módulos funcionales enriquecidos
- Copiar y pegar el enlace del servidor web cadena en un navegador web y abrir la página web de este servidor web. Buscar la lista para los genes CD3D y P53 y encontrar sus propiedades orquestadas usando la base de datos de cadena47. El mismo procedimiento puede realizarse utilizando otro servidor web, DAVID32.
Nota: Además de las mencionadas anotaciones de genes individuales, existen muchas herramientas de informática a gran escala investigar las propiedades de un grupo de genes. Un estudio reciente demostraron que genes marcadores individualmente mal podrían constituir un sistema gene mejorada5. Por lo tanto, vale la pena el coste computacional para detectar biomarcadores más complicados. La base de datos de cadena puede visualizar las conexiones de interacción conocidas o previstas, y el servidor de David puede detectar los módulos funcionales con fenotipo-asociaciones significativas en los genes consultado47,32. También hay varias otras herramientas de análisis de informática a gran escala.
11. exportación de los subconjuntos de biomarcadores generados y las parcelas de visualización
- Exportación de los subconjuntos de biomarcadores detectado como archivo de texto .tsv o CSV para su posterior análisis. Haga clic en el botón exportar la tabla de debajo de la mesa de todos los subconjuntos de biomarcadores detectados y elegir que formato de texto guardar como.
- Las parcelas de la visualización de la exportación como un archivo de imagen. Haga clic en el botón Guardar en cada parcela y elija formato de imagen de guardar como.
Nota: El software soporta el pixel formato .png y el vector formato .svg. Las imágenes de píxeles son buenas para la visualización en la pantalla del ordenador, mientras que las imágenes vectoriales se pueden convertir a cualquier resolución para fines de publicación de revista.
Resultados
El objetivo de este flujo de trabajo (figura 6) es detectar múltiples subconjuntos de biomarcadores con similar eficiencia de un conjunto de datos binarios de la clasificación. Todo el proceso es ilustrado por dos conjuntos de datos de ejemplo ALL1 y ALL2 extraído de una detección de biomarcadores recientemente publicado estudio12,48. Un usuario puede instalar kSolutionVis siguiendo las instruccio...
Discusión
Este estudio presenta un protocolo de detección y caracterización de fácil de seguir solución múltiples biomarcadores para un conjunto de datos de clasificación binario especificado por el usuario. El programa pone énfasis en la facilidad de uso e interfaces flexibles de importación y exportación de varios formatos de archivo, permitiendo que un investigador biomédico investigar sus datos fácilmente utilizando la interfaz gráfica del software. Este estudio también pone de relieve la necesidad de generar más...
Divulgaciones
No tenemos conflictos de interés relacionados con este informe.
Agradecimientos
Este trabajo fue apoyado por el programa de investigación prioridad estratégica de la Academia China de Ciencias (XDB13040400) y la subvención de puesta en marcha de la Universidad de Jilin. Revisores anónimos y usuarios pruebas biomédicos fueron apreciados por sus comentarios constructivos para mejorar la usabilidad y la funcionalidad de kSolutionVis.
Materiales
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
Referencias
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados