Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Diseño y síntesis de un ADN Reconfigurable acordeón Rack
* Estos autores han contribuido por igual
En este artículo
Resumen
Describimos el protocolo detallado para el diseño, simulación, wet-lab experimentos y análisis para un rack de acordeón ADN reconfigurable de mallas de 6 por 6.
Resumen
Sistemas mecánicos basados en nanoestructuras de ADN o DNA nanomáquinas, que producen el movimiento de nanoescala complejas en 2D y 3D en el nanómetro a ångström resolución, muestran gran potencial en diversos campos de la nanotecnología como los reactores moleculares, administración de fármacos, y sistemas de nanoplasmonic. El rack acordeón ADN reconfigurable, que colectivamente se puede manipular una red de nanoescala 2D o 3D de elementos, en múltiples etapas en respuesta a las entradas de ADN, se describe. La plataforma tiene potencial para aumentar el número de elementos que pueden controlar nanomáquinas de ADN de algunos elementos a una escala de red con múltiples etapas de reconfiguración.
En este protocolo se describe todo el proceso experimental de la rejilla de acordeón ADN reconfigurable de mallas de 6 por 6. El protocolo incluye un procedimiento de regla y simulación de diseño de las estructuras y un experimento de wet-lab para la síntesis y reconfiguración. Además, el análisis de la estructura utilizando met (microscopía electrónica de transmisión) y traste (transferencia de energía de resonancia de la fluorescencia) está incluido en el protocolo. Los nuevos métodos de diseño y simulación en este protocolo ayudará a los investigadores utilizar el canasto de acordeón de ADN para más aplicaciones.
Introducción
Sistemas mecánicos basados en nanoestructuras de ADN o DNA nanomáquinas en1,2,3,4,5 son únicos ya que producen movimiento de nanoescala complejas en 2D y 3D en el nanómetro a Ångström resolución, según varias biomoléculas estímulos2,3,6. Colocando materiales funcionales de estas estructuras y control de sus posiciones, estas estructuras pueden aplicarse a varias áreas. Por ejemplo, nanomáquinas de ADN se han propuesto para un reactor molecular7drogas entrega8y nanoplasmonic sistemas9,10.
Anteriormente, hemos introducido la rejilla acordeón del ADN reconfigurable, que puede manipular una red de nanoescala 2D o 3D de elementos11 (figura 1A). A diferencia de otros ADN nanomáquinas que sólo controlan una serie de elementos, la plataforma puede manipular colectivamente periódicamente vestida de elementos 2D o 3D en diferentes etapas. Anticipamos que una red de reacción química y biológica programable o un sistema de computación molecular se puede construir de nuestro sistema, aumentando el número de elementos controlables. La parrilla de acordeón de ADN es una estructura, en que la red de múltiples vigas de ADN está conectada a las articulaciones compuestas de ADN (figura 1B). Se reconfigura el acordeón rack generado por las vigas de ADN por las cerraduras de la DNA, que hibridan a la parte pegajosa de vigas y cambian el ángulo entre los rayos según la longitud de la parte puente de las esclusas (estado bloqueado). Además, multi-step reconfiguración se demuestra mediante la adición de nuevas cerraduras después de la formación del estado libre por desmontar cerraduras de ADN a través de la cadena basada en el punto de apoyo desplazamiento12,13.
En este protocolo se describe todo el proceso de diseño y síntesis de la rejilla acordeón reconfigurable de ADN. El protocolo incluye el diseño, simulación, wet-lab experimentos y análisis para la síntesis del ADN acordeón de 6 por 6 mallas y una reconfiguración de estos. La estructura de cubierta en el protocolo es el modelo básico de la investigación anterior11 y 65 nm por 65 nanómetro de tamaño, que consta de 14 vigas. En términos de diseño y simulación, el diseño estructural del soporte acordeón es diferente de ADN convencional14,de origami de la15 (es decir, firmemente embalado). Por lo tanto, se han modificado las reglas de diseño y simulación molecular de los métodos tradicionales. Para demostrar, mostramos la técnica de diseño utilizando el enfoque modificado de caDNAno14 y la simulación de la rejilla de acordeón con oxDNA16,17 de scripts adicionales. Por último, se describen ambos protocolos de TEM y traste para análisis de estructuras de rack acordeón configurado.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. diseño de 6 por 6 estante de acordeón de ADN con caDNAno14
- Descargar e instalar caDNAno 2.0 software14 para el diseño de un rack de acordeón de ADN (caDNAno 2.5 también está disponible en https://github.com/cadnano/cadnano2.5). Abrir caDNAno14 y haga clic en la Herramienta de cuadrado para añadir una nueva pieza con un enrejado de cuadrados.
- Número de cada viga de la parrilla de acordeón y dibujar en el panel izquierdo del enrejado del caDNAno14 (figura 2).
- Haga clic en la herramienta lápiz y dibujar cada haz en el panel derecho modificar el caDNAno14. Rotura de vigas cada 32 bp, que está para las uniones entre vigas adyacentes. Colocar grapas cruces en la misma posición de las articulaciones. Utilice la Herramienta lápiz y la Herramienta insertar en caDNAno14 a las articulaciones adicionales monocatenario crossovers.
- Haga clic en la Herramienta lápiz y conecte las articulaciones. Cada rayo tiene siete articulaciones.
- Generar cruces de andamio para combinar los andamios en el circuito simple utilizando el andamio previamente divulgado enrutamiento algoritmo11. No deje que el dominio obligatorio mínimo entre los filamentos de andamio y grapa de menos de 8 bp (figura 3).
- Colocar los andamios que no se utilizan en la Asamblea en los vértices situados en lados opuestos de la rejilla del acordeón, como se muestra en la figura 3.
- Haga clic en la herramienta de la rotura. Romper las cadenas donde hebras discontinuas son circulares o más de 60 bp.
- Diseño de los cerradura filamentos de la DNA.
- Haga clic en la herramienta de la rotura. Rotura 8 bp de una región de ADN básica para hacer una parte adhesiva y eliminar 8 bp de una región de ADN básica. Hay 18 partes pegajosas (figura 1) en el estante de acordeón de 6 por 6.
- Colocar secuencias inversas complementario a las partes pegajosas en ambos extremos de los hilos de la cerradura y conectarlos por una región puente, que consiste en filamentos de polietileno T de la longitud deseada (figura 1B).
- Para la reconfiguración, añadir 8 bp de las secuencias de punto de apoyo en el extremo de la DNA de cerraduras para el desplazamiento del filamento. La secuencia del punto de apoyo utilizada es en la tabla 2.
- Preparación de hilos de poli A que son inversa complementaria a la región de puente.
- Filamentos de diseño que son inversa complementaria a las cerraduras de la DNA para el experimento de reconfiguración.
- Haga clic en la Herramienta de secuencia y haga clic en ADN de andamio. Elegir el andamio estándar M13mp18. Haga clic en la Herramienta de exportar y guardar secuencia en formato csv (tabla 1).
2. simular la estructura con el oxDNA
- Descargar e instalar el oxDNA16,17. El último código fuente está disponible en https://sourceforge.net/projects/oxdna/files/.
- Hacer a partir los archivos de configuración desde el archivo14 de caDNAno usando python script 'cadnano_interface.py', que se proporciona en el paquete de17 oxDNA16,. El uso es el siguiente: 'cadnano_interface.py python cadnano_file.json sq'. Ahora se generan la topología archivo y el archivo de configuración.
Nota: El archivo de topología incluye cuántas hebras y nucleótidos en la estructura e información sobre bonos de la columna vertebral-columna vertebral entre los nucleótidos. El archivo de configuración incluye información general como rebotes, energía y tamaño de la caja. Información de orientación como el vector posición, vector de la base de la columna vertebral, vector normal, velocidad y velocidad angular de nucleótidos también está incluido (figura 4). - Cambiar la información en el archivo de configuración y topología de los caDNAno14 para reflejar la información estructural verdadera de la rejilla de acordeón. Los haces están dispuestos en paralelo cuando se visualizan los archivos de configuración y topología de caDNAno14 . Sin embargo, el acordeón rack es una estructura de enrejado por lo que la distancia entre los nucleótidos de servidumbre son ahora para la simulación (figura 5).
- Gire y mueva cada viga a la estructura deseada. Las nueve columnas de la izquierda en el archivo de configuración están el vector posición, vector de la base de la columna vertebral y vector normal (figura 4). Para girar de un rayo, rotar de posición, vectores base de la columna vertebral y normales usando transformación de rotación. Luego mover una viga cambiando el vector de posición para ubicar como se muestra en la figura 5.
- Relajar la estructura usando el script en el paquete de oxDNA (ver ejemplo en $oxDNA/ejemplos/RELAX_INITIAL_CONFIGURATION para más información).
- Ejecutar simulación de dinámica molecular para pasos 10 millones mediante el archivo de configuración relajada. El uso es como sigue: '. / oxDNA < input >' guardar los datos cada 5000 o 10000 pasos.
- Visualización
Nota: Las estructuras se visualizaron mediante cogli.- Descargar e instalar la última versión de la cogli (https://sourceforge.net/projects/cogli1/).
- Ejecute la cogli con los archivos de configuración y topología de la simulación de oxDNA. El uso es como sigue: '. / cogli1 -t < archivo topología >< archivo de configuración >'.
- Esconder la caja pulsando b.
3. síntesis de la estructura
Nota: El método de síntesis está adaptado desde el anterior protocolo15,18.
- Comprar las grapas de ADN diseñadas de un proveedor de oligonucleótidos.
- Ajustar la concentración de estas grapas de ADN a 100 μM con agua libre de nucleasa.
- Cada filamento de la DNA que constituye una estructura de 'estado libre' en un tubo de la piscina y ajustar la concentración a 2 μM de cada filamento.
- Piscina DNA filamentos de longitud y número de sitios de bloqueo en los tubos y ajustar la concentración a 2 μM de cada filamento. 18, 9 y 4 sitios de bloqueo se utilizan. Añadir hebras de A poli que son complementarias a la región de puente en la misma concentración.
- Filamentos de la piscina que son inversa complementaria al ADN cerradura filamentos de longitud en los tubos y ajustan la concentración a 2 μM de cada filamento.
- Preparar solución de MgCl2 de 300 nM mezclando 70 μL de agua libre de nucleasas y 30 μL de la solución de2 1 μM MgCl. Preparar un 5 x solución de Tris EDTA mezclando 95 μL de agua libre de nucleasas y 5 μL de la solución de Tris EDTA 100 x.
- Añadir 2 μL de la grapa ADN, 1.1 μL de solución de MgCl2 , 2 μL de solución de EDTA Tris, 7,6 μL de agua libre de nucleasas y 7.3 μL de DNA de andamio, de los cuales la concentración es de 110 nM a hacer 20 μL de acción mixta. Establecer la concentración final del andamio ADN a 40 nM, grapa ADN a 200 nM, MgCl2 a 16 mM y Tris EDTA solución 0,5 x.
- Rápidamente calentar la solución mixta en un termociclador a 80 ° C y enfriar a 60 ° C a razón de min 4 ° C y enfriar de 60 º C a 4 ° C a una velocidad de 40 min por ° C.
4. purificación de la estructura
Nota: Las muestras de todas las estructuras se purificaron antes del análisis. En esta sección, describimos el protocolo de purificación PEG, que es una adaptación de la literatura anterior19. La muestra también puede ser purificada por electroforesis en gel como se describe en la literatura anterior15,18.
- Preparar 5 M de NaCl y 100 x Tris-EDTA.
- Preparar precipitación-buffer mezclando 150 μL de PEG 8000, 500 μL de 100 x Tris EDTA y 101 μL de 5 M NaCl y 249 μL de agua libre de nucleasa.
- Preparar el búfer de destino mezclando 5.5 μL de solución 300 nM MgCl2 sección 3.3, 10 μL de solución de EDTA Tris x 5 de la sección 3.3 y 84,5 μL de agua libre de nucleasa.
- Mezcla 20 μL de la estructura sintetizada de la sección 3 y 20 μL de tampón de precipitación de la sección 4.2. Luego girar la acción mixta a 16000 x g a 4 ° C. Quite el sobrenadante y disolver el pellet en el buffer de destino de la sección 4.3.
5. reconfiguración del acordeón Rack de un «estado libre» a un 'estado bloqueado'
- Sintetizar la estructura sin candados de ADN para el experimento de configuración.
- Preparar ADN cerradura filamentos de sección 3.
- Añadir 2 μL de ADN cerradura de manera de la longitud deseada en 20 μL de la estructura sintetizada. Concentración de filamentos de la cerradura de DNA es cinco veces mayor que la estructura.
- Incubar la muestra de 0, 10, 25, 50 o 100 minutos para ver cómo rápida reconfiguración.
- Para la incubación de 100 minutos, incubar la muestra a 50 ° C durante 30 minutos y poco a poco enfriar hasta 25 ° C a una tasa de 0,33 ° C/minuto.
- Para la incubación de 50 minutos, incubar la muestra a 50 ° C durante 15 minutos y enfriar lentamente hasta 25 ° C a una velocidad de 0,66 º C/minuto.
- Para la incubación de 25 minutos, incubar la muestra a 50 ° C 7.5 minutos y poco a poco enfriar hasta 25 ° C a una velocidad de 1,32 ° C/minuto.
- Para la incubación de 10 minutos, incubar la muestra a 50 ° C durante 3 minutos y enfriar lentamente hasta 25 ° C a una velocidad de 3,3 ° C/minuto.
- Para la incubación de 0 minutos, guardar muestra a la derecha de 4 ° C después se agregan hebras de ADN cerraduras.
- Justo después del paso de atadura, enfriar rápidamente la muestra a 4 ° C para evitar la desnaturalización no deseada.
6. reconfiguración del Rack acordeón de un 'estado bloqueado' a un 'estado libre'
- Sintetizar la estructura con las cerraduras del ADN de la longitud deseada para el experimento de configuración.
- Preparar el inversos complementarios filamentos de sección 3.
- Añadir 2 μL de filamentos que son inversa complementaria a los hilos de la cerradura de la longitud deseada en 20 μL de la estructura sintetizada. Concentración de filamentos de la cerradura de DNA es cinco veces mayor que la estructura.
- Incubar la muestra de 0, 12, 60, 120, 240 minutos para ver cómo rápida reconfiguración.
- Para el 12, 60, 120, incubación de 240 minutos, calentar rápidamente la muestra a 40 ° C y enfriar lentamente hasta 20 ° C durante el tiempo correspondiente a cada uno. Justo después del paso de desprendimiento, enfriar rápidamente la muestra a 4 ° C para evitar la desnaturalización no deseada.
- Para la incubación de 0 minutos, guardar muestra a la derecha de 4 ° C después de reversas hebras complementarias se agregan.
7. proyección de imagen de TEM
Nota: Protocolo imagen TEM es una adaptación de la anterior literatura18,20.
- Preparar solución de NaOH de 1.25 M mezclando 87.5 μL de agua libre de nucleasas y 12,5 μL de solución de NaOH de 10 M.
- Añadir 1 μL de solución de NaOH de 1.25 M a 50 μL de la solución de formato de uranilo 2%.
- Vórtice de la solución durante 3 minutos y centrifugar a velocidad máxima durante 3 minutos. Depósito 3 μL de la muestra purificada en la rejilla TEM resplandor-descargado durante 3 minutos y lavar rápidamente con papel de filtro.
- Depósito 7 μL de solución de formato de uranilo preparado durante 30 segundos y Lave rápidamente con papel de filtro.
- Medir la longitud y el ángulo de la estructura de acordeón reflejada por TEM.
8. análisis del traste
- Utilizar Atto 550 y Atto 647N tinte, para que la distancia de Förster es 6.5 nm. Vuelva a colocar 58 grapa y grapa 117 en la tabla 1 con fluorescencia con filamentos. Entonces sintetizar la estructura con fluorescencia con hilos por el método descrito en la sección 3.
- Medir la concentración de la muestra purificada.
- Normalizar la muestra a 10 nM y carga 50 μL para las microplacas de 384 bien.
- Excitar la muestra con tintes donador y aceptor en 550 nm y medir el espectro de fluorescencia de 570 nm y 800 nm con un fluorómetro.
- Medir el espectro de fluorescencia de la muestra sólo donante de la misma manera.
- Excitar los tintes de la muestra de 650 nm y el espectro de fluorescencia y medida de 670 nm y 800 nm. Esto es para medir la concentración del aceptador.
- Obtener las desviaciones estándar, repitiendo el mismo experimento con muestras de tres, que se sintetizan y purificados por separado.
- Calcular la eficiencia de traste con la relación entre un método descrito por la ecuación a continuación21.
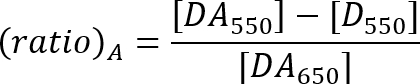
DA550: intensidad de la fluorescencia de pico del aceptador de la muestra con donador y aceptor en excitación nm 550.
D550: intensidad de la fluorescencia en el rango de emisión del aceptador de la donante sólo muestra excitación nm 550.
DA650: intensidad de la fluorescencia de pico del aceptador de la muestra con donador y aceptor en excitación nm 650.
Access restricted. Please log in or start a trial to view this content.
Resultados
La parrilla acordeón 6 diseñado por 6 ADN es simulada de la oxDNA16,17 y los resultados se muestran en la figura 6. El resultado de la simulación, se confirmó que la estructura prevista está formada sin distorsión de la estructura.
Las imágenes TEM en la figura 7 son de estructuras configuradas con una lo...
Access restricted. Please log in or start a trial to view this content.
Discusión
Este Protocolo introduce todo el proceso de diseño, simulación, síntesis y análisis de la rejilla acordeón de ADN 2D básica. El diseño modificado y las reglas de la simulación se han descrito porque la regla del diseño de origami de ADN estándar, difiere en cuanto a la rejilla de acordeón de ADN tiene nucleótidos adicionales en los crossovers para flexibilidad14,15. De esta forma, esperamos que el protocolo puede acelerar varias investigaciones utiliz...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar
Agradecimientos
Esta investigación fue parcialmente financiada por el programa Global del centro de desarrollo de investigación a través de la Fundación de investigación nacional de Korea(NRF) financiado por el Ministerio de ciencia y TIC (MSIT) (2015K1A4A3047345) y Nano· Programa de desarrollo de tecnología de materiales a través de la nacional investigación Fundación de Corea (NRF) financiado por el Ministerio de ciencia y TIC (MSIT) (2012M3A7A9671610). El Instituto de investigación de ingeniería en la Universidad Nacional de Seúl proporcionó instalaciones de investigación para este trabajo. Los autores reconocen con gratitud hacia Yoon Tae-Young (ciencias biológicas, Universidad Nacional de Seúl) en cuanto a la espectroscopia de fluorescencia para el análisis de traste.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| M13mp18 Single-stranded DNA | NEB | N4040s | |
| 1M MgCl2 Solution | Biosesang | M2001 | |
| Tris-EDTA buffer | Biosesang | T2142 | |
| Nuclease-Free Water | Qiagen | 129114 | |
| 5M Sodium Chloride solution | Biosesang | s2007 | |
| PEG 8000 | Sigma Aldrich | 1546605 | |
| 10N NaOH | Biosesang | S2038 | |
| Uranyl formate | Thomas Science | C993L42 | |
| Thermal cycler C1000 | Biorad | ||
| Nanodropic 2000 | Thermo Fisher Scientific | ||
| TEM (LIBRA 120) | Carl Zeiss | ||
| Fluorometer Enspire 2300 | Perkin-Elmer | ||
| Centrifuge | Labogene | LZ-1580 |
Referencias
- Andersen, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459 (7243), 73-76 (2009).
- Cha, T. -G., et al. Design principles of DNA enzyme based walkers: Translocation kinetics and photo-regulation. Journal of the American Chemical Society. 137 (29), 9429-9437 (2015).
- Gerling, T., Wagenbauer, K. F., Neuner, A. M., Dietz, H. Dynamic DNA devices and assemblies formed by shape-complementary, non-base pairing 3D components. Science. 347 (6229), 1446-1452 (2015).
- Pinheiro, A. V., Han, D., Shih, W. M., Yan, H. Challenges and opportunities for structural DNA nanotechnology. Nature nanotechnology. 6 (12), 763-772 (2011).
- Li, J., et al. Exploring the speed limit of toehold exchange with a cartwheeling DNA acrobat. Nature Nanotechnology. 1, (2018).
- Krishnan, Y., Simmel, F. C. Nucleic acid based molecular devices. Angewandte Chemie International Edition. 50 (14), 3124-3156 (2011).
- Liu, M., et al. A DNA tweezer-actuated enzyme nanoreactor. Nature communications. 4, 2127(2013).
- Douglas, S. M., Bachelet, I., Church, G. M. A logic-gated nanorobot for targeted transport of molecular payloads. Science. 335 (6070), New York, N.Y. 831-834 (2012).
- Kuzyk, A., et al. Reconfigurable 3D plasmonic metamolecules. Nature Materials. 13 (9), 862-866 (2014).
- Zhou, C., Duan, X., Liu, N. A plasmonic nanorod that walks on DNA origami. Nature communications. 6, 8102(2015).
- Choi, Y., Choi, H., Lee, A. C., Lee, H., Kwon, S. A Reconfigurable DNA Accordion Rack. Angewandte Chemie International Edition. 57 (11), 2811-2815 (2018).
- Chen, H., et al. Understanding the Mechanical Properties of DNA Origami Tiles and Controlling the Kinetics of their Folding and Unfolding Reconfiguration. Journal of the American Chemical Society. 136 (19), 6995-7005 (2014).
- Han, D., Pal, S., Liu, Y., Yan, H. Folding and cutting DNA into reconfigurable topological nanostructures. Nature Nanotechnology. 5 (10), 712-717 (2010).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Research. 37 (15), 5001-5006 (2009).
- Castro, C. E., et al. A primer to scaffolded DNA origami. Nature methods. 8 (3), 221-229 (2011).
- Ouldridge, T. E., Louis, A. A., Doye, J. P. K. DNA Nanotweezers Studied with a Coarse-Grained Model of DNA. Physical Review Letters. 104 (17), 178101(2010).
- Snodin, B. E. K., et al. Direct Simulation of the Self-Assembly of a Small DNA Origami. ACS Nano. 10 (2), 1724-1737 (2016).
- Amir, Y., Abu-Horowitz, A., Bachelet, I. Folding and Characterization of a Bio-responsive Robot from DNA Origami. Journal of Visualized Experiments. (106), e51272(2015).
- Stahl, E., Martin, T. G., Praetorius, F., Dietz, H. Facile and Scalable Preparation of Pure and Dense DNA Origami Solutions. Angewandte Chemie International Edition. 53 (47), 12735-12740 (2014).
- Wei, B., Vhudzijena, M. K., Robaszewski, J., Yin, P. Self-assembly of Complex Two-dimensional Shapes from Single-stranded DNA Tiles. Journal of Visualized Experiments. (99), e52486(2015).
- Clegg, R. M. Fluorescence resonance energy transfer and nucleic acids. Methods in enzymology. 211, 353-388 (1992).
- Kopperger, E., et al. A self-assembled nanoscale robotic arm controlled by electric fields. Science. 359 (6373), New York, N.Y. 296-301 (2018).
- Lauback, S., et al. Real-time magnetic actuation of DNA nanodevices via modular integration with stiff micro-levers. Nature Communications. 9 (1), 1446(2018).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados