Method Article
Explorar los efectos del vuelo espacial sobre fisiología del ratón utilizando la plataforma abierta de GeneLab de NASA de acceso
En este artículo
Resumen
La plataforma de la NASA GeneLab proporciona acceso sin restricciones a ómicas preciosos datos de experimentos biológicos vuelo espacial. Describimos cómo se realiza un experimento típico del ratón en el espacio y cómo se pueden acceder y analizar datos de dichos experimentos.
Resumen
Realizar experimentos biológicos en el espacio requiere adaptaciones especiales y procedimientos para asegurar que estas investigaciones se realizan con eficacia y eficiencia. Por otra parte, dada la poca frecuencia de estos experimentos es imprescindible maximizar su impacto. El rápido avance de las tecnologías ómicas ofrece una oportunidad para aumentar dramáticamente el volumen de datos producidos a partir de vuelos espaciales preciosas muestras. Para aprovechar esto, la NASA ha desarrollado la plataforma GeneLab para proporcionar acceso sin restricciones a los vuelos espaciales tripulados ómicas datos y fomentar su análisis extenso. Roedores (ratas y ratones) son organismos comunes del modelo utilizados por los científicos para investigar impactos biológicos relacionados con el espacio. El recinto que casa roedores durante vuelos espaciales se llaman hábitats de roedor (antes módulos de alojamiento de animales) y son substancialmente diferentes de jaulas vivero estándar en sus dimensiones, el flujo de aire y el acceso a agua y alimentos. Además, debido a las condiciones ambientales y atmosféricas en la estación espacial internacional (ISS), los animales están expuestos a una mayor concentración de CO2 . Recientemente nos informaron que ratones en los hábitats de roedor experimentan grandes cambios en su transcriptoma independientemente de si los animales estaban en el suelo o en el espacio. Además, estos cambios eran constantes con una respuesta hipóxica, potencialmente impulsada por mayores concentraciones de CO2 . Aquí describimos cómo se realiza un experimento típico de roedor en el espacio, cómo ómicas datos de estos experimentos se pueden acceder a través de la plataforma GeneLab y cómo identificar los factores claves en estos datos. Mediante este proceso, cualquier persona puede hacer descubrimientos importantes que podrían cambiar el diseño de actividades y misiones de espacio futuras.
Introducción
El objetivo general de este manuscrito es proporcionar una metodología clara de cómo utilizar GeneLab plataforma1 y cómo roedores experimentos realizados en espacio de la NASA se traducen a datos ómicas para el análisis. Los viajeros del espacio seres humanos están expuestos a numerosos riesgos para la salud de campos de gravedad alterada, radiación de espacio, aislamiento de la tierra y otros factores ambientales hostiles2,3,4,5, 6. experimentos biológicos realizados en el espacio y en el suelo han contribuido a definir y cuantificar estos riesgos7,8,9,10,11, 12 , 13 , 14. en el espacio, estos experimentos se han realizado en la estación espacial internacional (ISS), la lanzadera de espacio y otras plataformas orbitales. Realización de estos experimentos requiere hardware especializado y metodología dada la preocupación única de realizar experimentos en el espacio como el tiempo limitado del equipo y el ambiente de microgravedad. Varias plataformas ahora existen para realizar sofisticados experimentos en el espacio usando la planta, animal y microbiana modelos15.
Modelos de roedores han sido particularmente importantes para avanzar en nuestra comprensión de cómo los mamíferos, incluyendo a seres humanos, responden a vuelos espaciales. Estos incluyen el impacto de los vuelos espaciales tripulados del músculo estructura16,17,18 y las funciones inmunológicas19,20,21. Las jaulas vivero estándar utilizadas para los roedores de la vivienda en la tierra no son adecuadas para el vuelo espacial experimentos22,23. Por lo tanto, sobre los años ratones y ratas han sido voladas y alojados en jaulas diferentes, incluyendo la Agencia de exploración aeroespacial japonesa (JAXA) hábitat jaula24, animal llevando cápsulas de espacio usados en la M1 de BION no tripulado ruso vía satélite25 ,26,27, el sistema de cajón de ratones (MDS) diseñado por la Agencia Espacial Italiana28,29,30, el módulo de caja Animal de NASA (AEM) y ahora la NASA Roedor transportador y hábitats23. Experimentos de roedores comenzaron a bordo de la lanzadera de espacio usando jaulas que se refiere a como el módulo de caja de Animal (AEM). Este hardware se utilizó en 27 experimentos roedores en la lanzadera de espacio23. La AEM fue desarrollada originalmente para experimentos relativamente corto a bordo el transbordador (< 20 días). Desde el desarrollo de la ISS, las AEMs se han modificado para experimentos de duración más larga y ahora se denomina habitat roedor22,23. Los nuevos hábitats de roedores están diseñados para apoyar a las misiones de larga duración en la ISS con el acelere el procesamiento de los experimentos de interfaz de Rack de la estación espacial (EXPRESS). Hábitats de roedores son substancialmente diferentes de jaulas vivero estándar en sus dimensiones, el flujo de aire, filtro y sistema de escape y acceso a alimentos y agua (figura 1). Sin embargo, este hardware ha demostrado para ser una plataforma de investigación eficaz, permitiendo a ideas claves en los cambios inducidos por el vuelo espacial a fisiología mamífera19,31,32,33 ,34,35,36.
Ahora se pueden generar grandes volúmenes de datos ómicos de vuelo espacial biológico experimentos los realizados con roedores como. Recientemente, los datos de estos experimentos ómicos se hicieron disponibles al público a través de la NASA GeneLab plataforma1 que es un repositorio de datos y la plataforma de análisis que permite desarrollar hipótesis de los experimentos de vuelo espacial. GeneLab proporciona herramientas para el descubrimiento, acceso, intercambio y análisis de datos. Se utilizaron datos GeneLab para mostrar que las diferencias entre el vivero estándar jaulas y hábitats de roedor especializados utilizados en el espacio causan enormes diferencias en el transcriptoma de ratones36. Se analizaron cuatro diferentes disponibles conjuntos de datos, comparación de diferentes tejidos de roedores en el hábitat del roedor o jaulas vivero estándar. Usando un análisis de la biología de sistemas imparciales, determinamos que los controladores principales y vías que se han modificado eran constantes con una respuesta hipóxica debido a los altos niveles de2 CO causado por altas concentraciones de CO2 en la ISS, que lleva a mayor CO 2 concentraciones del hábitat del roedor ya que son sistemas pasivos que en el aire ambiente. Esto demuestra cómo los científicos pueden utilizar datos y herramientas de código abierto para generar resultados nuevos con implicación en cómo el ambiente de la ISS afecta salud de astronauta.
Aquí describimos cómo roedor experimentos se realizan en el espacio y cómo los datos de estos experimentos pueden accederse a través de un código abierto, omic plataforma relacionados con la biología del espacio. Discutimos la configuración de los hábitats de roedor utilizado para misiones espaciales, y cómo se procesan los tejidos de los vuelos espaciales tripulados. También describimos cómo vuelo espacial ómicas datos pueden ser descubiertos y accesible en GeneLab y claves cómo manejar la respuesta al vuelo espacial pueden ser identificado36. El ejemplo nos presentará cómo se implementa este Protocolo será comparar las diferencias biológicas que ocurren en los roedores alojados en hábitat de los roedores y los controles de vivero de Beheshti publicaron et al.36. Es importante tener en cuenta que suelo controles son esenciales para roedores experimentos de vuelo espacial. Como se describe en este protocolo, estos controles se realizan con ambas condiciones idénticas (es decir, CO2 condiciones, humedad, temperatura, dimensiones de la jaula, etc.) en los hábitats de roedor en la ISS y en jaulas vivero estándar que tienen el estándar medio ambiente (es decir,, CO2 condiciones, la humedad y temperatura) las condiciones en la tierra. Los roedores alojados en los controles de tierra Habitat roedor permiten la comparación directa a los roedores en el espacio. Mientras que los roedores alojados en jaulas vivero permiten la comparación biológica entre la carcasa diferente (por ejemplo, jaulas de vivero vs hardware de roedor). El hábitat del roedor es diferente que las jaulas vivero que tiene el flujo de aire constante (0,1 – 0,3 m/s), una larga duración y un filtro de escape secundario que capta y absorbe los residuos animales guiados para el filtro de salida por flujo de aire continuo en condiciones de microgravedad. Además, hábitats de roedor tienen sistemas pasivos y toma aire ambiente; por lo tanto, también tienen altas concentraciones de CO2 debido a niveles elevados en la cabina de la ISS (~ 5.000 ppm).
Protocolo
Los protocolos animal vivienda y procesamiento de tejido siguen las pautas estándar para el cuidado de animales de laboratorio y han sido aprobados por la NASA vuelo y de tierra Animal institucionalizados y comités del uso (IACUC).
1. configuración del hábitat del roedor
Nota: Los hábitats de roedor de la NASA (previamente AEMs) tienen diferentes características de las jaulas de vivero para las operaciones en el espacio (figura 1).
- Casa 10 ratones en cada hábitat de roedor (hasta 30 g por ratón). 5 ratones por compartimiento de la casa cuando el hábitat está configurado en dos compartimientos o 10 ratones si hay un solo compartimiento.
Nota: Hábitats de roedor de la NASA tienen una mayor superficie accesible por roedores que las jaulas estándar de vivero. - Para tierra controla animales, casa de ratones roedores Habitat interior simulador ambiental ISS (ISSES) en idénticas condiciones ambientales como los animales de vuelo incluyendo CO2 concentraciones, temperatura y humedad relativa.
- Proporcionar animales con acceso ad libitum a medida hecho NASA nutrientes actualizado roedor Foodbars (NuRFB) según requerimientos nutricionales de Consejo de investigación nacional (NRC) para ratones37, y de agua a través de la presión activa lixits.
- Monitorear los animales salud y comportamiento que se habilitará en el roedor con el 12:12 h luz ciclo similar a vivero jaulas en instalaciones estándar con iluminación LED durante el día y la iluminación infrarroja durante los controles de salud video que llevará a cabo durante el ciclo oscuro.
- Coloque cuatro cámaras en las jaulas de roedores Habitat para la supervisión diaria de salud y comportamiento y recogemos videos animales durante la noche con iluminación infrarroja.
- Entregar a los roedores a la ISS en un transportador (figura 2B) a bordo de la cápsula Dragon o similar vehículo de lanzamiento.
- Asegúrese de que los roedores son observados y examinados por el veterinario de vuelo de la NASA antes de ser cargado en el transportador para el lanzamiento y por los miembros del equipo entrenado a su llegada a la ISS y antes de la transferencia a los hábitats de roedor.
- Para este período de transición, casa hasta 20 ratones (10 en cada lado) o 12 ratas en el transportador.
Nota: Similares del hábitat del roedor, el transportador es una unidad pasiva de las condiciones ambientales. Durante este período de transición corto, esta unidad solo puede albergar hasta 20 ratones.
2. roedor para experimentos de vuelo espacial
- Adquirir roedores proveedores estándar.
Nota: Después del parto, grupo de roedores en jaulas estándar vivero y tienen los animales aclimatarse a la NASA NuRFB, lixits y suelos de alambre levantado hasta que los animales se cargan en el transportador. Dejando los roedores en jaulas permitirá adaptarse naturalmente de los animales. El manejo de ratones dentro y fuera del hábitat del roedor y jaulas vivero sigue protocolos comúnmente utilizados para los experimentos del roedor12,27,28. El sistema del hábitat del roedor (figura 1A) se utilizará para ambos misión de vuelo espacial en el STS y el ISS, respectivamente y para los controles de tierra simulando condiciones ambientales ISS o STS. - Para algunas misiones utilice jaulas vivero estándar (figura 1B) para el control de vivero. Usar 5 o 10 ratones por jaula vivero estándar.
- Para hábitats de roedor, coloque 10 ratones en dos compartimientos diferentes con 5 ratones por compartimento. Retire el divisor de la jaula para 10 ratones por hábitat en un solo compartimiento de la casa.
-
Utilizan tres componentes del hardware del roedor durante misiones de vuelo espacial como se describe a continuación (figura 2).
- Lugar de roedores en un transportador (figura 2B) para los viajes entre la tierra y la EEI o viceversa en doble densidad (10 ratones por el lado, 20 ratones por transportador).
- Una vez en ISS, fije el Animal acceso unidad (AAU) (figura 2) al transportador. Transferencia de roedores desde el transportador a los hábitats con cajas de transferencia de ratón (MTB) (5 ratones por MTB) (Figura 2D).
Nota: La AAU se utiliza para contener cualquier producto animal (e.g., las heces, orina, piel) de llegar a la cabina de la ISS. - Separar la AAU del transportador y coloque en el hábitat del roedor. Luego transferir los animales desde el MTB del hábitat del roedor (figura 2A) donde residen durante la duración de la misión.
Nota: La concentración de2 CO debido a niveles elevados en la cabina de la ISS para todos los habitat roedor es a 5.000 ppm.
- Monitorear la temperatura y la humedad de los hábitats de roedor, pero no hay ningún control térmico activo. Asegúrese de que el equipo de investigación de roedores funciona con ISS para mantener y controlar la temperatura de la cabina, que determina la temperatura del hábitat del roedor.
Nota: El ciclo de luz y oscuridad en los hábitats del roedor produce cada 12 h (por ejemplo,, 5:00 – 17:00 GMT, se enciende) y la tripulación de la ISS realiza cambio regular y frecuente de los alimentos (semanal o quincenal) y rellena el agua (cada ~ 28 días).
3. eutanasia de roedores y procesamiento del tejido
- Eutanasia, dar a roedor una sobredosis de anestesia general (ketamina/xilacina hasta 150/45 mg/kg cuerpo masa diluida en solución salina con tampón fosfato para un volumen total de 0,3 mL) por inyección intraperitoneal (IP) junto con un método secundario de eutanasia ( dislocación cervical o toracotomía).
-
Para experimentos en la ISS:
- Volver roedores ya sea viven, o
- Eutanasia en ISS.
- Congelación de cadáveres de roedores a-95 ± 2 ° C en el congelador de la ISS y volver a la tierra en el vehículo de retorno disponible (actualmente la cápsula SpaceX Dragon).
- Una vez que los roedores son devueltos a la tierra, diseccionar todos los órganos y tejidos (es decir,, hígado, riñón, piel, músculos, corazón, bazo, ojos, glándulas suprarrenales, los pulmones y cerebro) y almacenar a-80 ° C o en solución de estabilización de RNA.
- Siga los mismos procedimientos y tiempos para los experimentos de control de tierra como el experimento de vuelo con un desplazamiento de 3 – 5 días para que coincida con los datos de telemetría de la ISS.
- De los tejidos conservados aislar RNA, las proteínas y aislamiento de DNA usando protocolos estándar que se describen en detalle asociada a cada conjunto de datos en la plataforma GeneLab (genelab.nasa.gov).
Nota: Roedores tejidos no utilizados por el investigator(s) primario formar parte de la colección científica institucional de la NASA. Estas muestras se almacenan en el Ames Research Center (ARC) Biobanco no humanos donde están catalogados y a disposición para la petición por la comunidad científica. Los tejidos disponibles pueden encontrarse en las Ciencias de la vida datos archivo público sitio web en: https://Lsda.jsc.nasa.gov/Biospecimen.
4. generación de datos ómicos de RNA, DNA y proteínas extractos
- De las macromoléculas extraídas (ARN, ADN, proteína) utilizan protocolos estándar para generar datos ómicos. Éstos se describen en detalle en el estudio respectivos metadatos GeneLab.
5. GeneLab repositorio y presentación de datos
Nota: Espacio biología relacionados con datos ómicos son sometidos a GeneLab de depósito de datos. GeneLab acepta y organiza datos ómicos relacionadas con el espacio financiados por varias agencias espaciales del mundo.
-
Ómicas de generar datos que pueden ser alojados en el repositorio GeneLab relacionados.
- Enviar los datos generados a GeneLab, ya sea cuando el análisis es completo o en base a la discreción del investigador.
Nota: Datos a otras bases de datos públicas ómicas son importados y publicados en el repositorio GeneLab. GeneLab generada datos curados y publicados sin un periodo de embargo. GeneLab, específicamente la muestra proceso laboratorio, genera datos de varios experimentos de vuelo espacial usando protocolos de extracción optimizado y técnicas para aumentar los datos ómicos de los experimentos de vuelo espacial.
- Enviar los datos generados a GeneLab, ya sea cuando el análisis es completo o en base a la discreción del investigador.
-
Cuando los datos están listos para presentarse y el formato de transferencia de los datos y metadatos a GeneLab con el siguiente método (suplementario Figura 1):
- Utilizar las herramientas de ISAcreator para definir un estudio experimental y almacenar los metadatos.
Nota: ISAcreator la herramienta está disponible para descargar con un guía aquí tutorial38. - Consulte los datos de listan39 para entender los tipos de datos aceptados y formatos de archivos de datos crudos y procesados.
- Para optimizar la carga y almacenamiento, comprimir archivos de datos.
- Transferencia de los metadatos y los datos crudos o procesados a GeneLab curadores de datos mediante el espacio de trabajo40.
- Crear un nombre de usuario y contraseña y subir los datos.
- Utilizar las herramientas de ISAcreator para definir un estudio experimental y almacenar los metadatos.
- Una vez que los datos se han subido al espacio de trabajo, compartir datos con un Comisario GeneLab.
Nota: Pasos detallados sobre cómo subir y compartir archivos pueden encontrarse en la guía de presentación de datos41. - Cada presentación es verificado por un curador y publicado en el repositorio de GeneLab42.
6. encontrar conjuntos de datos para el análisis con funciones de búsqueda en GeneLab
-
Búsqueda de diferentes conjuntos de datos en GeneLab accediendo al enlace (suplementario Figura 2)38.
- Específicamente relacionado con una anterior publicación36, buscar los siguientes términos: 21 GLDS, GLDS-111, GLDS-25 y GLDS-63.
- Acceder a la página GeneLab haciendo clic en "Sistema de GeneLab de datos" en el lado izquierdo de la pantalla.
- Introduzca las palabras clave en el cuadro "buscar datos" para buscar áreas específicas de interés. En este caso introducir cada uno de los siguientes identificadores de conjunto de datos por separado: 21 GLDS, GLDS-111, GLDS-25 y GLDS-63.
- Además de buscar el repositorio GeneLab, búsquedas en otras bases de datos incluyendo GEO NIH, orgullo de la EBI y ANL MG-RAST seleccionando las casillas de verificación deseadas en la barra de búsqueda.
Nota: Actualmente solo para el repositorio GeneLab, puede buscar un usuario mediante el uso de las siguientes categorías de filtro: organismos, tipo ensayo, factores y tipo de proyecto.
7. almacenar y transferir archivos de interés para el análisis
Nota: El espacio de trabajo GeneLab está diseñado para almacenar y transferir archivos directamente desde la base de datos de GeneLab (suplementario Figura 3).
- Haga clic en "Workspace" en la parte superior el menú de sistemas de datos.
- Si nuevo usuario, registrar una nueva cuenta.
Nota: El espacio de trabajo GeneLab es alimentado por GenomeSpace43. - Acceso a detalladas instrucciones sobre cómo utilizar el espacio de trabajo seleccionando "Ayuda" en el menú superior y haciendo clic en Guía del usuario.
- Para cada usuario, acceder a todos los conjuntos de datos en el repositorio GeneLab seleccionando la carpeta "Público/genelab" en el menú de la izquierda.
-
Copiar las bases de datos de interés a un espacio de trabajo de directorio local por ir a la carpeta con los datos de interés. Haga clic derecho sobre el archivo específico, seleccione "copiar o mover" en el menú que aparece, seleccione la carpeta para copiar el archivo y haga clic en "copiar".
- Encontrar los siguientes conjuntos de datos relacionados a una anterior publicación36 como se indica arriba y copia a área de trabajo local: 21 GLDS, GLDS-111, GLDS-25 y GLDS-63.
8. acceso a metadatos y la descripción de cada estudio
Nota: Archivos de metadatos para cada conjunto de datos en el repositorio GeneLab son en la subcarpeta de datos de "Público/genelab" en el menú de la izquierda.
-
Encontrar la información de metadatos para el conjunto de datos de interés por acceder a uno o más archivos de metadatos contenidos en una subcarpeta de "metadatos" de cada conjunto de datos. Por ejemplo, GLDS-100, son 2 archivos en la subcarpeta "Público y genelab/GLDS-100/metadatos": "GLDS-100_metadata_RR1_BIOBANK-ojo-ISA.zip" y "GLDS-100_metadata_RR1ExpDesign.pdf".
- Asegúrese de que cada conjunto de datos tiene un único archivo comprimido que proporciona metadatos según la especificación ISATab (que subsume la MIAME, MIAPE y otras normas de marco MIBBI para los metadatos mínimos requisitos). Final siempre este tipo de nombre de archivo en "ISA.zip". Por ejemplo, GLDS-100, este archivo es "GLDS-100_metadata_RR1_BIOBANK-ojo-ISA.zip".
- Utilice la herramienta ISACreator del44 o un editor de texto para visualizar y acceder a los metadatos de ISATab, que contiene la descripción de texto para el estudio y análisis de metadatos para cada conjunto de datos.
Nota: Dentro de los metadatos de ISATab, las muestras se describen y asociadas con pruebas biológicas y pruebas biológicas se describen y asociados con los archivos de datos de salida. - Comprobar la presencia de los archivos de datos de ensayo de salida que se encuentran dentro de cada conjunto de datos en subcarpetas según el tipo de ensayo. Por ejemplo, GLDS-100, análisis de RNA-Seq archivos se encuentran en el "público/genelab/GLDS-100/transcriptómica /" carpeta.
9. Análisis de datos GeneLab
Nota: Varias tuberías pueden ser implementadas para varios datos ómicos. Aquí, lo ejemplo se centra en un gasoducto de transcriptómicos de Biología de sistemas imparciales que se utiliza para determinar los "drivers claves" del sistema en estudio.
- Compruebe previamente publicado literaturas36,45,46,47,48,49,50 para entender este gasoducto.
-
Una vez seleccionado un conjunto de datos específico de interés para el análisis, descargar los datos en un equipo local con el siguiente método:
- Haga clic en el conjunto de datos específico.
- Haga clic en la pestaña de "Archivos de estudio" en la extrema izquierda de los encabezados.
- Asegúrese de que todos los archivos de datos y metadatos están disponibles en este menú.
- Para descargar cada archivo, haga clic en los nombres de archivo específicos.
-
Para los conjuntos de datos de microarrays que se descargarán del GeneLab, siga los siguientes pasos de preprocesamiento.
- Proceso de los datos en bruto para cada conjunto de datos por separado usando fondo y Quantile normalizado utilizando RMAExpress51 para los microarrays comerciales.
- Crear parcelas de análisis (PCA) de componente de principio con R para determinar cómo se replica el biológico agrupados.
- Importar datos en el visor MultiExperiment52 y calcular importantes genes primero usando las estadísticas de tasa (FDR) falso descubrimiento a partir de FDR < 0.05. Si no hay genes importantes aparecieron con estadísticas FDR, utiliza pruebas t estándar a partir de un valor de p < 0.05 para determinar los genes importantes.
- Una vez que se han determinado los genes regulados estadísticamente significativos, implementar un corte de doble cambio de ≥ 1.2 o ≤ -1,2 para comparar las muestras experimentales con los controles.
-
Utilice análisis de enriquecimiento conjunto de Gene (GSEA)53 camino y predicciones funcionales.
- Utilice GSEA a través GenePattern54,55, directamente a través de GSEA, o usando el entorno de programación de R.
- Determinar las vías significativamente reguladas utilizando los siguientes grupos de genes: C2, C5 y señas de identidad.
- Realizar análisis de vanguardia sobre los conjuntos gen significativamente regulada y determinar vanguardia genes asociados con cada comparación experimental y Set de genes.
- Encontrar los genes de vanguardia que se superponen entre todos el gen establece para cada condición experimental.
-
Use otra plataforma para determinar funciones previstas y las vías que están siendo reguladas significativamente. En este caso utilice análisis de vía ingenio (IPA) para determinar los significativos reguladores de aguas arriba, biofunctions y caminos canónicos.
- Cargar la lista de genes con doblez-cambiar los valores de los genes estadísticamente determinados en el paso 9.4.4.
- Siga las instrucciones de la API para generar los reguladores aguas arriba, biofunctions y caminos canónicos para cada comparación experimental.
- Determinar que el gen asociado para reguladores de aguas arriba, biofunctions y caminos canónicos que tienen una activación puntaje z ≥ 2 (activación indicada) o ≤ -2 (indica inhibición).
- Encontrar los genes superpuestos relacionadas con todas las predicciones anteriores.
-
Determinar genes comunes/superposición entre pasos 9.4 y 9.5.
Nota: Estos genes son considerados como los genes clave/controlador que controla la mayoría de las actividades y funciones previstas con las condiciones experimentales analizadas. Estudios previos han demostrado que la anulación o promover estos genes hará que la condición experimental o sistema en estudio no funcional45,46,49.- Construir redes a través de IPA (o de cualquier software de montaje de red) para determinar la conectividad de los genes.
- Considerar el gen más conectado como eje central los genes claves de la conducción.
- Para determinar la conectividad entre los conjuntos de datos, grupo de todos los genes claves en una red y conectividad repetición prueba para determinar el eje central que está ocurriendo entre todos los genes claves de los conjuntos de datos analizados.
10. uso de galaxia56 interfaz en GeneLab para analizar datos transcriptómicos
Nota: Aquí se describe un protocolo para el uso de la interfaz de GeneLab Galaxy (disponible otoño 2018) para analizar los datos transcriptómicos de GeneLab. Abundan los tutoriales de la galaxia. Tutoriales de ejemplo sobre cómo utilizar Galaxy en general están disponibles elesewhere57,58.
- Usuarios pueden iniciar sesión en GeneLab utilizando credenciales de Google o la NASA. GeneLab galaxia herramientas se encuentran en el menú "Analizar".
-
Siga estas tres maneras de traer datos en la plataforma GeneLab Galaxy.
- Cargar datos desde el sistema de archivos local mediante la función "Cargar datos".
- Importar datos desde GeneLab GenomeSpace usando la herramienta importador de GenomeSpace en la sección "Obtener datos".
Nota: GeneLab todos los archivos de datos están disponibles en la carpeta "pública", organizada por el número de conjunto de datos (ver arriba). - Importación de datos aparece en la "historia" de la sección de análisis en el lado derecho. Los usuarios pueden tener múltiples historias, que se gestionan mediante botones de "Opciones de historial" o "Ver todas las historias" en la parte superior del panel historia.
- Herramientas para el análisis son listados y búsquedas en el lado izquierdo de la interfaz.
- Compruebe la aparición de conjuntos de datos que se han importado en la historia actual.
Nota: Muchos detalles con respecto a los datos están disponibles para la inspección para cada conjunto de datos. - Seleccione una herramienta del lado izquierdo para rellenar un formulario en el panel central, con opciones para el análisis y especificación de entradas de datos. Crear puestos de trabajo para ejecutar el análisis completando el formulario y pulsar "Ejecutar".
- Compruebe para trabajos presentaron que están representadas en la historia y codificadas por colores para indicar el estado de ejecución (cola, ejecución, terminados con o sin errores).
- Vincular las herramientas de flujos de trabajo complejos. Administrar flujos de trabajo a través de herramientas en el menú de "Flujos de trabajo". La figura 3 muestra un flujo de trabajo de ejemplo creado para procesar los datos de RNA-seq.
- Compartir conjuntos de datos, flujos de trabajo y las historias con los demás utilizando el menú de "Datos compartidos".
Resultados
Determinar la claves controladores de datos transcriptómicos de vuelo espacial ayudará a NASA con determinación de riesgos para la salud y el desarrollo de contramedidas potenciales para combatir los efectos negativos sobre la salud del astronauta. En nuestra reciente publicación, hemos seguido los pasos anteriores y GeneLab datos para mostrar con éxito una novela encontrando que las concentraciones de CO2 de la ISS pueden afectar salud36. También hemos usado la técnica anteriormente en otros estudios para determinar con éxito los factores claves que impulsan el sistema estudiado45,46,47,48,49,50 . Aquí mostraremos cómo los resultados del uso de este protocolo se pueden utilizar con éxito para determinar la clave.
En este estudio, nos centramos principalmente en las diferencias biológicas que se producen en los roedores en los controles de tierra de roedor de hábitos y el vivero. Como se describió anteriormente, es la clave para entender mejor estos dos hábitats, que nos proporcionará información sobre posibles factores de confusión que pueden afectar la salud debido al ambiente de la ISS. Para todos los experimentos de vuelo espacial de roedores, estos controles de tierra también son esenciales para determinar que factores biológicos están asociados directamente con el vuelo espacial o debido a las condiciones ambientales en la ISS. Como se indica en el protocolo, la condición ambiental para el habitat de vivero no está expuesta al mayor nivel de CO2 que está presente para el hábitat del roedor. El hábitat de vivero tiene CO2 nivel normal que está presente en la tierra (actualmente siendo 300 a 380 ppm). La temperatura y la humedad de ambos hábitats son similares.
Utilizamos los siguientes conjuntos de datos de la plataforma GeneLab para determinar los genes claves entre los roedores alojados en el roedor hábitat tierra controles y controles de planta de vivero que son responsables de conducir las diferencias entre lo dos hábitat: GLDS-21, GLDS-111, GLDS-25 y GLDS-63. Análisis para determinar los genes importantes se llevó a cabo como se describió anteriormente entre el hábitat del roedor (previamente AEM) y los controles de vivero independientemente para cada conjunto de datos. Agrupación de parcelas mostraron PCA de la biológica Replica (figura 4 muestra que la PCA parcelas GLDS-21). De los datos previamente procesados, determinamos los genes de la vanguardia de los distintos conjuntos de genes GSEA. Con los genes cambio 1.2 veces (log2), hemos sido capaces de predecir los genes involucrados con las predicciones para reguladores de aguas arriba, vías canónicas y biofunctions. Para cada conjunto de datos entonces encontramos los genes comunes/superposición involucrados para todos los genes (figura 5). Estos genes ahora se cree para ser la respuesta de conducción entre los roedores en el hábitat del roedor (o AEM) y los controles de vivero. Representación de la red de cómo conectan estos genes claves muestra que los ejes centrales para cada conjunto de datos que analizan (figura 6). Por ejemplo, MAPK1 es el eje central para STS-108 los tejidos de músculo esquelético de ratones (figura 6A). Esto se interpretaría como el gen que está impulsando los genes claves y es muy probable que el jugador central de causar diferencias biológicas para los ratones alojados en roedor hábitats frente a las jaulas de vivero. En nuestro trabajo anterior, se discute cómo estos genes claves se asocian con CO2 respuesta de la literatura científica existente y cómo estos genes pueden ser responsables de cambios biológicos observados en los ratones36.
Adoptar un enfoque de la biología de sistemas, luego determinamos un "regulador maestro" que conecta todos los conjuntos de datos/tejidos y es potencialmente responsable de efectos biológicos universales en roedores en AEMs comparados con jaulas de vivero. Esto se hizo determinando el gen de los conjuntos de datos que está más conectado al construir una red de todos los genes claves. Hemos sido capaces de demostrar que MAPK1 es el gene más conectada y el eje central de todos los genes claves (figura 7). Para confirmar si MAPK1 podrían ser responsables de cambios biológicos en ratones de los CO2 niveles más altos en AEMs, miramos a través de la literatura científica para apoyar la evidencia. Encontramos varios estudios que indican la correlación de MAPK1 con CO259 e hipoxia19,60,61.

Figura 1 : El hábitat del roedor (previamente AEM) comparado con las jaulas vivero. (A) imagen de la jaula AEM proporcionada por la NASA (créditos: NASA/Dominic Hart). (B) la jaula vivero estándar actualmente utilizado (Foto tomada por nuestro laboratorio). Esta figura ha sido modificada desde Beheshti et al.36. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2 : El sistema Habitat roedor con los tres diferentes módulos involucrados durante el transporte a y desde las misiones de espacio. El módulo izquierdo (A) es el módulo de hábitat del roedor (previamente AEM), el módulo del centro (B) es el transportador y el módulo de la derecha (C) es la unidad de acceso de animales (AAU). (D) La caja de transferencia de ratón (MTB). (Créditos: NASA/Dominic Hart). Haga clic aquí para ver una versión más grande de esta figura.
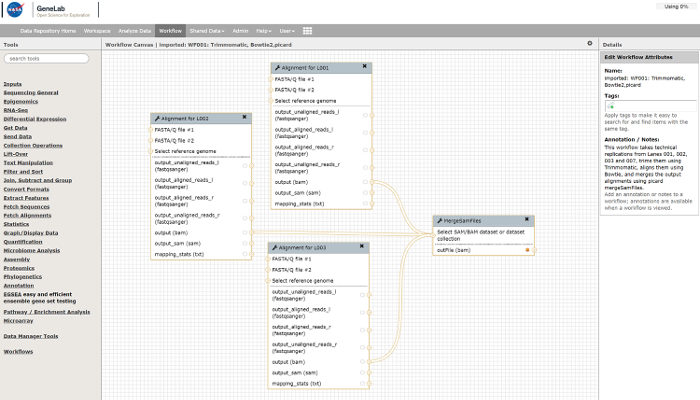
Figura 3 : Flujo de análisis del ejemplo que se puede utilizar en la interfaz de GeneLab Galaxy para RNA-seq datos. Haga clic aquí para ver una versión más grande de esta figura.
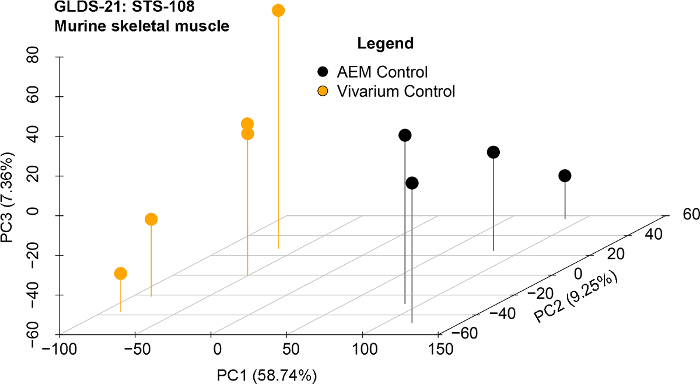
Figura 4 : Principales análisis de componentes (PCA) del conjunto de datos representativo después de pasos de pre-procesamiento. 21 GLDS dataset para AEM vs vivero la jaula se muestra para el músculo esquelético murine de la misión STS-118. Haga clic aquí para ver una versión más grande de esta figura.
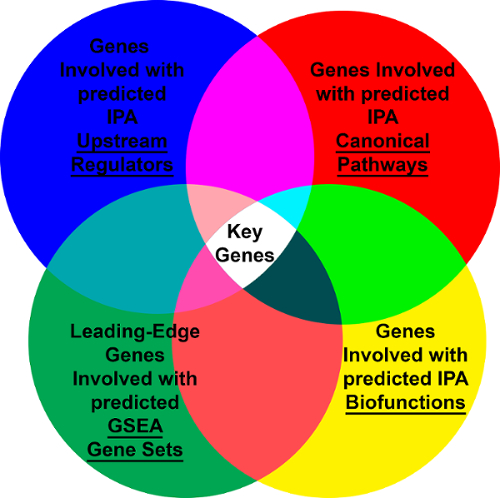
Figura 5 : Diagrama de Venn que representa qué genes clave se determinan usando herramientas de predicción de vía diferentes. Haga clic aquí para ver una versión más grande de esta figura.
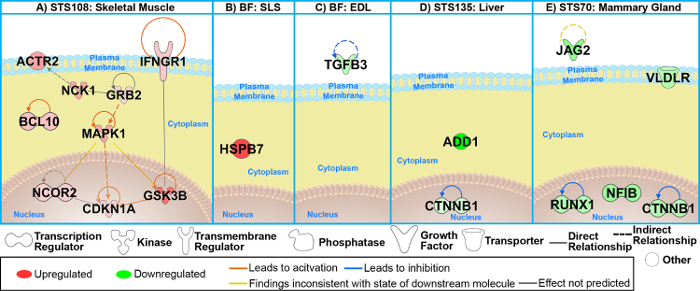
Figura 6 : Los genes claves para todas las condiciones y tejidos murinos entre las vs AEM . jaulas vivero. (A-E) Representación de los genes claves para cada conjunto de datos/roedor del tejido de la red. Registro2 fold-cambios (con un límite de cambio de 1.2 veces) a la expresión de genes fueron utilizados para obtener diferentes tonos de verde para doblez-cambio en los genes o, mientras que diferentes tonos de rojo representan doble cambio en genes de alza. Cuanto más oscura la sombra de color verde o rojo, mayor será el cambio de doblez. Esta figura ha sido modificada desde Beheshti et al.36. Haga clic aquí para ver una versión más grande de esta figura.
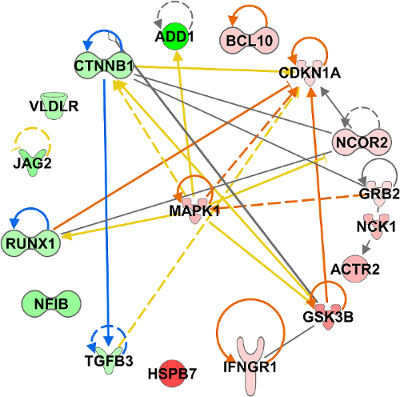
Figura 7 : Determinar el "regulador principal" para roedores en la vivienda del roedor hábitat comparado con jaulas vivero. Las conexiones entre todos los genes claves individuales (figura 6) se determina y muestra como una red a través de IPA. Red se representa como un diagrama radial con el gen clave más conectado, MAPK1, en el centro. Haga clic aquí para ver una versión más grande de esta figura.
Suplementario figura 1: integración GeneLab-GenomeSpace con ISACreator para optimizar las operaciones de procesamiento de datos. Haga clic aquí para descargar esta figura.
Suplementario figura 2: captura de pantalla del GeneLab búsquedas utilizando Federación e integración con bases de datos externas de Bioinformática heterogéneos (GEO, orgullo, MG-RAST). Por favor haga clic aquí para descargar esta figura
Suplementario figura 3: captura de pantalla del área de trabajo colaborativo GeneLab mostrar al usuario manejo de cuentas y controles de acceso (por ejemplo,, carpetas privadas, compartidos y públicos). Por favor haga clic aquí para descargar esta figura
Discusión
La plataforma de la NASA GeneLab es una plataforma de base de datos y análisis de omics integral que permitirá a la comunidad científica generar nuevas hipótesis relacionadas con la biología del espacio. Aquí presentamos un procedimiento general para roedores experimentos desde el principio del vuelo espacial para la generación de hipótesis de la novela de análisis de datos utilizando una plataforma de Biología de espacio disponible para el público. Además, también hemos proporcionado un protocolo extenso en un análisis de la biología de sistemas imparciales para la identificación de genes clave, conduciendo el sistema en estudio. Hemos utilizado nuestro reciente estudio36 como ejemplo de cómo este protocolo se utiliza con eficacia para generar una hipótesis novedosa para la biología del espacio. Esperamos que esta ayuda a los investigadores comprender mejor cómo se realizan los experimentos de vuelo espacial y cómo datos de ellos llevan a los datos disponibles en GeneLab y en última instancia permiten más clara interpretación de los datos de ómicas biología de espacio disponible para el público.
Hay varios pasos esenciales dentro de nuestro protocolo con respecto a los experimentos del roedor vuelo espacial y el análisis de los datos producidos. Comprensión del hábitat del roedor instalación es fundamental para desarrollar y diseñar el experimento óptima para el vuelo espacial. Esto implicaría concretamente el protocolo y la descripción que hemos proporcionado en el paso 1 de nuestro protocolo. Una vez que un investigador entiende perfectamente las diferentes condiciones existentes en el hábitat del roedor comparado con jaulas de vivero, los resultados biológicos interpretados pueden correlacionarse adecuadamente a las condiciones ambientales en el espacio. En las adiciones, modificaciones del hábitat del roedor no se puede hacer, ya que el hábitat del roedor ha sido óptimamente diseñado y aprobado por la NASA para el uso de vuelos espaciales tripulados.
Para interpretar los resultados biológicos, hemos proporcionado un protocolo exhaustivo de cada paso involucrado de subir sus datos a GeneLab al análisis de los datos para generar hipótesis de la novela de espacio de biología. Aunque todos los pasos son importantes para comprender cómo generar datos, los pasos más críticos para el análisis son pasos 9 y 10. Paso 9 proporciona un protocolo para analizar datos transcriptómicos utilizando un método de Biología de sistemas imparciales para determinar genes/vías que realmente conduce la condición experimental se analiza. Paso 10 es fundamental ya que proporciona a los usuarios con una metodología fácil para analizar ómicas GeneLab conjuntos utilizando la plataforma GeneLab. Modificaciones en el protocolo siempre se pueden hacer por algunas medidas en relación con el análisis de datos. Específicamente, se pueden hacer pasos 9.4-9.6 usando programación R o cualquier otras herramientas que el usuario prefiera. Según el conjunto de datos, estadísticas diferentes y doble cambio atajos pueden utilizarse para determinar los genes significativamente regulados. Además, para la determinación de los genes claves en los pasos 9.5 y 9.6, el usuario puede modificar este protocolo y utilizar cualquier herramienta que utiliza los genes significativamente regulados para predecir funciones. El concepto importante es que usando múltiples herramientas de predictivo ómicas funcional para la determinación de los genes involucrado en la mayoría de funciones regulada en el sistema en estudio.
La plataforma GeneLab continúa a desarrollar, mientras que los análisis aquí descritos se realizaron después de descargar datos, la siguiente fase del GeneLab permitirá análisis de omics datos directamente en plataforma GeneLab, que proporcionará un flujo de trabajo fácil para generar datos para el análisis de orden superior. Por otra parte, considerando que nos hemos centrado en un protocolo para la interpretación de datos transcriptómicos, GeneLab contiene una gran variedad de datos ómicos como proteómica, genómica, metabolómica y epigenómica. La eventual plataforma contendrá tuberías y normas para el análisis de estos diferentes tipos de omics. La última fase del GeneLab también pondrá en marcha una interfaz de visualización de nivel de sistema para permitir al usuario básico fácilmente generar hipótesis espacio biología.
Por último, nuestro análisis de la biología de sistemas proporciona un método único e imparcial para determinar la clave de conducir vías de genes en cualquier sistema de ser estudiado usando datasets ómicas. Hemos utilizado esta metodología en varios estudios independientes diferentes con gran éxito para determinar la clave involucrados36,45,46,47,48,49 ,50. En un cáncer relacionado con omics estudio, utilizando esta metodología hemos validado experimentalmente que nuestros genes/vías claves predichas realmente estaba manejando la respuesta al tratamiento medicamento al noquear a los genes claves en vitro45. Observamos, como habíamos predicho a través de este protocolo, que el tratamiento no fue efectivo ya debido a la ausencia de los genes claves. Creemos que este protocolo de Biología de sistemas imparcial puede ser una herramienta útil para determinar las vías claves para cualquier estudio de omics.
Este protocolo proporciona un método rápido y eficiente para la generación de nuevo espacio biología hipótesis. Los datos generados por GeneLab pueden aprovecharse por los investigadores para futuras oportunidades de financiamiento, validación experimental y potenciales dianas para el desarrollo de las contramedidas contra la radiación del espacio y la microgravedad. El protocolo que aquí permitirá futuro espacio biología investigaciones producir con eficiencia óptima para permitir misiones de espacio a largo plazo seguro.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Nos gustaría agradecer a Alison French en el archivo de datos de la NASA Ames Ciencias de la vida por su ayuda con la obtención del vídeo relacionado con los hábitats de roedor y ayuda general con la obtención de información relacionada de la jaula. También queremos agradecer a Marla Smithwick en el NASA Ames Research Center por su ayuda con la obtención de la información adecuada. Financiación de la investigación fue proporcionada por el proyecto GeneLab de NASA Ames Research Center, a través del programa de Biología del espacio de la NASA en la división de espacio de vida, investigación de ciencias físicas y aplicaciones (SLPSRA). Cualquier uso de nombres comerciales está para los propósitos descriptivos solamente y no implica la aprobación por el gobierno.
Materiales
| Name | Company | Catalog Number | Comments |
| C57BL/6 Mice | The Jackson Laboratoy | C57BL/6J | C57BL/6 mice were used for datasets related to Rodent Research-1 experiments |
| BALB/C Mice | Taconic | BALB | BALB/C mice were used for datasets related to Rodent Research-3 experiments |
| Vivarium Cages | Charles River Laboratory | Standard murine cages purchased from Charles River Laboratory | |
| Rodent Habitat | NASA | This cage and all components are built internally at NASA | |
| RNAlater | ThermoFisher Scientific | AM7020 | RNAlater is used to store the tissue for further RNA isolation |
Referencias
- Cortese, F. Vive la radioresistance!: converging research in radiobiology and biogerontology to enhance human radioresistance for deep space exploration and colonization. Oncotarget. 9 (18), 14692-14722 (2018).
- Beheshti, A. NASA GeneLab Project: Bridging Space Radiation Omics with Ground Studies. Radiation Research. , (2018).
- Fernandez-Gonzalo, R., Baatout, S., Moreels, M. Impact of Particle Irradiation on the Immune System: From the Clinic to Mars. Frontiers in Immunology. 8, 177 (2017).
- Bloomfield, S. A., Martinez, D. A., Boudreaux, R. D., Mantri, A. V. Microgravity Stress: Bone and Connective Tissue. Comprehensive Physiology. 6, 645-686 (2016).
- Giuliani, A. High-Resolution X-Ray Tomography: A 3D Exploration Into the Skeletal Architecture in Mouse Models Submitted to Microgravity Constraints. Frontiers in Physiology. 9, 181 (2018).
- Boice, J. D. The Final Frontier-Research Relevant to Mars. Health Physics. 112 (4), 392-397 (2017).
- Chancellor, J. C. Limitations in predicting the space radiation health risk for exploration astronauts. NPJ Microgravity. 4, 8 (2018).
- Cucinotta, F. A. Space radiation risks for astronauts on multiple International Space Station missions. PLoS One. 9 (4), e96099 (2014).
- Cucinotta, F. A. Review of NASA approach to space radiation risk assessments for Mars exploration. Health Physics. 108 (2), 131-142 (2015).
- Frippiat, J. P. Towards human exploration of space: The THESEUS review series on immunology research priorities. NPJ Microgravity. 2, 16040 (2016).
- Goel, N. Effects of sex and gender on adaptation to space: behavioral health. Journal of Women's Health (Larchmt). 23 (11), 975-986 (2014).
- Mortazavi, S. M. J., Bevelacqua, J. J., Fornalski, K. W., Welsh, J., Doss, M. Comments on "Space: The Final Frontier-Research Relevant to Mars". Health Physics. 114 (3), 344-345 (2018).
- Blottner, D. Morphological, physiological and behavioural evaluation of a 'Mice in Space' housing system. Journal of Comparative Physiology B. 179 (4), 519-533 (2009).
- Karouia, F., Peyvan, K., Pohorille, A. Toward biotechnology in space: High-throughput instruments for in situ biological research beyond Earth. Biotechnology Advances. 35 (7), 905-932 (2017).
- Shen, H. Effects of spaceflight on the muscles of the murine shoulder. The FASEB Journal. 31 (12), 5466-5477 (2017).
- Spatz, J. M. Sclerostin antibody inhibits skeletal deterioration in mice exposed to partial weight-bearing. Life Sciences in Space Research (Amst). 12, 32-38 (2017).
- Tascher, G. Proteome-wide Adaptations of Mouse Skeletal Muscles during a Full Month in Space. Journal of Proteome Research. 16 (7), 2623-2638 (2017).
- Pecaut, M. J. Is spaceflight-induced immune dysfunction linked to systemic changes in metabolism?. PLoS One. 12 (5), e0174174 (2017).
- Ward, C. Effects of spaceflight on the immunoglobulin repertoire of unimmunized C57BL/6 mice. Life Sciences in Space Research (Amst). 16, 63-75 (2018).
- Rettig, T. A., Ward, C., Pecaut, M. J., Chapes, S. K. Validation of Methods to Assess the Immunoglobulin Gene Repertoire in Tissues Obtained from Mice on the International Space Station. Gravitational and Space Research. 5 (1), 2-23 (2017).
- Allen, D. L. Effects of spaceflight on murine skeletal muscle gene expression. Journal of Applied Physiology (1985). 106 (2), 582-595 (2009).
- Moyer, E. L. Evaluation of rodent spaceflight in the NASA animal enclosure module for an extended operational period (up to 35 days). NPJ Microgravity. 2, 16002 (2016).
- Shimbo, M. Ground-based assessment of JAXA mouse habitat cage unit by mouse phenotypic studies. Experimental Animals. 65 (2), 175-187 (2016).
- Aseyev, N. Adaptive Changes in the Vestibular System of Land Snail to a 30-Day Spaceflight and Readaptation on Return to Earth. Frontiers in Cellular Neuroscience. 11, 348 (2017).
- Markina, E., Andreeva, E., Andrianova, I., Sotnezova, E., Buravkova, L. Stromal and Hematopoietic Progenitors from C57/BI/6N Murine Bone Marrow After 30-Day "BION-M1" Spaceflight. Stem Cells and Development. , (2018).
- Radugina, E. A. Exposure to microgravity for 30 days onboard Bion M1 caused muscle atrophy and impaired regeneration in murine femoral Quadriceps. Life Sciences in Space Research (Amst). 16, 18-25 (2018).
- Albi, E. Reinterpretation of mouse thyroid changes under space conditions: the contribution of confinement to damage. Astrobiology. 14 (7), 563-567 (2014).
- Cancedda, R. The Mice Drawer System (MDS) experiment and the space endurance record-breaking mice. PLoS One. 7 (5), e32243 (2012).
- Neutelings, T. Skin physiology in microgravity: a 3-month stay aboard ISS induces dermal atrophy and affects cutaneous muscle and hair follicles cycling in mice. NPJ Microgravity. 1, 15002 (2015).
- Anselm, V., Novikova, S., Zgoda, V. Re-adaption on Earth after Spaceflights Affects the Mouse Liver Proteome. International Journal of Molecular Sciences. 18 (8), (2017).
- Baqai, F. P. Effects of spaceflight on innate immune function and antioxidant gene expression. Journal of Applied Physiology (1985). 106 (6), 1935-1942 (2009).
- Blaber, E. A., Pecaut, M. J., Jonscher, K. R. Spaceflight Activates Autophagy Programs and the Proteasome in Mouse Liver. International Journal of Molecular Sciences. 18 (10), (2017).
- Jonscher, K. R. Spaceflight Activates Lipotoxic Pathways in Mouse Liver. PLoS One. 11 (4), e0152877 (2016).
- Moskaleva, N. Spaceflight Effects on Cytochrome P450 Content in Mouse Liver. PLoS One. 10 (11), e0142374 (2015).
- Beheshti, A., Cekanaviciute, E., Smith, D. J., Costes, S. V. Global transcriptomic analysis suggests carbon dioxide as an environmental stressor in spaceflight: A systems biology GeneLab case study. Scientific Reports. 8 (1), 4191 (2018).
- National Resource Council. . Nutrient Requirements of Laboratory Animals, Fourth Revised Edition, 1995. , (1995).
- . NASA GeneLab Data System Available from: https://genelab-data.ndc.nasa.gov/genelab/ (2018)
- . GeneLab FAQ Available from: https://genelab.nasa.gov/faq/#6 (2018)
- . GeneLab Workspace Available from: https://genelab.nasa.gov/faq/#6 (2017)
- . GeneLab Data Submission Guide Available from: https://genelab-data.ndc.nasa.gov/genelab/help/GeneLab_Submission_Guide_2.0.pdf (2017)
- . GeneLab repository Available from: https://genelab-data.ndc.nasa.gov/genelab/projects (2017)
- Qu, K. Integrative genomic analysis by interoperation of bioinformatics tools in GenomeSpace. Nature Methods. 13 (3), 245-247 (2016).
- . ISACreator Available from: https://isa-tools.org/category/isacreator/index.html (2014)
- Ravi, D. Proteasomal Inhibition by Ixazomib Induces CHK1 and MYC-Dependent Cell Death in T-cell and Hodgkin Lymphoma. Cancer Research. 76 (11), 3319-3331 (2016).
- Wage, J. Proton irradiation impacts age-driven modulations of cancer progression influenced by immune system transcriptome modifications from splenic tissue. Journal of Radiation Research. 56 (5), 792-803 (2015).
- Beheshti, A. Tumor-host signaling interaction reveals a systemic, age-dependent splenic immune influence on tumor development. Oncotarget. 6 (34), 35419-35432 (2015).
- Beheshti, A., Neuberg, D., McDonald, J. T., Vanderburg, C. R., Evens, A. M. The Impact of Age and Sex in DLBCL: Systems Biology Analyses Identify Distinct Molecular Changes and Signaling Networks. Cancer Informatics. 14, 141-148 (2015).
- Beheshti, A. Host age is a systemic regulator of gene expression impacting cancer progression. Cancer Research. 75 (6), 1134-1143 (2015).
- Beheshti, A., Peluso, M., Lamont, C., Hahnfeldt, P., Hlatky, L. Proton irradiation augments the suppression of tumor progression observed with advanced age. Radiation Research. 181 (3), 272-283 (2014).
- Bolstad, B. M., Irizarry, R. A., Astrand, M., Speed, T. P. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics. 19 (2), 185-193 (2003).
- Saeed, A. I. TM4 microarray software suite. Methods in Enzymology. 411, 134-193 (2006).
- Subramanian, A. Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles. Proceedings of the National Academy of Sciences of the United States of America. 102 (43), 15545-15550 (2005).
- Kuehn, H., Liberzon, A., Reich, M., Mesirov, J. P. Using GenePattern for gene expression analysis. Current Protocols in Bioinformatics. , (2008).
- Reich, M. GenePattern 2.0. Nature Genetics. 38 (5), 500-501 (2006).
- Afgan, E. The Galaxy platform for accessible, reproducible and collaborative biomedical analyses: 2016 update. Nucleic Acids Research. 44 (W1), W3-W10 (2016).
- Xu, Y. J., Elimban, V., Dhalla, N. S. Suppression of phosphorylated MAPK and caspase 3 by carbon dioxide. Molecular and Cellular Biochemistry. 436 (1-2), 23-28 (2017).
- Sang, N. MAPK signaling up-regulates the activity of hypoxia-inducible factors by its effects on p300. Journal of Biological Chemistry. 278 (16), 14013-14019 (2003).
- Seta, K. A., Kim, R., Kim, H. W., Millhorn, D. E., Beitner-Johnson, D. Hypoxia-induced regulation of MAPK phosphatase-1 as identified by subtractive suppression hybridization and cDNA microarray analysis. Journal of Biological Chemistry. 276 (48), 44405-44412 (2001).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados