Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Cuantificación automatizada basada en imágenes de trampas extracelulares de neutrófilos mediante NETQUANT
En este artículo
Resumen
Aquí, presentamos un protocolo para generar trampas extracelulares de neutrófilos (NET) y operar NETQUANT, una opción de software totalmente automática para la cuantificación de NETs en imágenes de inmunofluorescencia.
Resumen
Las trampas extracelulares de neutrófilos (NET) son estructuras antimicrobianas similares a la web que consisten en proteínas antimicrobianas derivadas del ADN y del gránulo. La microscopía de inmunofluorescencia y los métodos de cuantificación basados en imágenes siguen siendo herramientas importantes para cuantificar la formación de trampas extracelulares de neutrófilos. Sin embargo, existen limitaciones clave en los métodos basados en la inmunofluorescencia que actualmente están disponibles para cuantificar los NET. Los métodos manuales de cuantificación de NET basada en imágenes suelen ser subjetivos, propensos a errores y tediosos para los usuarios, especialmente los usuarios no experimentados. Además, las opciones de software disponibles actualmente para la cuantificación son semiautomáticas o requieren capacitación antes de la operación. Aquí, demostramos la implementación de un método automatizado de cuantificación de imágenes basado en inmunofluorescencia para evaluar la formación NET denominada NETQUANT. El software es fácil de usar y tiene una interfaz gráfica de usuario (GUI) fácil de usar. Considera parámetros biológicamente relevantes como un aumento en la superficie y la relación de proteína de marcador DNA:NET, y la deformación nuclear para definir la formación de NET. Además, esta herramienta se construye como una aplicación de libre disposición, y permite la cuantificación y el análisis de la resolución de una sola celda.
Introducción
Los neutrófilos son mediadores cruciales de las respuestas de defensa innatas del huésped contra una amplia variedad de patógenos microbianos1. Ejecutan sus funciones antimicrobianas liberando sus gránulos que contienen una amplia gama de proteínas antimicrobianas2,produciendo especies reactivas de oxígeno (ROS) e hipoclorito1,y a través de fagocitosis3. Además, Brinkmann et al. 4 describió las trampas extracelulares de neutrófilos (NET) como un mecanismo novedoso por el cual los neutrófilos atrapan y eliminan los patógenos invasores. Desde su descubrimiento hace poco más de una década4, los NETs se han implicado en una amplia variedad de infecciosos5,6 y no infeccioso7 morbilidades. La formación de REDES es un proceso activo y da como resultado la extrusión de ADN de cromatina recubierto con proteínas antimicrobianas derivadas de gránulos8. Algunos de los cambios clave en la morfología celular y nuclear asociados con la formación de REDES incluyen la pérdida de morfología nuclear, la descondensación de cromatina, la movilización de proteínas de gránulos desde el citoplasma al núcleo y un aumento en el diámetro nuclear y celular8,9.
Los NET similares a la web, que pueden aparecer como estructuras difusas ligeramente más grandes que la célula o como estructuras varias veces más grandes que un solo neutrófilo se consideran indicadores de NETosis5,10. Mediante la microscopía de fluorescencia, las NET se pueden detectar sondeando el ADN con una sonda fluorescente como 4',6-diamidino-2-phenylindole (DAPI) y por la tinción de inmunofluorescencia contra proteínas ligadas a redes netasas como la aslastasa de neutrófilos. La cuantificación de áreas superpuestas de tinción para ADN y proteínas ligadas a REDES determina el área total bajo NETs en una imagen11.
Hay varias opciones de análisis de imágenes disponibles para realizar la cuantificación basada en imágenes por fluorescencia de los NET11,12. Pero estas opciones de software presentan limitaciones para no ser fáciles de usar y/ o totalmente automatizados. En este artículo, demostramos el funcionamiento de NETQUANT13, una aplicación de libre disponibilidad que puede realizar una cuantificación NET basada en imágenes basada en imágenes de microscopía de inmunofluorescencia totalmente automatizada imparcial. La aplicación tiene una interfaz gráfica fácil de usar (GUI) y puede realizar análisis de una sola célula. El software cuantifica la NETosis en una imagen mediante la detección de los cambios morfológicos en el área del marcador enlazado a LA RED de ADN, la deformación asociada a la descondensación de cromatina del núcleo y el aumento de la relación de proteínas enlazadas dnalo:NET. En conjunto, los múltiples criterios de definición de NET permiten una cuantificación estricta de NET en varios conjuntos de datos de forma imparcial.
Protocolo
El comité de ética de la Universidad de Lund aprobó la recolección de sangre venosa de voluntarios sanos de acuerdo con la Declaración de Helsinki (2013/728). Todos los voluntarios proporcionaron su consentimiento informado por escrito.
1. Aislamiento de neutrófilos de sangre periférica usando Centrifugación Density-Gradient
- Recoger sangre venosa humana en tubos que contienen heparina y permitir que los tubos alcancen la temperatura ambiente.
Nota: Se requiere un mínimo de 16 ml de sangre de un donante sano para producir un pellet de células suficientemente grande. - Mezclar la sangre con un volumen de 2% de dextran en solución salina (0,9% NaCl) y dejar sedimentar a temperatura ambiente durante 30 minutos en un tubo de centrífuga cónica estéril de 50 ml.
- Aspirar el sobrenadante en un tubo de centrífuga cónica estéril de 50 ml y centrífuga a 200 x g durante 10 min a 4 oC.
- A partir de este paso, continúa el aislamiento a 4oC o sobre hielo.
- Resuspender el pellet en 5 ml de salina helada y capa en la parte superior de 5 ml de gradiente de aislamiento de leucocitos (9,1% diatrizoato de sodio con 5,7% de dextran, p/v) en un tubo de centrífuga cónico estéril de 15 ml.
- Centrífuga durante 30 min a 400 x g a 4oC.
- Aspira rinde al sobrenadante y deséchalo.
- Lyse glóbulos rojos mediante la reanimación del pellet en 3 ml de agua helada durante 30 s. Añadir inmediatamente 1 mL de 3.6% NaCl y luego llenar con 10 mL de salina fría de hielo.
- Centrifugar la suspensión celular durante 10 min a 350 x g.
- Retire el sobrenadante, recoja el pellet celular y resuspenderlo en 1 ml de salina. Reservar 10 l en un tubo de microcentrífuga para la evaluación del número de celda y la viabilidad utilizando el azul de tripa en una cámara de B-rker.
- Añadir 10 sl de suspensión celular a 90 ml de solución azul trypan al 0,4%. Tomar 10 l de la suspensión de la célula en una cámara de B-rker. Cuente las celdas en los 4 cuadrados enlazados por 3 líneas en cada esquina de la cámara. Las células que aparecen de color azul oscuro a la suanada de tinte no son viables, excluyémoslas del número total de células.
- Exprese el número de celda como celdas/ml según lo definido por la ecuación siguiente.
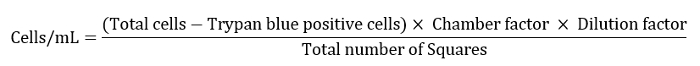
Nota: Aquí el factor de cámara era 10.000, el factor de dilución era 10 y el número total de cuadrados era 4. - Diluir la suspensión de celda restante a 10 ml para un paso de lavado final.
- Centrífuga durante 5 min a 200 x g.
- Resuspender los neutrófilos en RPMI-1640 con albúmina sérica humana (HSA) inactivada por calor de 2 mg/ml a una concentración de 5 x 105 células/ml.
2. Preparación de Coverslips y Estimulación de Neutrófilos
- Coloque un cubreobjetos (10 mm, #1) en cada pocal de una placa de 12 pocillos y cubra el cubreobjetos añadiendo 200 ml de solución de poli-l-lisina al 0,01% y déjelo a 37 oC durante la noche.
- Lave los cubreobjetos con solución salina tampón de fosfato de 300 l (PBS) una vez y déjelo secar.
- Añadir 400 éL de 5 x 105 neutrófilos/ml a cada pocil e incubar a temperatura ambiente durante 15 min.
- Mover la placa que contiene neutrófilos a una incubadora a 37 oC con 5% deCO2 durante 15 min.
- Retire el sobrenadante. Añadir 400 l de medio RPMI-1640 precalentado con 2 mg/ml de HSA a los controles. Añadir 300 l de RPMI precalentado con 20 nM phorbol 12-myristate 13-acetate (PMA) para la estimulación.
- Estimular los neutrófilos durante 150 min a 37 oC con 5% co2.
3. Visualización de NETs
- Retire el sobrenadante y lave las muestras 2 veces con 200 s de PBS.
- Corrija las muestras añadiendo 200 s de paraformaldehído (PFA) al 4% en PBS durante 20 min a 37 oC.
Nota: La PFA es tóxica y debe manipularse con cuidado. - Las muestras de lavado 3 x con 200 ml de PBS.
- Permeabilizar las muestras añadiendo 50 sL de 0.5% Triton X-100 para 30 s.
- Lave las muestras 3 veces con 200 ml de PBS.
- Bloquear las muestras con un 5% de suero de cabra en PBS durante 1 h a 37 oC.
- Añadir 300 ml de elastasa de neutrófilos antihumanosa de conejo primario en solución de bloqueo a una dilución de 1:500 durante 90 min a 37 oC.
- Lave las muestras 3 veces con 300 s de PBS.
- Añadir 300 ml de anticuerpo fluorescente anticonejo de cabra secundario a una dilución de 1:1000 durante 90 min a 37 oC.
- Lavar los cubreobjetos 3x con 300 sl de PBS.
- Retire los cubreobjetos de los pozos y monte el cubreobjetos con un medio de montaje de 10 l que contenga DAPI. Conservar durante la noche a temperatura ambiente en la oscuridad para secar las muestras.
Nota: La tinción de ADN con DAPI es sin duda un paso crítico en el método. Los usuarios también pueden solucionar problemas mediante la adición de una solución de DAPI exógena en un rango de concentración final de 0,1 u20120,5 g/ml durante 2 u20123 min, seguido de 3 pasos de lavado con 300 ol pbS. - Adquiera imágenes con un microscopio de fluorescencia de campo ancho utilizando un objetivo 20X.
4. Análisis y cuantificación de NETs utilizando NETQUANT
Nota: NETQUANT se puede descargar haciendo clic en el archivo de instalación que se encuentra en el archivo Zenodo Github o en el sitio web de Nordenfelt Lab (https://nordlab.med.lu.se/?page_id=34).
- Importación de conjuntos de datos para análisis, nomenclatura de canales y conversión de imágenes
- Abra la pestaña Configuración en NETQUANT.
- Elija la carpeta de origen para el análisis haciendo clic en la opción Obtener ruta en el menú de origen y seleccione la carpeta que contiene las secuencias de imágenes que se van a analizar.
- Haga clic en la opción Obtener ruta en el menú de destino y seleccione la carpeta para guardar los datos después del análisis de imagen.
- Nombre los canales de modo que "canal de ADN" se corresponda con la tinción de ADN(por ejemplo,ADN o DAPI) y "canal NET" representa la tinción de proteína ligada a LA RED(porejemplo, NET, elastasa de neutrófilos) en las imágenes. Para el buen funcionamiento del software, (recomendado) nombre la carpeta que contiene los archivos de imagen de control como "control".
Nota: El canal NET hace referencia únicamente a la tinción del marcador de proteína de gránulo enlazado a NET. - Alimentar los metadatos de la imagen en el software haciendo clic en el botón Cargar información de imagen en el submenú Información de imagen.
- Seleccione el orden de canal correcto contenido en las imágenes en el submenú Orden de canal. Esta opción se ha incluido como a prueba de fallos para evitar discrepancias accidentales.
- Adquiera propiedades de imagen principal de los datos sin procesar y convierta las imágenes haciendo clic en el botón Preparar datos. Las imágenes convertidas aparecen en el submenú Tipo de ejemplo. Haga clic en el menú Tipo de ejemplo para mostrar y seleccionar todos los conjuntos de datos adquiridos para su análisis.
- Seleccione una imagen del submenú Tipo de muestra y haga clic en el botón Mostrar datos de imagen para mostrar las imágenes divididas en el canal DNA y NET respectivamente.
- Segmentación de células en el canal de ADN y el canal NET
- Seleccione el método de segmentación haciendo clic en el submenú Método en el canal de ADN y el canal NET.
Nota: El método predeterminado de segmentación se establece en adaptativo y es la configuración recomendada. Otras opciones también están disponibles, incluyendo global, edge y Chan-Vese. También se incluye una opción de cuenca hidrográfica para ayudar a distinguir entre células o NET colocadas de cerca. - Introduzca la pestaña Segmentación para segmentar las celdas de control primero en ambos canales haciendo clic en la opción Muestras de control de segmento.
- Seleccione PMA en el submenú de tipo de muestra y haga clic en la opción Lote (recomendado) para comenzar la segmentación de todas las imágenes incluidas en el conjunto de datos. Seleccione las imágenes en el menú de tipo de muestra y haga clic en el botón Mostrar datos de imagen para visualizar y validar las máscaras de imagen binarias (máscara de ADN y máscara NET) generadas después de la segmentación.
- Seleccione el método de segmentación haciendo clic en el submenú Método en el canal de ADN y el canal NET.
- Análisis de una sola celda de propiedades identificables
- Introduzca la pestaña de análisis y analice las muestras de control haciendo clic en el botón Determinar umbral.
- Cambie el tipo de muestra a PMA y haga clic en el botón Obtener propiedades de celda para completar el análisis de muestras estimuladas.
- Seleccione una imagen del submenú Tipo de muestra y haga clic en el botón Mostrar datos de imagen para mostrar la superposición y el número de celdas y celdas de formación de REDES en la imagen.
- Comparación de propiedades de celda para identificar celdas formadoras de NET
- Seleccione la muestra del submenú de tipo de muestra y haga clic en el botón Analizar NET para completar el análisis. Las imágenes individuales se pueden seleccionar en el submenú de tipo de muestra para el análisis o se puede analizar todo el lote de imágenes seleccionando la opción de lote (recomendado).
- Ajuste los criterios de NET manualmente para obtener resultados óptimos para una muestra determinada. Compare los NET identificados con las imágenes originales para evaluar la calidad de la identificación.
Nota: Los criterios de NET se pueden utilizar en todas las imágenes del conjunto de datos. Cualquier cambio en los criterios de NET se aplica simultáneamente en todos los ejemplos de control. Esto limita la posibilidad de cualquier diferencia potencial que pueda surgir debido al exceso de ajuste de los parámetros de NET. La configuración de los criterios NET se puede ajustar según los requisitos del usuario. La relación entre las tasas de detección falsas y NETQUANT se ha explorado anteriormente13. Los rangos típicos para el aumento de área son de 2 u20124, la circularidad será de 0,7 u20120.9 y la relación ADN/NET será de 0,6 a 2,0. - Inspeccione el resumen de datos en el submenú Datos de celda donde se muestra nifique el número de imágenes, el número de celdas por imagen y el porcentaje de NET por imagen.
Nota: El porcentaje total de NET en todo el conjunto de datos se muestra mediante el "indicador NET". El recuento total de imágenes, el recuento de celdas, el porcentaje de NET en la muestra (NET%) y NETs% en el ejemplo de control se muestran en la tabla de estadísticas de resumen debajo del indicador NET. Recomendamos que los datos de control se notifiquen junto con los datos obtenidos de muestras estimuladas.
- Resultados de resultados
- Introduzca la pestaña Salida para seleccionar y ver las salidas de resultados.
- Explore y compare las diversas salidas de datos generadas a partir del análisis de control y PMA seleccionando la forma de la salida y haciendo clic en el botón Resultados de salida.
Nota: Todos los datos generados después del análisis para controles y estimulaciones se guardan en la carpeta de análisis como se selecciona en el submenú de destino. Los datos se guardan en formatos .csv o .pdf. - Inicie el archivo Method para obtener la versión del software y los criterios de NET utilizados para el análisis (que se incluirán en la sección de métodos con fines de publicación).
- Haga clic en la tabla De resultados para visualizar los puntos de datos individuales de una muestra determinada.
- Visualice la distribución del área NET y la relación DNA:NET en las muestras. La línea roja indica el valor de umbral en los gráficos.
- Determine el área NET frente a la forma del ADN haciendo clic en el archivo de distribución Bivariate.
- Carga de análisis anteriores y todos los pasos de lote
- Cargue la configuración de análisis previamente correcta en NETQUANT mediante el botón Cargar análisis anterior.
- Utilice el botón Lote todos los pasos incluidos en el menú Configuración para ejecutar los pasos 5-u201212(Figura 1, Figura 2, Figura 3, Figura 4, Figura 5) directamente para obtener la salida final.
Resultados
5 x 105 neutrófilos/ml fueron sembrados sobre los cubreobjetos colocados en una placa de 12 pocillos y estimulados con PMA de 20 nM o sin estimular durante 150 min. Las muestras se teñían entonces utilizando anticuerpos primarios anti-humanos neutrófilos elastasa, anticuerpos conjugados secundarios con fluoróforo anticonejo de cabra y DAPI - un tinte fluorescentemente etiquetado que mancha el ADN (Ver la Tabla de Materiales para más detalles). A continuac...
Discusión
La formación NET es una adición relativamente reciente al diverso armamentario de neutrófilos4 y ha habido una notable oleada de interés para estudiar la implicación de los NETs en una amplia gama de áreas de investigación5,7,14,15. La adquisición de imágenes mediante microscopía de inmunofluorescencia y la cuantificación posterior basada en imágenes es un m?...
Divulgaciones
TM y PN tienen una patente pendiente relacionada con los algoritmos utilizados en NETQUANT.
Agradecimientos
El trabajo fue financiado por la Fundación Crafoord (TM y PN), la beca sueca de investigación gubernamental (PN, TM), el Consejo de Investigación Sueco (PN) y la Fundación Groschinsky (TM, PN).
Materiales
| Name | Company | Catalog Number | Comments |
| BD Vacutainer Heparinised plastic tubes | BD Biosciences | 367885 | |
| Lymphoprep | Axis-Shield | 114547 | |
| RPMI-1640 with L-Glutamine | Gibco | 11835-030 | |
| 50mL conical flasks | Sarstedt | 62.547.004 | |
| 15mL conical flasks | Sarstedt | 62.554.002 | |
| 12-well Tissue culture plates | Falcon | 10626491 | |
| #1 Coverslips 10mm | Menzel Glaser | CS10100 | |
| Glass slides | Menzel Glaser | 631-0098 | |
| Primary anti-human elastase | DAKO | DAKO rabbit 1373, contract immunization | |
| Secondary fluorophore conjugated goat anti-rabbit | Life technologies | A-11072, A-11070 | |
| PROLONG-Gold Antifade reagent with DAPI | Life technologies | P36930 | Mounting medium |
| Goat serum | Sigma-Aldrich | G9023 | |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma-Aldrich | 79346 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Nikon Ti-E Epifluorescence microscope | Nikon | ||
| CCD camera | Andor Zyla | ||
| Plan Apochromat 20x, 40x objectives | Nikon | ||
| Windows 10 | Microsoft | Operating system | |
| macOS Sierra 10.12 | Apple | Operating system | |
| MATLAB | Mathworks |
Referencias
- Nauseef, W. M., Borregaard, N. Neutrophils at work. Nature Immunology. 15 (7), 602-611 (2014).
- Borregaard, N., Cowland, J. B. Granules of the human neutrophilic polymorphonuclear leukocyte. Blood. 89 (10), 3503-3521 (1997).
- Nordenfelt, P., Tapper, H. Phagosome dynamics during phagocytosis by neutrophils. Journal of Leukocyte Biology. 90 (2), 271-284 (2011).
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- Sorensen, O. E., Borregaard, N. Neutrophil extracellular traps - the dark side of neutrophils. Journal of Clinical Investigation. 126 (5), 1612-1620 (2016).
- Yipp, B. G., Kubes, P. NETosis: how vital is it?. Blood. 122 (16), 2784-2794 (2013).
- Jorch, S. K., Kubes, P. An emerging role for neutrophil extracellular traps in noninfectious disease. Nature Medicine. 23 (3), 279-287 (2017).
- Fuchs, T. A., et al. Novel cell death program leads to neutrophil extracellular traps. J Cell Biol. 176 (2), 231-241 (2007).
- Mohanty, T., et al. A novel mechanism for NETosis provides antimicrobial defense at the oral mucosa. Blood. 126 (18), 2128-2137 (2015).
- Hakkim, A., et al. Activation of the Raf-MEK-ERK pathway is required for neutrophil extracellular trap formation. Nature Chemical Biology. 7 (2), 75-77 (2011).
- Brinkmann, V., Goosmann, C., Kuhn, L. I., Zychlinsky, A. Automatic quantification of in vitro NET formation. Frontiers in Immunology. 3, 413 (2012).
- Coelho, L. P., et al. Automatic determination of NET (neutrophil extracellular traps) coverage in fluorescent microscopy images. Bioinformatics. 31 (14), 2364-2370 (2015).
- Mohanty, T., Sorensen, O. E., Nordenfelt, P. NETQUANT: Automated Quantification of Neutrophil Extracellular Traps. Frontiers in Immunology. 8, 1999 (2017).
- Kaplan, M. J., Radic, M. Neutrophil extracellular traps: double-edged swords of innate immunity. Journal of Immunology. 189 (6), 2689-2695 (2012).
- Cedervall, J., Olsson, A. K. Immunity Gone Astray - NETs in Cancer. Trends in Cancer. 2 (11), 633-634 (2016).
- Ginley, B. G., et al. Computational detection and quantification of human and mouse neutrophil extracellular traps in flow cytometry and confocal microscopy. Scientific Reports. 7 (1), 17755 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados