Method Article
Un sistema de cultivo timático tridimensional para generar emigrantes timémicos específicos de células madre pluripotentes inducidos por la murino
* Estos autores han contribuido por igual
En este artículo
Resumen
Este artículo describe un método novedoso para generar antígenos tumorales inducidos por células madre pluripotentes inducidas por antígenos timáticos (iTE) por un sistema de cultivo timico tridimensional (3D). iTE son un subconjunto homogéneo de células T estrechamente relacionadas con células T ingenuas con la capacidad de proliferación, formación de la memoria y supresión tumoral.
Resumen
La herencia de los receptores de linfocitos T pre-reorganizados (TPR) y su rejuvenecimiento epigenético hacen que las células T derivadas de células madre pluripotentes inducidas (iPSC) sean una fuente prometedora para la terapia adoptiva de células T (ACT). Sin embargo, los métodos in vitro clásicos para producir células T regeneradas a partir de iPSC dan como resultado células T innatas o diferenciadas terminalmente, que son fenotípicamente y funcionalmente distintas de las células T ingenuas. Recientemente, se desarrolló un nuevo sistema de cultivo trímico tridimensional (3D) para generar un subconjunto homogéneo de células T específicas del antígeno CD8+ con un fenotipo funcional similar a la célula T ingenuo, incluyendo la capacidad de proliferación, formación de la memoria , y la supresión tumoral in vivo. Este protocolo evita los aberrantes destinos de desarrollo, permitiendo la generación de células T derivadas del iPSC clínicamente relevantes, designadas como emigrantes timicos derivados de iPSC (iTE), al tiempo que proporciona una potente herramienta para dilucidar las funciones posteriores necesarias para la maduración de células T después de la selección timica.
Introducción
La terapia adoptiva de células T (ACT) puede ser un tratamiento eficaz para algunos pacientes con cáncer avanzado. Desafortunadamente, muchos pacientes no experimentan regresión tumoral, y las células transferidas no persisten después de la perfusión. Esto puede deberse a la calidad de las células T infundidas. Un modelo de ratón ACT mostró que en comparación con las células T de memoria central ingenuas o menos diferenciadas, las células efectoras diferenciadas terminalmente son menos potentes debido a la persistencia in vivo deficiente1, una observación también apoyada por datos clínicos2, 3.
En un esfuerzo por mejorar la eficacia de la ACT actual, las células madre pluripotentes inducidas por células T (T-iPSC) se han estudiado extensamente4,5. Cuando las células T se reprograman en T-iPSC y se vuelven a diferenciar en células T, la configuración reorganizada de los genes TCR es heredada por T-iPSC, y posteriormente las células T rediferenciadas. Por lo tanto, la capacidad de T-iPSC para someterse a una expansión in vitro ilimitada permite la reproducción eficiente de células T inmaduras que transportan los receptores de linfocitos T específicos de neoantígenos (TCR) cuando dichas células están diseñadas a partir de células T específicas de antígeno tumor6 ,7. Sin embargo, el método preciso para la diferenciación de T-iPSC en células T maduras, lo que permitiría la producción de células T específicas del antígeno canceroso con un fenotipo menos diferenciado y una mejor potencia antitumoral, sigue siendo aclarado.
La diferenciación T-iPSC que emplea el cocultivo de células estromales de OP9 que sobreexpresan el ligando de muesca humano DLL1 es un método bien establecido para producir células T in vitro6,7. En ratones y humanos, este sistema de cocultivo puede diferenciar consistentemente iPSC, recapitulando así los eventos de desarrollo desde la etapa del blastocisto hasta la etapa de linaje inmaduro de células T6,7. A pesar de estos avances biotecnológicos, la diferenciación fisiológica después de la etapa CD4+CD8+ doble positivo (DP) sigue siendo difícil de lograr. Una de las razones es que in vivo CD4+CD8- y CD4-CD8+ células T únicas positivas (SP) se generan en el timo, un órgano responsable de la maduración y selección de células T que tienen especificidad antógeno extraño pero no la reactividad automática8. Estos procesos selectivos se definen como selección positiva y negativa, respectivamente. Sin embargo, la mayoría de los mecanismos moleculares necesarios para madurar las células T en el timo todavía no se entienden completamente, lo que dificulta la reconstrucción de este proceso in vitro. En un intento de superar este obstáculo fisiológico, varios grupos han estimulado el complejo TCR utilizando anticuerpos anti-CD3 o péptidos agonistas. Estas técnicas in vitro generan productos celulares que expresan marcadores clave de células T, como CD3, CD8, TTC y CD62L, sin dejar de conservar la especificidad del antígeno tumoral. Desafortunadamente, las células T generadas por estos métodos extraitmicos constituyen una amplia población heterogénea de células caracterizada por una selección positiva incompleta, características innatas, matanza no específica de TCR, incapacidad para la formación de la memoria y efectos antitumorales no persistentes in vivo8,9,10,11. Estas anomalías han suscitado preocupaciones de que estas células puedan desencadenar una variedad de efectos secundarios, incluyendo linfoma y anormalidades cutáneas y óseas, si se utilizan para aplicaciones terapéuticas12,13,14 .
Para recrear las señales fisiológicas que faltan en los sistemas actuales de diferenciación in vitro, se diferenciaron T-iPSC específicos del antígeno tumoral utilizando un timo cosechado. El sistema clásico de cultivo de órganos de timo fetal (FTOC), que fue diseñado para estudiar el desarrollo intra-timico de las células T, se mejoró mediante el uso de un sistema de cultivo 3D que produjo con éxito células T que completaron la educación timica. Estas células T post-tímicas, que fueron designadas como emigrantes timicos derivados de iPSC (iTE), exhibieron propiedades ingenuas15. iTE mostró proliferación, formación de la memoria, y efectos antitumorales adecuados en un modelo de ratón contra tumores de melanoma B16 establecidos. Este artículo describe en detalle el protocolo de este nuevo sistema FTOC utilizando un sistema de cultivo 3D (Figura 1).
Protocolo
Todos los experimentos con animales fueron aprobados por los Comités Institucionales de Cuidado y Uso Animal del Instituto Nacional del Cáncer (NCI) y realizados de acuerdo con las directrices de los NIH.
1. Preparación de células OP9/DLL1 para cocultura con iPSC
- Cultivo de células OP9/DLL1 en medios OP9 (medio esencial mínimo [-MEM] + 20% suero bovino fetal no inactivado por calor [FBS] + 1x penicilina-estreptomicina + ácido ascórbico [50 ng/mL] y mono-tioglicerol [100 nM]) a 37oC. Cuando las células OP9/DLL1 alcancen una confluencia del 80-95%, lave una vez con 1x magnesio, calcio y fosfato libre de fenol solución salteada (en adelante, PBS).
- Añadir 4 mL de 0,05% de trippsina e incubar durante 5 min a 37oC. Luego agregue 4 ml de medios OP9, disocie la capa celular pipeteando para hacer una suspensión de una sola célula.
- Transfiera la suspensión celular a un tubo cónico de 50 ml a través de un colador de células de 100 m. Centrifugar a 300 x g durante 5 min a 4oC, aspirar el sobrenadante y resuspender en 12 ml de soporte OP9.
- Placa 2 mL de suspensión de celda OP9/DLL1 sobre un nuevo plato Petri de cultivo celular de 10 cm y añadir 8 ml adicionales de medios OP9. Repita el pasaje cada 2–3 días.
NOTA: La calidad del FBS y las condiciones de cultivo son fundamentales para mantener la expansión de las células OP9/DLL1 sin perder su capacidad de admitir la diferenciación iPSC. Por lo tanto, se recomienda preevaluar el lote de FBS y pasar consistentemente al 80% de confluencia para evitar la diferenciación celular y la senescencia. También es importante hacer suficiente stock congelado de células OP9/DLL1 y descongelar un nuevo stock cada 4-6 semanas.
2. In Vitro Diferenciación de iPSC en Células T Inmaduras
-
El día 0, comience la cocultura de iPSC en platos confluentes OP9/DLL1.
- Cosecha iPSC como suspensión de una sola célula por trippsinización (5 min en 0.05% trippsina a 37oC), recoge las células y centrifuga a 300 x g durante 5 min a 4oC.
- Aspirar las células sobrenadant asitonantes y resuspender a 1.0 x 105 iPSC por 10 mL de medios OP9. Placa 1.0 x 105 iPSC sobre un plato confluente OP9/DLL1 de 10 cm.
NOTA: Los platos OP9/DLL1 de 10 cm se utilizan para la diferenciación iPSC cuando alcanzan una confluencia del 90-100%. Las diferencias en la confluencia pueden afectar a la eficiencia de la diferenciación iPSC.
- En el día 3, aspirar medios antiguos y reemplazar con 10 mL de medios OP9 frescos.
-
En el día 6, celdas de paso.
- Lave cada plato OP9 confluente de 10 cm con 10 ml de PBS. Añadir 3 ml de 0,05% de trippsina por plato e incubar durante 3-5 min a temperatura ambiente (RT).
- Agregue 4 ml de medios OP9 y recoja las células mediante pipeteo suave. Pasar las células a través de un colador de células de 100 m y centrífuga a 300 x g durante 5 min a 4 oC. Deseche el sobrenadante.
- Resuspender las células en 10 ml de medios de diferenciación (medios OP9 con 5 ng/ml de ratón Flt3 ligando [FLT3L] y 5 ng/mL ratón IL-7) y suspensión de celda de placa en una nueva placa de confluente OP9/DLL1 de 10 cm.
- En el día 9, aspirar medios antiguos y reemplazarcon 10 ml de medios de diferenciación frescos.
-
El día 11, cuando se observan cardiomiocitos en colonias de iPSC, separa mecánicamente las células no adherentes pipeteando y filtra a través de un colador de células de 100 m. Girar a 300 x g durante 5 min a 4 oC.
- Aspirar el sobrenadante y resuspender en 24 ml de medios de diferenciación. Placa iPSC en una placa confluente OP9/DLL1 de 6 pocillos (4 ml/pozo).
-
El día 15, recoja todas las células no adherentes y filtre a través de un colador de células de 40 m.
- Girar a 300 x g durante 5 min a 4 oC.
- Continúe el paso de células no adherentes cada 3-4 días repitiendo el paso 2.5.1.
3. Cultivo de órganos timicos 3D para generar iTE
- Coser los lóbulos tiémicos fetales del ratón y desplegar linfocitos endógenos mediante el tratamiento con desoxiguanosina (dGUO) como se describió anteriormente16.
- En el día 7 del tratamiento dGUO, tomar cuatro nuevos platos de 10 cm y llenar cada uno con 20 ml de medios completos (Rosswell Park Memorial Institute Media 1640 [RPMI 1640] + 10% FBS + 1x L-alasilo-L-glutamina + 1x piruvato sódico + 1x medio esencial mínimo con aminoácidos no esenciales (MEM-NEAA) + 1x penicilina-estreptomicina + [1:1000] 2-mercapto etanol).
- Transfiera todas las membranas de nitrocelulosa con lóbulos timáticos en un plato de 10 cm. Separar los lóbulos individuales de la membrana con fórceps, lo que permite sumergirlos en los medios. Deseche las membranas. Incubar durante 1 h a RT.
- Transfiera los lóbulos timicos a un nuevo plato de 10 cm con medios completos e incubar durante 1 h en RT. Repita este paso 2 veces más.
- Usando fórceps, fije los lóbulos timáticos al plato (uno a la vez), y con la otra mano haga una incisión profunda de 100-200 m en el centro y extendiendo la mitad del diámetro del lóbulo para facilitar la migración del progenitor de células T en el lóbulo.
- Transfiera los lóbulos timicos a un nuevo plato de 10 cm lleno de medios de diferenciación completos (medios completos + ratón de 5 ng/ml IL-7 + ratón de 5 ng/mL FLT3L + 5 ng/mL SCF).
- Opcionalmente, si utiliza placas de cultivo 3D con rejillas de nivel inferior y superior, llene ambas rejillas con PBS estéril para evitar la evaporación y el secado de las gotas colgantes.
- Transfiera 30 ml de medios completos que contengan un lóbulo timic tratado con dGuo del paso 3.6 a cada pozo de placa de cultivo 3D.
- Recoja las células de linaje T no adherentes (células T inmaduras derivadas de iPSC) del cocultivo OP9/DLL1 (días 16-21) (paso 2.6.2) y resuspenda a células de linaje de 2-5 x 103 T por medio de 20 l.
- Añadir 20 ml de suspensión de células de linaje T a cada lóbulo timico en la placa de cultivo 3D. Incubar durante la noche a 37oC con 5% deCO2.
- Ajuste el pipeta P200 a 30 l y aspirar el medio después de pipetear varias veces de cada poca para eliminar todas las células que rodean los lóbulos timáticos. Deseche los medios y añada 30 s de medios completos. Repita este procedimiento de 5 a 7 veces para eliminar cualquier célula T inmadura adicional que no migre a los lóbulos. Cambiar de 25 a 30 ml de medios diariamente a partir de entonces.
- Confirmar la formación de un halo de emigrantes timicos derivados de iPSC (iTE) alrededor de los lóbulos a partir del día 4-5 mediante microscopía ligera.
- Recoger iTE diariamente mediante la pipeteo de medios sin interrupción del lóbulo. Cambie los medios todos los días y continúe la recopilación hasta aproximadamente 12 días.
- Los iTE cosechados están listos para su uso para análisis moleculares (Figura2, Figura 3, Figura 4y Figura 5)o experimentos de trasplante in vivo.
4. Preparación de células que presentan antígenos (APC)
- Sacrificar un ratón C57BL/6 por luxación cervical y colocar en una estera de empapada de laboratorio como se describió anteriormente.
- Retire el bazo y colóquelo en un colador de células de 100 m. Comprima el bazo en el colador con un émbolo de jeringa de 12 ml para hacer una suspensión de una sola célula.
- Transfiera la suspensión celular a través de un colador de células estéril de 40 m. Centrifugar la suspensión a 300 x g durante 5 min a 4 oC para peletizar las células.
- Aspirar el sobrenadante y resuspender el pellet celular en 2 ml de tampón de lisis de amonio-cloruro-potasio (ACK) para excluir los glóbulos rojos (RBC). Incubar durante 5 min a RT.
- Afirme el búfer de lisis ACK agregando 10 ml de PBS. Peletizar las células por centrifugación a 300 x g durante 5 min a 4oC.
- Aspirar el sobrenadante y resuspender el pellet celular en 10 ml de medios completos y transferirlo a un plato petri estéril de 10 cm.
- Irradiar esplenocitos con 3500 rad utilizando un dispositivo de irradiación (radiación) para prevenir la proliferación celular.
- Inmediatamente devolver las células irradiadas a una incubadora y cultivo de 37oC durante la noche.
- Utilice células irradiadas como APC o se congele en el banquero celular.
5. Pulsación de APC con antígeno
- Cuente el APC irradiado en vivo usando un hemocitómetro Neubauer y un tinte azul trypan. Incubar APC con péptidos (hgp100) o nucleoproteína durante 30 min a 37oC.
- Lave el APC dos veces con 10 ml de PBS para eliminar cualquier péptido adicional.
- Cuente iTE y mezcle con APC en una proporción de 1:1 en medios completos con 100 UI IL-2 y 5 ng/mL IL-7. Alícuota 100 l de la mezcla de células (concentración total: 1 x 106 células/ml) en cada pocal de un extremo U ultra-bajo 96 placa de pozo y cultivo durante 48 h a 37 oC.
- Después de 48 h, transfiera las células a una placa nueva usando una pipeta multicanal y pasaje cada 2-3 días después.
-
En el día 3, analizar el perfil de secreción de citoquinas manteniendo las células con anticuerpos intracelulares y analizar por citometría de flujo (Figura3).
- Añadir 0,67 l/ml de inhibidor de transporte de proteínas (p. ej., GolgiStop) e incubar a 37 oC durante 6 h para mejorar la acumulación intracelular de citoquinas. Lavar con 10 ml de PBS.
- Resuspenda las células en PBS frío (4oC) y agregue lentamente 1 ml de solución fría de paraformaldehído (PFA) al 4%.
- Después de 10 min, espíre las células a 300 x g durante 5 min a 4 oC, deseche el sobrenadante y lave con 10 ml de PBS.
- Resuspender las células en 1 mL PBS + 1% FBS + 0.1% tensioactivo no iónico, y colocar en 4 oC durante 10-15 min.
- Añadir anticuerpos, proteger las muestras de la luz y colocar en 4 oC durante 30 min.
- Gire las células a 300 x g durante 5 min a 4 oC, deseche el sobrenadante y lave con 10 ml de PBS.
- Gire las células a 300 x g durante 5 min a 4 oC y resuspenda las células en 1 ml de PBS. Las celdas están listas para ser analizadas en un catómetro de flujo.
Resultados
Los timoes fetales co-cultivados fueron seccionados para analizar si las células de linaje T derivadas de iPSC pueden migrar a los lóbulos timicos. Los lóbulos de control no sembrados tenían una arquitectura tisular caracterizada por una red epitelial timómica similar a la astrocíta17,desplegada de células ENDógenas CD3+. Por otro lado, los lóbulos timáticos sembrados con células T inmaduras derivadas de iPSC fueron repoblados con células CD3+ mononucleares, lo que indica la migración de células T inmaduras derivadas de iPSC a los lóbulos (Figura2A).
Células T que migranen y maduraron dentro del microambiente timico posteriormente salidadas como iTE. Para probar su caracterización fenotípica, se realizó un análisis citométrico de flujo de timocitos C57BL6, células T inmaduras derivadas de Pmel iPSC (extratímica) y células que salen de los lóbulos timicos (iTE). Las células T extratímicas en OP9/DLL1 mostraron células T CD4+CD8+ (DP) y células T CD8SP sin expresión del marcador de selección positivo MHC-I, mientras que iTE tenía una población clara de fenotipo de células CD8-SP MHC-I+ T, lo que indica su paso exitoso a través de la selección positiva antes de salir de los lóbulos timáticos. iTE expresa constantemente MHC-I y CD62L, que son marcadores asociados con alta competencia proliferativa, producción de citoquinas, supervivencia periférica, y afectación linfoide18,19,20. Este fenotipo es consistente con los timocitos M2 SP que son la población más madura de células T positivas individuales en el timo20,lo que sugiere que iTE ha pasado por un programa de desarrollo timémico normal (Figura3). Para monitorear la eficiencia de la generación de iTE, se aislaron las células que habían expulsado de lóbulos timicos individuales. En el día 7, los lóbulos timáticos generaron un promedio de 1 x 103 CD8SP en vivo CD45.1+ CD3+ iTE por día (Figura3B). Una tasa similar de producción de iTE se observa desde el día 6 hasta el día 12 de la cocultura timmica 3D.
Se analizaron la activación dependiente del antígeno y la secreción de citoquinas para observar las propiedades funcionales de las células T inmaduras derivadas del iPSC educadas tímicamente. En presencia de un péptido irrelevante (nucleoproteína), Pmel-iTE no liberó cantidades significativas de TNF-o, IL-2 o IFN-o. Cuando se estimuló con el péptido cognado para las células T de Pmel (hgp100), Pmel-iTE liberó cantidades robustas de TNF-- e IL-2, mientras que también produce cantidades bajas de IFN-o (Figura4), lo que indica que iTE con educación timica puede reconocer su péptido cognado y secretamente citkines efector con un perfil similar al de los emigrantes timicos recientes naturales (RTE).
Para examinar las diferencias transcripcionales entre las células de linaje T derivadas de iPSC diferenciadas en OP9/DLL1 con o sin educación timica (es decir, iTE frente a células T extratímicas), se realizó un análisis ARN-seq en estas dos poblaciones y se comparó a la de las células de linaje DP T diferenciadas usando OP9/DLL1 (DP) y células primarias ingenuas CD8+ Pmel T. La expresión de 102 genes que desempeñan un papel crucial en la ontogenia de células T, la activación de thymocitos y la formación de la memoria se analizaron15,20,21,22. Un análisis de componentes principales de esas cuatro poblaciones estudiadas demostró que las células T DP y CD8SP generadas extralíticamente se agruparon, mientras que iTE se agruparon más cerca de las células T ingenuas (Figura5). Colectivamente, estos datos demuestran que iTE tiene un fenotipo más cercano a las células T ingenuas que las células de linaje T generadas por métodos extratímicos.
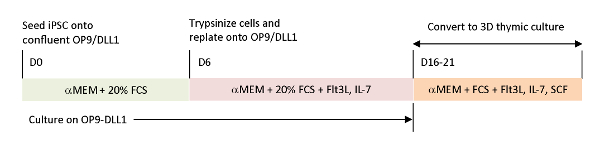
Figura 1 : Descripción esquemática de la diferenciación de iPSC a iTE utilizando OP9/DLL1 y cultivo timico 3D. El protocolo implica tres pasos de diferenciación separados; (Izquierda) de células iPSC a células de linaje hematopoyético en OP9/DLL1 (día 0 a 6), (Medio ) de células de linaje hematopoyéticas a células T inmaduras en OP9/DLL1 con citoquinas (días 6 a 16–21), y (derecha) de células T inmaduras (día 16–21) a iTE utilizando un sistema de cultivo timico 3D. Haga clic aquí para ver una versión más grande de esta figura.
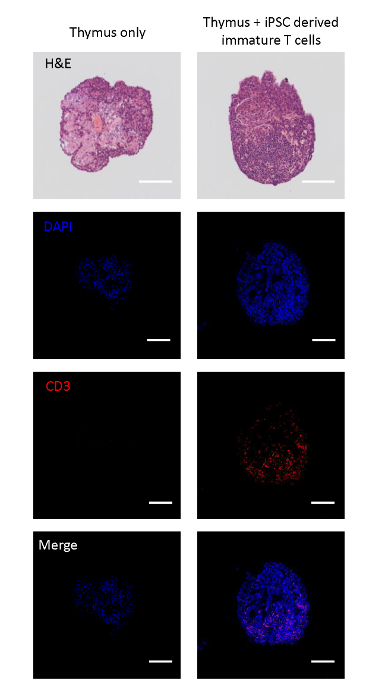
Figura 2 : Inmunohistoquímica de lóbulos timáticos sembrados con células T inmaduras derivadas de iPSC. Parte superior: Tinción de H&E de un lóbulo timico con y sin sembración de células T inmaduras derivadas de iPSC. De la segunda parte superior a la inferior: imágenes confocales de los lóbulos seccionados teñidos con DAPI (núcleo), CD3 (célula T) y fusión. Barras de escala a 100 m. Por favor, haga clic aquí para ver una versión más grande de esta figura.
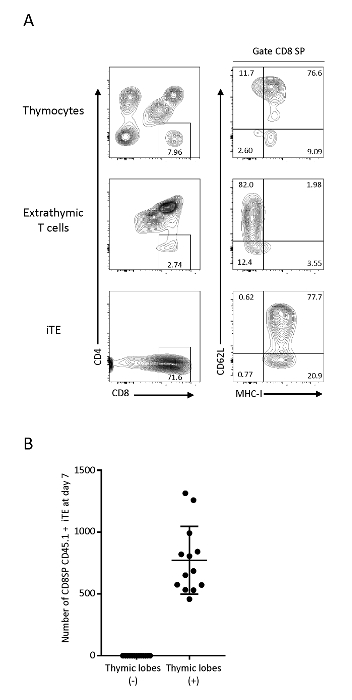
Figura 3 : iTE muestra un fenotipo de células T post-tímicos. (A) análisis FACS de timocitos, células T extratímicas (sistema de cocultivo OP9/DLL1) y Pmel-iTE. Las células vivas estaban cerradas en CD45 congénico+. Las poblaciones de CD8 SP se analizaron más a fondo para la expresión CD62L y MHC-I. (B) Número medio de CD8SP CD45.1 iTE producido durante la noche por lóbulo 7 días después de la pre-sembración. Se recopilaron datos de 12 experimentos independientes. Haga clic aquí para ver una versión más grande de esta figura.
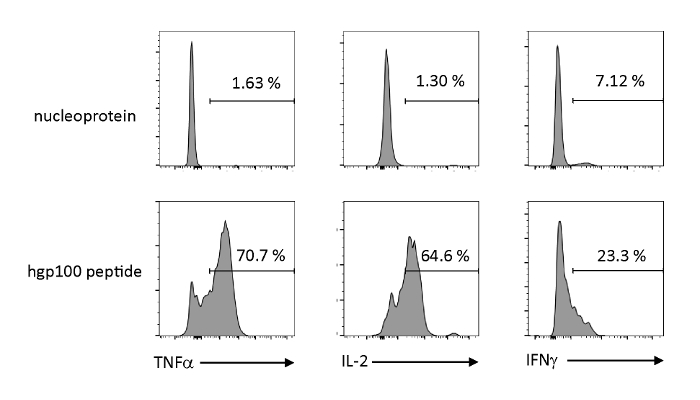
Figura 4 : iTE produce varias citoquinas mediante estimulación específica del antígeno. Análisis FACS de la producción intracelular de citoquinas por iTE. iTE fueron co-cultivados con APC precargados con péptido irrelevante (nucleoproteína) o cognado (hgp100) durante tres días. Los números mostrados en los cuadrantes superiores derecho indican los porcentajes de citoquinas productoras de iTE. Haga clic aquí para ver una versión más grande de esta figura.
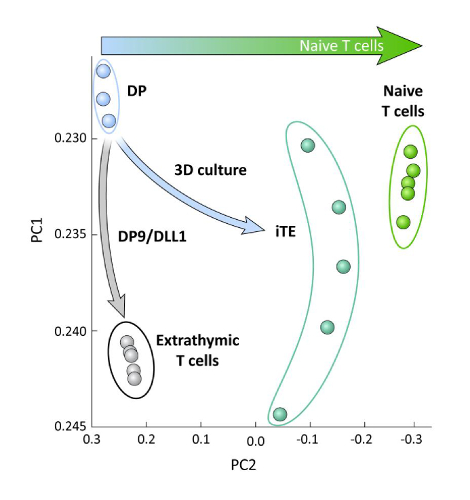
Figura 5 : El análisis del transcriptoma entero revela un cambio en la expresión del gen iTE hacia un CD8 ingenuo + Programa de células T. Análisis de componentes principales (PCA) de datos RNA-seq de células DP, CD8 extraitmicos SP, iTE y células T ingenuas. (Análisis de 102 genes relacionados con la diferenciación timmica utilizando la base de datos pública GSE105110) 15. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El uso de T-iPSC para regenerar células T específicas del antígeno tumoral puede superar muchos de los obstáculos actuales de ACT mediante la generación de células jóvenes con una mejor persistencia. Aunque se han notificado varios métodos que utilizan el sistema de cocultivo OP9/DLL1 para generar células CD8 SP6,7,10,13 que expresan moléculas cd8 y TPR específicos de antígeno tumoral, gen global los patrones de expresión y el análisis funcional muestran que estas células CD8 SP regeneradas extraimiánicamente son diferentes de las células T ingenuas (Figura4). Aquí, describimos un sistema de cultivo timico 3D que puede generar emigrantes timicos derivados de iPSC (iTE) con alta fidelidad y homogeneidad de la murino T-iPSC. iTE se asemeja a células T ingenuas en el patrón global de expresión génica y en la funcionalidad, como la formación de la memoria y el efecto antitumoral in vivo contra el tumor establecido15.
El sistema CTOC clásico es una forma de recapitular la selección timmica invitro. Se ha utilizado para el estudio del desarrollo intra-timico de timiocitos23,y hay algunos informes de FTOC que se utiliza para generar RTE24. Sin embargo, el sistema FTOC tiene varias limitaciones. Para hacer frente a la falta de oxígeno en un cultivo de órganos artificiales, varios grupos han utilizado un cultivo basado en membrana semi-seca23,o sistemas de cultivo de inmersión de alto oxígeno25. Sin embargo, ningún método actual puede generar constantemente una población homogénea de células T post-timicas. Para superar las limitaciones del sistema Clásico FTOC, diseñamos un sistema de cultivo timico 3D que proporciona mejoras técnicas sobre los métodos convencionales15. Por ejemplo, utilizando nuestro método de cultivo timático 3D, el intercambio máximo de oxígeno y la ausencia de estrés mecánico del lóbulo superficial mantienen los lóbulos timáticos en un entorno más fisiológico. Además, el cultivo a largo plazo permite que las células T maduras saquen naturalmente de los lóbulos timáticos. Por último, la observación en tiempo real y la micromanipulación permiten el intercambio de medios y una colección constante de iTE sin alterar físicamente los lóbulos timicos. Por lo tanto, el método de cultivo timático 3D proporciona mejoras técnicas significativas, así como una vía para estudiar células T naé seleccionadas con timmática que no estaban disponibles anteriormente.
Hay varios puntos clave para la exitosa generación de iTE utilizando este sistema de cultivo timico 3D. La calidad del FBS y las condiciones de cultivo es fundamental para mantener la expansión de las células OP9/DLL1 sin perder su capacidad de admitir la diferenciación iPSC. Por lo tanto, recomendamos la preevaluación del lote FBS, así como el paso constante en 80% de confluencia para evitar la diferenciación celular y la senescencia. Además, se requiere un cultivo OP9/DLL1 confluente para la diferenciación in vitro de iPSC en células T inmaduras, ya que las diferencias en la confluencia pueden afectar su eficiencia. Por último, la era embrionaria de los lóbulos timicos es crucial para la generación de iTE. Recomendamos el uso de los lóbulos timicos E14.5 - 15.5.
Al igual que con cualquier nuevo protocolo, este método tiene limitaciones y está sujeto a mejoras. La técnica de cultivo presentada aquí genera aproximadamente 1000 iTE por lóbulo timico por día durante un período de dos semanas. El aumento de la generación de iTE puede ser posible con más modificaciones, incluyendo la optimización de la concentración de oxígeno, el volumen de medios y el tipo de placa de cultivo 3D. La adición o eliminación de citoquinas, así como los cambios en la concentración de citoquinas, también pueden contribuir a mejorar el rendimiento de iTE.
Dado que el sistema de cultivo timico 3D presentado aquí puede generar emigrantes timicos en un sistema completamente ex vivo, esta técnica se puede aplicar a una variedad de proyectos de investigación de transferencia celular inmunológica y adoptiva, incluyendo, pero no limitado a T diferenciación celular, maduración de células T post-timicas y generación de células T específicas de antígenos a partir de progenitores hematopoyéticos o células madre. Aunque este método no es directamente aplicable a las muestras humanas, iTE y el sistema de cultivo timico 3D tienen un gran potencial para esclarecer los mecanismos moleculares de selección positiva y negativa y pueden facilitar la creación de un sistema de cultivo que permita la generación de células T ingenuas específicas del antígeno tumoral clínicamente relevantes para ACT.
Divulgaciones
Los autores Raúl Vizcardo, Nicholas D. Klemen y Nicholas P. Restifo son inventores de la solicitud internacional de patente pendiente PCT/US2017/65986, presentada el 13 de diciembre de 2017, titulada "Métodos de preparación de una población aislada o purificada de células de emigrantes ímicos y métodos de tratamiento usando lo mismo."
Agradecimientos
Agradecemos a Hiroshi Kawamoto y Kyoko Masuda por proporcionar amablemente la línea celular OP9/DLL1. Agradecemos a Alan B. Hoofring y Erina Z. El para la asistencia gráfica. Esta investigación fue apoyada por el Programa de Investigación Intramuros del Instituto Nacional del Cáncer de los Estados Unidos (ZIA BC010763) y el programa Cancer Moonshot para el Centro de Terapia Basada en Células en el NCI, NIH. El trabajo también fue apoyado por la Milstein Family Foundation.
Materiales
| Name | Company | Catalog Number | Comments |
| Chemicals, Peptides and Recombinant Proteins | |||
| 2-deoxyguanosine | Sigma-Aldrich | 312693-72-4 | |
| 2-Mercaptoethanol (1,000x) | Thermo Fisher Scientific | 21985-023 | |
| ACK Lysing Buffer | Gibco | A1049201 | |
| Ascorbic acid | Sigma-Aldrich | A8960 | |
| Blasticidin | Thermo Fisher Scientific | R21001 | |
| FBS | Gemini | 100-500 | |
| Flt-3 ligand | R&D Systems | 427-FL | |
| GlutaMAX (100x) | Thermo Fisher Scientific | 35050-061 | |
| hgp100 | Genscript | 282077-1, KVPRNQDWL | |
| Interleukin-2 | R&D Systems | 402-ML | |
| Interleukin-7 | R&D Systems | 407-ML | |
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| MEM powder | Gibco | 61100061 | |
| Monothioglycerol | Sigma-Aldrich | M-6145 | |
| Nucleoprotein | Global Peptides | ASNENMETM | |
| Penicillin/streptomycin | Thermo Fisher Scientific | 15140-122 | |
| Phosphate buffered saline pH 7.4 (1x) | Thermo Fisher Scientific | 10010-023 | |
| Puromycin | Thermo Fisher Scientific | A1113803 | |
| RPMI 1640 | Gibco | 11875093 | |
| Sodium Pyruvate | Thermo Fisher Scientific | 11360-070 | |
| Stem Cell Factor (SCF) | R&D Systems | 455-MC | |
| Stemfactor LIF, Mouse Recombinant | STEMGENT | 03-0011-100 | |
| Trypsin-EDTA (0.05%), phenol red | Thermo Fisher Scientific | 25300-062 | |
| Cell Culture Vessels and others | |||
| 10 cm dish | Corning, Inc. | 353003 | |
| 12 mL Syringe | Covidien Monoject | 22-652-090 | |
| 6 well plate | Corning/Coster | 3516 | |
| Cell strainer 100 μm | Fisher Scientific | 22-363-549 | |
| Cell strainer 40 μm | Fisher Scientific | 22-363-547 | |
| Forceps | DUMONT | 0108-5PO | |
| Lab soaker mat | Versi-Dry | Cat. EF2175CX 74018-00 | |
| Membrane filters ( 0.8 μm, 47diam) | Whatman | WHA7408004 ALDRICH | |
| Perfecta3D Hanging Drop Plate | Sigma-Aldrich | HDP1096 | |
| U Bottom 96 well plate | Corning/Coster | 3799 | |
| Experimental Cell lines | |||
| CD3-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| MEF-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| Mouse Embryonic Fibroblasts (MEF) | ATCC | SCRC-1040; RRID:MGI:5007926 | |
| OP9/N-DLL1 | Riken Bioresource center | Cat# RCB2927; RRID:CVCL_B220 | |
| Pmel-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| Experimental mouse models | |||
| B6.SJL-PtprcaPepcb/BoyCrCrl | Charles River | Strain Code 564; RRID:IMSR_CRL:564 | |
| C57BL/6N | NCI/Charles River | N/A | |
| Pmel-1 mice | Overwijk et al. | J Exp Med 198(4):569-80 | |
| Antibodies | |||
| Anti-aTCR | Biolegend | 109202; RRID:AB_313425 | |
| Anti-CD3 | abcam | ab11089; RRID:AB_369097 | |
| Anti-CD4 | BD Biosciences | 553730; RRID:AB_395014 | |
| Anti-CD44 | BD Biosciences | 559250; RRID:AB_398661 | |
| Anti-CD45.1 | BD Biosciences | 553775; RRID:AB_395043 | |
| Anti-CD45.2 | BD Biosciences | 553772; RRID:AB_395041 | |
| Anti-CD62L | BD Biosciences | 560516; RRID:AB_1645257 | |
| Anti-CD69 | BD Biosciences | 552879; RRID:AB_394508 | |
| Anti-CD8a | BD Biosciences | 557959; RRID:AB_396959 | |
| Anti-CD8b | BD Biosciences | 550798; RRID:AB_393887 | |
| Anti-H-2Kb | BD Biosciences | 553570; RRID:AB_394928 | |
| Anti-IFN-g | BD Biosciences | 557998; RRID:AB_396979 | |
| Anti-IL-2 | BD Biosciences | 554428; RRID:AB_395386 | |
| Anti-TCRb | Thermo Fisher Scientific | 35-5961-81; RRID:AB_469741 | |
| Anti-TCRVb13 | BD Biosciences | 553204; RRID:AB_394706 | |
| Anti-TNFa | BD Biosciences | 557644; RRID:AB_396761 |
Referencias
- Gattinoni, L., et al. Wnt signaling arrests effector T cell differentiation and generates CD8+ memory stem cells. Nature Medicine. 15 (7), 808-813 (2009).
- Rosenberg, S. A., et al. Durable complete responses in heavily pretreated patients with metastatic melanoma using T-cell transfer immunotherapy. Clinical Cancer Research. 17 (13), 4550-4557 (2011).
- Singh, N., Perazzelli, J., Grupp, S. A., Barrett, D. M. Early memory phenotypes drive T cell proliferation in patients with pediatric malignancies. Science Translational Medicine. 8 (320), 320-323 (2016).
- Crompton, J. G., Clever, D., Vizcardo, R., Rao, M., Restifo, N. P. Reprogramming antitumor immunity. Trends of Immunology. 35 (4), 178-185 (2014).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Vizcardo, R., et al. Regeneration of human tumor antigen-specific T cells from iPSCs derived from mature CD8(+) T cells. Cell Stem Cell. 12 (1), 31-36 (2013).
- Nishimura, T., et al. Generation of rejuvenated antigen-specific T cells by reprogramming to pluripotency and redifferentiation. Cell Stem Cell. 12 (1), 114-126 (2013).
- Takada, K., Kondo, K., Takahama, Y. Generation of Peptides That Promote Positive Selection in the Thymus. Journal of Immunology. 198 (6), 2215-2222 (2017).
- Yamagata, T., Mathis, D., Benoist, C. Self-reactivity in thymic double-positive cells commits cells to a CD8 alpha alpha lineage with characteristics of innate immune cells. Nature Immunology. 5 (6), 597-605 (2004).
- Themeli, M., et al. Generation of tumor-targeted human T lymphocytes from induced pluripotent stem cells for cancer therapy. Nature Biotechnology. 31 (10), 928-933 (2013).
- Serwold, T., Hochedlinger, K., Inlay, M. A., Jaenisch, R., Weissman, I. L. Early TCR expression and aberrant T cell development in mice with endogenous prerearranged T cell receptor genes. Journal of Immunology. 179 (2), 928-938 (2007).
- Lei, F., et al. In vivo programming of tumor antigen-specific T lymphocytes from pluripotent stem cells to promote cancer immunosurveillance. Cancer Research. 71 (14), 4742-4747 (2011).
- Maeda, T., et al. Regeneration of CD8alphabeta T Cells from T-cell-Derived iPSC Imparts Potent Tumor Antigen-Specific Cytotoxicity. Cancer Research. 76 (23), 6839-6850 (2016).
- Serwold, T., et al. T-cell receptor-driven lymphomagenesis in mice derived from a reprogrammed T cell. Proceedings of the National Academy of Sciences of the United States of America. 107 (44), 18939-18943 (2010).
- Vizcardo, R., et al. Generation of Tumor Antigen-Specific iPSC-Derived Thymic Emigrants Using a 3D Thymic Culture System. Cell Reports. 22 (12), 3175-3190 (2018).
- Jenkinson, W., Jenkinson, E., Anderson, G. Preparation of 2-dGuo-treated thymus organ cultures. Journal of Visualized Experiments. (18), (2008).
- Hamazaki, Y., Sekai, M., Minato, N. Medullary thymic epithelial stem cells: role in thymic epithelial cell maintenance and thymic involution. Immunological Reviews. 271 (1), 38-55 (2016).
- Vivier, E., et al. Innate or adaptive immunity? The example of natural killer cells. Science. 331 (6013), 44-49 (2011).
- Rosen, S. D. Ligands for L-selectin: homing, inflammation, and beyond. Annual Review of Immunology. 22, 129-156 (2004).
- Hogquist, K. A., Xing, Y., Hsu, F. C., Shapiro, V. S. T Cell Adolescence: Maturation Events Beyond Positive Selection. Journal of Immunology. 195 (4), 1351-1357 (2015).
- Best, J. A., et al. Transcriptional insights into the CD8(+) T cell response to infection and memory T cell formation. Nature Immunology. 14 (4), 404-412 (2013).
- Schmitz, I., Clayton, L. K., Reinherz, E. L. Gene expression analysis of thymocyte selection in vivo. International Immunology. 15 (10), 1237-1248 (2003).
- Nitta, T., Ohigashi, I., Takahama, Y. The development of T lymphocytes in fetal thymus organ culture. Methods in Molecular Biology. 946, 85-102 (2013).
- Ueno, T., et al. Role for CCR7 ligands in the emigration of newly generated T lymphocytes from the neonatal thymus. Immunity. 16 (2), 205-218 (2002).
- Watanabe, Y., Katsura, Y. Development of T cell receptor alpha beta-bearing T cells in the submersion organ culture of murine fetal thymus at high oxygen concentration. European Journal of Immunology. 23 (1), 200-205 (1993).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados