Method Article
Basado en el ARN la reprogramación de fibroblastos primarios de humanos en células madre pluripotentes inducidas
En este artículo
Resumen
Aquí describimos un método clínicamente relevante, alta eficiencia y libre de alimentador para reprogramar fibroblastos primarios humanos en células pluripotentes inducidas, usando modificado mRNAs que codifican factores de reprogramación y microRNA maduro-367/302 imita. También se incluyen métodos para evaluar la eficiencia de reprogramación, ampliar las colonias clonales iPSC y confirmar la expresión de los marcadores de pluripotencia TRA-1-60.
Resumen
Células madre pluripotentes inducidas (iPSCs) han demostrado para ser una herramienta valiosa para el estudio de la enfermedad y el desarrollo humano. Más avance de iPSCs como un regenerador terapéutico requiere un seguro, robusto y conveniente protocolo de reprogramación. Aquí, presentamos un protocolo clínico relevante, paso a paso para la extremadamente alta eficiencia reprogramación de fibroblastos dérmicos humanos en iPSCs usando un acercamiento no-integración. El núcleo del protocolo consiste en expresar factores de pluripotencia (SOX2, KLF4, cMYC, LIN28A, NANOG, fusión de OCT4-MyoD) en vitro ARN mensajero transcrito sintetizado con modificado nucleótidos (mRNAs modificado). Los mRNAs modificados reprogramación son transfectados en fibroblastos primarios cada 48 h junto con madurados embrionarios células madre específicas microARN-367/302 imitadores durante dos semanas. Las colonias de iPSC resultante pueden entonces aisladas y directamente ampliadas en condiciones libres de alimentador. Para maximizar la eficiencia y la consistencia de nuestro protocolo de reprogramación a través de muestras de fibroblastos, hemos optimizado varios parámetros incluyendo el régimen de la transfección de RNA, sincronización de transfecciones, condiciones de cultivo y densidades de siembra. Lo importante, nuestro método genera iPSCs de alta calidad procedentes de la mayoría del fibroblasto, incluyendo muestras enfermas, mayores o senescentes difícil reprogramar.
Introducción
Reprogramación de células somáticas en células madre pluripotentes inducidas (iPSCs) requiere extendida expresión de un conjunto básico de factores de transcripción que son importantes en el mantenimiento de pluripotencia1,2. Cuando se produce iPSCs para aplicaciones clínicas, es esencial que la carga mutacional en células de entrada se reduce al mínimo durante el proceso y la eficiencia de generación de iPSC se mantiene en un nivel relativamente alto en las muestras del paciente. Sin embargo, la mayoría de reprogramación métodos, incluyendo los protocolos de integración-libre, sufre de muy baja eficiencia reprogramación, que límite de utilidad clínica de éstos acerca3. La baja eficiencia de reprogramación también puede promover la reprogramación selectiva de las células con mutaciones preexistentes, aumentando la carga mutacional en iPSCs resultante. Además, todos basados en ADN reprogramación los métodos, como enfoques basados en lentivirus y episomal, sufren la preocupación de seguridad que DNA al azar puede integrar en el genoma y crear la posibilidad de mutagénesis de insertional perjudicial y no deseados ( potencialmente oncogénica) expresión de los genes de la pluripotencialidad en aguas abajo del tejido derivados4.
Un enfoque prometedor para lograr inducción eficiente de pluripotencia en las células somáticas y reducir la carga mutacional en iPSCs resultante es utilizar sintético con mensajero RNAs que contiene modificado nucleobases (mRNAs modificado) para la reprogramación de5. La eficacia de enfoques reprogramación modificados de mRNA puede mejorarse aún más mediante la adición de células madre embrionarias (ESC)-específicos microRNAs (miRNAs)-367/302s3, que se han demostrado para reprogramar células somáticas con aumento de la eficiencia6 ,7. Sin embargo, incluso con la adición de miRNAs-367/302s, el enfoque reprogramación de ARNm modificado a menudo falla durante la aplicación a las células recién aisladas de pacientes3. A inconsistencias de la dirección de este enfoque modificado basado en el mRNA, hemos divulgado recientemente una estrategia optimizada y libre integración que induce pluripotencia en fibroblastos humanos primarios con una alta tasa de éxito y utiliza ambos mRNAs modificados de codificación reprogramación de factores y miRNA maduros-367/302 imita8. En nuestro método, el ARNm modificado reprogramación cóctel incluye una versión modificada de OCT4 fusionado con el MyoD transactivation dominio (denominado M3O)9 y cinco otros factores de reprogramación (SOX2, KLF4, cMYC, LIN28A y NANOG). Combinando el modificado mRNA que codifican los factores de pluripotencia con la miRNA imitadores parecen tener un efecto sinérgico sobre la reprogramación de eficiencias en el presente Protocolo. Optimizaciones adicionales del régimen de la transfección de RNA, la célula de siembra y condiciones de cultivo también eran necesarias para aumentar la eficiencia de reprogramación de la aproximación a un muy alto nivel8.
A diferencia de muchos otros protocolos, nuestro enfoque de reprogramación requiere típicamente solamente algunos mil entrada los fibroblastos. Además, muchos no integrar estrategias comunes utilizando plásmidos episomal, virus Sendai o auto-replicadoras del ARN implican extenso pases para diluir el vector reprogramación en iPSCs generado. Por el contrario, mRNA modificado e imitadores de miRNA maduros tienen una vida media corta y se eliminan rápidamente de las células. Tomados en conjunto, la cantidad de tiempo de la cultura de célula acumulativo entre recogida de muestras de pacientes y la generación de iPSCs utilizable es mínima en este enfoque, limitando con eficacia la acumulación de la mutación en iPSCs resultante y mejorar rentabilidad.
Aquí, presentamos el protocolo paso a paso detallado para lograr alta eficiencia la reprogramación de fibroblastos humanos adultos en iPSCs usando nuestro combinatoria modificado mRNA/miRNA-enfoque basado en la8. Este protocolo de reprogramación RNA-basado proporciona un método sencillo, rentable y sólido para la generación de iPSCs libre integración de investigación y potencial aplicación clínica. Además, es aplicable a la reprogramación de una variedad de líneas de fibroblastos como difícil-a-reprogramar, asociado a la enfermedad, los fibroblastos envejecidos y senescentes. Figura 1muestra un esquema del protocolo para la reprogramación de fibroblastos humanos. El protocolo específicamente describe un método para reprogramar tres pozos de fibroblastos humanos de primarios adultos en una placa de la pozo 6 formato. Dos pozos producen típicamente un número suficiente de colonias de iPSC de alta calidad. En muchos casos, sólo uno es necesario, y el tercero también puede utilizarse para el análisis de la eficiencia de reprogramación. Si es necesario, el número de pozos puede ampliarse.
Protocolo
Trabajar en condiciones libres de RNasa y utilizar técnicas asépticas cuando sea posible. Realizar todas las manipulaciones relacionadas con el cultivo celular en un gabinete usando técnicas asépticas de seguridad biológica. Siga las normas de bioseguridad institucional para el trabajo con células humanas.
1. reactivos y equipos para preparación de reprogramación iniciación
- Preparar los factores de reprogramación codificación Cóctel de ARNm modificado.
- Seguir el protocolo previamente publicados10 para realizar en vitro transcripción, tapado y desfosforilación procedimientos para cada ARNm modificado codificación individual factor de reprogramación. Después del paso de la purificación final, procederá a la elución del mRNA modificado con agua libre de nucleasa, complementar la solución purificada de ARNm modificado con 1 inhibidor de la Rnasa U/μl, cuantificar los mRNAs modificados usando un espectrofotómetro y almacenar a-80 ° C hasta por 6 meses. Preparar varias alícuotas de cada mRNA modificada para reducir al mínimo el número de ciclos hielo-deshielo.
Nota: Protocolos similares para modificado mRNA producción han sido registrados en otras partes5,8,11. Plantillas para la transcripción en vitro pueden ser generadas por amplificación por PCR de las correspondientes plantillas de plásmido usando las cartillas enumeradas en la Tabla de materiales según el protocolo publicado anteriormente10. Plantillas de plásmido para cada factor de reprogramación (M3O9, SOX2, KLF4, cMYC, NANOG, LIN28A) y mWasabi (control de transfección) están disponibles en Addgene, un repositorio de plásmido sin fines de lucro. - Mezclar todos los mRNAs modificados en una proporción molar de 3:1:1:1:1:1 (M3O: SOX2: KLF4: cMYC: NANOG: LIN28A) y 10% mWasabi modificado mRNA como control de la eficiencia de transfección. Ajustar la concentración del mRNA modificada completa reprogramación de mezcla a una concentración final de 100 ng/μl añadiendo complementado con 1 inhibidor de la Rnasa U/μl de agua libre de nucleasa. Preparar siete 33 alícuotas de μl de las completas modificación Cóctel de mRNA. Almacenar las alícuotas reprogramación mixtas a-80 ° C.
Nota: para cada transfección, 1.000 ng del cóctel de ARNm modificado se agrega por pozo (es decir, un total de 3.000 ng para 3 pozos). Cada 33 μl alícuota está dimensionada para transfectar 3 pocillos de una placa de la pozo 6 formato e incluye 3 μl de volumen de exceso para tener en cuenta errores de pipeteo. Preparar siete 33 alícuotas μl es suficiente para completar una reprogramación de fibroblastos completo de 3 pozos de una placa de la pozo 6 formato.
- Seguir el protocolo previamente publicados10 para realizar en vitro transcripción, tapado y desfosforilación procedimientos para cada ARNm modificado codificación individual factor de reprogramación. Después del paso de la purificación final, procederá a la elución del mRNA modificado con agua libre de nucleasa, complementar la solución purificada de ARNm modificado con 1 inhibidor de la Rnasa U/μl, cuantificar los mRNAs modificados usando un espectrofotómetro y almacenar a-80 ° C hasta por 6 meses. Preparar varias alícuotas de cada mRNA modificada para reducir al mínimo el número de ciclos hielo-deshielo.
- Preparar el cóctel de reprogramación miRNA imitadores.
- Imitadores de miRNA disolver liofilizado (Syn-ha-miR-302a - 3P, Syn-ha-miR-302b - 3P, Syn-ha-miR - c 302-3p, Syn-ha-miR-302d-3 p, Syn-ha-miR-367-3 p) a una concentración final de 5 pmol/μl (5 μm) en agua libre de nucleasa, complementado con 1 U/μl de inhibidor de la Rnasa. Prepare varias alícuotas de cada imitador de miRNA y almacenar a-80 ° C para almacenamiento a largo plazo.
- Mezclar todos los imitadores de miRNA en una proporción molar de 1:1:1:1:1 a una concentración final de 5 pmol/μl (5 μm). Preparar siete 14 alícuotas de μl de la miRNA imita mix. Almacenar las mezcladas alícuotas a-80 ° C.
Nota: Para cada transfección, 20 pmol de la mezcla de los imitadores de miRNA se agrega por pozo (es decir, un total de 60 pmol de 3 pozos). Cada 14 μl alícuota está dimensionada para transfectar 3 pocillos de una placa de la pozo 6 formato e incluye 2 μl de volumen de exceso para tener en cuenta errores de pipeteo. Preparar siete 14 alícuotas μl es suficiente para completar una reprogramación de fibroblastos completo de 3 pozos de una placa de la pozo 6 formato.
- Preparar el buffer de transfección.
- Caliente previamente una botella de 500 mL y un frasco de 100 mL de medio fresco de suero reducido a temperatura ambiente (RT) por aproximadamente 2 h. No use un baño de agua.
- Transferencia de un medidor de pH a un gabinete de bioseguridad. Lave el electrodo de vidrio del medidor con agua libre de nucleasa. Calibrar el medidor de pH según las instrucciones del fabricante. Lave el electrodo otra vez con agua libre de nucleasa.
- Dos botellas de suero reducido medio de transferencia en el gabinete de seguridad de la biotecnología. Use la botella de 500 mL RT para ajustar el pH.
- Medir el pH base del suero reducido medio insertando el electrodo de vidrio del medidor de pH en el búfer. Esperar 1 min antes de leer el pH en el medidor.
- Agregar 3 – 4 mL de 1 M NaOH en 500 mL de suero reducido medio, cierre el frasco y mezclar bien. Esperar hasta 5 minutos antes de abrir la botella para medición del pH. Introduzca el electrodo del medidor de pH en el búfer y espere hasta que se estabilice la lectura en el medidor de pH.
- Seguir añadiendo pequeñas cantidades de 1 M NaOH hasta que el pH del suero reducido medio alcance 8.15 – 8.17. Calibrar el pH-metro varias veces durante el proceso. Si en cualquier momento el pH llega a ser superior a 8.18, reducir mediante la adición de medio fresco de suero reducido del frasco con 100 mL (paso 1.3.1).
Nota: El pH del buffer suero reducido basado en el medio de transfección es crítico. Reprogramación tendrá éxito si el pH del buffer de la transfección es aproximadamente 8,2 ± 0.05 pero puede fallar si el pH es superior a 8.25. Por lo tanto, se aconseja dejar de añadir NaOH una vez que pH del tampón alcanza 8.15 – 8.17. Esto asegurará que el pH final del búfer de la transfección es aproximadamente 8.2 – 8.22 después de la esterilización. - Filtro de esterilizar el búfer de transfección con un sistema de filtración de vacío 0.22 μm. Alícuota del tampón esterilizado en tubos de 5 o 15 mL con un mínimo espacio de aire.
- Guardar alícuotas del búfer de la transfección por hasta 3 meses a 4 ° C. Medir el pH de las alícuotas periódicamente. Deseche las alícuotas si el pH es superior a 8.25. Limitar uso de alícuota a 2 transfecciones solamente puesto que la exposición del búfer de la transfección a aire atmosférico aumenta el pH del buffer.
- Preparar el medio de expansión del fibroblasto (FEM): medio esencial mínimo suplementado con 10% suero fetal bovino (FBS), 1 x aminoácidos no esenciales, suplemento de glutamina x 1, 55 μm 2-Mercaptoetanol, 1 x lápiz/strep/fungizone. Tienda la FEM a 4 ° C.
- Prepare el soporte de reprogramación: DMEM/F12 (no HEPES) suplementado con 20% nocaut suero reemplazo (KOSR), 0,5 x aminoácidos no esenciales, suplemento de glutamina x 0.5, 55 μm 2-Mercaptoetanol, 50 μg/mL de ácido ascórbico, 1 x lápiz/strep/fungizone, bFGF de 100 ng/mL y 200 ng/mL B18R. Preparar el medio en el inicio de la reprogramación sin bFGF y B18R y almacenar a 4 ° C. No use más allá de 1 mes después de la preparación. Añadir bFGF y B18R inmediatamente antes de cada uso a una parte alícuota de tamaño para el uso de ese día.
- Prepare el soporte de placas mediante la adición de 5% inactivado con calor (HI) equipos en el medio de reprogramación que contiene 20% KOSR sin B18R de paso 1.5 y el bFGF. Preparar el medio fresco el día de uso. Añadir bFGF y B18R inmediatamente antes de su uso en el día 0 de reprogramación.
- Calibrar los incubadores de cultivo de tejidos antes de iniciar la reprogramación según las instrucciones del fabricante utilizando un analizador digital mano de CO2 . Uso de una incubadora de baja O2 de los pasos de reprogramación para iPSC exitosa generación.
2. cultivo de fibroblastos para reprogramar
- Preparar los fibroblastos antes de la iniciación de reprogramación (día -2).
- Capa una nueva placa de cultivo de tejidos de 10 cm con 5 mL de 0.1% de gelatina. Toque o agitar la placa para asegurarse de que está cubierta toda la superficie. Incubar por 15 min a 37 ° C. Aspire la gelatina y añadir 10 mL de FEM. conjunto a un lado. No permita que la superficie cubierta de la placa se seque antes de agregar el FEM.
- Aspirar cuidadosamente el medio gastado de los fibroblastos. Enjuague las celdas una vez con 5 mL de DPBS para eliminar el suero residual. Añadir 3 mL de tripsina-EDTA. Muévalo suavemente la placa para asegurar la cobertura completa sobre las células.
- Aspire el exceso de líquido, dejando ~ 500 μl de la tripsina. No aspirar demasiado, ya que esto puede secar y matar a los fibroblastos. Incubar los fibroblastos con tripsina-EDTA durante 3 min a 37 ° C.
- Retire la placa de la incubadora y firme pero suavemente golpee el lado de la placa para desalojar a las células. Compruebe las células bajo el microscopio. Si las células son separadas y flotante, proceder. Si las células están todavía Unidas, incubar durante otros 3 minutos.
- Rápidamente enjuague recopilar las células separadas usando 5 mL de FEM para neutralizar el tripsina-EDTA. Pasar la suspensión de fibroblastos a un tubo cónico de 15 mL. Asegúrese de que las células estén bien mezcladas.
- Mezclar suavemente la suspensión celular para romper grumos grandes de las células, y luego contarlas en un hemocitómetro. Transferencia de 2.5 x 105 células en el plato 10 cm recubierta de gelatina preparada en el paso 2.1.1. Incube las células durante la noche de la 37 ° C, 5% CO2 cultura de tejido regular humidificado incubadora.
Nota: La densidad celular es crítica para lograr alta eficiencia de reprogramación, ya que es importante que los fibroblastos son saludables y dividir rápidamente. Galjanoplastia de 2.5 x 105 células produce una confluencia de 40-60% 2 días más adelante para la mayoría de las muestras del fibroblasto. Ajustar en consecuencia la cantidad de células senescentes o enfermas alcanzar la deseada 40-60% de confluencia 48 h más tarde. - Reemplazar el medio con 10 mL de FEM fresco al día siguiente (día -1).
- La placa de los fibroblastos para iniciar la reprogramación (día 0).
- Verifique que los fibroblastos a introducirse en la confluencia de 40 a 60% (figura 2, día 0). Si las células son excesivo o debajo-confluentes, paso los fibroblastos como se describe en el paso 2.1 y ajustar en consecuencia la densidad de placas. Cultura por otros 2 días.
- Transferir 4 mL de DPBS en un tubo cónico de 15 mL. Añada 100 μl del laminin-521 recombinante humano (rh). Pipetee hacia arriba y hacia abajo para mezclar bien.
- Añada 1 ml por pozo de rhLaminin-521 diluido en 3 pocillos de una placa de 6 pozos. Incube la placa revestida a 37 ° C por 2 h.
- Calentar 6 mL de FEM y 4 mL de placas medio a 37 ° C. Complementar el medio forro con bFGF a una concentración final de 100 ng/mL y B18R a una concentración final de 200 ng/mL.
- Aspirar cuidadosamente el medio gastado de fibroblastos (paso 2.2.1). Lavar las células con 5 mL de DPBS y añadir 3 mL de tripsina-EDTA. Muévalo suavemente la placa para cubrir las células con tripsina-EDTA.
- Aspire el exceso de líquido, dejando ~ 500 μl de tripsina, como en el paso 2.1.3. Incubar los fibroblastos con tripsina-EDTA durante 3 min a 37 ° C. Retire la placa de la incubadora y firme pero suavemente golpee el lado de la placa para desalojar a las células. Compruebe las células bajo el microscopio. Si las células son separadas y flotante, proceder. Si las células están todavía Unidas, incubar durante otros 3 minutos.
- Enjuague/recoger las células separadas usando 5 mL de FEM para neutralizar el tripsina-EDTA. Pasar la suspensión de fibroblastos a un tubo cónico de 15 mL. Asegúrese de que las células están bien mezcladas y contarlas en un hemocitómetro. Pipetear las 12.000 células en 4 mL de medio de placas precalentadas de paso 2.2.4.
Nota: No centrifugar las células en cualquier momento. El número de células plateados se puede ajustar hacia arriba o hacia abajo según se requiera para las líneas o rápida-lenta, respectivamente. - Retire la placa de cubierta de la incubadora y Aspire rhLaminin-521 diluido de los pocillos recubiertos. No permita que la superficie de los pozos se sequen. Resuspender suavemente las células en el medio de la galjanoplastia. Pipetear 1 mL de la suspensión celular en cada cubierto bien (es decir, 3.000 células por pocillo).
- Coloque las células plateadas en una incubadora de cultivo de tejidos de tri-gas con O2 a 5% (bajo-O2). Una vez la placa de, suavemente pero completamente dispersa células alternando entre un movimiento de arriba/abajo y derecha/izquierda. Repita los movimientos 2 veces más. Incube las células durante la noche. No agitar la placa para mezclar.
- Pipetear 4 mL de medio a un tubo cónico de 15 mL de reprogramación y colóquelo en la incubadora O bajo2 con una tapa suelta para equilibrar durante la noche. No agregue bFGF y B18R hasta el día siguiente.
3. Inicio de la reprogramación (día 1)
Nota: Una vez iniciada la reprogramación, mantenimiento diario se requiere aproximadamente 1 mes. Asegúrese de planificar en consecuencia. Todas las incubaciones celular posterior deben ser realizadas bajo condiciones de baja O2 en 37 ° C, 5% CO2, incubadora de cultivo de tejidos de 5% O2 tri-gas humidificado.
- Reemplazar el medio forro por lo menos 1 h antes de la transfección.
- Retire el medio equilibrado de reprogramación de la incubadora O bajo2 . Añadir bFGF a una concentración final de 100 ng/mL y B18R a una concentración final de 200 ng/mL. Mezclar bien.
- Proceder con 1 bien a la vez, utilice una pipeta de 1 mL para retirar el medio gastado y colocar 1 ml de reprogramación suplementado con bFGF y B18R. Repita esto para cada pozo. No use aspiración que excesivamente seca las células, causa estrés y reduce la eficiencia de reprogramación. Mueva hacia atrás la placa con las células en la incubadora O bajo2 .
- Transfectar fibroblastos con 5 μl de reactivo de transfección de RNAiMax por 1 μg de ARNm modificado y 1 μl de reactivo de transfección por 6 pmol de miRNA imita por transfección.
Nota: Los resultados óptimos se obtienen cuando transfecciones proceden para 16-20 h. El reactivo de transfección es diluido 10 x; los 100 ng/μl cóctel modificado mRNA es diluido x 5; y los imitadores de miRNA pmol/μl 5 mix es 8.33 x con el tampón de transfección antes de complejación. Vea la tabla 1 para un resumen de la configuración de transfección.- Mientras prepara la transfección mezcla, minimizar la exposición potencial de los reactivos a Rnasa siguiendo prácticas de laboratorio estándar.
- Equilibrar una alícuota de la solución de transfección (paso 1.3) por aproximadamente 1 h a TA. No utilice un 37 º C baño María o incubadora para calentar el búfer de transfección.
- Quitar una sola alícuota de mRNAs modificado (33 μL) y miRNA imita (14 μL) de-80 ° C y calentarlos RT para aproximadamente 3-5 min, hasta que el descongelado. Descongelar las alícuotas a 37 ° C. Los hace girar hacia abajo brevemente en una microcentrífuga.
- Caliente el reactivo de transfección para RT aproximadamente 3-5 minutos. No use un baño de agua de 37 ° C o incubadora. Invertir el tubo cerrado de 2 a 3 veces para mezclar el reactivo. Girar hacia abajo brevemente en una microcentrífuga.
- Transferir 279 μl del buffer de transfección de RT en un tubo de microcentrífuga libre de Rnasa. Añadir 31 μl del reactivo de transfección para alcanzar un volumen final de 310 μl. mezcla bien por pipeteo. No no agitar con vortex. Incubar el tubo a temperatura ambiente durante 1 minuto.
Nota: Este volumen del reactivo diluido de la transfección es suficiente complejo tanto el mRNA y miRNA modificado imita alícuotas de paso 3.2.3. - Añadir 132 μl del buffer de transfección de RT a la alícuota del 33 μl de ARNm modificado. Pipetee suavemente para mezclar: volumen final es 165 μl.
- Añadir 102,6 μl del buffer de transfección de RT a la alícuota de 14 μl de imitadores de miRNA. Pipetee suavemente para mezclar: volumen final es 116,6 μl.
- Añadir 165 μl del reactivo de transfección diluido del paso 3.2.5 a la diluida modificado mRNA de paso 3.2.6. Pipeta para mezclar: volumen final es 330 μl.
- Añadir 116,6 μl del reactivo diluido de la transfección de paso 3.2.5 a la mezcla de imitadores miRNA diluido del paso 3.2.7. Mezclar bien (el volumen final es 233,2 μL). Incubar por 15 min a temperatura ambiente para permitir que el tampón de la transfección con los mRNAs modificados y miRNA imita.
- Retire la placa con las células de la incubadora O bajo2 . Añada 100 μl (1 μg) de la acomplejada modificado mezcla de transfección de mRNA para cada pozo, gota a gota a través del pozo. Dispersar los complejos de la transfección por suavemente pero bien agitar la placa con un movimiento de arriba/abajo y derecha/izquierda. No agitar la placa para mezclar.
- Añadir 66,7 μl (20 pmol) de la mezcla de transfección miRNA complejado imita a cada pocillo, gota a gota a través del pozo. Dispersar el complejos de transfección por suavemente pero bien agitar la placa con un movimiento de arriba/abajo y derecha/izquierda. No agitar la placa para mezclar.
- Coloque las células transfected en la incubadora de tri-gas con O2 a 5% (bajo-O2). Una vez la placa de, se dispersan los complejos de transfección otra vez por suavemente pero bien agitar la placa con un movimiento de arriba/abajo y derecha/izquierda.
- Pipetear 4 mL de medio de reprogramación en un tubo cónico de 15 mL y colóquelo en la incubadora O bajo2 con tapa suelta para equilibrar durante la noche. No agregue bFGF y B18R hasta el día siguiente.
| Tubo 1 - dilución de RNAiMax (mix 1) | ||
| Reactivo de | Concentración | Volumen |
| Buffer de transfección | 279 ΜL | |
| RNAiMax (Añadir 2 º) | 10 x | 31 ΜL |
| Total: 310 μl | ||
| (incubar 1 minuto a temperatura ambiente) | ||
| Tubo 2 - ARNm modificado (mezcla 2) | ||
| Reactivo de | Concentración | Volumen |
| modificado mRNA de mezcla | 100 ng/μl (5 x) | 33 ΜL |
| Buffer de transfección | ΜL DE 132 | |
| Total: 165 μl | ||
| (añadir un volumen igual de RNAiMax diluido de 1 tubo) | ||
| Tubo 3 - miRNA imitadores (mezclan 3) | ||
| Reactivo de | Concentración | Volumen |
| miRNA imita mix | 5 pmol/μl (8.33 x) | 14 ΜL |
| Buffer de transfección | 102,6 ΜL | |
| Total: 116,6 μl | ||
| (añadir un volumen igual de RNAiMax diluido de 1 tubo) | ||
Tabla 1: Preparación de la mezcla de transfección.
4. sustitución de reprogramación medio entre transfecciones (días 2, 4, 6, 8, 10 y 12)
- Reemplace la transfección posterior mediana 16-20 h según se describe en 3.1.1–3.1.2. Añadir bFGF a una concentración final de 100 ng/mL y B18R a una concentración final de 200 ng/mL en alícuotas de medio reprogramación.
- Monitor mWasabi expresión diariamente para confirmar la calidad de la transfección con un microscopio configurado para visualizar EGFP.
Nota: mWasabi expresión debe ser mínimamente aparente en el día 2 y aumento de brillo con cada transfección adicional.
5. otros-diario transfecciones (días 3, 5, 7, 9, 11 y 13)
- Realizar la transfección como se describe en el paso 3.2. No cambie el medio en los días de la transfección.
- Preparar una alícuota de 4 mL de medio de reprogramación y colóquelo en la incubadora O bajo2 para equilibrar el cambio medio en el día siguiente. No agregue bFGF y B18R hasta el día siguiente.
6. procedimientos a realizar después de la Final de la transfección
- Realizar cambios de media diarias desde el día 14 a través de aproximadamente día 17. Caliente 7 mL de reprogramación medio a 37 ° C. Añadir bFGF a una concentración final de 100 ng/mL.
Nota: B18R no es necesario después de la final de la transfección. Más allá del día 14, ya no es necesario equilibrar el medio durante la noche en la incubadora O bajo2 . - Retire el medio de todos los pozos con una pipeta serológica. Seguir evitar el uso de la aspiración. Añadir 2 mL de medio suplementado con bFGF por pozo de reprogramación.
- Los días 17 y 18, analizar los pozos reprogramados en un microscopio invertido o disección. Si las colonias se forman cerca uno al otro debido a la alta eficiencia de reprogramación y no se puede aisladas manualmente, separar las colonias mediante la realización de un pases opcional de pozos reprogramadas que se describe en el paso 7 a continuación. Si las colonias son escasas y bien separados, elegir manualmente los clones directamente en el reprogramado como se describe en el paso 8 y subcultura iPSCs según protocolos estándar.
7. procedimiento opcional: Pases de células reprogramadas pozos utilizando EDTA
- Preparar 0,5 mM EDTA (EDTA) en DPBS diluyendo la solución madre de EDTA de 0,5 M. Filtro de esterilización EDTA utilizando un sistema de filtración de vacío de 0,22 μm.
- Prepare el soporte de la célula de vástago (PSC) de pluripotentes libre de alimentador (por ejemplo, mTeSR1) según las instrucciones del fabricante. Caliente previamente 32 mL de medio de PSC a 37 ° C.
- Cubra todos los pozos de dos placas de formato 6-bien con hESC-calificado de matriz extracelular (ECM) siguiendo las instrucciones del fabricante. Sellar las placas con la película de parafina e Incube por 1 h a TA.
- Aspirar la solución ECM de las placas precalentadas y reemplazarlo con 2 mL de medio PSC por pozo. No permita que la superficie de los pozos se sequen. Colóquelos a un lado.
- Aspirar el medio de la reprogramación de 2 pozos reprogramadas en un día cuando las colonias son grandes y claramente formado (generalmente día 18). Enjuague una vez con 1 mL de EDTA y aspirado.
- Añadir 1 mL de EDTA por pozo. Incubar durante 4 min a 37 ° C.
- Suavemente Retire la placa de la incubadora y coloque en la bioseguridad gabinete.
Nota: en este punto, las células pueden ser adheridos muy libremente y fácilmente desalojada. - Aspirar cuidadosamente el EDTA de ambos pozos. Añadir 3 mL de medio PSC precalentado a cada uno bien tratada con EDTA. Use un raspador de célula para suavemente pero completamente desalojar las células de ambos pozos.
- Utilice una pipeta serológica para pipetear la suspensión de células y romper grumos grandes. No pipetear hasta que todos los grupos de células se han ido. Conservar grupos de iPSC.
Nota: Galjanoplastia grumos grandes no afectará a consecuencia de Colonia de iPSC. Si existen grupos excesivamente grandes de la célula, simplemente evitar la transferencia en el siguiente plato. - Con una pipeta serológica, distribuir las células de cada pozo tratada con EDTA por pipetear 0.5 mL en cada pocillo de la placa de la pozo 6 revestida de ECM.
Nota: No combinar las células de los pozos reprogramadas (es decir, reprogramadas bien 1 debe distribuirse uniformemente en la primera placa de 6 pozos y reprogramado 2 bien debe distribuirse uniformemente en la segunda placa de 6 pozos). - Volver las células plateadas a la incubadora O bajo2 . Para distribuir uniformemente las células, agitar cada placa y hacia atrás y de lado a lado. Remolino no. Reemplazar el medio PSC diariamente.
8. cosecha iPSC colonias
- Caliente previamente 15 mL de medio PSC a 37 ° C. Cubrir todos los pocillos de una placa de 6 pozos solo con ECM hESCs cualificado siguiendo las instrucciones del fabricante. Sellar las placas con la película de parafina e Incube por 1 h a TA.
- Aspire el medio de cultivo de los pozos se utiliza para recoger iPSCs. Enjuague una vez con 1 mL de EDTA y aspirado. Añadir 1 mL de EDTA por pozo. Incubar durante 4 min a 37° C. Mientras que las células están incubando, aspirar la solución ECM de las placas precalentadas y reemplazar con 2 mL de medio PSC por pozo. Fijar las placas a un lado.
- Suavemente Retire la placa con iPSCs para ser recogido de la incubadora y coloque en la bioseguridad gabinete.
Nota: en este punto, las células pueden ser adheridos muy libremente y fácilmente desalojada. - Aspirar cuidadosamente el EDTA. Muy suavemente, añadir 3 mL de medio precalentado de PSC, teniendo cuidado de no para desalojar las colonias de iPSC.
- Mover la placa a un invertido o disección alcance para mejor visualizar las colonias. Preparar una pipeta de 1 mL con una punta estéril. Presione el émbolo completamente, después utilizar la punta para raspar suavemente una colonia mientras poco a poco líquido en la punta para recoger la Colonia. Drenaje como medio poco como sea posible mientras la colonia de iPSC.
- Para transferir la Colonia, pipetear arriba y abajo 3-4 veces en un solo pozo de la placa revestida de ECM de paso 8.2. Repita hasta 6 colonias han sido recogidas y transferidos en pocillos individuales. Elegir no más de 2 colonias de cualquier bien individual si un pases opcional que se describe en el paso 7 se ha realizado.
- Utilizar un protocolo estándar humano célula de vástago para congelar a todos los pozos restantes. Descongelar y volver a placa acciones congeladas Si más colonias deben ser recogidas posteriormente.
9. Caracterización de iPSCs
- Realizar TRA-1-60 de tinción para análisis de reprogramación eficiencia (día 18).
Nota: 2 de 3 pozos reprogramadas son pasados para cosecha futura Colonia. Los restantes pueden ser utilizados para la tinción de TRA-1-60. Este procedimiento puede realizarse en la mesa de laboratorio.- Lave el reprogramado con 1 mL de PBS. Fijar las células con metanol helado a-20 ° C durante 5 minutos.
- Aspire metanol. Seco bien durante aproximadamente 2 minutos tenga cuidado de no secar demasiado. Las células han secado lo suficiente cuando se convierten en un color transparente/mate.
- Lave el bien 3 veces con 1 mL de PBS durante 5 minutos con agitación suave. Durante los lavados, preparar 3 mL de peróxido de hidrógeno 3% en PBS.
- Aspire el PBS. Añadir 2 mL de solución diluida de peróxido. Incubar por 15 min a temperatura ambiente con agitación suave. Preparar 4 mL de la solución de bloqueo: 10% albúmina de suero bovino (BSA) en PBS.
- Aspire la solución de peróxido. Lave bien 2 veces con 1 mL de PBS durante 5 minutos.
- Aspire PBS y añadir 2 mL de solución de bloqueo en el pozo. Incubar durante 1 h a TA.
- Lave el bien 3 veces con 1 mL de PBS durante 5 minutos con agitación suave.
- Diluir el anticuerpo primario anti-TRA-1-60 al 1: 100 en la solución de bloqueo con 0.05% de azida sódica. Preparar 1 mL de la dilución del anticuerpo 1 bien de un plato de formato 6-bien. Agregar la dilución de anticuerpo al pozo y envolver el plato con parafilm para evitar la evaporación. Incubar durante una noche a 4 ° C con agitación suave.
Nota: Si es necesario, puede hacerse la incubación con el anticuerpo primario por 1 h a temperatura ambiente con agitación suave. La dilución del anticuerpo primario puede ser reutilizada hasta 5 veces. - Tras la incubación con el anticuerpo primario, lave el bien 3 veces con 1 mL de PBS durante 5 minutos con agitación suave.
- Diluir el anticuerpo conjugado con HRP de anti-ratón secundario en el 1: 200 en la solución de bloqueo. Incubar el pozo con la dilución del anticuerpo secundario por 2 h a temperatura ambiente con agitación suave.
- La dilución del anticuerpo secundario de aspirar y lavar el bien 3 veces con 1 mL de PBS durante 5 minutos con agitación suave.
- Durante el tercer lavado, preparar la solución de substrato siguiendo las instrucciones del fabricante. Después del último lavado, Aspire PBS. Añadir 1 mL de la solución sustrato e incubar hasta que el color deseado convierte (aproximadamente 10 min).
- Aspirar la solución de sustrato. Enjuague bien con agua durante 5 minutos agitando suavemente.
- Contar las colonias, si lo desea. Reprogramación de eficiencia = (número de colonias) / (número de fibroblastos plateados) x 100%.
- Para el almacenamiento a largo plazo, aspirar el agua de la placa teñida y deje secar al aire durante la noche en el sello de excelencia la placa seca con parafilm y almacenar a 4 ° C hasta por 2 años evaluar la eficiencia de reprogramación más adelante.
Resultados
Por lo general tarda aproximadamente 5-6 semanas desde el inicio de la reprogramación de fibroblastos a la congelación de los primeros frascos de iPSCs (figura 1). El protocolo de reprogramación se puede categorizar generalmente en dos fases. Fase 1 incluye el cultivo de fibroblastos y siete transfecciones, con el ARN reprograma cóctel realizados cada 48 h. fase 2 incluye aislamiento, expansión y caracterización de las colonias de iPSC.
Antes de iniciar el protocolo, debe garantizarse que los fibroblastos a introducirse son de buena calidad. Fibroblastos sanos deben aparecer en forma de huso, bipolar y refractile con un tiempo de duplicación de aproximadamente 24 horas. Día 0, 250.000 células plateadas en un plato de 10 cm en el día -2 deben crecer hasta confluencia de 40 a 60% (figura 1, día 0) y rinden aproximadamente 6 – 10 x 105 células. Células proliferando a un ritmo más lento pueden compensarse por la galjanoplastia en una densidad más alta en el día -2 y en el día 0 para reprogramación.
Fibroblastos deben aparecer muy escasas placas siguientes en un pozo de un plato bien 6 formato de reprogramación (día 1,figura 2). Veinticuatro horas después de la primera de la transfección, fibroblastos perderá su forma de huso y adoptar una morfología más redondeada (figura 2, día 2), que se mantiene a través del resto de reprogramación. Fluorescencia verde de mWasabi mRNA debe mínimamente observables en el día 2 y constante aumento de brillo para ser claramente visible por el día 4. La capacidad para detectar la fluorescencia mWasabi puede depender de la configuración y alcance sensibilidad. Densidad celular constantemente y gradualmente aumentará a lo largo de los tres primeros transfecciones (días 1-6), con una aparente explosión en proliferación que ocurre entre los días 7 y 8. Las células deben aparecer en gran parte confluentes por día 10 (figura 2, día 10). Las primeras colonias de iPSC pueden aparecer tan temprano como el día 11 (figura 2, el día 11); sin embargo, las colonias pueden no ser observables hasta tan tarde como el día 18. Generalmente, días 15 a 18, habrá colonias de iPSC grande y obvio que son claramente distintas de las circundantes, incompleto reprogramados fibroblastos (figura 2, el día 15 y la figura 3, día 17). Immunostaining para el marcador de pluripotencia TRA-1-60 puede realizarse para evaluar la eficiencia de reprogramación (figura 3, día 17, TRA-1-60). En nuestra experiencia, la mayoría líneas de fibroblastos producen cientos de colonias por reprogramado bien (figura 3, recuadro B).
Densidad de placas subóptima es la razón más común para la reducida eficiencia de reprogramación en nuestro protocolo y se asocia frecuentemente con fibroblastos enfermos, senescente o paso alto. Si placas de densidad es muy baja, habrá grandes manchas acelulares estériles al final de reprogramación (figura 4), y colonias de iPSC no pueden formar (figura 4). Células reprogramadas deben ser muy confluentes día 14 (Comparar Figura 4A y 4B a figura 2, día 14). Del mismo modo, si las células son demasiado densamente plateadas o proliferan demasiado rápido, eficiencia de reprogramación se reduce dramáticamente.
Para mantener la homogeneidad de iPSCs derivados del paciente, es importante ampliar líneas de células de una colonia única. Puesto que reprogramación eficiencia es muy alta en nuestro protocolo, las vecinas colonias iPSC pueden formar en proximidad cercana a y crecer en cada otra (figura 3, días 15 – 17). Esto a veces hace difícil separar mecánicamente una sola Colonia de expansión clonal. Hemos encontrado que es útil primer paso un reprogramado bien y diluir en un área más grande de la cultura. Una relación de buen paso consiste en dividir uniformemente una placa 6-pozo-formato reprogramada bien a través de una placa de 6 pozos toda.
Tras el paso de la dilución, iPSCs crecen como colonias y son fácilmente distinguibles de los fibroblastos (figura 5, el día 18). Inicialmente, iPSC colonias pueden ser embaladas libremente, y las células individuales tienen un área citoplásmico relativamente grande. En el transcurso de 4 a 7 días, las iPSCs proliferan y forman una colonia característica apretada con bordes definidos. Las células individuales dentro de la colonia tienen una fracción nuclear grandes con nucleolos prominentes (figura 5, el día 22). Debe haber muchas colonias que se forman en cada pozo, y sólo aquellos con morfología de iPSC classic deben ser recogidos para la expansión.
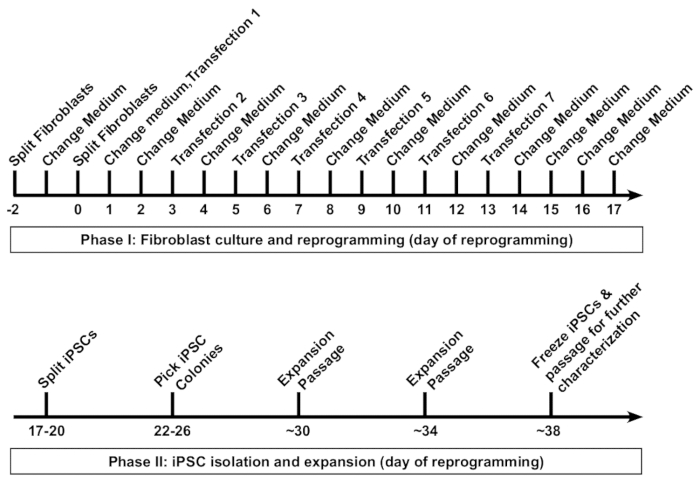
Figura 1 : Reprogramación de fibroblastos humanos en inducida por células madre pluripotentes (iPSCs). Se presenta un protocolo de manual de reparacion para la reprogramación de fibroblastos humanos. Los fibroblastos son pasados primero a una baja densidad en un pozo de un plato de formato 6-bien, seguido por siete transfecciones realizadas a intervalos de 48 h. Medio es reemplazado 16 – 20 h después de cada transfección. IPSCs reprogramadas son pasados primero en colonias 18 y clonal de aproximadamente día son recogidas por día 26. Por lo general, líneas de iPSC derivados del fibroblasto pueden ser congeladas para almacenamiento a largo plazo por día 38. Haga clic aquí para ver una versión más grande de esta figura.
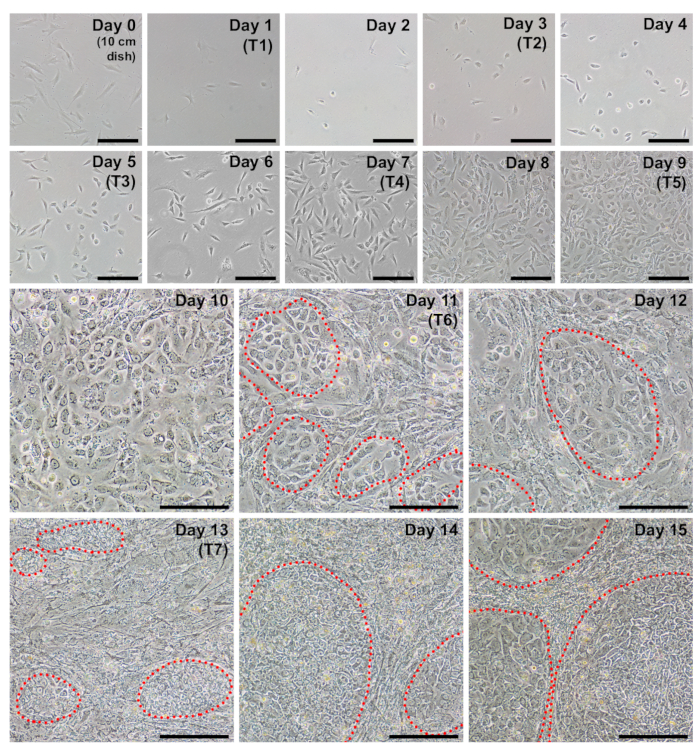
Figura 2 : Representante imágenes diarias durante cada día de reprogramación. Fibroblastos deben ser aproximadamente de 40 – 60% confluente en el paso para iniciar la reprogramación (día 0, las células se platean en un plato de 10 cm). La transfección primera (T1) se produce el día 1, y las células deben aparecer muy escasas en este punto. Al día siguiente (día 2), una morfología más redondeada debería ser evidente. Las células continuará aumentando en densidad a lo largo del Protocolo con racimos de iPSC comienzan a aparecer tan pronto como el día 11 (un círculo rojo). Día 15, iPSC colonias será grandes con límites discretos. Barra de escala = 200 μm. haga clic aquí para ver una versión más grande de esta figura.
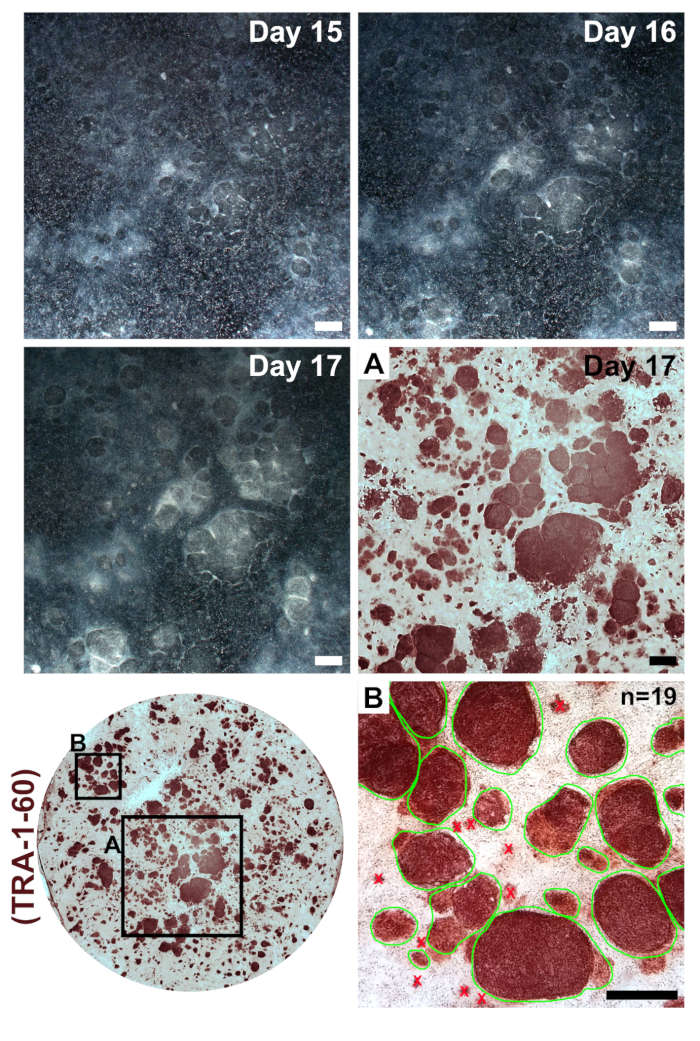
Figura 3 : Formación de la Colonia después de transfecciones con reprogramación modificado mRNAs y miRNA imita. Bajo magnificación imágenes fueron tomadas de un representante de reprogramación en los días 15 y 17. Tras el final de la transfección, reprogramadas iPSCs formarán colonias claras con límites definidos que expansión en tamaño y condensan para llegar a ser claramente distinta de incompleto reprogramados fibroblastos circundantes. Immunostaining para el marcador de pluripotencia TRA-1-60 indica la presencia de iPSCs (recuadro A) y puede ser utilizado para el cálculo de la eficiencia de reprogramación por contar todas las colonias dentro de un pozo único (recuadro B, ejemplos de colonias contables un círculo verde). Barra de escala = 1 mm. haga clic aquí para ver una versión más grande de esta figura.
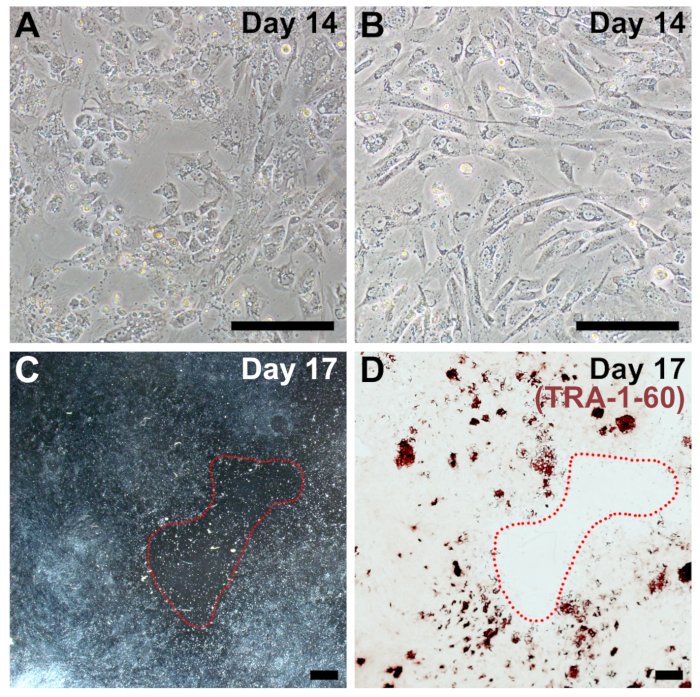
Figura 4 : Imágenes representativas de la densidad de placas óptimo para reprogramación. (A, B) Ejemplos de los fibroblastos que son demasiado escasos durante el día 14 de reprogramación (Comparar figura 2, día 14). (C) imagen de ampliación baja el día 17 de reprogramación con un parche grande, árido en un círculo en rojo. (D) el pozo mismo era fijo y mancharon para que el TRA-1-60 confirmar los pobres total reprogramación eficiencia debido a la densidad celular baja. Barras de escala = 200 μm (A, B) y 1 mm (C, D). Haga clic aquí para ver una versión más grande de esta figura.
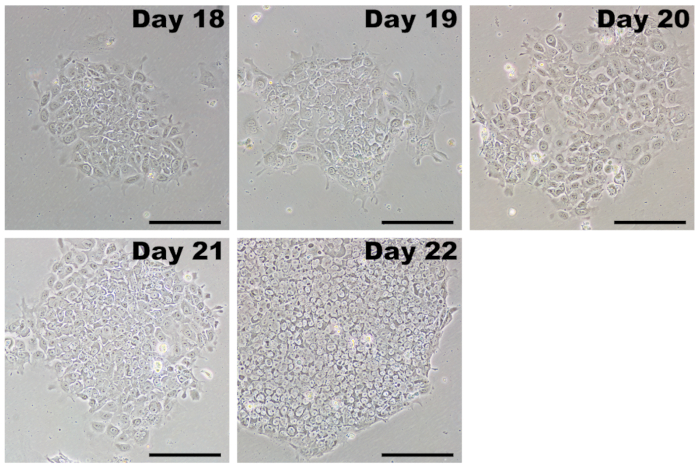
Figura 5 : Imágenes representativas de iPSCs después inicial paso. Después de completar las transfecciones con reprogramación mRNAs modificado y miRNA imitadores, las células reprogramadas son pasados por los días 17-20. iPSCs tienen una ventaja de crecimiento en medio PSC y superar rápidamente cualquier fibroblastos que fueron reprogramados. Inicialmente, iPSCs forman colonias que pueden aparecer con bordes mal definidos. Dentro de varios días, las células rápidamente proliferan y toman a la morfología característica de células apretadas con una alta relación núcleo-citoplasma, clustering firmemente en colonias con distintas fronteras. Barra de escala = 200 μm. haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este protocolo describe un método clínicamente relevantes, no integración, basada en el RNA que permite la reprogramación de las líneas de fibroblastos humanos normales y enfermedad asociada en iPSCs con una ultra alta eficiencia. Hasta la fecha, cada línea de fibroblastos humanos que hemos tratado de reprogramar con el protocolo descrito ha rendido un número satisfactorio de iPSCs de alta calidad para aplicaciones posteriores. IPSCs resultante inmediatamente puede transferir y ampliado en condiciones de cultivo libre de alimentador.
Calidad de los fibroblastos para la reprogramación:
Reprogramación de éxito es fuertemente dependiente de la calidad de a partir de fibroblastos. Idealmente, reprogramación se debe iniciar con los fibroblastos de pasaje más bajo disponibles para alcanzar la mayor eficiencia. Eficiencia de reprogramación es mejor con fibroblastos de pasaje 2 – 4. Reprogramación de todavía puede trabajar con alto-paso (paso 5 – 8), incluso senescentes fibroblastos, aunque con una eficacia reducida. A veces los fibroblastos pasaje baja son de carácter o muestras de los pacientes tienen una mutación genética que impide el crecimiento sano. En este caso, optimización de la densidad de placas inicial puede ser requerido. La reprogramación de líneas comprometidas del fibroblasto se asocia generalmente a muerte creciente de la célula en transfecciones de RNA. Como resultado, las células en la reprogramación bien parecerá estar escasos por 10 – 14 días de reprogramación. Grandes áreas acelulares también será visibles en el pozo. Si este es el caso, el protocolo de reprogramación tendrá que ser nuevamente iniciados con un mayor número inicial a partir de fibroblastos. Galjanoplastia 3.000 entradas células por pocillo de un plato bien 6 formato trabaja constantemente para adultas más líneas de fibroblastos. Sin embargo, aumentar el número de placas a 5.000-10.000 (50.000 líneas senescentes) puede ayudar a mejorar la reprogramación de las muestras asociadas a la enfermedad, como ha sido reportado en nuestra anterior publicación8. Por el contrario, las células alcanzan confluencia demasiado pronto también pueden ser resistentes a la reprogramación. Si las células que experimentan transfecciones con reprogramación RNAs proliferan demasiado rápido (como ocurre a veces con fibroblastos neonatales primarios), iniciar la reprogramación de 500 fibroblastos por pozo de un pozo de 6 formato plato8.
Manejo de la memoria intermedia de transfección:
El pH del buffer de transfección (suero reducido medio ajustado a pH de 8.2) es fundamental para lograr la eficiencia de transfección óptima requerida para este protocolo de reprogramación. Por esta razón, se recomiendan varias precauciones en relación con el manejo del buffer de transfección. Hemos encontrado que incluso corta exposición del búfer de la transfección a aire atmosférico afecta el pH del buffer. Por lo tanto, el búfer de transfección debe alícuotas en un recipiente de tapón de rosca con un mínimo espacio de aire (usamos 5 ó 15 mL tapón cónico los tubos). Para minimizar aún más la exposición al aire, utilizar cada búfer de transfección alícuota hasta un máximo de dos transfecciones. Por último, desde pH de efectos de temperatura, es fundamental que el búfer de la transfección es equilibrado a RT para el ensamblaje de los complejos de transfección. Se recomienda no calentar el búfer de transfección a 37 ° C.
Pases de iPSCs:
Mientras que muchos anteriormente publicados protocolos recomiendan escoger colonias individuales de iPSC en el extremo de reprogramación, esto puede ser difícil de lograr cuando la eficiencia de reprogramación es muy alta o si las colonias se agrupan juntos, como sucede a menudo en nuestra Protocolo. Por lo tanto, si iPSC colonias están cerca uno al otro, le recomendamos que primero pases las células repartidas iPSCs reprogramado antes de la recolección manual colonias. Hay varias ventajas al realizar este paso paso temprano. Extendiéndose a las colonias les da más espacio para crecer, produciendo colonias mucho más grandes para recoger de otra manera podría ser alcanzado en el pozo original. Esto mejora la tasa de éxito en el establecimiento de una línea de iPSC de un clon seleccionado. También encontramos que el tiempo de la cultura adicional con fibroblastos, aunque en una proporción de diluido, parece mejorar la calidad promedio de colonias de iPSC escogido. Los fibroblastos incompleto reprogramados pueden proporcionar apoyo factores paracrinos, que siguen para ayudar a establecer las iPSCs y facilitar la transición directa en condiciones de cultivo de célula libre de alimentador. Fibroblastos por casualidad tienen una desventaja selectiva del crecimiento comparada con iPSCs cuando cultivadas en mTeSR1. Por lo tanto, la población de células de fibroblastos contaminante se diluye rápidamente a cantidades insignificantes dentro de 3 o 4 pasos.
Inhibidores de la roca como Y-27632 se utilizan con frecuencia para el cultivo rutinario de iPSCs humana. Hemos encontrado que frecuente y extendida cultura de algunas líneas de iPSC con Y-27632 puede tener efectos deletéreos sobre la calidad en general. Cuando se utiliza un grupo de pases de método, como con EDTA, Y-27632 no es necesario para mantener la viabilidad de iPSC después de partir. Hemos eliminado completamente medios que suplen con Y-27632 de todo aislamiento de iPSC, expansión o cultura rutinaria.
Limitaciones del Protocolo:
Una limitación del enfoque de reprogramación descrito basado en RNA es el costo inicial y la complejidad asociados con la preparación de los reactivos de reprogramación. Aunque los procedimientos preparatorios para generar mRNA reactivos son todas de la rutina y han sido previamente describen (PCR, transcripción en vitro , tratamiento de DNasa, tapado, desfosforilación, purificación), acumulativamente la producción de reactivos de mRNA es un proceso relativamente largo y no trivial. El otro reto importante de este protocolo es la necesidad de transfectar células cada 48 h, incrementando la intensidad de trabajo del Protocolo de reprogramación. Estas consideraciones pueden ser prohibitivas si se desea la reprogramación sólo unos pocos de muestras de pacientes. Sin embargo, si la consideración primaria es la generación de iPSCs clínicamente relevante o muy alta eficiencia de reprogramación, el enfoque de reprogramación descrito basado en el RNA es ideal.
En Resumen, el alta eficiencia basado en RNA reprogramación método descrito se basa en eficiencia de transfección optimizado del tipo de célula somática a introducirse como se describe en nuestra anterior publicación8. El protocolo de la transfección de RNA presentado en este estudio es altamente sintonizado para fibroblastos primarios humanos pero potencialmente puede adaptarse a otros tipos celulares para mejorar la eficiencia de reprogramación de células somáticas diversas.
Divulgaciones
I.K. y G.B. son inventores Co en una patente titulada "Métodos y composiciones para reprogramación de células", no. de solicitud PCT PCT/US2016/063258.
Agradecimientos
Estamos agradecidos por el apoyo financiero de los institutos nacionales de salud (T32AR007411-33) y el centro de la Universidad de Colorado piel enfermedades investigación Core (P30AR057212). También agradecemos a la Asociación de investigación de epidermolisis Bullosa (EB), Fundación de investigación médica de EB, Cure EB caridad, distrófico de la epidermolisis investigación Asociación (DEBRA) internacional, de la Fundación Rey Balduino Vlinderkindje fondo y puertas Fondo de las fronteras.
Materiales
| Name | Company | Catalog Number | Comments |
| Plasmid templates for PCR | |||
| pcDNA3.3_KLF4 | Addgene | 26815 | |
| pcDNA3.3_SOX2 | Addgene | 26817 | |
| pcDNA3.3_c-MYC | Addgene | 26818 | |
| pcDNA3.3_LIN28A | Addgene | 26819 | |
| pCR-Blunt_hM3O | Addgene | 112638 | |
| pCR-Blunt_hNANOG | Addgene | 112639 | |
| pCR-Blunt_mWasabi | Addgene | 112640 | |
| Modified mRNA in vitro transcription and miRNA mimics | |||
| Forward Primer | Integrated DNA Technologies | TTGGACCCTCGTACAGAAGC TAATACG | |
| Reverse Primer (Ordered as ultramer, 4nmol scale) | Integrated DNA Technologies | TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT TTTTTTTTTTTTTTTTTTTTTTTT CTTCCTACTCAGGCTTTATTCA AAGACCA | |
| (ARCA Cap) 3´-0-Me-m7G(5')ppp(5')G | New England Biolabs | S1411S | |
| Pfu Ultra II Hotstart 2x Master Mix | Agilent | 600850-51 | |
| 5-Methylcytidine-5'-Triphosphate | Trilink Biotechnologies | N-1014 | |
| Antarctic Phosphatase | New England Biolabs | M0289L | |
| DNase I | NEB | M0303S | |
| MEGAscript T7 Transcription Kit | ThermoFisher Scientific | AM1334 | |
| Pseudouridine-5'-Triphosphate | Trilink Biotechnologies | N-1019 | |
| Riboguard RNase Inhibitor | Lucigen | RG90910K | |
| RNA Clean & Concentrator | ZymoResearch | R1019 | |
| Syn-hsa-miR-302a-3p miScript miRNA Mimic | Qiagen | MSY0000684 | |
| Syn-hsa-miR-302b-3p miScript miRNA Mimic | Qiagen | MSY0000715 | |
| Syn-hsa-miR-302c-3p miScript miRNA Mimic | Qiagen | MSY0000717 | |
| Syn-hsa-miR-302d-3p miScript miRNA Mimic | Qiagen | MSY0000718 | |
| Syn-hsa-miR-367-3p miScript miRNA Mimic | Qiagen | MSY0000719 | |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9937 | Use to dilute modified mRNAs and miRNA mimics |
| Fibroblast culture and reprogramming | |||
| 0.1% Gelatin in H2O | StemCell technologies | #07903 | |
| Stericup-GV Sterile Vacuum Filtration System | EMD Millipore | SCGVU05RE | Use to sterilize the transfection buffer and 0.5 mM EDTA in DPBS |
| 2-Mercaptoethanol | ThermoFisher Scientific | 21985023 | |
| 6-well plates (tissue culture treated) | Corning | 3516 | |
| DMEM/F12 | ThermoFisher Scientific | 11320033 | |
| Fetal Bovine Serum | Fisher | 26-140-079 | |
| FGF Basic | ThermoFisher Scientific | phg0263 | |
| GlutaMax Supplement | ThermoFisher Scientific | 35050061 | Glutamine supplement used for the prepration of media |
| Heat Inactivated FBS | Gibco Technologies | 10438026 | |
| KnockOut Serum Replacement | ThermoFisher Scientific | 10828010 | |
| Lipofectamine RNAiMAX Transfection Reagent | ThermoFisher Scientific | 13778500 | The protocol is optimized for the Lipofectamine RNAiMax transfection reagent |
| MEM | ThermoFisher Scientific | 11095080 | |
| MEM Non-essential amino acids | ThermoFisher Scientific | 11140050 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985070 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 500 mL |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 31985062 | Reduced-serum medium, use to make the transfection buffer by adjusting the pH to 8.2, 100 mL |
| 10 M NaOH | Sigma-Aldrich | 72068 | Make a 1 M solution by diluting in nuclease free water and use for pH adjustment |
| Water (Nuclease free, Not DEPC-treated) | Fisher | AM9932 | Use to dilute NaOH and wash a pH meter |
| Pen/Strep/Fungizone | GE Healthcare | SV30079.01 | |
| rhLaminin-521 | ThermoFisher Scientific | A29248 | Supplied at a concentration of 100 µg/mL, use as a matrix for the reprogramming procedure |
| Dulbecco's phosphate-buffered saline (DPBS) | Life Technologies | 14190144 | |
| Trypsin-EDTA 0.25% Phenol Red | Life Technologies | 2520056 | |
| Vaccinia Virus B18R (CF) | ThermoFisher Scientific | 34-8185-86 | |
| iPSC culture | |||
| Corning Matrigel hESC-Qualified Matrix | Corning | 354277 | Extracellular matrix (ECM) for culturing iPSCs |
| EDTA, 0.5 M stock solution | K&D Medical | RGF-3130 | Dilute to 0.5 mM in DPBS, filter sterilize and use for iPSC passaging |
| mTeSR1 | StemCell Technologies | 85850 | Pluripotent stem cell (PSC) medium, provides growth advantage to iPSCs over fibroblasts |
| Antibodies and Detection | |||
| Rabbit anti Mouse (HRP conjugated) | Abcam | ab97046 | |
| Tra-1-60 (mouse anti human) | Stemgent | 09-0010 | |
| Hydrogen Peroxide (30%) | LabChem | LC154301 | Dilute to 3% with PBS |
| Bovine Serum Albumin (BSA) | Fisher | BP9703100 | |
| Phosphate-buffered salin (PBS) | Hyclone | SH30258.02 | Supplied as 10x, dilute to 1x |
| VECTOR NovaRED Peroxidase Substrate Kit | Vector Laboratories | SK-4800 | |
| Special Equipment | |||
| Description | Notes | ||
| Biological safety cabinet | |||
| Regular humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 37 °C. | ||
| Tri-gas humidified tissue culture incubator | Calibrate CO2 using digital meter, fyrite, or equivalent. Equilibrate incubator to 5% CO2, 5% O2, 37 °C. Use for the reprogramming procedure. | ||
| pH Meter | Must have resolution to two decimal places. Designate to RNA work if possible. | ||
| Inverted microscope | Microscope configured to visualize EGFP for monitoring transfection efficiency. |
Referencias
- Takahashi, K., Yamanaka, S. Induction of Pluripotent Stem Cells from Mouse Embryonic and Adult Fibroblast Cultures by Defined Factors. Cell. 126 (4), 663-676 (2006).
- Takahashi, K., et al. Induction of Pluripotent Stem Cells from Adult Human Fibroblasts by Defined Factors. Cell. 131 (5), 861-872 (2007).
- Schlaeger, T. M., et al. A comparison of non-integrating reprogramming methods. Nature Biotechnology. 33 (1), 58-63 (2015).
- Hacein-Bey-Abina, S., et al. LMO2-Associated Clonal T Cell Proliferation in Two Patients after Gene Therapy for SCID-X1. Science. 302 (5644), 415-419 (2003).
- Warren, L., et al. Highly efficient reprogramming to pluripotency and directed differentiation of human cells with synthetic modified mRNA. Cell Stem Cell. 7 (5), 618-630 (2010).
- Judson, R. L., Babiarz, J. E., Venere, M., Blelloch, R. Embryonic stem cell-specific microRNAs promote induced pluripotency. Nature Biotechnology. 27 (5), 459-461 (2009).
- Anokye-Danso, F., et al. Highly efficient miRNA-mediated reprogramming of mouse and human somatic cells to pluripotency. Cell Stem Cell. 8 (4), 376-388 (2011).
- Kogut, I., et al. High-efficiency RNA-based reprogramming of human primary fibroblasts. Nature Communications. 9 (1), (2018).
- Hirai, H., et al. Radical acceleration of nuclear reprogramming by chromatin remodeling with the transactivation domain of MyoD. Stem Cells. 29 (9), 1349-1361 (2011).
- Mandal, P. K., Rossi, D. J. Reprogramming human fibroblasts to pluripotency using modified mRNA. Nature Protocols. 8 (3), 568-582 (2013).
- Avci-Adali, M., et al. In vitro synthesis of modified mRNA for induction of protein expression in human cells. Journal of Visualized Experiments. , e51943 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados