Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Uso en transferencia de energía de resonancia de la fluorescencia de Vitro para el estudio de la dinámica de los complejos de la proteína en una escala de tiempo de milisegundos
En este artículo
Resumen
Las interacciones proteína-proteína son críticas para los sistemas biológicos, y estudios de la cinética de Unión proporcionan penetraciones en la dinámica y función de complejos de la proteína. Se describe un método que cuantifica los parámetros cinéticos de una proteína compleja mediante la transferencia de energía de resonancia de la fluorescencia y la técnica de flujo detenido.
Resumen
Las proteínas son los principales operadores de los sistemas biológicos, y generalmente interactúan con otros macro o pequeñas moléculas para llevar a cabo sus funciones biológicas. Estas interacciones pueden ser altamente dinámicas, lo que significa las subunidades interactúan constantemente asociadas y disociadas en determinados tipos. Medir la afinidad usando técnicas tales como desplegable cuantitativa revela la fuerza de la interacción, estudiando la cinética de Unión proporciona penetraciones en cómo rápidamente se produce la interacción y cuánto tiempo puede existir cada complejo. Además, medir la cinética de la interacción en presencia de un factor adicional, como un factor de intercambio de proteínas o una droga, ayuda a revelar el mecanismo por el cual la interacción está regulada por el factor conocimiento importante para la avance de la investigación biológica y médica. Aquí, describimos un protocolo para la medición de la cinética de unión de una proteína compleja que tiene una tasa alta asociación intrínseca y puede disociarse rápidamente por otra proteína. El método utiliza la transferencia de energía de resonancia de la fluorescencia para divulgar la formación del complejo de la proteína in vitro, y permite la rápida asociación y disociación del complejo en tiempo real en un Fluorímetro de flujo detenido. Usando este análisis, se cuantifican las constantes de tarifa de asociación y disociación de la proteína del complejo.
Introducción
En última instancia se llevan a cabo actividades biológicas de proteínas, más que interactuar con otros para las funciones biológicas apropiadas. Usando un enfoque computacional, el importe total de las interacciones proteína-proteína en humanos se estima que 650.000 ~1, y la interrupción de estas interacciones a menudo conduce a enfermedades2. Debido a su papel esencial en el control de procesos celulares y organismos, se han desarrollado numerosos métodos para estudiar interacciones de proteínas, tales como levadura dos-híbrido, complementación bimolecular de la fluorescencia, split-luciferase complementación y co-inmunoprecipitación ensayo3. Mientras que estos métodos son buenos para descubrir y confirmar las interacciones proteína-proteína, son generalmente no-cuantitativos y así proporcionar información limitada sobre la afinidad entre los socios de proteínas interactuantes. Cuantitativas desplegables pueden utilizarse para medir la afinidad de unión (por ejemplo, la constante de disociación Kd), pero no mide la cinética de la Unión, ni se puede aplicar cuando el Kd es muy baja debido a una inadecuada relación señal a ruido4. Espectroscopia de la resonancia (SPR) de plasmón superficial cuantifica la cinética de Unión, pero se requiere una superficie específica y la inmovilización de un reactivo en la superficie, que puede potencialmente cambiar la propiedad de la fijación del reactivo5. Por otra parte, es difícil para el SPR medir rápida asociación y disociación tarifas5y no es apropiado utilizar SPR para caracterizar el evento de intercambio de subunidades de la proteína en un complejo proteico. Aquí, describimos un método que permite medir las tasas de proteínas complejo montaje y desmontaje en una escala de tiempo de milisegundos. Este método era fundamental para determinar el papel de Cullin -unsociado -Nedd8 -dissociated proteína 1 (Cand1) como el F-box proteína exchange factor6,7.
Cand1 regula la dinámica de Skp1•Cul1•F-caja proteina (SCF) E3 ligasas, que pertenecen a la gran familia de ligasas de ubiquitina Cullin-anillo. Contratando consiste en el cullin Cul1, que une a la proteína de dominio de anillo Rbx1, y una proteína F-box intercambiable, que recluta a los sustratos y se une Cul1 a través de la proteína adaptador Skp18. Como una ligasa E3, SCF cataliza la conjugación de ubiquitina a su sustrato, y se activa cuando el sustrato es reclutado por la proteína F-box, y cuando Cul1 es modificado por la proteína ubiquitina-como Nedd89. Cand1 se une Cul1 sin modificar, y en Unión, altera tanto la Asociación de la proteína Skp1•F-caja con Cul1 y la conjugación de Nedd8 Cul110,11,12,13. Como resultado, Cand1 parece ser un inhibidor de la actividad SCF in vitro, pero la deficiencia de Cand1 en organismos causó defectos que sugiere un papel positivo de Cand1 en la regulación de las actividades SCF en vivo14,15,16 , 17. esta paradoja fue finalmente explicada por un estudio cuantitativo que reveló las interacciones dinámicas entre proteína Cul1, Cand1 y caja de Skp1•F. Usando análisis de transferencia (traste) de energía de resonancia de la fluorescencia que detectan la formación de los complejos SCF y Cul1•Cand1, la asociación y disociación tasa constantes (kel y kfuera, respectivamente) fueron medido individualmente. Las mediciones revelaron que Cand1 y caja de Skp1•F forma muy apretado complejo proteico con Cul1 pero el kfuera de SCF es aumentado por Cand1 y el kfuera de Cul1•Cand1 es aumentado por proteínas de Skp1•F-caja de6,7. Estos resultados proporcionan la ayuda inicial y fundamental para definir el papel de Cand1 como un factor de intercambio de proteína, que cataliza la formación de nuevos complejos SCF a través del reciclaje Cul1 de los viejos complejos SCF.
Aquí, presentamos el procedimiento de desarrollar y utilizar el ensayo de traste para estudiar la dinámica de los complejos de Cul1•Cand17, y el mismo principio puede aplicarse para estudiar la dinámica de diversas biomoléculas. TRASTE se produce cuando un donante está entusiasmado con la longitud de onda adecuada, y con el espectro de excitación superposición el espectro de emisión del donador aceptor está presente dentro de una distancia de 10-100 Å. El estado excitado se transfiere al aceptador, de tal modo disminuyendo la intensidad de donantes y aumentar la intensidad aceptador del18. La eficacia de traste (E) depende del radio de Förster (R0) y la distancia entre el donador y aceptor fluoróforos (r) y está definida por: E = R06/ (R0 6 + r6). El radio de Förster (R0) depende de algunos factores, incluyendo la orientación angular del dipolo, la superposición espectral de la pareja donante-receptor y la solución utilizada19. Para aplicar el ensayo de traste en un Fluorímetro de flujo detenido, que supervisa el cambio de la emisión del donador en tiempo real y permite medidas de rápida kel y kfuera, es necesario establecer eficiente traste que resultados en una reducción significativa de emisiones de donantes. Por lo tanto, diseño eficiente traste seleccionando el par apropiado de tintes fluorescentes y los sitios de las proteínas de la blanco para fijar los tintes es importante y se discutirá en el presente Protocolo.
Protocolo
1. diseño del ensayo de traste.
- Descargar el archivo de estructura de la Cul1•Cand1 complejo de Protein Data Bank (archivo 1U6G).
- Ver la estructura de la Cul1•Cand1 complejo en PyMOL.
- Utilice la función de medición bajo el menú Asistente de PyMOL para estimar la distancia entre el primer aminoácido de Cand1 y el último aminoácido de Cul1 (figura 1).
- El visor de espectros en línea de carga (véase Tabla de materiales) y los espectros de emisión de 7-amino-4-methylcoumarin (AMC) y FlAsH y excitación al mismo tiempo (figura 2). Tenga en cuenta que AMC es el donante de traste y FlAsH es el aceptador de traste.
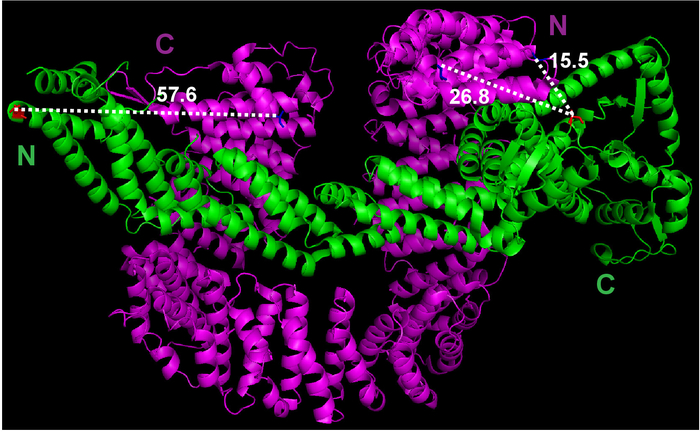
Figura 1: la estructura cristalina de Cul1•Cand1 y medición de la distancia entre potencial etiquetado sitios. El archivo de estructura de cristal fue descargado de Protein Data Bank (archivo 1U6G) y han consultado en PyMOL. Mediciones entre átomos seleccionados fueron realizadas por PyMOL. Haga clic aquí para ver una versión más grande de esta figura.
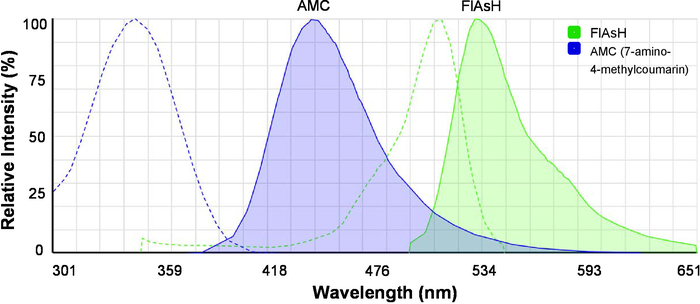
Figura 2: la excitación y espectros de emisión de los tintes fluorescentes para traste. Se muestran espectros de AMC (7-amino-4-methylcoumarin) y FlAsH. Las líneas punteadas indican los espectros de excitación, y líneas sólidas indican espectros de emisión. La imagen fue originalmente generada por el SpectraViewer de fluorescencia y fue modificada para mejor claridad. Haga clic aquí para ver una versión más grande de esta figura.
2. preparación de Cul1AMC•Rbx1, la proteína del donante de traste
- La construcción de plásmidos para expresar humana Cul1sortase•Rbx1 en células de e. coli . Tenga en cuenta que los dos plásmidos expresar Co Cul1•Rbx1 humano en células de e. coli se describen en detalle en un informe anterior de20.
- Agregar una secuencia de ADN codificación "LPETGGHHHHHH" (etiqueta sortase su6 ) para el extremo 3' de la secuencia de codificación de Cul1 mediante PCR estándar y clonación métodos21,22.
- El nuevo plásmido para confirmar la inserción del gen es correcta la secuencia.
- Conjunto express Cul1sortase•Rbx1 en células de e. coli . El método se deriva de un informe anterior de20.
- Mezcla 100 ng de cada de la dos plásmidos con BL21 (DE3) células químicamente competentes para la transformación mediante el calor del choque método23. Cultivar células en LB placa de agar que contiene 100 μg/mL ampicilina y cloranfenicol 34 μg/mL a 37 ° C durante la noche.
- Sembrar 50 mL de cultivo LB con colonias recién transformadas y crecer durante la noche a 37 ° C con agitación de 250 rpm. Esto da una cultura de arrancador.
- Sembrar 6 frascos, cada uno con 1 L de medio LB, con cultura de arrancador 5 mL cada uno y crecen a 37 ° C con 250 rpm de agitación hasta que el OD600 ~ 1.0. Enfriar la cultura a 16 ° C y añadir Isopropil-β-D-thiogalactoside (IPTG) a 0,4 milímetros. Conservar el cultivo a 16 ° C durante la noche con agitación de 250 rpm.
- Cosechar las células de e. coli por centrifugación a 5.000 x g durante 15 min y recoger el pellet celular en tubos cónicos de 50 mL.
Nota: Los pellets de células pueden ser procesados para la purificación de proteínas o ser congelados a-80 ° C antes de proceder a los pasos de purificación de proteínas.
- Purificación de la Cul1sortase•Rbx1 complejo. Este método se deriva de un informe anterior de20.
- Añadir 50 mL de tampón de lisis (30 mM Tris-HCl, 200 mM NaCl, 5 mM TDT, 10% glicerol, 1 tableta de inhibidor de la proteasa coctel, pH 7,6) para el sedimento de células de e. coli expresan Cul1sortase•Rbx1.
- Lyse las células en hielo con sonicación con amplitud del 50%. Alternar entre 1 segundo y 1 segundo apaga y funcionar durante 3 minutos.
- Repita el paso 2.3.2 x 2-3.
- Transferir el lisado de células en un tubo de centrifugación de 50 mL y eliminar los desechos celulares por centrifugación a 25.000 x g durante 45 minutos.
- Incube las células claras lisado con 5 mL de granos de glutatión a 4 ° C por 2 h.
- Centrifugar la mezcla de granos-lisado en 1.500 x g durante 2 min a 4 ° C. Eliminar el sobrenadante.
- Lavar los granos con 5 mL de tampón de lisis (con no inhibidores de la proteasa) y eliminar el sobrenadante después de centrifugación a 1.500 x g durante 2 min a 4 ° C.
- Repita el paso 2.3.7 x 2.
- Añadir 3 mL de tampón de lisis a los granos lavados y transferir la mezcla de grano en una columna vacía.
- Añadir 5 mL de tampón de elución (50 mM Tris-HCl, 200 mM NaCl, 10 mM reducida glutatión, pH 8.0) en la columna. Incubar por 10 min y recoger el eluido.
- Repita el paso 2.3.10 x 3-4.
- Añadir 200 μL de la trombina de 5 mg/mL (véase Tabla de materiales) para el efluente de los granos de glutatión e incubar durante la noche a 4 ° C.
Nota: El protocolo se puede detener aquí. - Diluir la muestra de proteína con el tampón A (25 mM HEPES, 1 mM TDT, glicerol al 5%, pH 6.5) tres veces.
- Equilibrar una columna de cromatografía de intercambio catiónico (véase Tabla de materiales) en un sistema FPLC con Buffer A.
- Carga de la muestra de proteína a la columna de cromatografía de intercambio catiónico equilibrado a un flujo de 0,5 mL/min.
- Eluir la proteína con un gradiente de NaCl en 40 mL de mezcla de tampón A y 0 a 50% Buffer B (25 mM HEPES, 1 M NaCl, 1 mM TDT, glicerol al 5%, pH 6.5) a un flujo de 1 mL/min.
- Compruebe la proteína eluída en distintas fracciones mediante SDS-PAGE24.
Nota: El protocolo se puede detener aquí. - Las fracciones de eluato que contiene Cul1sortase•Rbx1 de la piscina.
- Concentrar la muestra colectiva de •Rbx1 desortaseCul1 a 2,5 mL pasando el búfer a través de una membrana de ultrafiltración (corte de 30 kDa).
- Añadir AMC a la c-terminal de Cul1 a mediado sortase transpeptidation21,22.
- Cambiar el búfer en el Cul1sortase•Rbx1 muestra en el búfer del sortase (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7,5) utilizando una columna de desalación (véase Tabla de materiales).
- Equilibrar una desalación columna con 25 mL de tampón sortase.
- Carga 2,5 mL de Cul1sortase•Rbx1 muestra en la columna. Deseche el flujo a través.
- Eluir la muestra con 3,5 mL de tampón sortase. Recoge el flujo a través.
- En 900 μl de la solución de •Rbx1 Cul1sortaseen el buffer del sortase, añada 100 μl de una solución de 600 μm purificada sortase y 10 μl de péptido GGGGAMC 25 mM. Incubar la mezcla de reacción a 30 ° C en la noche oscura. Nota que este paso generará Cul1AMC•Rbx1.
PRECAUCIÓN: Tintes fluorescentes son sensibles a la luz, así que evite exponerlos a la luz ambiental durante la preparación muestra y proteínas tanto como sea posible.
Nota: El protocolo se puede detener aquí. - Añadir 50 μl de agarosa granos de Ni-NTA a la mezcla de reacción e incubar a temperatura ambiente durante 30 minutos.
- Las perlas de agarosa de Ni-NTA de pellets por centrifugación a 5.000 x g durante 2 min y recoger el sobrenadante.
- Equilibrar una columna de cromatografía de exclusión de tamaño (véase Tabla de materiales) con tampón (30 mM Tris-HCl 100 mM NaCl, 1 mM TDT, 10% glicerol) en el sistema FPLC.
- Cargar el Cul1AMC•Rbx1 muestra en la columna de cromatografía de exclusión de tamaño. Eluir con volumen de columna de 1.5 x de buffer.
- Compruebe las fracciones de eluato por SDS-PAGE24.
- Piscina las fracciones de eluato que contiene Cul1AMC•Rbx1.
- Medir la concentración de proteínas mediante su absorbancia a 280 nm con un espectrofotómetro.
- Alícuota de la solución de la proteína y conservar a-80 ° C.
Nota: El protocolo se puede detener aquí.
- Cambiar el búfer en el Cul1sortase•Rbx1 muestra en el búfer del sortase (50 mM Tris-HCl, 150 mM NaCl, 10 mM CaCl2, pH 7,5) utilizando una columna de desalación (véase Tabla de materiales).
3. preparación de FlAsHCand1, la proteína FRET aceptor
Nota: La mayoría de los pasos en esta parte es los mismos como paso 2. Condiciones diferentes se describen en detalle a continuación.
- Construir el plásmido de expresión humana TetraCysCand1 en células de e. coli .
- Añadir secuencia de la DNA codificación "CCPGCCGSG" (tetracysteine/TetraCys etiqueta) antes de los 15th aminoácido de Cand1 regular de PCR25 (secuencias de la cartilla: TGCTGTCCGGGCTGCTGCGGCAGCGGCATGACATCCAGCGACAAGGACTTTAG; CTAACTAGTGTCCATTGATTCCAAG).
- Inserte el producto de la polimerización en cadena en un vector pGEX-4T-2. La secuencia del plásmido para confirmar la inserción del gen es precisa y el marco.
- Expresar TetraCysCand1in e. coli las células de la misma manera como paso 2.2, salvo que el plásmido se transforma en células competentes de Rosetta.
- Purificación de Cand1 TetraCysde las células de e. coli .
- Lyse los pellets de células de e. coli y extraer TetraCysCand1using glutatión granos. Estos pasos son los mismos que 2.3.1–2.3.12 pasos.
- Diluir el efluente de la proteína de los granos de glutatión con el amortiguamiento C (50 mM Tris-HCl, 1 mM TDT, 5% de glicerol, pH 7,5) por tres pliegues. Equilibrar una columna de cromatografía de intercambio aniónico (véase Tabla de materiales) en el sistema FPLC con amortiguamiento C y carga de la muestra de proteína diluido a un flujo de 0,5 mL/min.
- Eluir la proteína con un gradiente de NaCl en 40 mL mediante la mezcla de Buffer C y 0 a 50% Buffer D (50 mM de Tris-HCl, 1 M NaCl, 1 mM TDT, 5% de glicerol, pH 7.5) con un caudal de 1 mL/min. Compruebe la proteína eluída en distintas fracciones mediante SDS-PAGE24y las fracciones que contienen TetraCysCand1 la piscina. Observe que Cand1 TetraCystiene un mayor volumen de retención que GST gratis.
- Concentrar la muestra colectiva de Cand1 TetraCyspasando el búfer a través de una membrana de ultrafiltración (corte de 30 kDa).
- Equilibrar la columna de cromatografía de exclusión de tamaño con tampón etiquetado (20 mM Tris-HCl 100 mM NaCl, 2 mM TCEP, 1 mM EDTA, 5% de glicerol) en el sistema FPLC. Carga Cand1 TetraCysmuestra (500 ml cada vez) y compruebe las fracciones de eluato por SDS-PAGE24.
- Todas las fracciones que contienen TetraCysCand1 la piscina y concentrar la proteína ~ 40 μm pasando el búfer a través de una membrana de ultrafiltración (corte de 30 kDa). Calcular la concentración de proteínas mediante su absorbancia a 280 nm. Almacenar la proteína como alícuotas de μl 50 a-80 ° C.
Nota: El protocolo se puede detener aquí.
- Preparación de Cand1 FlAsH.
- Añadir 1 μl de solución de FlAsH (véase Tabla de materiales) a TetraCysCand1 solución a 50 μl.
- Mezcla bien e incubar la mezcla a temperatura ambiente en la oscuridad durante 1-2 h para obtener FlAsHCand1.
Nota: El protocolo se puede detener aquí.
4. elaboración de Cand1, la proteína de chase de traste
Nota: El protocolo de preparación de la proteína es similar al paso 3, con las siguientes modificaciones.
- Introduzca la secuencia de codificación de Cand1 cuerpo entero en el vector pGEX-4T-2.
- Cambie el búfer utilizado en paso 3.3.5 a un búfer que contiene 30 mM Tris-HCl 100 mM NaCl, 1 mM TDT, 10% glicerol.
- Eliminar pasos 3.3.7 y 3.3.8.
5. probar y confirmar el ensayo de traste
- Preparar el tampón de traste que contiene 30 mM Tris-HCl 100 mM NaCl, 0.5 mM DTT, ovoalbúmina de 1 mg/mL, pH 7,6 y utilizar a temperatura ambiente.
- Prueba el traste entre Cul1AMC•Rbx1 y Cand1 de FlAsHen un Fluorímetro.
- En 300 μL de tampón de traste, añadir Cul1AMC•Rbx1 (donante del traste) a una concentración final de 70 nM. Transferir la solución en una cubeta.
- Coloque la cubeta en el soporte de la muestra de un Fluorímetro. Excitar la muestra con la luz de excitación de 350 nm y analizar las señales de emisión de 400 nm a 600 nm en incrementos de nm 1.
- Repita el paso 5.2.1 pero cambio Cul1AMC•Rbx1 a Cand1 FlAsH(FRET aceptor). Analizar la muestra de Cand1 de FlAsHutilizando el mismo método que en el paso 5.2.2.
- (Opcional) Excitar el Cand1 FlAsHcon la luz de excitación de 510 nm y la señal de emisión de 500 nm a 650 nm.
- En 300 μL de tampón de traste, agregue ambos Cul1AMC•Rbx1 y FlAsHCand1 a una concentración final de 70 nM. Analizar la muestra de la misma manera como en el paso 5.2.2.
- Confirmar el traste entre Cul1AMC•Rbx1 y Cand1 de FlAsHmediante la adición de la proteína de chase (Cand1) (figura 3).
- En 300 μL de tampón de traste, añadir 70 nM Cul1AMC•Rbx1 y 700 nM Cand1. Analizar la emisión de la muestra como en el paso 5.2.2.
- En 300 μL de tampón de traste, añadir 70 nM Cand1 FlAsHy 700 nM Cand1. Analizar la emisión de la muestra como en el paso 5.2.2.
- En 300 μL de tampón de traste, añadir 70 nM Cul1AMC•Rbx1 y 70 nM Cand1 FlAsHe incubar la muestra a temperatura ambiente durante 5 minutos. Luego añadir 700 nM Cand1 e inmediatamente después de la adición, la emisión de la muestra como en el paso 5.2.2). Tenga en cuenta que este paso es similar al paso 5.2.5.
- En 300 μL de tampón de traste, secuencia agregar 70 nM Cul1AMC•Rbx1, 700 nM Cand1 y 70 nM Cand1 FlAsH. Incubar la muestra a temperatura ambiente durante 5 minutos y analizar la emisión de la muestra como en el paso 5.2.2. Tenga en cuenta que se trata de la muestra de chase (línea verde en la figura 3).
6. medir la constante de velocidad de asociación (ka) de Cul1•Cand1
Nota: Detalles de un Fluorímetro de flujo detenido de funcionamiento se ha descrito en un anterior informe26.
- Preparar el flujo dejado Fluorímetro para medición.
- Encienda el Fluorímetro de flujo detenido según las instrucciones del fabricante.
- Ajuste la luz de excitación a 350 nm y uso un filtro band-pass que permite emisión de nm 450 luz pasar y bloquea la emisión de 500-650 nm luz.
- Mantener las válvulas de la muestra en la posición de llenar y conectar una jeringa de 3 mL con agua. Lave las dos jeringas de la muestra (A y B) con el agua moviendo la unidad jeringa de muestra arriba y abajo varias veces. Desechar toda el agua utilizado en esta etapa.
- Mantener las válvulas de la muestra en la posición de llenar y conectar una jeringa de 3 mL con buffer de traste. Lave las muestra dos jeringas con el tampón de traste moviendo la unidad jeringa de muestra por vario tiempo. Elimine todo el tampón de traste utilizado en esta etapa.
- Tomar una medida de control (figura 4).
- Conecte una jeringa de 3 mL y jeringa A la carga con 100 nM Cul1AMC•Rbx1 en el buffer de traste. Gire la válvula de la muestra a la posición de conducir .
- Conecte una jeringa de 3 mL y jeringa B de carga con el tampón de traste. Gire la válvula de la muestra a la posición de conducir .
- Utilizar el Panel de Control en la adquisición del software para tomar cinco tiros (mezcla de igual volumen de muestras de la jeringa A y la jeringa B) en el Fluorímetro de flujo detenido sin grabar los resultados.
- Abra el Panel de Control en la adquisición del software y programa para grabar la emisión de Cul1AMC sobre 60 s. Luego tomar un solo tiro.
- Repita el paso 6.2.4 x 2.
- Gire la válvula de la muestra a la posición de llenar . Vacíe la jeringa B y lavar con el tampón de traste.
- Observada constantes de la tarifa de la Asociación (kobs) de Cul1•Cand1 de medida (Figura 4B).
- Mantener la muestra en la jeringa A la misma como en el paso 6.2.1.
- Conecte una jeringa de 3 mL y jeringa B de carga con 100 nM Cand1 FlAsHen el búfer de traste. Gire la válvula de la muestra a la posición de conducir .
- Utilizar el Panel de Control en la adquisición del software para tomar cinco tiros sin grabar los resultados.
- Abra el Panel de Control en la adquisición del software y programa para grabar la emisión de Cul1AMC sobre 60 s. Luego tomar un solo tiro.
- Repita el paso 6.3.4 x 2.
- Vacíe la jeringa B y lavar con el tampón de traste.
- Repita los pasos 6.3.1–6.3.6 varias veces con el aumento de las concentraciones de Cand1 FlAsHen el búfer de traste.
- Ajustar el cambio (disminución) en señales fluorescentes medidas en el tiempo de cada toma a una sola curva exponencial. Esto le dará kobsen cada medición, y la unidad es s-1. Tenga en cuenta que la base de este cálculo se ha discutido también en un anterior informe27.
- Calcular la media y desviación estándar de kobspara cada concentración de Cand1 FlAsHutilizado. Parcela la media kobscontra concentración de Cand1 (figura 4), y la pendiente de la recta representa la ken el de Cul1•Cand1, con una unidad de M-1 s-1.
7. medir la constante de velocidad de disociación (kde) de Cul1•Cand1 en presencia de la proteína Skp1•F-caja.
Nota: Este paso es similar al paso 6, con las siguientes modificaciones.
- En la jeringa A, debajo de la posición de llenar , cargar una solución de 100 nM Cul1AMC•Rbx1 y 100 nM Cand1 FlAsHen el búfer de traste. Gire la válvula de la muestra en la posición "DRIVE".
- En jeringa B, debajo de la posición de llenar , cargar una solución de Skp1•Skp2 (preparada a raíz de un informe anterior de20). Gire la válvula de la muestra en la posición "DRIVE".
- Abra el Panel de Control en la adquisición del software y programa para grabar la emisión de Cul1AMC 30 s. Luego tomar un solo tiro. Las señales fluorescentes aumentan con el tiempo después de mezclar las soluciones de la jeringa A y B de la jeringa (figura 5).
Resultados
Para probar el traste entre Cul1AMC y Cand1 FlAsH, primero determinamos la intensidad de emisión de 70 nM Cul1AMC (el donante) y 70 nM Cand1 FlAsH(el aceptador), respectivamente (Figura 3A-C, azul líneas). En cada análisis, solamente un pico de emisión era presente y la emisión de Cand1 FlAsH(el aceptador) fue bajo. Cuando 70 nM de Cul1AMC y destelloCand1 se mez...
Discusión
TRASTE es un fenómeno físico que es de gran interés para el estudio y comprensión de los sistemas biológicos19. Aquí, presentamos un protocolo para probar y usar trastes para estudiar la cinética de unión de dos proteínas interactuantes. Al diseñar el traste, se consideraron tres factores principales: el solapamiento espectral entre donantes emisión y aceptador de la excitación, la distancia entre los dos fluoróforos y la orientación del dipolo de los fluoróforos28...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Ou Shu Shan (California Institute of Technology) perspicaz discusión sobre el desarrollo de la prueba de traste. M.G., Y.Z. y los X.L. fueron financiados por fondos de inicio de la Universidad de Purdue a Y.Z. y X.L.This trabajo fue apoyado en parte por una subvención de semillas del centro de la Universidad de Purdue para Biología de plantas.
Materiales
| Name | Company | Catalog Number | Comments |
| Anion exchange chromatography column | GE Healthcare | 17505301 | HiTrap Q FF anion exchange chromatography column |
| Benchtop refrigerated centrifuge | Eppendorf | 2231000511 | |
| BL21 (DE3) Competent Cells | ThermoFisher Scientific | C600003 | |
| Calcium Chloride | Fisher Scientific | C78-500 | |
| Cation exchange chromatography column | GE Healthcare | 17505401 | HiTrap SP Sepharose FF |
| Desalting Column | GE Healthcare | 17085101 | |
| Floor model centrifuge (high speed) | Beckman Coulter | J2-MC | |
| Floor model centrifuge (low speed) | Beckman Coulter | J6-MI | |
| Fluorescence SpectraViewer | ThermoFisher Scientific | https://www.thermofisher.com/us/en/home/life-science/cell-analysis/labeling-chemistry/fluorescence-spectraviewer.html | |
| FluoroMax fluorimeter | HORIBA | FluoroMax-3 | |
| FPLC | GE Healthcare | 29018224 | |
| GGGGAMC peptide | New England Peptide | custom synthesis | |
| Glutathione beads | GE Healthcare | 17075605 | |
| Glycerol | Fisher Scientific | G33-500 | |
| HEPES | Fisher Scientific | BP310-100 | |
| Isopropyl-β-D-thiogalactoside (IPTG) | Fisher Scientific | 15-529-019 | |
| LB Broth | Fisher Scientific | BP1426-500 | |
| Ni-NTA agarose | Qiagen | 30210 | |
| Ovalbumin | MilliporeSigma | A2512 | |
| pGEX-4T-2 vector | GE Healthcare | 28954550 | |
| Protease inhibitor cocktail | MilliporeSigma | 4693132001 | |
| Reduced glutathione | Fisher Scientific | BP25211 | |
| Refrigerated shaker | Eppendorf | M1282-0004 | |
| Rosetta Competent Cells | MilliporeSigma | 70953-3 | |
| Size exclusion chromatography column | GE Healthcare | 28990944 | Superdex 200 10/300 GL column |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-500 | |
| Stopped-flow fluorimeter | Hi-Tech Scientific | SF-61 DX2 | |
| TCEP·HCl | Fisher Scientific | PI20490 | |
| Thrombin | MilliporeSigma | T4648 | |
| Tris Base | Fisher Scientific | BP152-500 | |
| Ultrafiltration membrane | MilliporeSigma | UFC903008 | Amicon Ultra-15 Centrifugal Filter Units, Ultra-15, 30,000 NMWL |
Referencias
- Stumpf, M. P. H., et al. Estimating the size of the human interactome. Proceedings of the National Academy of Sciences of the United States of America. 105 (19), 6959-6964 (2008).
- Kuzmanov, U., Emili, A. Protein-protein interaction networks: probing disease mechanisms using model systems. Genome Medicine. 5 (4), 37 (2013).
- Titeca, K., Lemmens, I., Tavernier, J., Eyckerman, S. Discovering cellular protein-protein interactions: Technological strategies and opportunities. Mass Spectrometry Reviews. , 1-33 (2018).
- Lapetina, S., Gil-Henn, H. A guide to simple, direct, and quantitative in vitro binding assays. Journal of Biological Methods. 4 (1), 62 (2017).
- Zheng, X., Bi, C., Li, Z., Podariu, M., Hage, D. S. Analytical methods for kinetic studies of biological interactions: A review. Journal of Pharmaceutical and Biomedical Analysis. 113, 163-180 (2015).
- Pierce, N. W., et al. Cand1 promotes assembly of new SCF complexes through dynamic exchange of F box proteins. Cell. 153 (1), 206-215 (2013).
- Liu, X., Reitsma, J. M., Mamrosh, J. L., Zhang, Y., Straube, R., Deshaies, R. J. Cand1-Mediated Adaptive Exchange Mechanism Enables Variation in F-Box Protein Expression. Molecular Cell. 69 (5), 773-786 (2018).
- Zheng, N., et al. Structure of the Cul1-Rbx1-Skp1-F boxSkp2 SCF ubiquitin ligase complex. Nature. 416 (6882), 703-709 (2002).
- Kleiger, G., Deshaies, R. Tag Team Ubiquitin Ligases. Cell. 166 (5), 1080-1081 (2016).
- Zheng, J., et al. CAND1 binds to unneddylated CUL1 and regulates the formation of SCF ubiquitin E3 ligase complex. Molecular Cell. 10 (6), 1519-1526 (2002).
- Hwang, J. W., Min, K. W., Tamura, T. A., Yoon, J. B. TIP120A associates with unneddylated cullin 1 and regulates its neddylation. FEBS Letters. 541 (1-3), 102-108 (2003).
- Min, K. W., Hwang, J. W., Lee, J. S., Park, Y., Tamura, T., Yoon, J. B. TIP120A associates with cullins and modulates ubiquitin ligase activity. Journal of Biological Chemistry. 278 (18), 15905-15910 (2003).
- Goldenberg, S. J., et al. Structure of the Cand1-Cul1-Roc1 complex reveals regulatory mechanisms for the assembly of the multisubunit cullin-dependent ubiquitin ligases. Cell. 119 (4), 517-528 (2004).
- Chuang, H. W., Zhang, W., Gray, W. M. Arabidopsis ETA2, an apparent ortholog of the human cullin-interacting protein CAND1, is required for auxin responses mediated by the SCF(TIR1) ubiquitin ligase. Plant Cell. 16 (7), 1883-1897 (2004).
- Feng, S., et al. Arabidopsis CAND1, an Unmodified CUL1-Interacting Protein, Is Involved in Multiple Developmental Pathways Controlled by Ubiquitin/Proteasome-Mediated Protein Degradation. the Plant Cell Online. 16 (7), 1870-1882 (2004).
- Cheng, Y., Dai, X., Zhao, Y. AtCAND1, a HEAT-repeat protein that participates in auxin signaling in Arabidopsis. Plant Physiology. 135 (June), 1020-1026 (2004).
- Lo, S. -. C., Hannink, M. CAND1-Mediated Substrate Adaptor Recycling Is Required for Efficient Repression of Nrf2 by Keap1. Molecular and Cellular Biology. 26 (4), 1235-1244 (2006).
- Okamoto, K., Sako, Y. Recent advances in FRET for the study of protein interactions and dynamics. Current Opinion in Structural Biology. 46, 16-23 (2017).
- Hussain, S. A. An introduction to fluorescence resonance energy transfer (FRET). arXiv preprint. , (2009).
- Li, T., Pavletich, N. P., Schulman, B. A., Zheng, N. High-level expression and purification of recombinant SCF ubiquitin ligases. Methods in Enzymology. 398 (1996), 125-142 (2005).
- Popp, M. W., Antos, J. M., Grotenbreg, G. M., Spooner, E., Ploegh, H. L. Sortagging: A versatile method for protein labeling. Nature Chemical Biology. 3 (11), 707-708 (2007).
- Antos, J. M., Ingram, J., Fang, T., Pishesha, N., Truttmann, M. C., Ploegh, H. L. Site-Specific Protein Labeling via Sortase-Mediated Transpeptidation. Current Protocols in Protein Science. 89, (2017).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. Journal of Visualized Experiments. , (2007).
- Simpson, R. J. SDS-PAGE of Proteins. Cold Spring Harbor Protocols. 2006 (1), (2006).
- Kleiger, G., Saha, A., Lewis, S., Kuhlman, B., Deshaies, R. J. Rapid E2-E3 Assembly and Disassembly Enable Processive Ubiquitylation of Cullin-RING Ubiquitin Ligase Substrates. Cell. 139 (5), 957-968 (2009).
- Biro, F. N., Zhai, J., Doucette, C. W., Hingorani, M. M. Application of Stopped-flow Kinetics Methods to Investigate the Mechanism of Action of a DNA Repair Protein. Journal of Visualized Experiments. (37), 2-8 (2010).
- Patel, J. T., Belsham, H. R., Rathbone, A. J., Friel, C. T. Use of Stopped-Flow Fluorescence and Labeled Nucleotides to Analyze the ATP Turnover Cycle of Kinesins. Journal of Visualized Experiments. (92), 1-6 (2014).
- Bajar, B. T., Wang, E. S., Zhang, S., Lin, M. Z., Chu, J. A guide to fluorescent protein FRET pairs. Sensors (Switzerland). 16 (9), 1-24 (2016).
- Chen, A. K., Cheng, Z., Behlke, M. A., Tsourkas, A. Assessing the sensitivity of commercially available fluorophores to the intracellular environment. Analytical Chemistry. 80 (19), 7437-7444 (2008).
- Lin, C. T., Rorabacher, D. B. Mathematical approach for stopped-flow kinetics of fast second-order reactions involving inhomogeneity in the reaction cell. Journal of Physical Chemistry. 78 (3), 305-308 (1974).
- Toseland, C. P., Geeves, M. A. Rapid Reaction Kinetic Techniques. Fluorescent Methods for Molecular Motors. , 49-65 (2014).
- Adams, S. R., et al. New biarsenical ligands and tetracysteine motifs for protein labeling in vitro and in vivo: Synthesis and biological applications. Journal of the American Chemical Society. 124 (21), 6063-6076 (2002).
- Lin, C. W., Ting, A. Y. Transglutaminase-catalyzed site-specific conjugation of small-molecule probes to proteins in vitro and on the surface of living cells. Journal of the American Chemical Society. 128 (14), 4542-4543 (2006).
- Yin, J., et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase. Proceedings of the National Academy of Sciences of the United States of America. 102 (44), 15815-15820 (2005).
- Diaspro, A., Chirico, G., Usai, C., Ramoino, P., Dobrucki, J. Photobleaching. Handbook Of Biological Confocal Microscopy. , 690-702 (2006).
- Aoki, K., Kamioka, Y., Matsuda, M. Fluorescence resonance energy transfer imaging of cell signaling from in vitro to in vivo: Basis of biosensor construction, live imaging, and image processing. Development, Growth & Differentiation. 55 (4), 515-522 (2013).
- Kilic, S., et al. Single-molecule FRET reveals multiscale chromatin dynamics modulated by HP1α. Nature Communications. 9 (1), (2018).
- Shen, K., Arslan, S., Akopian, D., Ha, T., Shan, S. O. Activated GTPase movement on an RNA scaffold drives co-translational protein targeting. Nature. 492 (7428), 271-275 (2012).
- Bajar, B. T., et al. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports. 6 (February), 1-12 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados