Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Generación, amplificación y titulación de virus sincitiales respiratorios recombinantes
En este artículo
Resumen
Se describe un método para generar y amplificar genéticamente modificados virus sincitiales respiratorios (RSVs) y un ensayo de placa optimizada para RSVs. Ilustramos este protocolo mediante la creación de dos virus recombinantes que respectivamente permiten cuantificación de replicación RSV y análisis de cuerpos de inclusión de RSV y gránulos asociada a cuerpos de inclusión dinámica en vivo.
Resumen
La utilización de virus recombinantes se ha vuelto crucial en virología básica o aplicada. Genética inversa ha demostrado para ser una tecnología muy potente, tanto para descifrar los mecanismos de replicación viral y a estudiar antivirales o proporcionar la plataforma de desarrollo de vacunas. La construcción y manipulación de un sistema de genético reverso para un negativo-strand RNA virus como un virus sincitial respiratorio (VSR), sin embargo, sigue siendo delicadas y requiere conocimientos técnicos especiales. El genoma del RSV es un ARN monocatenario, de sentido negativo de aproximadamente 15 kb que sirve como plantilla para la replicación del RNA viral y transcripción. Nuestro sistema de genética reversa utiliza una copia del cDNA del genoma humano de largo cepa de RSV (HRSV). Este cDNA, así como de los cDNAs que codifican las proteínas virales de la polimerasa compleja (L, P, N y M2-1), se colocan en vectores de expresión individual bajo secuencias de control de polimerasa de T7. La transfección de estos elementos en células BSR-T7/5, que estable expresan T7 polimerasa, permite la replicación citoplasmática y transcripción de la RSV recombinante, dando lugar a viriones modificados genéticamente. Una RSV nueva, que está presente en la superficie celular y en el sobrenadante de cultivo de BSRT7/5, se reunió para infectar las células humanas de HEp-2 para la amplificación viral. Dos o tres rondas de amplificación son necesarios para obtener las poblaciones virales que contiene 1 x 106 a 1 x 107 unidades formadoras de placa (PFU) / mL. Métodos para la óptima recolección, congelación y valoración de las poblaciones virales se describen aquí en detalle. Ilustramos el protocolo que se presenta aquí mediante la creación de dos virus recombinantes que expresan respectivamente gratis proteína verde fluorescente (GFP) (RSV-GFP) o M2-1 viral fusionada a GFP (RSV-M2-1-GFP). Mostramos cómo utilizar RSV-GFP para cuantificar replicación RSV y la RSV-M2-1-GFP para visualizar estructuras virales, así como la dinámica de la proteína viral en células vivas, mediante técnicas de microscopia video.
Introducción
RSV humano es la principal causa de hospitalización por infección respiratoria aguda en niños en todo el mundo1. Además, RSV se asocia con una sustancial morbilidad en adultos comparables a la gripe, con la mayor parte de la hospitalización y la carga de mortalidad en los ancianos2. No hay vacunas o antivirales específicos disponibles sin embargo contra el VRS, pero promete nuevos fármacos están en desarrollo3,4. La complejidad y la pesadez de las técnicas de cuantificación de la multiplicación de RSV impiden la búsqueda de antivirales o vacunas a pesar de esfuerzos considerables. La cuantificación de la multiplicación de RSV in vitro se basa generalmente en métodos laboriosos, lentos y costosos, que consisten sobre todo en el análisis del efecto citopático por microscopia, immunostaining, reducción de placa ensayos cuantitativos (qRT) de la transcriptasa reversa-reacción en cadena de polimerasa (PCR) y enzima-ligado del inmunosorbente pruebas de ensayo. Virus con genomas modificados y la expresión de genes reporteros, tales como codificación de la GFP, son más convenientes para tales proyecciones. Junto a la utilización de lectores de placa automatizada, virus recombinantes portadores de gen reportero pueden hacer estos ensayos más conveniente para los propósitos de estandarización y alto rendimiento.
RSV es un virus ARN de sentido negativo envuelto, que pertenece al género Orthopneumovirus de Pneumoviridae familiar, orden Mononegavirales5. El genoma del RSV es un ARN monocatenario, de sentido negativo de unos 15 kb, que contiene una región noncoding en el extremos 3' y 5' llamado líder y Trailer y 10 unidades transcripcionales que codifican 11 proteínas. Los genes se ordenan como sigue: 3'-NS1, NS2, N, P, M, SH, G, F, M2 (codificación de las proteínas de 1 M2 y M2-2) y L-5'. El ARN genómico firmemente empaquetado por la nucleoproteína N. usando el genoma de ARN genómico como plantilla, viral RNA polimerasa dependiente de ARN (RdRp) se asegurará de transcripción y replicación del ARN viral. RdRp viral se compone de la proteína grande L que lleva la actividad de polimerasa nucleótidos por sí, su cofactor obligatorio la fosfoproteína P y la proteína M2-1 que funciona como una transcripción viral factor6. En las células infectadas, RSV induce la formación de inclusiones citoplasmáticas llamadas cuerpos de inclusión (IBs). Inclusiones citoplásmicas morfológicamente similares se han observado varios Mononegavirales7,8,9,10. Recientes estudios sobre el virus de la rabia, virus de la estomatitis vesicular (VSV), virus de Ebola y RSV demostrada que la síntesis de RNA viral ocurre en IBs, que pueden considerarse así como fábricas virales8,9,11, 12. las fábricas de virus concentrado el ARN y las proteínas virales necesarias para la síntesis de RNA viral y también contienen proteínas celulares13,14,15,16, 17. IBs exhiben un subcompartment funcional llamado gránulos asociada a IB (IBAGs), que se concentran los recién sintetizado naciente mRNA viral junto con la proteína M2-1. El RNA genómico y la L, P y N no son detectados en IBAGs. IBAGs son pequeñas estructuras esféricas dinámicas dentro de IBs que exhiben las propiedades de organelos líquido12. A pesar del papel central del SCI en la multiplicación viral, muy poco se sabe acerca de la naturaleza, estructura, formación y funcionamiento de estas fábricas virales.
La expresión del genoma de un virus de la polio de un cDNA permitieron la producción de la primera copia viral infecciosa en 198118. Para negativo virus de ARN monocatenario, no fue hasta 1994 que la producción de un primer virus de la rabia después de la transfección de plásmidos en células19 ocurrió. El primer plásmido sistema basado en inversa genético para RSV fue publicado en 199520. Genética reversa ha conducido a importantes avances en el campo de la virología. La posibilidad de introducir modificaciones específicas en el genoma viral ha proporcionado penetraciones críticas en la replicación y patogenia del virus del RNA. Esta tecnología también ha facilitado el desarrollo de vacunas por lo que permite la atenuación específica a través de una serie específica de modificaciones. Modificaciones del genoma que permite una cuantificación rápida de la multiplicación viral grandemente mejoraron del antiviral proyección y estudio de su modo de acción.
Aunque descrito previamente, obtención de transgénicos RSVs sigue siendo delicado. Aquí detallamos un protocolo para crear dos tipos de HRSV recombinante, respectivamente expresan GFP RSV o RSV-M2-1-GFP. En este protocolo, describimos las condiciones de la transfección a rescatar los nuevos virus recombinantes, así como su amplificación para obtener poblaciones virales con alto título, apto para experimentaciones reproducibles. La construcción de vectores de la genética reversa propiamente se describe aquí. Describir métodos de cosecha óptima y la congelación de las poblaciones virales. El método más preciso para cuantificar partículas infecciosas virales sigue siendo el ensayo de placa. Las células son infectadas con diluciones seriadas de la suspensión analizada y se incubaron con un recubrimiento que impide la difusión de partículas virales libres en el sobrenadante. En esas condiciones, el virus infectará sólo celdas contiguas formando una "placa" para cada partícula infecciosa inicial. En el análisis convencional de la titulación de RSV, placas son reveladas por inmunotinción y contadas bajo observación microscópica. Este método es costoso y desperdiciador de tiempo. Aquí describimos un protocolo muy simple para un ensayo de placa de RSV mediante recubrimiento de celulosa microcristalina que permite la formación de placas visibles al ojo desnudo. Mostramos cómo se puede utilizar RSV-GFP a medida RSV replicación y, por tanto, para cuantificar el impacto de antivirales. Combinación de genética reversa y en tecnología de imagen, demostramos cómo RSV-M2-1-GFP permite a los científicos visualizar M2-1 en células vivas y seguir la dinámica de estructuras virales intracelulares, como IBs.
Protocolo
1. material preparación
- Compra de medios de comunicación de la célula (suero reducido soporte, mínimo esencial [MEM], 10 x MEM y Dulbecco de modificado medio de águila [DMEM]), reactivo de transfección y celulosa microcristalina (véase Tabla de materiales).
- Obtener los siguientes vectores de genética reversa: vector(s) genómica y los vectores de expresión que codifican la proteína N y las proteínas del complejo polimerasa. Los vectores de la genómicos contienen el cDNA completo genoma del RSV-GFP (GFP-RSV-p) y del RSV-M2-1GFP (p-RSV-M2-1GFP) aguas abajo de la promotora de bacteriófago T7 ARN polimerasa (pol T7). Los vectores de expresión (designado como p-N, p p -P,-L y p-M2-1) contiene la secuencia de codificación de N, P, L o M2-1 abajo el pol T7 (véase Rincheval et al12 y Rameix Welti et al21 para más detalles sobre las construcciones de plásmido).
- Preparar los medios de comunicación para un cultivo de células en un ambiente estéril y para la transfección y la infección. Uso DMEM con 2 mM L-glutamina complementado con 10% de suero fetal de ternero (FCS), 1.000 unidades/mL de penicilina y estreptomicina de 1 mg/mL (o sin antibióticos) y MEM con 2 mM L-glutamina suplementada con 0%, 2% o 10% FCS, 1.000 unidades/mL de penicilina y 1 mg/mL estreptomicina, señalado como "medio completo" en el siguiente protocolo.
- Obtención de BSRT7/522 células y hacer acciones en completo suplementado con 10% dimetil sulfóxido (DMSO) en 1 a 2 x 106 células/mL. Conservar las poblaciones de células en nitrógeno líquido. Obtener células HEp-2. Cultura BSRT7/5 células en DMEM completo y células HEp-2 en MEM completa a 37 ° C y 5% de CO2 en un ambiente estéril.
- Preparar una RSV 10 x conservación solución (0,5 M HEPES y 1 M MgSO4 [pH 7.5] en agua) en un ambiente estéril.
- Obtener un microscopio de fluorescencia invertido compatible con mediciones de la fluorescencia de GFP y compatible con vivo de imagen si es necesario controlar una infección. Obtener un lector de microplacas compatible con las mediciones de la fluorescencia de GFP para la cuantificación de la replicación RSV-GFP.
2. rescate y el primer paso de Virus recombinantes
Nota: Realizar los siguientes pasos en un ambiente estéril, usando una clase de seguridad II gabinete.
- El día antes de la transfección, hacer una suspensión de la línea celular de BSRT7/5 5 x 105 células/ml en medio completo. Distribuir 2 mL de la suspensión celular por pozo en una placa de 6 pozos. Preparar bien por virus, que va a ser rescatado y un pozo adicional para control negativo. Incubar la placa a 37 ° C y 5% CO2. Comprobar que las células están en una confluencia del 80% – 90% al día siguiente.
- Desbloquear la genética reversa vectores (en el paso 1.2) p-RSV-GFP y p-RSV-M2-1-GFP, así como p-N, p p -P,-L y p-M2-1. Mezcla, para cada virus rescatar, 1 μg de p-N y p -P, 0.5 μg de p-L, 0,25 μg de p-M2-1 y 1.25 μg de p-RSV (GFP o GFP M2-1) en un tubo.
Nota: Vectores de expresión diferente de N, P, L y M2-1 pueden ser utilizados; sin embargo, la proporción entre las proteínas tiene que ser mantenido. Realizar el control negativo sustituyendo el vector p-RSV con un vector vacío. -
Proceder a la transfección, siguiendo el protocolo del fabricante de reactivo de transfección (véase tabla de materiales).
- Añadir 250 μl de medio de suero reducido a los vectores mixtos. En otro tubo, diluir 10 μl del reactivo de transfección en 250 μl de medio de suero reducido. Suavemente mezclar los tubos y espere 5 minutos mezcla el contenido de ambos tubos y esperar 20 min a temperatura ambiente.
- Enjuague las células de BSRT7/5 con 1 mL de medio de suero reducido y distribuir 1,5 mL de MEM con 10% FCS sin antibióticos por pozo. Si es necesario, incubar a 37 ° C y 5% de CO2 hasta completa la incubación descrita en el paso 2.3.1.
- Añadir 500 μl de la mezcla de transfección preparada en paso 2.3.1 a un pozo, cuando el tiempo de incubación de 20 minutos. Colocar las células en la incubadora a 37 ° C y 5% CO2 durante 3 días. No cambie el medio de cultivo de las células durante la transfección.
- Observar la fluorescencia de GFP (excitación a 488 nm) y emisión a 515-535 nm en un microscopio de fluorescencia invertido con 20 aumentos 1 x al día para monitorear la eficacia del rescate, usando la GFP filtro.
- En el segundo día después de la transfección, las células para el primer paso de los virus rescatados de la semilla. Preparar una suspensión de células HEp-2 en 5 x 105 células/mL en medio completo. Distribuir 2 mL de la suspensión celular por pozo en una placa de 6 pozos (uno bien por virus para rescate y un control negativo).
- En el tercer día de la transfección, las células scratch en cada pocillo de la placa de 6 pozos de BSRT7/5 transfected, usando un raspador diferentes para cada pozo. Transfiera cada contenido bien (células y sobrenadante) a un tubo de microcentrífuga estéril de 2 mL. Vortex cada tubo vigorosamente durante al menos 30 s para liberar el virus rescatado de las membranas celulares.
Nota: Esto corresponde al paso 0 (P0) rescatado del virus de la (figura 1). -
Utilizar la suspensión fresca de P0 viral para realizar la primera amplificación de los virus rescatados.
- Retire el medio de cultivo de la placa de 6 pozos de HEp-2 sembrados el día anterior (ver paso 2.5) y rápidamente agregar 500 μl de la suspensión de P0 (del paso 2.6) por pozo. Coloque la placa HEp-2 a 37 ° C en un eje de balancín subibaja para agitación suave por 2 h.
- Retire y deseche el 500 μl del inóculo y añadir 2 mL de MEM con FCS 2%. Incubar la placa a 37 ° C y 5% de CO2 durante 3 días. Esto producirá el primer paso (P1) de los virus rescatados (figura 1).
- Añadir 1/10 del volumen de solución de conservación de RSV de 10 x (0.5 M HEPES y 1 M MgSO4 [pH 7.5]) en la suspensión restante de P0 (del paso 2.6). Vórtice los microtubos vigorosamente durante 5 s y alícuota el contenido en tubos criogénicos etiquetados con las etiquetas resistente al alcohol. Sumerja los tubos durante al menos 1 hora en alcohol preenfriado a-80 ° C y almacenar a-80 ° C.
- Valorar la acción P0 (ver paso 2.6) de cada virus rescatados (ver sección 4 para la valoración de la celulosa microcristalina).
- Observar la fluorescencia de GFP (excitación a 488 nm) y emisión a 515-535 nm de las células HEp-2 infectadas con la suspensión de P0 bajo un microscopio de fluorescencia invertido con 20 aumentos 1 x al día para controlar la infección. Observar bajo un microscopio de campo luminoso la aparición de pequeños sincicios y desprendimiento que refleja el efecto citopatógeno RSV (CPE) de la célula (ver figura 2).
- Tenga en cuenta que el rescate ha fracasado si la fluorescencia ni CPE es visible después de 2-3 días.
- Recoger el primer paso (P1) en día 3 o 4 como se describe en el paso 2.6. En Resumen, raspar las células, recoger las células y sobrenadante junto, vortex, la solución de conservación como se describe en paso 2.8 alícuotas de la muestra y congelarla.
- Valorar (ver sección 4 para el ensayo de titulación) y ampliar el primer paso (ver sección 3 para la amplificación).
3. amplificación de los virus rescatados
Nota: El siguiente protocolo describe la amplificación de los virus rescatados en un matraz de2 75 cm. Adaptar el tamaño de matraz el volumen necesario y la necesaria multiplicidad de infección (MOI). Tabla 1 indica los volúmenes de diferentes frascos. Realice todos los pasos siguientes en un entorno estéril en una clase de seguridad II gabinete.
- Preparar una suspensión de células HEp-2 en 5 x 105 células/mL en medio completo, el día antes de la amplificación. Distribuir 15 mL de la suspensión cada frasco de 75 cm2 e incube los frascos a 37 º C y 5% CO2. Preparar un frasco por virus para amplificar.
- El día después del comienzo de la incubación, comprobar que las células son confluentes de 80 – 100%.
- Diluir la suspensión viral de paso 2.12 en MEM sin FCS para obtener una suspensión de 3 mL en 50.000 PFU/mL (correspondiente a una MOI de 0.01 UFP/célula).
- Quite el medio y agregar rápidamente la suspensión viral de 3 mL. Coloque el frasco a 37 ° C en un eje de balancín subibaja para agitación suave por 2 h.
- Retire y deseche el inóculo y añadir 15 mL de MEM con FCS 2%. Incubar a 37 ° C y 5% CO2 para 2-4 días.
- Comprobar la morfología de la célula y la fluorescencia de GFP (excitación a 488 nm) y emisión a 515-535 nm en un microscopio de fluorescencia invertido con 20 aumentos para estimar el momento de cosechar los virus. Tenga en cuenta que esto es generalmente cuando 50 – 80% de la capa de células HEp-2 se desconecta debido a la CPE de RSV que se produce entre 48 y 72 h postinfection (p.i.) (ver figura 3).
- Raspe todas las celdas con un raspador celular. Reunir las células y el sobrenadante y transferir a un tubo de centrífuga de 50 mL.
- Añadir el 1/10 del volumen de los 10 x solución de conservación de RSV (0,5 M HEPES y 1 M MgSO4 [pH 7.5]). Vórtex los tubos vigorosamente durante 5 s y aclarar la suspensión por un centrifugado de 5 minutos a 200 x g.
- Transfiera el sobrenadante a un tubo de 50 mL. Mezclar brevemente y alícuota de la suspensión en tubos criogénicos etiquetados con las etiquetas resistente al alcohol. Sumerja los tubos en alcohol preenfriada-80 ° C durante al menos 1 h y almacenarlas a-80 ° C.
- Liberar una de las alícuotas para valorar la suspensión viral (ver sección 4).
4. placa ensayo de titulación
- Preparar placas de 12 pocillos para valoración el día antes de que se realice el ensayo de titulación (seis pozos deberá valorar por medio de un tubo de virus). Los pozos con 1 mL de células HEp-2 en 5 x 105 células/mL en medio completo de la semilla.
-
Al día siguiente, preparar una suspensión estéril de celulosa microcristalina (2.4% [p/v] de agua) (véase tabla de materiales).
- Dispersar a 2,4 g de polvo de celulosa microcristalina en 100 mL de agua destilada, utilizando un agitador magnético estándar, hasta disolución completa del polvo (generalmente 4-12 h). Autoclave de la suspensión a 121 ° C por 20 min y almacenar a temperatura ambiente antes de usar.
Nota: Bajo tales condiciones, la suspensión es estable por 1 año. - Después de abrir la solución en un ambiente estéril, almacenar a 4 ° C durante 6 meses. Siempre mezcle la suspensión antes de usar (por mano agitación o vórtex) para asegurarse de que es homogénea.
- Dispersar a 2,4 g de polvo de celulosa microcristalina en 100 mL de agua destilada, utilizando un agitador magnético estándar, hasta disolución completa del polvo (generalmente 4-12 h). Autoclave de la suspensión a 121 ° C por 20 min y almacenar a temperatura ambiente antes de usar.
- Preparar los 2 x MEM en un ambiente estéril. Diluir comercial MEM 10 x con agua destilada y añadir L-glutamina, 1.000 unidades/mL de penicilina y estreptomicina de 1 mg/mL. Agite vigorosamente la dilución y almacenar a 4 ° C.
Nota: Realice los pasos 4.4, 4.10 en un ambiente estéril utilizando una clase de seguridad II gabinete. - Preparar seis tubos con 900 μl de MEM sin FCS por virus para titularse (la valoración). Descongelar las alícuotas de virus, tubo de vortex vigorosamente por 5 s y transferir 100 μl de la primera valoración.
- Realizar una dilución por diez 6 x, como sigue. Añadir 100 μL de virus a 900 μL del medio en el primer tubo, ponga la tapa en el tubo y mezclar su contenido con un vórtex durante unos segundos. Cambiar la punta de la pipeta, añada 100 μl de la primera dilución a 900 μl del medio en el segundo tubo, ponga la tapa en el tubo y el vórtice. Repita el procedimiento hasta el sexto tubo.
Nota: Es muy importante cambiar la punta para cada dilución. - Escriba el virus de nombre y las diluciones en las placas de 12 pozos de HEp-2. Agregar una marca para que coincida con la placa y la cubierta, ya que pueden ser separados durante la tinción (paso 4.9). Retire el medio de las placas y distribuir 400 μL de una dilución por pozo. Incubar las placas a 37 ° C por 2 h, para la adsorción del virus.
Nota: Cambiar la punta de la pipeta entre cada inóculo o proceder de menor a mayor concentración con la punta del mismo. Inocular una serie limitada de placas (1 a 2) al mismo tiempo para evitar que las células de secado. -
Preparar el recubrimiento de celulosa microcristalina en la adsorción de virus (preparación extemporánea). Para obtener 100 mL de superposición, mezclar 10 mL de 2.4% de celulosa microcristalina suspensión, 10 mL de 2 x MEM y 80 mL de MEM con FCS 2%.
- Ajustar el pH de los 2 x MEM para alrededor de 7.2 con una solución de bicarbonato de sodio estéril al 7.5%, siguiendo el indicador de color. Añadir la suspensión de celulosa microcristalina y el MEM y mezclar vigorosamente.
- Al final de la incubación de 2 h, añadir 2 a 3 mL de superposición en cada pocillo de las placas 12-bien sin retirar el inóculo. Tenga cuidado para evitar la contaminación de los pozos adyacentes con inóculo alto título viral. Incubar la placa a 37 ° C y 5% de CO2 durante 6 días. No mueva la placa y no mover la incubadora durante la incubación.
-
Proceder a la tinción de las células, utilizando solución de violeta cristal (violeta de cristal 8% [v/v] 2% formaldehído [v/v] y 20% [v/v] de etanol en agua).
- Proteger la superficie de trabajo de la seguridad de la biotecnología con una hoja (el violeta de cristal colores fuertemente las superficies).
- Agite suavemente las placas para sacar el recubrimiento de celulosa microcristalina. Eliminar el sobrenadante y lave las células 2 x 1 x solución salina tamponada con fosfato (PBS). Manejar los platos uno por uno para evitar que las células de secado. Añadir 1-2 mL de la solución de cristal violeta y esperar 10-15 minutos retirar la solución, que puede ser reutilizada para la tinción de la placa posterior.
- Sumergir las placas y las tapas en lejía fresca por unos segundos y, luego, lavarlos bien con agua del grifo. Tenga en cuenta que las placas y cubiertas son descontaminadas por el blanqueador.
- Poner las placas y las tapas en toallas de papel y deje secar. Secar las placas a una temperatura ambiente el agua de enjuague y después almacenar a temperatura ambiente. Para largos períodos de almacenamiento (meses), mantener las placas protegidas de la luz para proteger el color. Tenga en cuenta que si las células pierden su coloración, pueden ser teñidos otra vez con cristal violeta.
- Calcular los títulos de virus. Contar las placas en los pocillos de las placas secas, que son visibles a simple vista. Compruebe que el número de las placas de las diluciones diferentes es coherente (factor de 10 entre cada dilución). Elegir el bien en que las placas son los más fáciles de contar. Evaluar el número de placas versus el volumen de inóculo y la dilución.
Nota: En el ejemplo proporcionado en la figura 4, 21 placas son contadas en la dilución 10-5 . Estos corresponden a un título de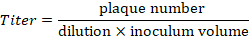
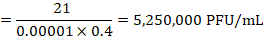
5. el uso de Virus recombinante GFP HRSV para controlar la Replicación Viral en células tratadas con ARN interferente pequeño o antivirales
Nota: Realizar todos los pasos excepto el 5.1 y 5.2.5 en un ambiente estéril utilizando una clase de seguridad II gabinete.
-
El efecto de silenciamiento sobre multiplicación de RSV del gen celular de monitoreo
Nota: El protocolo de la transfección depende del reactivo (véase Tabla de materiales).- Preparar placas de 96 pocillos para la medición de la GFP. Dos días antes del ensayo, para dado ARN interferente pequeño (siRNA), preparar una solución de medios de comunicación reducido suero que contiene siRNA en una concentración de 100 nM y un reactivo de transfección de siRNA diluido al 1/500. Incubar la solución durante 30 min a temperatura ambiente.
- Añadir 25 μl de la solución a los pozos de la placa preparada en 5.1.1 (por triplicado). Semilla de los pozos con 75 μl de una suspensión de células A549 en 4 x 105 células/mL en medio completo sin antibióticos para obtener una concentración celular final de 3 x 105 células/mL. Incube la placa durante 48 h a 37 ° C y 5% CO2.
Nota: La concentración final de siRNA es de 25 nM y el volumen de reactivo de transfección final es 0,5 μL/pocillo). - Infectar a las células como sigue. Retire el medio de los pozos. Añadir 100 μl de suspensión de RSV-GFP en 50.000 PFU/mL e incubar por 2 h a 37 ° C y 5% CO2. Retirar la suspensión viral y añadir 100 μl de DMEM con 2% de FCS y sin rojo de fenol. Incubar la placa a 37 ° C y 5% CO2.
- En 24 h y 48 h p.i., medir la fluorescencia, utilizando un spectrofluorometer a longitudes de onda de excitación y emisión de 488 y 520 nm, respectivamente (fluorescencia se expresa en unidades de fluorescencia relativa). Utilizar células A549 como estándares para los niveles de fluorescencia y el fondo.
Nota: Las células deben fijarse con 4% paraformaldehido (PFA) antes de medir sin la tapa de la placa.
-
Evaluación de la inhibición de la droga con RSV-GFP
- Preparar placas de 96 pocillos para la medición de la GFP. El día antes del ensayo, la semilla los pocillos con 100 μl de una suspensión de células HEp-2 en 5 x 105 células/mL en medio completo sin rojo de fenol.
- Preparar una dilución seriada de la probado drogas (AZ4316 en este ejemplo) en MEM complementado con 2% de FCS y los antibióticos (50 μL por pocillo). Preparar una suspensión viral 10.000 PFU/ml de MEM sin factor vascular stromal (SVF) y sin rojo de fenol (50 μL por pocillo).
- Retire el medio de la HEp-2 de 96 pocillos de la placa y añadir 50 μl de la suspensión de droga y 50 μl de la suspensión viral (por triplicado). Realizar una simulacro infección paralelamente como control.
Nota: La dilución de la droga y suspensión viral pueden ser mezclados antes de añadir en las celdas, o se puede añadir secuencialmente. - Incube la placa durante 48 h a 37 ° C y 5% CO2.
- Medida de la fluorescencia, usando un spectrofluorometer como se describe en el paso 5.1.4. Utilizar células HEp-2 infectadas con mofa como estándares para los niveles de fondo de fluorescencia.
6. Caracterización de la M2-1 localización en Vivo con el Virus recombinante VSR-M2-1-GFP
Nota: Realice los pasos 6.1 y 6.2 en un ambiente estéril, usando una clase de seguridad II gabinete.
- Preparar una suspensión de células HEp-2 en 5 x 105 células/mL en medio completo. Semillas de 1,5 mL de la suspensión celular en un 35 mm plato de Petri permeant a CO2 y adaptada para viven imágenes.
- Realice la infección el día después de la siembra con el virus RSV-M2-1-GFP en MOI 1, como se describe en pasos 3.3-3.5 (quitarlo del medio, Añadir 500 μl a 1 mL de inóculo, incubar la muestra a 37 º C agitando suavemente por 2 h; eliminar el inóculo y añadir 1,5 mL de MEM con 2 % FCS). Incube las células a 37 ° C y 5% CO2 durante el tiempo deseado (IBs empezarán a aparecer desde 10 h p.i.).
- La cámara de incubación de un microscopio invertido, dotado de 40 x a 100 objetivos x a 37 ° C, antes de colocar la placa de Petri conteniendo las células infectadas en la etapa de precalentamiento. Abra la fuente de CO2 y espere para la estabilización del foco.
- Realizar la proyección de imagen con filtros compatibles con GFP, debajo de una frecuencia de excitación baja intensidad y la imagen (de 1 a 0.1 imágenes por minuto) para minimizar la fototoxicidad.
Resultados
En este trabajo, describe un protocolo detallado para producir virus RSV recombinantes que expresan una proteína fluorescente (figura 2). PRSV-GFP, el gene GFP se introdujo entre los genes P y M, como se describe para el gene de cerezo en el trabajo anteriormente publicado21. En el pRSV-M2-1-GFP, el gen M2 fue tocado y un gen adicional que codifica M2-1-GFP fue insertado entre SH y G genes12. El primer paso, co...
Discusión
Aquí presentamos un método de rescate de recombinante RSVs de cinco plásmidos y su amplificación. La habilidad de manipular el genoma de los virus ha revolucionado la investigación de virología para probar mutaciones y expresar un gen adicional o una etiquetado proteína viral. El RSV hemos descrito y utilizado como ejemplo en este artículo es un virus que expresa un gen reportero, RSV-GFP (inédito) y expresa una proteína M2-1 fusionada a GFP etiqueta12. Rescate de RSV es difícil y requi...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores agradecen el Dr. Qin Yu de AstraZeneca R & D Boston, MA, Estados Unidos, para proporcionar la droga AZD4316. Los autores agradecemos a la plataforma de Cymages acceso al microscopio ScanR Olympus, que fue apoyado por subvenciones de la región Ile-de-France (DIM una salud). Los autores reconocen apoyo del INSERM y la Universidad de Versailles Saint-Quentin.
Materiales
| Name | Company | Catalog Number | Comments |
| 35 mm µ dish for live cell imaging | Ibidi | 81156 | |
| A549 | ATCC | ATCC CCL-185 | |
| Avicel RC-591 | FMC BioPolymer | Avicel RC-591 | Technical and other information on Avicels is available at http://www.fmcbiopolymer.com. Store at room temperature. Protocol in step 4 is optimized for this reagent. |
| BSRT7/5 | not commercially available | See reference 22. Buchholz et al. 1999 | |
| Crystal violet solution | Sigma | HT90132 | |
| Fluorescence microscope for observations | Olympus | IX73 Olympus microscope | |
| Fluorescence microscope for videomicroscopy | Olympus | ScanR Olympus microscope | |
| HEp-2 | ATCC | ATCC CCL-23 | |
| HEPES ≥99.5% | Sigma | H3375 | |
| L-Glutamine (200 mM) | ThermoFisher Scientific | 25030024 | |
| LIPOFECTAMINE 2000 REAGENT | ThermoFisher Scientific | 11668019 | Protocol in step 2.3. is optimized for this reagent. |
| MEM (10x), no glutamine | ThermoFisher Scientific | 11430030 | |
| MEM, GlutaMAX Supplement | ThermoFisher Scientific | 41090-028 | |
| MgSO4 ReagentPlus, ≥99.5% | Sigma | M7506 | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher Scientific | 51985-026 | |
| Paraformaldehyde Aqueous Solution, 32%, EM Grade | Electron Microscopy Sciences | 15714 | |
| Penicillin-Streptomycin (10,000 U/mL) | ThermoFisher Scientific | 15140122 | |
| Plasmids | not commercially available | See reference 21. Rameix-Welti et al. 2014 | |
| See Saw Rocker | VWR | 444-0341 | |
| Si RNA GAPDH | Dharmacon | ON-TARGETplus siRNA D-001810-10-05 | SMARTpool and 3 of 4 individual siRNAs designed by Dharmacon. |
| Si RNA IMPDH2 | Dharmacon | ON-TARGETplus siRNA IMPDH2 Pool- Human L-004330-00-0005 | SMARTpool of 4 individual siRNAs designed by Dharmacon. Individual references and sequences J-004330-06: GGAAAGUUGCCCAUUGUAA; J-004330-07: GCACGGCGCUUUGGUGUUC; J-004330-08: AAGGGUCAAUCCACAAAUU; J-004330-09: GGUAUGGGUUCUCUCGAUG; |
| Si RNA RSV N | Dharmacon | ON-TARGETplus custom siRNA | UUCAGAAGAACUAGAGGCUAU and UUUCAUAAAUUCACUGGGUUA |
| SiRNA NT | Dharmacon | ON-TARGETplus Non-targeting Pool | |
| SiRNA transfection reagent | Dharmacon | DharmaFECT 1 Ref: T-2001-03 | Protocol in steps 5.1.and 5.1.2 are optimized for this reagent. |
| Sodium Bicarbonate 7.5% solution | ThermoFisher Scientific | 25080094 | |
| Spectrofluorometer | Tecan | Tecan infinite M200PRO |
Referencias
- Shi, T., et al. Global, regional, and national disease burden estimates of acute lower respiratory infections due to respiratory syncytial virus in young children in 2015: a systematic review and modelling study. The Lancet. 390 (10098), 946-958 (2017).
- Falsey, A. R., Hennessey, P. A., Formica, M. A., Cox, C., Walsh, E. E. Respiratory Syncytial Virus Infection in Elderly and High-Risk Adults. The New England Journal of Medicine. 352 (17), 1749-1759 (2005).
- DeVincenzo, J. P., et al. Activity of Oral ALS-008176 in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 373 (21), 2048-2058 (2015).
- DeVincenzo, J. P., et al. Oral GS-5806 Activity in a Respiratory Syncytial Virus Challenge Study. The New England Journal of Medicine. 371 (8), 711-722 (2014).
- Afonso, C. L., et al. Taxonomy of the order Mononegavirales: update 2016. Archives of Virology. 161 (8), 2351-2360 (2016).
- Collins, P. L., Hill, M. G., Cristina, J., Grosfeld, H. Transcription elongation factor of respiratory syncytial virus, a nonsegmented negative-strand RNA virus. Proceedings of the National Academy of Sciences of the United States of America. 93 (1), 81-85 (1996).
- Hoenen, T., et al. Inclusion bodies are a site of ebolavirus replication. Journal of Virology. 86 (21), 11779-11788 (2012).
- Heinrich, B. S., Cureton, D. K., Rahmeh, A. A., Whelan, S. P. Protein expression redirects vesicular stomatitis virus RNA synthesis to cytoplasmic inclusions. PLoS Pathogens. 6 (6), e1000958 (2010).
- Lahaye, X., et al. Functional Characterization of Negri Bodies (NBs) in Rabies Virus-Infected Cells: Evidence that NBs Are Sites of Viral Transcription and Replication. Journal of Virology. 83 (16), 7948-7958 (2009).
- Kolesnikova, L., Mühlberger, E., Ryabchikova, E., Becker, S. Ultrastructural organization of recombinant Marburg virus nucleoprotein: comparison with Marburg virus inclusions. Journal of Virology. 74 (8), 3899-3904 (2000).
- Dolnik, O., Stevermann, L., Kolesnikova, L., Becker, S. Marburg virus inclusions: A virus-induced microcompartment and interface to multivesicular bodies and the late endosomal compartment. European Journal of Cell Biology. 94 (7-9), 323-331 (2015).
- Rincheval, V., et al. Functional organization of cytoplasmic inclusion bodies in cells infected by respiratory syncytial virus. Nature Communications. 8 (1), 563 (2017).
- Santangelo, P. J., Bao, G. Dynamics of filamentous viral RNPs prior to egress. Nucleic Acids Research. 35 (11), 3602-3611 (2007).
- Lifland, A. W., et al. Human Respiratory Syncytial Virus Nucleoprotein and Inclusion Bodies Antagonize the Innate Immune Response Mediated by MDA5 and MAVS. Journal of Virology. 86 (15), 8245-8258 (2012).
- Garcia, J., Garcia-Barreno, B., Vivo, A., Melero, J. A. Cytoplasmic inclusions of respiratory syncytial virus-infected cells: formation of inclusion bodies in transfected cells that coexpress the nucleoprotein, the phosphoprotein, and the 22K protein. Virology. 195 (1), 243-247 (1993).
- Brown, G., et al. Evidence for an association between heat shock protein 70 and the respiratory syncytial virus polymerase complex within lipid-raft membranes during virus infection. Virology. 338 (1), 69-80 (2005).
- Radhakrishnan, A., et al. Protein analysis of purified respiratory syncytial virus particles reveals an important role for heat shock protein 90 in virus particle assembly. Molecular & Cellular Proteomics. 9 (9), 1829-1848 (2010).
- Racaniello, V. R., Baltimore, D. Cloned poliovirus complementary DNA is infectious in mammalian cells. Science. 214 (4523), 916-919 (1981).
- Schnell, M. J., Mebatsion, T., Conzelmann, K. K. Infectious rabies viruses from cloned cDNA. The EMBO Journal. 13 (18), 4195-4203 (1994).
- Collins, P. L., et al. Production of infectious human respiratory syncytial virus from cloned cDNA confirms an essential role for the transcription elongation factor from the 5' proximal open reading frame of the M2 mRNA in gene expression and provides a capability for vaccine. Proceedings of the National Academy of Sciences of the United States of America. 92 (25), 11563-11567 (1995).
- Rameix-Welti, M. -. A., et al. Visualizing the replication of respiratory syncytial virus in cells and in living mice. Nature Communications. 5, 5104 (2014).
- Buchholz, U. J., Finke, S., Conzelmann, K. K. Generation of bovine respiratory syncytial virus (BRSV) from cDNA: BRSV NS2 is not essential for virus replication in tissue culture, and the human RSV leader region acts as a functional BRSV genome promoter. Journal of Virology. 73 (1), 251-259 (1999).
- Cianci, C., Meanwell, N., Krystal, M. Antiviral activity and molecular mechanism of an orally active respiratory syncytial virus fusion inhibitor. Journal of Antimicrobial Chemotherapy. 55 (3), 289-292 (2005).
- Derscheid, R. J., et al. Human respiratory syncytial virus memphis 37 grown in HEp-2 cells causes more severe disease in lambs than virus grown in vero cells. Viruses. 5 (11), 2881-2897 (2013).
- McKimm-Breschkin, J. L. A simplified plaque assay for respiratory syncytial virus - direct visualization of plaques without immunostaining. Journal of Virological Methods. 120, 113-117 (2004).
- Matrosovich, M., Matrosovich, T., Garten, W., Klenk, D. New low-viscosity overlay medium for viral plaque assays. Virology Journal. 7, 1-7 (2006).
- Novina, C. D., Sharp, P. A. The RNAi revolution. Nature. 430 (6996), 161-164 (2004).
- Sintchak, M. D., Nimmesgern, E. The structure of inosine 5'-monophosphate dehydrogenase and the design of novel inhibitors. Immunopharmacology. 47 (2-3), 163-184 (2000).
- Beaucourt, S., Vignuzzi, M. Ribavirin: A drug active against many viruses with multiple effects on virus replication and propagation. Molecular basis of ribavirin resistance. Current Opinion in Virology. 8, 10-15 (2014).
- Hruska, J. F., Bernstein, J. M., Douglas, R. G., Hall, C. B. Effects of Ribavirin on Respiratory Syncytial Virus in vitro. Antimicrobial Agents and Chemotherapy. 17 (5), 770-775 (1980).
- Simões, E. A. F., et al. Past, Present and Future Approaches to the Prevention and Treatment of Respiratory Syncytial Virus Infection in Children. Infectious Diseases and Therapy. 7 (1), 87-120 (2018).
- Alvarez, R., et al. RNA interference-mediated silencing of the respiratory syncytial virus nucleocapsid defines a potent antiviral strategy. Antimicrobial Agents and Chemotherapy. 53 (9), 3952-3962 (2009).
- DeVincenzo, J., et al. A randomized, double-blind, placebo-comtrolled study of an RNAi-based therapy directed against respiratory syncytial virus. Proceedings of the National Academy of Sciences of the United States of America. 107 (19), 8800-8805 (2010).
- Zhou, Y., et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Nikolic, J., et al. Negri bodies are viral factories with properties of liquid organelles. Nature Communications. 8 (1), 58 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados