Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Plataforma de vectores lentivirales para la entrega eficiente de las herramientas de edición de epigenoma en humanos inducida por modelos de enfermedad derivados de células madre pluripotentes
En este artículo
Resumen
A ADN epigenoma edición representa un potente enfoque terapéutico. Este protocolo describe la producción, purificación y concentración de vectores lentivirales all-in-one que el transgén CRISPR-dCas9-DNMT3A para aplicaciones de edición de epigenoma en células pluripotentes inducidas humanas (hiPSC)-derivado de las neuronas.
Resumen
El uso de células derivadas de hiPSC representa un valioso enfoque para el estudio de enfermedades neurodegenerativas humanas. Aquí, describimos un protocolo optimizado para la diferenciación de hiPSCs derivada de un paciente con la triplicación del lugar geométrico del gene (SNCA) de alfa-sinucleína en la enfermedad de Parkinson (EP)-las poblaciones neuronales dopaminérgicas pertinentes. La acumulación de pruebas ha demostrado que altos niveles de SNCA son causales para el desarrollo de PD. Reconociendo la insatisfecha necesita establecer nuevos enfoques terapéuticos para la EP, especialmente aquellos dirigidos a la regulación de la expresión del SNCA , recientemente desarrolló un sistema CRISPR/dCas9-metilación-basado en el ADN para modular epigenéticamente SNCA transcripción por enriquecer los niveles de metilación en la región reguladora del intrón 1 de SNCA . Para entregar el sistema, que consta de un muerto (desactivado) versión de Cas9 (dCas9) fusionado con el dominio catalítico de la 3A de enzima metiltransferasa de ADN (DNMT3A), se utiliza un vector lentivirales. Este sistema se aplica a las células con la triplicación del lugar geométrico del SNCA y reduce los niveles de proteína y SNCA-mRNA por cerca de 30% a través de la metilación del ADN específica del SNCA intrón 1. El downregulation del afinado de los niveles SNCA rescata fenotipos celulares relacionados con la enfermedad. En el protocolo actual, nuestro objetivo es describir un procedimiento paso a paso para diferenciarse hiPSCs en células progenitoras neurales (NPCs) y el establecimiento y validación de los análisis de Pirosecuenciación para la evaluación del perfil de metilación en el SNCA intrón 1. Para describir más detalladamente el sistema de lentivirus-CRISPR/dCas9 utilizado en estos experimentos, este protocolo describe cómo producir, purificar y concentrar vectores lentivirales y destacar su idoneidad para aplicaciones de edición de genoma y epigenoma utilizando hiPSCs y NPCs. El protocolo es fácilmente adaptable y puede utilizarse para producir lentivirus de alto título para aplicaciones in vitro e in vivo.
Introducción
Múltiples plataformas de edición de epigenoma han desarrollado recientemente para cualquier secuencia de ADN en las regiones que controlan la expresión de gene1,2. Las herramientas de edición de epigenoma creadas están diseñadas para (i) regular la transcripción, (ii) alterar modificaciones post-traduccionales de las histonas, (iii) modificar la metilación del ADN y (iv) modular las interacciones del elemento regulador. El enfoque para la fijación de los modificadores de la transcripción de la cromatina a un Cas9 desactivados (muertos) (dCas9) levantado de previamente desarrolladas plataformas de edición de epigenoma, como zinc dedo de proteínas (ZFPs) y efectores como activador de transcripción (cuentos), albergar un dominio potente efector transcripcional (ED) fusionados con el diseño dominio DNA-que atan (DBD)3. Los resultados del fenotipo deseado como activación o represión es definido por la molécula efectora anclada a los loci endógenos (figura 1). Para crear activadores transcripcionales programables, dCas9/gRNA módulos están vinculados con VP164,5,6 (figura 1A), un dominio de activación viral que recluta Pol II y la maquinaria de transcripción general. La modificación de este sistema ha incluido VP64, un tetrámero de dominios VP16, proporcionando una activación más robusto tipo5,6. El sistema ha sido empleado con éxito para activar regiones de codificación y los dirigidos a promotores y elementos reguladores. Lo importante es que, aunque las moléculas VP64 no modifican directamente la estructura de la cromatina en la región de destino, recluta a modificadores de la cromatina que se unen los resultados en la deposición de las marcas activa (eucromatina), como la acetilación de H3/H4 y H3 K4 di / Tri-metilación5,6. Además VP64, la subunidad p65 del humano complejo NF-kB ha sido anclada a la dCas9/gRNA módulo7. Curiosamente, la inmovilización de estos efectores a las regiones corriente arriba de los sitios de inicio de transcripción (TSSs) a promotores da como resultado una inducción del gen fuerte. Sin embargo, efectores VP64 y p65 también pueden ejercer efectos activatory al ser enlazadas a las regiones ubicadas aguas abajo de TSSs y potenciadores distal7,8. Para provocar una respuesta transcripcional más robusta, múltiples fusiones VP64 dCas9 o dCas9-p65 deben reclutarse a un solo objetivo locus9,10. Así, el reciente desarrollo de próxima generación activadores, que contratar varios dominios efector por un complejo dCas9-gRNA sola, como SunTag, ha dado como resultado una capacidad de activación más fuerte comparación con dCas9-VP64 de contrapartes fusion11 , 12. una mejor activación transcripcional ha sido obtenida mediante la fusión de VP64, p65 y Rta (VPR), un dominio de transactivación de gamma-herpesvirus, el C-terminal de dCas913 (figura 1A). Sistemas similares de CRISPR/dCas9 se han desarrollado para la represión de destino específico (figura 1B).
Represión del gen endógeno puede lograrse con fusiones de represor dirigido a través de una variedad de mecanismos (figura 1B). Se ha demostrado que CRISPR/dCas9 sistemas, relacionados con el represor DBD (incluso sin un dominio efector/s), eficaz pueden silenciar genes mientras que atado a un promotor o upstream, downstream-TSS regiones3,6 ,14. Los efectos sobre la transcripción es causada por la interferencia estérica de unión del factor de transcripción y el procesamiento de ARN polimerasa. Sin embargo, se necesitan enfoques más integrales, como represión génica por impedimento estérico sola a menudo no es suficiente para silenciar robusto. El reciente desarrollo de la próxima generación de silenciadores basados en sistemas de CRISPR/dCas9 con dominios represor transcripcional (TRD), modificadores de la histona (H3-K9 di-/ tri-metilación, H3-K27 di-/ tri-metilación; H3-K36 di-/ tri-metilación, desacetilación H3/H4) y la metilación del ADN (GPC) dirigida a la construcción de herramientas epigenéticas permitiendo más robusto silenciamiento efectos4,5,15,16, 17,18,19,20. Se ha demostrado que el reclutamiento de estos modificadores epigenéticos en el ADN puede conducir a la formación de cromatina más condensada y cerrada, que normalmente generan un silenciamiento más potente resultado21,22. El dominio silenciamiento más comúnmente utilizado con DBD es la caja asociada a Krüppel (Cascarudo)4,5. La contratación del factor se ha demostrado para corresponder con los cambios de la cromatina; sin embargo, los mecanismos de estas modificaciones deben ser elucidadas16,17,18. Recientemente, se ha demostrado que la localización de KRAB al ADN puede promover al montaje de la histona metiltransferasa SETDB1 y los complejos de NuRD histona desacetilación (HDAC), sugiriendo la posibilidad de que estas interacciones median la formación de condensación de la cromatina y silenciamiento transcripcional del3,13. Como un enfoque alternativo, los dominios efectores se pueden fusionar a DBD para crear una proteína silenciamiento epigenética personalizada. Este sistema cataliza directamente represivas marcas de ADN o modificaciones de las histonas.
Recientemente, el uso de sistemas CRISPR/dCas9 sintéticos anclada a la enzima DNMT3A ha sido repurposed para desactivación transcripcional. DNMT3A cataliza la metilación del ADN que ejerce represión transcripcional durante la formación de la heterocromatina en promotores de genes endógenos y otras regiones reguladoras (figura 1B)18,20. McDonald et al.18 y20 de Vojta et al fueron los primeros autores para informar que la metilación del ADN puede utilizarse para silenciamiento de epigenoma o represión, demostrando que el sistema de fusión de plásmido-entrega dCas9-DNMT3A potentemente puede mejorar metilación de la citosina en el TSS18,20. McDonald ' s y compañeros de trabajo demostraron que el empleo de la estrategia puede resultar en una reducción significativa (alrededor del 40%) en un gen supresor de tumores, los niveles de mRNA de CDKN2A 18. Del mismo modo, dirigida a la región unmethylated promotor de los genes de BACH o IL6ST muestra mayor metilación CpG que se ha correlacionado con una reducción de doble en la expresión génica del20. Nuestro laboratorio recientemente ha reutilizar el uso de la metilación del ADN para atenuar los resultados patológicos de SNCA sobreexpresión (figura 2)23. La estrategia se basa en la mejora selectiva en la metilación del ADN en la región del intrón 1 de SNCA , como fue divulgado previamente para ser hypomethylated en EP y demencia de Lewy (DLB) los cuerpos cerebros24,25, 26. Esta hipometilación se ha relacionado con sobreexpresión del SNCA , ofreciendo así un atractivo objetivo para la intervención terapéutica24,27,28. Nos recientemente mostró un bajo nivel de metilación del ADN en el intrón 1 del SNCA región en NPCs dopaminérgico derivado hiPSC Obtenido de un paciente de PD con el SNCA la triplicación de la23. La ventaja de este modelo experimental es que los PNJs pueden ser propagados enérgicamente en la cultura o más diferenciados en neuronas maduras, lo que permite una prueba eficiente identificar factores genéticos que median en fenotipos celulares, incluyendo oxidativo estrés y apoptosis29. Además, este modelo de sistema permite a los científicos a recapitular los acontecimientos del desarrollo que ocurrieron antes de la aparición de los síntomas en los pacientes. Además, NPCs hiPSC derivados representan una gran herramienta para poner a prueba las vías celulares y moleculares asociadas con la expresión génica. Lo importante, NPCs hiPSC derivados combinados con tecnología CRISPR/Cas9-epigenoma pueden facilitan enormemente el desarrollo de "drogas de última generación" para muchas enfermedades neurodegenerativas.
Para reducir niveles patológicos de expresión SNCA, recientemente desarrollamos un lentivirus sistema basado en que una proteína de fusión dCas9-DNMT3A y gRNA a específicamente la metilación CpG objetivo dentro del SNCA intrón 1 (figura 2A)23. Este protocolo describe vectores lentivirales (LV) diseño y producción en detalle. LVs representan un medio eficaz de ofrecer componentes CRISPR/dCas9 por varias razones, a saber: (i) su capacidad para llevar la DNA voluminoso inserciones, (ii) una alta eficiencia de transducción de una amplia gama de células, incluyendo células divisorias y nondividing30 y (iii) su capacidad para inducir respuestas inmunogénicas y citotóxicas mínima. Recientemente, aplicó el sistema del LV a neuronas dopaminérgicas hiPSC derivado de un paciente con la triplicación del lugar geométrico del SNCA y demostró el potencial terapéutico de LVs para la entrega de la edición de epigenoma metilación herramientas23 ( Figura 2B). De hecho, un sistema del LV-gRNA/dCas9-DNMT3A causa un aumento significativo en la metilación del ADN en la región del intrón 1 del SNCA . Este aumento se corresponde con la reducción de los niveles de mRNA y proteína SNCA 23. Por otra parte, downregulation del SNCA rescata fenotipos relacionados con la PD en el SNCA triplicación/hiPSC-derivado dopaminérgico neuronas (p. ej., mitocondrial ROS producción celular viabilidad y)23. Importante aún, hemos demostrado que la reducción en la expresión de SNCA por el sistema del LV-gRNA-dCas9-DMNT3A es capaz de revertir los fenotipos característicos de neuronas dopaminérgicas hiPSC derivado de un paciente de PD que llevó a la SNCA triplicación, como ROS mitocondriales producción y célula viabilidad23. El objetivo de este protocolo es 1) para indicar el protocolo de producción y concentración de una plataforma de LV optimizada para generar alta cuchicheaba preparaciones virales y 2) para describir la diferenciación de hiPSCs en NPCs con motivos para ser dopaminérgico maduro las neuronas31,32 y la caracterización de los niveles de metilación de la región específica dentro del SNCA intrón 1.
Lentivirales plataformas tienen una gran ventaja sobre la plataforma más popular de vector, es decir, adeno-asociado vectores (AAVs), que es la capacidad del primero para dar cabida a grandes inserciones genética33,34. Aireación puede generarse rendimientos significativamente más altos pero poseen una capacidad de envasado bajo (< 4,8 kb), comprometer su uso para la entrega de sistemas de CRISPR/Cas9 all-in-one. Así pues, parece que el LVs sería la plataforma de elección en las aplicaciones involucradas en la entrega de herramientas CRISPR/dCas9. Por lo tanto, el protocolo descrito aquí será una herramienta valiosa para los investigadores que deseen entregar eficazmente epigenoma edición componentes a las células y órganos. El protocolo adicional describe la estrategia para aumentar las capacidades de producción y expresión de los vectores a través de una modificación en cis de los elementos en el vector expresión cassette30,35. La estrategia se basa en la novela el sistema desarrollado y estudiado en nuestro laboratorio y destaca su habilidad para producir partículas virales en el rango de 1010 unidades virales (VU) / ml30,35.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. diseño y producción de Virus
- Construcción y diseño de plásmido
Nota: La construcción de un vector de LV-gRNA-dCas9-DNMT3A all-in-one se realiza utilizando una producción- y optimizado de expresión expresión casete publicada por Ortinski et al.30. El cassette de vector lleva una repetición del sitio de reconocimiento del factor de transcripción Sp1 y una canceladura de vanguardia dentro del sin traducir (U3') región de una repetición terminal 3'-larga (LTR) (figura 2A)30,36. La columna vertebral del vector se ha encontrado para ser eficaz en la entrega y expresando CRISPR/Cas930,35.- Obtener la versión de desactivar (muertos) de SpCas9 (dCas9) mediante mutagénesis sitio-dirigida (datos no mostrados). Vuelva a colocar el clon que mutaciones D10A y H840A en el HNH y RuvC dominios catalíticos de la enzima, respectivamente, con la activa Cas9 en pBK30129 mediante el intercambio entre fragmentos BamHI de africanas (figura 3).
- El dominio catalítico DNMT3A derivan pdCas9-DNMT3A-eGFP (véase Tabla de materiales) amplificando la DNMT3A porción BamHI-429/R 5 '- GAGCGGATCCCCCTCCCG - 3' 5 de BamHI-429/L' - CTCTCCACTGCCGGATCCGG - 3' (figura 3). Para amplificar la región conteniendo DNMT3A, utilice las siguientes condiciones: (1) 95 ° C durante 60 s, (2) 95 ° C durante 10 s, (3) 60 ° C durante 20 s, (4) 68 ° C por 60 s. Repita las condiciones 2 y 4 30 x. Para la extensión final, uso de 68 ° C por 3 min y mantener a 4 ° C.
- Copia el fragmento DNMT3A, digerido por una enzima de restricción BamHI, en el sitio de BamHI del vector modificado pBK301 con dCas9. Verificar la clonación directa Sanger secuenciación. Tenga en cuenta que el plásmido resultante alberga transgen dCas9-DNMT3A-p2a-puromicina. El plásmido expresa gRNA andamio del promotor humano del U6 (figura 3).
- Reemplazar el gen reportero de puromicina con proteína verde fluorescente (GFP) para crear dCas9-DNMT3A-p2a-GFP. Digerir el plásmido dCas9-DNMT3A-p2a-Puro con FseI. Purificar el fragmento de vector usando un método de purificación del gel. Preparar el relleno por digerir pBK201a (pLenti-GFP) con FseI. Copia el fragmento FseI en el vector. El plásmido resultante pBK539 alberga dCas9-DNMT3A-p2a-GFP transgen (figura 3).
- Cultivo de células HEK-293T y células para la transfección de la galjanoplastia
Nota: Riñón embrionario humano 293T (HEK-293T) las células se cultivan en completa alta-glucosa Dulbecco de modificado medio de águila (DMEM; 10% de suero de ternera, 1 x de antibiótico-antimicótico, piruvato de sodio x 1, 1 x aminoácido no esencial, 2 mM L-glutamina) a 37 ° C con 5% CO 2. para la reproducibilidad del Protocolo, se recomienda probar el suero cuando se cambia a un lote diferente. Hasta seis 15 cm placas se necesitan para la producción de lentivirales.- Utilizar células de paso bajo para iniciar una nueva cultura (inferior a paso 20). Una vez que las células alcanzan confluencia de 90 – 95%, los medios de comunicación de Aspire y lave suavemente con estéril de 1 x solución salina tamponada con fosfato (PBS).
- Añadir 2 mL de tripsina-EDTA (0.05%) e incubar a 37 ° C durante 3 – 5 minutos. Para inactivar el reactivo de disociación, añada 8 mL de DMEM completo de alta glucosa y pipeta 10 x x-15 con una pipeta serológica de 10 mL para crear una suspensión unicelular de 4 x 106 células/mL.
- Para las transfecciones, capa de placas de 15 cm con 0.2% de gelatina. Añadir 22,5 mL de medio de alta glucosa y las células de semillas mediante la adición de 2,5 mL de suspensión celular (total ~ 1 x 107 células/placa). Incubar las placas a 37 ° C con 5% CO2 hasta llega a confluencia de 70% – 80%.
- Transfección de células HEK-293T
- Preparar 2 x solución tampón BES BBS y 1 M de CaCl2, según Vijayraghavan y Kantor35. Filtrar las soluciones pasando a través de un filtro de 0,22 μm y almacenar a 4 ° C. La mezcla de transfección tiene que quedar claro antes de su adición a las células. Si la mezcla se convierte en turbia durante la incubación, preparar fresco 2 x BBS (pH = 6,95).
- Para preparar la mezcla de plásmido, utilice la cuatro plásmidos que se enumeran (la siguiente mezcla es suficiente para una placa de 15 cm): 37,5 μg del vector CRISPR/dCas9-transferencia (pBK492 [DNMT3A-Puro-NO-gRNA] o pBK539 [DNMT3A-GFP-NO-gRNA]); 25 μg de pBK240 (psPAX2); 12.5 μg de pMD2.G; 6.25 μg de pRSV-rev (Figura 4A). Calcular el volumen de lo plásmidos basado en las concentraciones y añadir las cantidades requeridas en un tubo cónico de 15 mL. Añadir 312.5 μl de 1 M de CaCl2 y llevar el volumen final a 1,25 mL, utilizando estéril dd-H2O. suavemente añadir 1,25 mL de solución x BBS 2 mientras Vortex la mezcla. Incubar durante 30 min a temperatura ambiente. Las células están listas para la transfección una vez confluentes de 70 – 80%.
- Aspire los medios de comunicación y sustituirla por 22,5 mL de recién preparada alta glucosa DMEM sin suero. Añadir 2,5 mL de la mezcla de transfección gota a gota a cada placa 15 cm. Remolino las placas e incubar a 37 ° C con 5% CO2 para 2-3 h.
- Después de 3 horas, añadir 2,5 mL (10%) de suero por la placa e incubar toda la noche a 37 ° C con 5% CO2.
- El día 1 después de la transfección, observar las células para asegurarse de que no hay ninguna o muerte celular mínima y que las células formaron una cultura confluente (100%).
- Cambiar el medio mediante la adición de 25 mL de suero recién preparado de DMEM y 10% de glucosa alta en cada placa.
- Incubar a 37 ° C con 5% CO2 durante 48 h.
- Cosecha del virus
- Recoger el sobrenadante de todas las células transfected y piscina en tubos cónicos de 50 mL. Centrifugar a 400 – 450 x g durante 10 minutos filtra el sobrenadante a través de una unidad de vacío del filtro 0.45 μm. Después de la filtración, el sobrenadante puede conservarse a 4 ° C para almacenamiento a corto plazo (hasta 4 días). Para el almacenamiento a largo plazo, preparar alícuotas y almacenarlas a-80 ° C.
Nota: Se espera que las preparaciones virales dispuso a ~ 2 x 107 3 x 107 vu/ml (ver sección 1.5 para la determinación del título). Se recomienda preparar alícuotas de un solo uso, ya que múltiples ciclos de congelación y descongelación se traducirá en una pérdida de 10% – 20% en títulos funcionales.
- Recoger el sobrenadante de todas las células transfected y piscina en tubos cónicos de 50 mL. Centrifugar a 400 – 450 x g durante 10 minutos filtra el sobrenadante a través de una unidad de vacío del filtro 0.45 μm. Después de la filtración, el sobrenadante puede conservarse a 4 ° C para almacenamiento a corto plazo (hasta 4 días). Para el almacenamiento a largo plazo, preparar alícuotas y almacenarlas a-80 ° C.
- Concentración de partículas virales
Nota: Para la purificación, un método de sacarosa de doble de dos etapas que implican un paso gradiente de sacarosa y un amortiguador de la sacarosa es realizada (Figura 4B).- Para crear un gradiente de sacarosa, preparar los tubos cónicos ultracentrifugación en el siguiente orden: 0,5 mL de sacarosa al 70% en PBS 1 x, 0,5 mL de sacarosa al 60% en DMEM, 1 mL de sacarosa al 30% en DMEM y 2 mL de sacarosa al 20% en PBS 1 x.
- Añadir cuidadosamente el sobrenadante, recolectado de acuerdo con la sección 1.4, al gradiente. Puesto que el volumen total de cuatro placas de 15 cm es de 100 mL, utilizar seis tubos de ultracentrifugación para procesar el sobrenadante viral.
- Igualmente distribuimos el sobrenadante viral entre cada tubo de ultracentrifugación. Para evitar la rotura del tubo durante la centrifugación, llenar los tubos de ultracentrifugación a menos de tres cuarto de su capacidad de volumen total. Equilibrar los tubos con PBS 1 x. Centrifugar las muestras a 70.000 x g durante 2 h a 17 ° C.
Nota: Para mantener la capa de sacarosa durante la aceleración y desaceleración, permiten la ultracentrífuga lentamente acelerar y decelerar el rotor de 0 a 200 x g y 200 g x 0 durante el primero y la último 3 minutos de la vuelta, respectivamente. - Suavemente recoger fracciones de sacarosa 30% – 60% en tubos limpios (Figura 4B). Añadir 1 x (frío) hasta PBS en 100 mL de volumen total. Mezclar mediante pipeteo varias veces.
- Estratificar cuidadosamente la preparación viral sobre un cojín de sacarosa añadiendo 4 mL de sacarosa al 20% (de 1 x PBS) al tubo. Continuar por pipeteo ~ 20-25 mL de la solución viral por cada tubo. Llenar los tubos con PBS 1 x, si el volumen de su contenido es menos de tres cuarto por el tubo. Equilibrar cuidadosamente los tubos. Centrifugar a 70.000 x g durante 2 h a 17 ° C. Vaciar el sobrenadante e invierta los tubos sobre toallas de papel para permitir que el resto de líquido a drenar.
- Aspirar cuidadosamente el líquido restante para quitar todo el líquido. Tenga en cuenta que, en este paso, gránulos que contienen el virus apenas son visibles como pequeñas manchas translúcidas. Añadir 70 μl de 1 x PBS al primer tubo para Resuspender el precipitado. Fondo pipetear la suspensión y transferir al tubo siguiente hasta que los pellets estén correctamente suspendidos.
- Lavar los tubos con un adicional 50 μl de 1 x PBS y mezcla como antes. Tenga en cuenta que, en este paso, el volumen de la suspensión definitiva es ~ 120 μl y aparece ligeramente lechoso. Para obtener una suspensión clara, proceder con una centrifugación s 60 a 10.000 x g. Transferir el sobrenadante a un tubo nuevo, hacer 5 alícuotas μl y almacenarlos a-80 ° C.
Nota: Preparación de vectores lentivirales es sensibles a los ciclos repetidos de congelación y descongelación. Además, se sugiere que los pasos restantes en la contención de cultivo de tejidos o en áreas calificadas en términos de estar en niveles adecuados de las normas de bioseguridad (Figura 4B) designados.
- Cuantificación de títulos virales
Nota: La estimación de títulos virales se realiza utilizando el método de ensayo (ELISA) de enzima-ligado del inmunosorbente p24 (p24gag ELISA) y según el programa de vacunas del SIDA de institutos nacionales de salud (NIH) protocolo para el análisis captura del antígeno p24 de VIH-1 con ligeras modificaciones.- Utilizar 200 μL de 0.05% Tween 20 en frío 1 x PBS (PBS-T) para lavar los pocillos de una placa de 96 pocillos 3 x.
- Para cubrir la placa, utilice 100 μl del anticuerpo monoclonal anti-p24 diluido en 1:1,500 en PBS 1 x. Incube la placa durante la noche a 4 ° C.
- Preparar el reactivo de bloqueo (1% albúmina de suero bovino [BSA] en PBS 1 x) y agregar 200 μl a cada pocillo para evitar atascamiento no específico. Utilizar 200 μL de PBS-T para lavar el pozo 3 x durante al menos 1 h a temperatura ambiente.
- Proceder con la preparación de la muestra. Cuando se trabaja con preparaciones de vector concentrado, diluir el vector a 1: 100 con 1 μl de la muestra, 89 μl de dd-H20 y 10 μl de Tritón X-100 (a una concentración final de 10%). Para las preparaciones dispuso, diluya las muestras en 1:10.
- Obtener estándares de VIH-1 mediante una dilución seriada del doble (la concentración inicial es de 5 ng/mL).
- Diluir las muestras concentradas (preparadas en el paso 1.6.4) en RPMI 1640 suplementado con 0.2% Tween 20 y 1% de BSA para obtener 1:10, 000, 1:50, 000 y las diluciones de 1: 250,000. Del mismo modo, diluya las muestras dispuso (preparadas en el paso 1.6.4) en RPMI 1640 suplementado con 0.2% Tween 20 y 1% de BSA para establecer 1: 500, 1:2, 500 y las diluciones 1:12,500.
- Agregar los estándares y las muestras en la placa en triplicado. Incubar durante una noche a 4 ° C.
- Al día siguiente, lave los pocillos de 6 x.
- Añada 100 μl del anticuerpo policlonal de conejo anti-p24 diluido en 1:1,000 en RPMI 1640, 10% suero bovino fetal (FBS), 0.25% de BSA y suero de ratón normal 2% (SNM) e incubar a 37 ° C por 4 h.
- Lavar los pozos 6 x. Añadir peroxidasa cabra anti-conejo IgG diluido al 1: 10,000 en RPMI 1640 suplementado con 5% de suero normal de cabra 2% NMS, 0.25% de BSA y 0.01% Tween 20. Incubar a 37 ° C durante 1 h.
- Lavar los pozos 6 x. Añadir el sustrato de la peroxidasa TMB e incubar a temperatura ambiente durante 15 minutos.
- Para detener la reacción, añada 100 μl de HCl N 1. En un lector de microplacas, medir la absorbancia a 450 nm.
- Medición de intensidad de reportero fluorescente
- Utilizar la suspensión viral para obtener una dilución seriada 10 veces (de 10-1 a 10-5) en PBS 1 x.
- Placa de 5 x 105 HEK-293T células en cada pozo de una placa de 6 pozos. Aplicar 10 μl de cada dilución viral a las células e incubar a 37 ° C con 5% CO2 durante 48 h.
- Proceder a la celular activado por fluorescencia clasificación análisis (FACS) como sigue: separar las células mediante la adición de 200 μL de solución de tripsina-EDTA 0,05%. Incubar las células a 37 ° C por 5 min y resuspender en 2 mL de medio DMEM (con suero). Recolectar las muestras a un tubo cónico de 15 mL y centrifugar a 400 x g a 4 ° C. Resuspender el precipitado en 500 μl de PBS 1 x fría.
- Fijar las células añadiendo 500 μl del 4% paraformaldehido (PFA) e incubar 10 min a temperatura ambiente.
- Centrifugar a 400 x g a 4 ° C y resuspender el precipitado en 1 mL de PBS 1 x. Analizar la expresión de GFP utilizando un instrumento de FACS, como se describe en Ortinski et al.30. Para determinar el título funcional del virus, utilice la siguiente fórmula.

Aquí,
TG = número de células GFP-positivas;
TN = número total de células;
N = número total de células transduced;
V = volumen utilizado para la transducción (en microlitros).
- Conteo células GFP-positivas
Nota: Determinar la multiplicidad de infección (MOI) que se emplea para la transducción. Probar una amplia gama de MOIs (de MOI = 1 a MOI = 10).- 3 x 105 a 4 x 105 HEK-293T células por cada pocillo de una placa de 6 pozos de la semilla.
- Cuando las células alcanzan > 80% de confluencia, transduce con el vector en el MOI de interés.
- Incubar a 37 ° C con 5% CO2y monitorizar la señal GFP en las células de 1 a 7 días.
- Contar el número de células GFP-positivas. Emplear un microscopio de fluorescencia (el Plan 4 x objetivo, 0.1 N.A., aumento x 40) con un filtro GFP (longitud de onda de excitación = 470 nm, longitud de onda de emisión = 525 nm). Utilice las células untransduced para establecer la población control de células GFP-negativas.
- Emplear la siguiente fórmula para determinar el título funcional del virus.
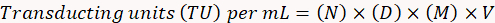
Aquí,
N = número de células GFP-positivas;
D = factor de dilución;
M = factor de aumento;
V = volumen de virus utilizado para la transducción.
Nota: Calcular los resultados siguiendo este ejemplo: para 10 células GFP-positivas (N) en una dilución (D) de 10-4 (1:10, 000) con aumento 20 x (M) en una muestra de 10 μl (V), TU por mililitro será (10 x 104) x (20) x (10) x (100) = 2 x 108 vu/mL.
2. diferenciación de las células progenitoras neuronales dopaminérgicas
- Cultivo hiPSCs
Nota: hiPSCs de un paciente con la triplicación del lugar geométrico del SNCA, ND34391, se obtuvieron de la Instituto Nacional de desórdenes neurológicos y movimiento (NINDS) catálogo (véase tabla de materiales).- Cultura hiPSCs condiciones alimentador independiente en medio de cultivo libre de alimentador de ESC-iPSC (véase Tabla de materiales) sobre la membrana de la matriz básica calificados de hESCs (BMM)-recubierto de placas (véase Tabla de materiales). Lavar las colonias confluentes con 1 mL de DMEM/F12, añadir 1 mL de reactivo de disociación (véase Tabla de materiales) e incubar 3 min a temperatura ambiente.
- Aspirar el reactivo de disociación y añadir 1 mL de medio de cultivo libre de alimentador de ESC-iPSC.
- Raspar la placa mediante un elevador de la célula y resuspender colonias en 11 mL de medio de cultivo libre de alimentador de ESC-iPSC por pipeteo x-5 4 x utilizando pipetas de borosilicato.
- 2 mL de la suspensión de la Colonia en las placas revestidas de BMM de la placa y colocar la placa a 37 ° C con 5% CO2. Realizar un cambio de medio día y dividir las celdas cada 5 – 7 días.
- Diferenciación en las células progenitoras neuronales dopaminérgicas
Nota: La diferenciación de hiPSCs en las células dopaminérgicas progenitoras neurales (MD PNJ) se realiza mediante un protocolo medio de inducción neural disponible comercialmente por las instrucciones del fabricante, con ligeras modificaciones ()32 31, Véase Tabla de materiales). El primer día de la diferenciación se considera como día 0. HiPSCs de alta calidad son necesarios para la diferenciación neuronal eficiente. La inducción de NPCs de MD es performedusing un cuerpo embryoid (EB)-basado en el protocolo.- Antes de comenzar la diferenciación de hiPSCs, preparar una placa de cultivo de micropocillos (véase Tabla de materiales) según las instrucciones del fabricante.
- Después de preparar la placa de cultivo de pocillos, agregar 1 mL de medio de inducción neural (NIM; véase Tabla de materiales) suplementado con 10 μm Y-27632.
- Coloque la placa a un lado hasta que esté listo para usar.
- Lavar hiPSCs con DMEM/F12, añadir 1 mL de solución de separación celular (véase Tabla de materiales) e incubar por 5 min a 37 ° C con 5% CO2.
- Resuspender las células en DMEM/F12 y centrifugar a 300 x g durante 5 minutos.
- Cuidadosamente aspirar el sobrenadante y resuspender las células en NIM + 10 μm Y-27632 para obtener una concentración final de 3 x 106 células/mL.
- Añadir 1 mL de la suspensión unicelular a un único pozo de la placa de micropocillos cultura y centrifugar la placa a 100 x g durante 3 minutos.
- Examinar la placa bajo el microscopio para asegurar una distribución uniforme de las células entre el pocillo e incubar las células a 37 ° C con 5% CO2.
- En los días 1 – 4, realizar un cambio de medio parcial diario.
- Usando una micropipeta de 1 mL, 1,5 mL de medio de Quite y deseche. Lentamente, añadir 1,5 mL de NIM fresco sin Y-27632.
- Repita el paso 2.2.10 hasta el día 4.
- Día 5, un pocillo de una placa de 6 pozos con BMM. de la capa
- Colocar un filtro reversible de 37 μm (véase Tabla de materiales) en la parte superior un tubo cónico de 50 mL (residuos). Punto de la flecha hacia arriba del filtro reversible.
- Retire el medio de la placa de micropocillos cultura sin molestar a los EBs formado.
- Añadir 1 mL de DMEM/F12 y rápidamente recoger del EBs con la pipeta de borosilicato y filtrarlas a través de la coladera.
- Repita paso 2.2.15 hasta EBs todas se quitan de la placa de cultivo de micropocillos.
- Invertir el colador sobre un nuevo tubo cónico de 50 mL y añadir 2 mL de NIM para recoger todo el EBs.
- Placa de 2 mL de la suspensión de la EB en un único pozo de la placa revestida de BMM con una pipeta de borosilicato. Incubar el EBs a 37 ° C con 5% CO2.
- Día 6, preparar 2 mL de NIM + 200 ng/mL SHH (véase Tabla de materiales) y realizar un cambio de media diario.
- El día 8, examinar el porcentaje de inducción neuronal.
- Cuenta todos EBs y, específicamente, determinar el número de cada EB individual que está llena de rosetas de nervios. Cuantificar la inducción neural roseta utilizando la siguiente fórmula.
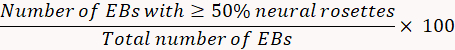
Nota: Si la inducción neural es < % 75, selección neural roseta puede ser ineficiente. - El día 12, preparar 250 mL de medio de N2B27 con 119 mL de medio de neurobasal, 119 mL de medio DMEM/F12, 2,5 mL de GlutaMAX, 2,5 mL de AANE, 2,5 mL de suplemento para N2, 5 mL de B27 sin vitamina A, 250 μL de gentamicina (50 mg/mL) y 19. 66 μL de BSA (7 mg/mL).
- Para preparar 50 mL de medio completo de N2B27, añadir 3 μM CHIR99021, μM 2 SB431542, 20 ng/mL bFGF, EGF ng/mL 20 y 200 ng/mL SHH.
Nota: Es importante preparar el medio justo antes de usar. - Medio de los pozos que contienen las neuronales rosetas de aspirar y lavar con 1 mL de DMEM/F12.
- Anuncio 1 mL de reactivo de selección neural roseta (véase Tabla de materiales) e incubar a 37 ° C con 5% CO2 para 1 h.
- Eliminar el reactivo de selección y, utilizando una pipeta de 1 mL, apuntar directamente a los racimos de la roseta.
- Añadir la suspensión a un tubo cónico de 15 mL y repetir pasos 2.2.25 y 2.2.26 hasta la mayoría de los grupos neuronales roseta se han recogido.
Nota: Para evitar la contaminación con tipos celulares nonneuronal, no overselect. - Centrifugue la suspensión de la roseta a 350 x g por 5 min aspirar el sobrenadante y resuspender las neuronales rosetas en N2B27 + 200 ng/mL SHH. Añadir la suspensión de roseta neural a un pozo cubierto de BMM e incubar la placa a 37 ° C con 5% CO2.
- En los días 13 y 17, realizar un cambio medio diario por medio de N2B27 terminado. Paso de las células cuando las culturas son confluentes de 80 – 90%.
- Para dividir las células, prepare una placa revestida de BMM.
- Lavar las células con 1 mL de DMEM/F12, aspirar el medio y añadir 1 mL de reactivo de disociación (véase Tabla de materiales).
- Incubar por 5 min a 37 ° C, añadir 1 mL de DMEM/F12 y desalojar células conectadas mediante pipeteo arriba y abajo. Recoger la suspensión de la APN en un tubo cónico de 15 mL. Centrifugar a 300 x g durante 5 minutos.
- Aspirar el sobrenadante y resuspender las células en 1 mL de N2B27 completa + 200 ng/mL SHH.
- Contar las células de las placa a una densidad de 1,25 x 105 células/cm2e Incube las células a 37 ° C con 5% CO2.
- Cambiar el medio cada otro día, con completa N2B27 + 200 ng/mL SHH.
Nota: En este pasaje, NPCs consideran como paso (P) 0. SHH puede ser retirada del medio N2B27 en P2. - Paso de las células una vez que lleguen a 80 – 90% de confluencia.
- En esta etapa, confirman que las células expresan marcadores Nestin y FoxA2 mediante inmunocitoquímica y qPCR. Este protocolo permite la generación de las células doble positivas de 85% para los marcadores Nestin y FoxA2.
- Para pases de células, repita los pasos 2.2.31–2.2.36.
- Congelación de las células, a partir de paso P2. Para la congelación de las células, repita los pasos 2.2.31–2.2.36 y resuspender el precipitado de células en 2 x 106 a 4 x 106 células/mL mediante frío progenitoras neurales medio de congelación (véase Tabla de materiales).
- Transfiera 1 mL de la suspensión celular en cada cryovial y congelar las células mediante un sistema de refrigeración estándar lento-velocidad-controlado. Para el almacenamiento a largo plazo, mantener las células en nitrógeno líquido.
- Descongelación de MD de NPCs
- Preparar una placa revestida de BMM y caliente completa N2B27. Añadir 10 mL de DMEM/F12 caliente a un tubo cónico de 15 mL. Colocar un cryovial en un bloque de calor de 37 ° C por 2 min.
- Transferencia de las células de la cryovial en el tubo que contenga DMEM/F12. Centrifugar a 300 x g durante 5 minutos.
- Aspirar el sobrenadante, resuspender las células en 2 mL de N2B27 y añadir la suspensión de células a un pocillo de la placa revestida de BMM. Incube las células a 37 ° C con 5% CO2.
- Preparar una placa revestida de BMM y caliente completa N2B27. Añadir 10 mL de DMEM/F12 caliente a un tubo cónico de 15 mL. Colocar un cryovial en un bloque de calor de 37 ° C por 2 min.
3. transducción de NPCs de MD y el análisis de los cambios de metilación
-
Transducción de NPCs MD
- Transduce MD NPCs en confluencia de 70% con LV-gRNA/dCas9-DNMT3A vectores en MOI = 2. Vuelva a colocar el posttransduction de N2B27 medio 16 h.
- Añadir N2B27 con puromicina de 5 μg/mL, 48 horas después de la transducción. Las células de la cultura durante 3 semanas en N2B27 plus puromicina para obtener las líneas MD NPC estables. Las células están preparadas para aplicaciones posteriores (DNA, RNA, análisis de proteína, caracterización fenotípica23, congelación y pases).
-
Caracterización del perfil de metilación del intrón 1 de SNCA
- Extraer el ADN de cada célula estable transduced línea mediante una extracción de DNA kit (véase Tabla de materiales).
- Uso 800 ng de ADN para realizar una conversión de bisulfito, utilizando comercialmente disponible kit (véase Tabla de materiales). Después de la conversión de bisulfito, eluir la DNA bisulfito-convertido a 20 ng/μl.
-
PCR para el análisis de pirosecuenciación
- Preparar la mezcla principal de PCR en un tubo libre de nucleasas. Para cada reacción, se utiliza 0,4 μL de cebador inverso (10 μm), 0.4 μL de cebador hacia adelante (10 μm), 1,6 μl de MgCl2 (25 mM), 2 μl de 10 x CoralLoad concentrado, 10 μl de la mezcla principal de 2 x PCR, 4 μL de 5 x Q-solución, 1 μl de ADN y 0,6 μl de agua libre de nucleasa.
- Transferir la placa de reacción a un termociclador y realizar la polimerización en cadena usando las siguientes condiciones: 95 ° C durante 15 min, 50 ciclos de 94 ° C por 30 s, 56 ° C durante 30 s y 72 ° C por 30 s, con un paso de extensión final de 10 min a 72 ° C. Cebadores utilizados para la pirosecuenciación del intrón 1 de SNCA se enumeran en tabla 1y Figura 7A figura 1 complementaria.
- Después de la amplificación, visualizar los amplicones con 2 μl del producto de PCR con bromuro de etidio coloración, siguiente electroforesis en gel de agarosa.
- Análisis de pirosecuenciación se validan mediante el uso de mezclas de unmethylated (U) y metilado (M) convertido a bisulfito DNAs en las proporciones siguientes, a saber, 100U:0M, 75U:25M, 50U:50M, 25U:75M y 0U:100M (véase Tabla de materiales).
- Conducta de pirosecuenciación pirosecuenciación reactivos (véase Tabla de materiales) y calcular los valores de metilación para cada sitio de GPC mediante pirosecuenciación software. Un protocolo detallado de la pirosecuenciación, consulte Bassil et al37.
Access restricted. Please log in or start a trial to view this content.
Resultados
Validación de los títulos de la producción de los vectores de LV-dCas9-DNMT3A-GFP/Puro en comparación con las contrapartes GFP de ingenuo
Realizamos p24gag ELISA para comparar entre títulos físicos de LV-dCas9-DNMT3A-GFP/Puro con las contrapartes GFP/Puro ingenuo. Resultados representativos, presentados en la figura 5A, demuestran que los rendimientos físicos de lo...
Access restricted. Please log in or start a trial to view this content.
Discusión
LVs han comenzado a emerger como el vehículo de elección para la edición de epigenoma, especialmente en el contexto de enfermedades genéticas, debido principalmente a su capacidad para (i) grandes cargas útiles de DNA y (ii) transducen eficazmente una amplia gama de dividir y células nondividing. La eficacia de empaquetado grande de las LVs es especialmente beneficiosa para las aplicaciones de embalaje de los sistemas CRISPR/dCas9 que son de gran tamaño. Desde esta perspectiva, LVs representan la plataforma de ele...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Universidad de Duke presentó una solicitud de patente provisional relacionada con este estudio.
Agradecimientos
Este trabajo fue financiado en parte por el Kahn Neurotecnología desarrollo Premio (C.O.), institutos de salud nacional Instituto Nacional de desórdenes neurológicos y derrame cerebral (NINDS/NIH) (NS085011 R01 para C.O.).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| 0.22 μM filter unit, 1 L | Corning | 430513 | |
| 0.45-μm filter unit, 500 mL | Corning | 430773 | |
| 100 mm TC-Treated Culture Dish | Corning | 430167 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 50 mL conical centrifuge tubes | Corning | 430291 | |
| 6-well plates | Corning | 3516 | |
| Aggrewell 800 | StemCell Technologies | 34811 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| BD FACS | Becton Dickinson | 338960 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| Eppendorf Cell Imaging Slides | Eppendorf | 30742060 | |
| High-binding 96-well plates | Corning | 3366 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Reversible Strainer | StemCell Technologies | 27215 | |
| SW32Ti rotor | Beckman Coulter | 369650 | |
| VWR® Disposable Serological Pipets, Glass, Nonpyrogenic | VWR | 93000-694 | |
| VWR® Vacuum Filtration Systems | VWR | 89220-694 | |
| xMark™ Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| Accutase | StemCell Technologies | 7920 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| Anti-FOXA2 Antibody | Abcam | Ab60721 | |
| Anti-Nestin Antibody | Abcam | Ab18102 | |
| Antibiotic-antimycotic solution, 100x | Sigma Aldrich | A5955-100ML | |
| B-27 Supplement (50x), minus vitamin A | Thermo Fisher Scientific | 12587010 | |
| BES | Sigma Aldrich | B9879 - BES | |
| Bovine Albumin Fraction V (7.5% solution) | Thermo Fisher Scientific | 15260037 | |
| CHIR99021 | StemCell Technologies | 72052 | |
| Corning Matrigel hESC-Qualified Matrix | Corning | 08-774-552 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| DMEM-F12 | Lonza | 12-719 | |
| DMEM, high glucose media | Gibco | 11965 | |
| DNeasy Blood & Tissue Kit | Qiagen | 69504 | |
| EpiTect PCR Control DNA Set | Qiagen | 596945 | |
| EZ DNA Methylation Kit | Zymo Research | D5001 | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Gentamicin | Thermo Fisher Scientific | 15750078 | |
| Gentle Cell Dissociation Reagent | stemCell Technologies | 7174 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Human Recombinant bFGF | StemCell Technologies | 78003 | |
| Human Recombinant EGF | StemCell Technologies | 78006 | |
| Human Recombinant Shh (C24II) | StemCell Technologies | 78065 | |
| MEM Non-Essential Amino Acids Solution (100x) | Thermo Fisher Scientific | 11140050 | |
| mTeSR1 | StemCell Technologies | 85850 | |
| N-2 Supplement (100x) | Thermo Fisher Scientific | 17502001 | |
| Neurobasal Medium | Thermo Fisher Scientific | 21103049 | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| PyroMark PCR Kit | Qiagen | 978703 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| SB431542 | StemCell Technologies | 72232 | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| STEMdiff Neural Induction Medium | StemCell Technologies | 5835 | |
| STEMdiff Neural Progenitor Freezing Medium | StemCell Technologies | 5838 | |
| TaqMan Assay FOXA2 | Thermo Fisher Scientific | Hs00232764 | |
| TaqMan Assay GAPDH | Thermo Fisher Scientific | Hs99999905 | |
| TaqMan Assay Nestin | Thermo Fisher Scientific | Hs04187831 | |
| TaqMan Assay OCT4 | Thermo Fisher Scientific | Hs04260367 | |
| TaqMan Assay PPIA | Thermo Fisher Scientific | Hs99999904 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| Y27632 | StemCell Technologies | 72302 | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Goat serum, Sterile, 10 mL | Sigma | G9023 | Working concentration 1:1000 |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Normal mouse serum, Sterile, 500 mL | Equitech-Bio | SM30-0500 | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| pMD2.G | Addgene | 12253 | |
| pRSV-Rev | Addgene | 52961 | |
| psPAX2 | Addgene | 12259 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsmBI | New England Biolabs | R0580S | |
| BsrGI | New England Biolabs | R0575S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
Referencias
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Gaj, T., Gersbach, C. A., Barbas, C. F. 3rd ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology. 31 (7), 397-405 (2013).
- Thakore, P. I., Black, J. B., Hilton, I. B., Gersbach, C. A. Editing the epigenome: technologies for programmable transcription and epigenetic modulation. Nature Methods. 13 (2), 127-137 (2016).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Gilbert, L. A., et al. Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. Cell. 159 (3), 647-661 (2014).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nature Methods. 10 (10), 973-976 (2013).
- Chavez, A., et al. Highly efficient Cas9-mediated transcriptional programming. Nature Methods. 12 (4), 326-328 (2015).
- Horlbeck, M. A., et al. Nucleosomes impede Cas9 access to DNA in vivo and in vitro. eLife. 5, (2016).
- Chavez, A., et al. Comparison of Cas9 activators in multiple species. Nature Methods. 13 (7), 563-567 (2016).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21 (3), 440-446 (2018).
- Tanenbaum, M. E., Gilbert, L. A., Qi, L. S., Weissman, J. S., Vale, R. D. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell. 159 (3), 635-646 (2014).
- Konermann, S., et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature. 517 (7536), 583-588 (2015).
- Holtzman, L., Gersbach, C. A. Editing the Epigenome: Reshaping the Genomic Landscape. Annual Review of Genomics and Human Genetics. , (2018).
- Perez-Pinera, P., et al. Synergistic and tunable human gene activation by combinations of synthetic transcription factors. Nature Methods. 10 (3), 239-242 (2013).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nature Methods. 12 (12), 1143-1149 (2015).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- McDonald, J. I., et al. Reprogrammable CRISPR/Cas9-based system for inducing site-specific DNA methylation. Biology Open. 5 (6), 866-874 (2016).
- Huang, Y. H., et al. DNA epigenome editing using CRISPR-Cas SunTag-directed DNMT3A. Genome Biology. 18 (1), 176(2017).
- Vojta, A., et al. Repurposing the CRISPR-Cas9 system for targeted DNA methylation. Nucleic Acids Research. 44 (12), 5615-5628 (2016).
- Razin, A., Kantor, B. DNA methylation in epigenetic control of gene expression. Progress in Molecular and Subcellular Biology. 38, 151-167 (2005).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proceedings of the National Academy of Sciences of the United States of America. 106 (44), 18786-18791 (2009).
- Kantor, B., et al. Downregulation of SNCA Expression by Targeted Editing of DNA Methylation: A Potential Strategy for Precision Therapy in PD. Molecular Therapy. , (2018).
- Jowaed, A., Schmitt, I., Kaut, O., Wullner, U. Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. Journal of Neuroscience. 30 (18), 6355-6359 (2010).
- Wang, Y., et al. A DNA methyltransferase inhibitor, 5-aza-2'-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson's disease-related genes in dopaminergic neurons. CNS Neuroscience & Therapeutics. 19 (3), 183-190 (2013).
- Matsumoto, L., et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson's disease. PLOS One. 5 (11), e15522(2010).
- Desplats, P., et al. Alpha-synuclein sequesters Dnmt1 from the nucleus: a novel mechanism for epigenetic alterations in Lewy body diseases. Journal of Biological Chemistry. 286 (11), 9031-9037 (2011).
- Ai, S. X., et al. Hypomethylation of SNCA in blood of patients with sporadic Parkinson's disease. Journal of the Neurological Sciences. 337 (1-2), 123-128 (2014).
- Tagliafierro, L., Chiba-Falek, O. Up-regulation of SNCA gene expression: implications to synucleinopathies. Neurogenetics. 17 (3), 145-157 (2016).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Molecular Therapy - Methods & Clinical Development. 5, 153-164 (2017).
- Tagliafierro, L., et al. Genetic analysis of alpha-synuclein 3' untranslated region and its corresponding microRNAs in relation to Parkinson's disease compared to dementia with Lewy bodies. Alzheimer’s & Dementia. 13 (11), 1237-1250 (2017).
- Tagliafierro, L., Zamora, M. E., Chiba-Falek, O. Multiplication of the SNCA locus exacerbates neuronal nuclear aging. Human Molecular Genetics. , (2018).
- Kantor, B., McCown, T., Leone, P., Gray, S. J. Clinical applications involving CNS gene transfer. Advances in Genetics. 87, 71-124 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Advances in Genetics. 87, 125-197 (2014).
- Vijayraghavan, S., Kantor, B. A Protocol for the Production of Integrase-deficient Lentiviral Vectors for CRISPR/Cas9-mediated Gene Knockout in Dividing Cells. Journal of Visualized Experiments. (130), e56915(2017).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Molecular Therapy. 16 (12), 1968-1976 (2008).
- Bassil, C. F., Huang, Z., Murphy, S. K. Bisulfite pyrosequencing. Methods in Molecular Biology. 1049, 95-107 (2013).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Research. 43 (13), 6450-6458 (2015).
- Nishimasu, H., et al. Crystal Structure of Staphylococcus aureus Cas9. Cell. 162 (5), 1113-1126 (2015).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. Journal of Virology. 68 (4), 2632-2648 (1994).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. Journal of Virology. 71 (8), 6113-6127 (1997).
- Goffin, V., et al. Transcription factor binding sites in the pol gene intragenic regulatory region of HIV-1 are important for virus infectivity. Nucleic Acids Research. 33 (13), 4285-4310 (2005).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. Journal of Biological Chemistry. 280 (22), 21545-21552 (2005).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Molecular Therapy. 19 (3), 547-556 (2011).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados