Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación del riesgo y la prevalencia del cáncer colorrectal mediante la detección de la integridad del ADN de las heces
En este artículo
Erratum Notice
Resumen
El kit de diagnóstico de FL-DNA presentado es un método fácil de ahorrar tiempo y fácil de usar para determinar la probabilidad confiable de la presencia de lesiones por cáncer colorrectal.
Resumen
Hoy en día, el ADN de las heces puede ser aislado y analizado por varios métodos. Los fragmentos largos de ADN en las heces pueden ser detectados por un ensayo qPCR, que proporciona una probabilidad confiable de la presencia de lesiones colorrectales pre-neoplásicas o neoplásicas. Este método, llamado ADN largo de fluorescencia (FL-DNA), es un procedimiento rápido y no invasivo que mejora el sistema de prevención primaria. Este método se basa en la evaluación de la integridad del ADN fecal mediante la amplificación cuantitativa de objetivos específicos del ADN genómico. En particular, la evaluación de fragmentos de ADN de más de 200 bp permite la detección de pacientes con lesiones colorrectales con muy alta especificidad. Sin embargo, este sistema y todas las pruebas de ADN de heces disponibles actualmente presentan algunas cuestiones generales que deben abordarse (por ejemplo, la frecuencia con la que se deben llevar a cabo las pruebas y el número óptimo de muestras de heces recogidas en cada punto de tiempo para cada individuo). Sin embargo, la principal ventaja de FL-DNA es la posibilidad de utilizarlo en asociación con una prueba utilizada actualmente en el programa de cribado de CRC, conocido como análisis de sangre oculta en heces basada en inmunoquímicos (iFOBT). De hecho, ambas pruebas se pueden realizar en la misma muestra, reduciendo los costos y logrando una mejor predicción de la presencia final de lesiones colorrectales.
Introducción
El cáncer colorrectal (CRC) deriva de un proceso de varios pasos en el que el epitelio sano se convierte lentamente en adenomas o pólipos, que progresan en carcinomas malignos con el tiempo1,,2. A pesar de la alta tasa de incidencia de la CRC, se ha observado una tendencia a la baja en el porcentaje de muertes en la última década3. De hecho, las herramientas de diagnóstico temprano adoptadas en los programas de cribado han llevado a la detección y eliminación temprana de adenomas pre-neoplásicos o pólipos4. Sin embargo, debido a los diferentes límites técnicos, ninguno de estos métodos es óptimo. De hecho, con el fin de mejorar la sensibilidad y la especificidad, muchas pruebas de ADN de heces se han propuesto solo o en combinación con las pruebas diagnósticas de rutina actuales5,6.
Típicamente, la mucosa sana se arroja en los colonocitos apoptóticos de la corriente fecal, mientras que la mucosa enferma exfolia los colonocitos no apoptóticos. Fragmentos de 200 bp o más de longitud caracterizan el ADN no apoptótico. Este ADN se denomina ADN largo (ADN-L) y se ha convertido en un biomarcador utilizable para el diagnóstico temprano de la CRC. El L-DNA puede ser aislado de la muestra de heces y cuantificado por qPCR utilizando un kit de ADN FL de diagnóstico in vitro7,8,9,10,11,12.
La prueba consta de dos ensayos para la detección de fragmentos de FL-DNA que van desde 138 bp a 339 bp. Cada ensayo permite la amplificación de FL-DNA (FAM) así como el ADN de pico (HEX). Para garantizar una amplificación óptima de todos los fragmentos, la prueba se ha dividido en dos ensayos (denominados "A" y "B"). El ensayo A detecta dos regiones del exón 14 del gen APC (NM_001127511) y un fragmento del exón 7 del gen TP53 (NM_001276760). El ensayo B detecta un fragmento del exón 14 del gen APC (NM_001127511) y dos regiones de los exones 5 y 8 del gen TP53 (NM_001276760). Los ensayos no distinguen entre las regiones detectadas. El ADN de espiga corresponde al ADN salmón Oncorhynchus keta y permite verificar que el procedimiento se ha realizado correctamente y comprueba la posible presencia de inhibidores, lo que puede producir resultados falsos negativos. La concentración de FL-DNA se evalúa mediante cuantificación absoluta utilizando el método de curva estándar y se expresa como ng/reacción.
El método FL-DNA es una prueba de ADN de heces no invasiva y económica que, combinada con el análisis de sangre oculta en heces (iFOBT) basado en inmunoquímicos, se utiliza actualmente en los programas de cribado de CRC y permite mejores predicciones de lesiones de CRC y/o adenoma de alto riesgo12.
Protocolo
Los pacientes fueron reclutados en el Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) de Meldola (FC, Italia) entre 2013 y 2015. Los pacientes inscritos estaban en el protocolo IRSTB002, aprobado por el Comité de ética del IRST - IRCCS AVR (25/10/2012, ver. 1). Todos los métodos se realizaron de acuerdo con las directrices y regulaciones pertinentes. Se obtuvo el consentimiento informado por escrito de todos los pacientes.
1. Extracción de ADN de heces
- Utilice un kit para preparar muestras de heces (consulte Tabla de materiales). Seleccione y trate el material fecal realizando la extracción de acuerdo con las instrucciones del fabricante. Amplificar el ADN purificado directamente o almacenar a -20 oC para su posterior análisis.
2. Preparación de control positivo, estándares, ADN con clavos y muestras clínicas
- Preparación de normas y muestras
- Para preparar el control positivo, las normas, el ADN con espiga y todas las muestras clínicas, centrifugar una alícuota de control positivo, estándares y ADN con espiga, y luego resuspender cada reactivo añadiendo la cantidad correcta de agua proporcionada (ver más abajo). Luego, vórtice cuidadosamente el control positivo, estándar, y spike-in ADN, luego centrífuga para 10 s. Para lograr una resuspensión completa de los reactivos secos, almacene los reactivos líquidos a temperatura ambiente (RT) durante 30 minutos antes de su uso.
- El control positivo es el ADN humano en un formato seco. Resuspender cada alícuota con 750 ml de agua.
- El ADN de espiga es el ADN de salmón (Oncorhynchus keta), que se utiliza como un control interno exógeno para verificar la posible presencia de inhibidores en muestras de ADN extraídas de heces. Resuspender cada alícuota con 100 ml de agua.
- Para preparar la curva estándar, produzca cuatro diluciones 1:5 a partir de la solución de stock. Los puntos estándar deben ser 10 ng/reacción, 2 ng/reacción, 0,4 ng/reacción y 0,08 ng/reacción.
- Para preparar el control positivo, las normas, el ADN con espiga y todas las muestras clínicas, centrifugar una alícuota de control positivo, estándares y ADN con espiga, y luego resuspender cada reactivo añadiendo la cantidad correcta de agua proporcionada (ver más abajo). Luego, vórtice cuidadosamente el control positivo, estándar, y spike-in ADN, luego centrífuga para 10 s. Para lograr una resuspensión completa de los reactivos secos, almacene los reactivos líquidos a temperatura ambiente (RT) durante 30 minutos antes de su uso.
- Preparación del ADN de 1x espiga en
- Prepare el control de ADN de pico directamente antes de usarlo.
- Preparar el control de ADN de 1x pico-en mezclando 5 s de pico FL-DNa con 20 ml de agua estéril. El número de muestras de control de ADN 1x spike-in se preparará de acuerdo con el número de muestras a analizar, más el control positivo.
- Preparación de muestras
- Mezclar 75 l de las muestras (muestras clínicas o control positivo) con 25 ml de ADN de 1x espiga, lo que produce un volumen total de 100 l.
3. Amplificación y determinación del valor FL-DNA utilizando qPCR Easy PGX
NOTA: Las mezclas de amplificación completas que contienen imprimaciones y sondas específicas dirigidas al ADN humano y el control interno se proporcionan en un formato liofilizado en 8 tiras de pozo para FL-DNA Mix A y FL-DNA Mix B. Estándares, controles positivos y negativos, y las muestras deben amplificarse con ambas mezclas liofilizadas. Las muestras clínicas solo deben amplificarse por duplicado con ambas mezclas liofilizadas.
- Consulte la Tabla de materiales para el instrumento qPCR y el software operativo.
- Abra el software de funcionamiento y configure la placa y el perfil térmico:
- Configure la placa como se muestra en la Tabla 1.
- Establezca el tipo de pozo para las ocho posiciones de la columna 1 como Estándar.
- Establezca el tipo de pozo para los pozos A2 y B2 como NTC.
- Establezca el tipo de pozo para C2 y D2 (los controles positivos) como Desconocido.
- Establezca el tipo de pozo para todas las demás posiciones como Desconocido.
- Seleccione las 96 posiciones y agregue los tintes FAM y HEX. Haga clic en Sincronizar placa.
- Establezca el perfil térmico según la Tabla 2.
- Configure la placa como se muestra en la Tabla 1.
- Centrifugar el número necesario de tiras durante 10 s para llevar el contenido a la parte inferior del tubo.
- Retire suavemente los sellos de las tiras, mientras presta atención a retener el contenido, y añadir a las tiras respectivas: control negativo: 20 l de agua; muestra: 20 l de ADN; curva estándar: 20 l de 1, 2, 3 o 4 de serie; control positivo: 20 ml de control positivo.
- Cierre cuidadosamente todas las tiras utilizando las tapas ópticas planas de 8 tiras y el vórtice durante unos segundos.
- Centrifugar las tiras durante 10 s y cargarlas en el instrumento. Entonces, comienza la carrera.
- Abra el software de funcionamiento y configure la placa y el perfil térmico:
4. Análisis de datos
NOTA: El análisis de datos se puede realizar de forma automática o manual en función del software (consulte Tabla de materiales).
- Al final de la ejecución, seleccione las columnas A, C, E, G para "FAM: FL-DNA-A" y"HEX: IC"y las columnas B, D, F, H para "FAM: FL-DNA-B" y"HEX: IC".
- Establezca lo siguiente para el Importe de inicio de cantidades estándar:10 ng/reacción para pozos A1 y B1, 2 ng/reacción para C1 y D1, 0,4 ng/reacción para E1 y F1, y 0,08 ng/reacción para G1 y H1.
- Establezca los valores de fluorescencia de umbral en 150 para los canales FAM (FL-DNA A y FL-DNA B) y HEX (IC).
- En el cuadro Tabla de resultados, haga clic en Opciones de columna . Seleccione Todo ? Ok para obtener los resultados en ambos canales con sus respectivos valores Cq (R) y R.
NOTA: Estos valores son suministrados por el software de instrumentos PCR en tiempo real. El último valor de fluorescencia corresponde al último ciclo de amplificación. - En el cuadro Tabla de resultados, haga clic con el botón derecho en la tabla para abrir el menú contextual y haga clic en Enviar a Excel para exportar los datos sin procesar.
- Compruebe los valores de las normas para verificar la idoneidad de la curva estándar.
- Para cada mezcla de FL-DNA, compruebe la R2 ["R2 (columna R)" y la eficiencia ["Eficiencia (%)" columna]. Si se encuentran en un rango aceptable, es posible proceder con el análisis de acuerdo con las instrucciones del fabricante (Tabla 3).
- Si los resultados del canal FAM no están en el rango esperado, omita un punto de la curva estándar y vuelva a analizar la ejecución.
- Determine los valores de los controles negativos y positivos con la siguiente fórmula, considerando los valores "No Cq" como cero:
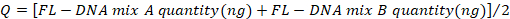
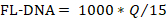
- Compare los valores obtenidos con los reportados en la Tabla 4.
- Si los controles de reacción están en el rango de valores esperados, proceda con el análisis de las muestras.
NOTA: Compruebe que los valores Cq obtenidos se generan a partir de una reacción de amplificación real (curva de fluorescencia sigmoidal) y no de un artefacto (curva de fluorescencia lineal). - Para analizar la idoneidad de la muestra para cada mezcla FL-DNA, compare los valores Cq del canal HEX. Si el valor es 16, proceda con el análisis de las muestras. Si el valor es <16 o no hay Cq, es probable que se deba a un error de dosificación del pico FL-DNA. Por lo tanto, no es posible analizar las muestras.
- Calcule el promedio de los valores Cq en el canal "HEX" del control positivo utilizando la siguiente fórmula:

- Calcule el promedio de los valores Cq en el canal "HEX" de las réplicas de muestra utilizando la siguiente fórmula:
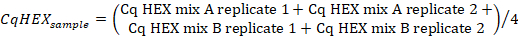
- Calcule los valores de CqHEX de acuerdo con la siguiente fórmula:
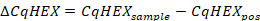
- Compare los valores de CqHEX de las muestras con los notificados en la Tabla 5.
- Para cada mezcla (Mix A y Mix B), compare los valores Cq del canal FAM con los reportados en la Tabla 6.
- Para determinar el valor FL-DNA de cada muestra adecuada, utilice la siguiente fórmula, considerando los valores "No Cq" como cero:
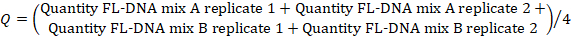
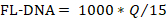
NOTA: El riesgo y la prevalencia del cáncer colorrectal es una función de las evaluaciones iFOBT y FL-DNA de acuerdo con los resultados del nomograma de Fagan obtenidos por Rengucci et al.12 (Tabla 7).
Resultados
El flujo de trabajo de este protocolo se muestra en la figura 1. El flujo de trabajo proporciona dos pasos de control y acciones diferentes según estos resultados de paso. En primer lugar, si una muestra presenta controles inadecuados, la amplificación debe repetirse. En segundo lugar, si se inhibe la amplificación, la muestra debe ser reprocesada desde el principio o clasificada como no valiosa.
La Figura 2 muestra las curvas de fluorescencia producidas po...
Discusión
Estudios anteriores han demostrado que el análisis de la integridad del ADN de las heces extraídas por enfoques manuales y semiautomáticos puede representar una herramienta alternativa para la detección temprana de lesiones colorrectales7,8,9,10,11,12. A lo largo de los años se han desarrollado pruebas de detección mole...
Divulgaciones
Maura Menghi es empleada a tiempo completo de Diatech Pharmacogenetics srl.
Agradecimientos
Los autores no tienen reconocimientos.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL and 2 mL polypropylene twist-lock tubes (DNase-, RNase-, DNA-, PCR inhibitor-free) | Consumables required for DNA extraction and Real Time PCR | ||
| Absolute Ethanol (quality of analytical degree) | Reagent required for DNA extraction | ||
| Benchtop centrifuge | Maximum speed of 20000 x g. Instrument required for DNA extraction | ||
| EasyPGX analysis software version 2.0.0 | Diatech Pharmacogenetics | RT800-SW | Analysis software |
| EasyPGX centrifuge/vortex 8-well strips | Diatech Pharmacogenetics | RT803 | Instrument recommended for the Real Time PCR assay |
| EasyPGX qPCR instrument 96 | Diatech Pharmacogenetics | RT800-96 | Instrument recommended for the Real Time PCR assay |
| EasyPGX ready FL-DNA | Diatech Pharmacogenetics | RT029 | Kit required for the Real Time PCR assay |
| Micropipettes (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Powder-free disposable gloves | Consumables required for DNA extraction and Real Time PCR | ||
| QIAamp Fast DNA Stool | Qiagen | 51604 | Kit recommended for the DNA extraction and purification from stool |
| Sterile filter tips DNase-, RNase-free (volumes from 1 to 1.000 µL) | Consumables required for DNA extraction and Real Time PCR | ||
| Thermal block e.g. EasyPGX dry block | Diatech Pharmacogenetics | RT801 | Instrument required for DNA extraction |
| Vortex e.g. EasyPGX centrifuge/vortex 1.5 ml | Diatech Pharmacogenetics | RT802 | Instrument required for DNA extraction |
Referencias
- Fearon, E. R. Molecular Genetics of Colorectal Cancer. Annual Review of Pathology. 6, 479-507 (2011).
- Sears, C. L., Garrett, W. S. Microbes, Microbiota, and Colon Cancer. Cell Host and Microbe. 15, 317-328 (2014).
- Levin, B., et al. Screening and Surveillance for the Early Detection of Colorectal Cancer and Adenomatous Polyps, 2008: A Joint Guideline From the American Cancer Society, the US Multi-Society Task Force on Colorectal Cancer, and the American College of Radiology. Gastroenterology. 134, 1570-1595 (2008).
- Bosch, L. J., et al. Molecular tests for colorectal cancer screening. Clinical Colorectal Cancer. 10, 8-23 (2011).
- Ahlquist, D. A. Molecular detection of colorectal neoplasia. Gastroenterology. 138, 2127-2139 (2010).
- Calistri, D., et al. Fecal multiple molecular tests to detect colorectal cancer in stool. Clinical Gastroenterology and Hepatology. 1, 377-383 (2003).
- Calistri, D., et al. Detection of colorectal cancer by a quantitative fluorescence determination of DNA amplification in stool. Neoplasia. 6, 536-540 (2004).
- Calistri, D., et al. Quantitative fluorescence determination of long-fragment DNA in stool as a marker for the early detection of colorectal cancer. Cellular Oncology. 31, 11-17 (2009).
- Calistri, D., et al. Fecal DNA for noninvasive diagnosis of colorectal cancer in immunochemical fecal occult blood test-positive individuals. Cancer Epidemiology Biomarkers and Prevention. 19, 2647-2654 (2010).
- De Maio, G., et al. Circulating and stool nucleic acid analysis for colorectal cancer diagnosis. World Journal of Gastroenterology. 20, 957-967 (2014).
- Rengucci, C., et al. Improved stool DNA integrity method for early colorectal cancer diagnosis. Cancer Epidemiology Biomarkers and Prevention. 23, 2553-2560 (2014).
Erratum
Formal Correction: Erratum: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection
Posted by JoVE Editors on 9/28/2020. Citeable Link.
An erratum was issued for: Evaluation of Colorectal Cancer Risk and Prevalence by Stool DNA Integrity Detection. An affiliation was updated.
The first affiliation was updated from:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST)
to:
Istituto Scientifico Romagnolo per lo Studio e la Cura dei Tumori (IRST) IRCCS, Meldola, Italy
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados