Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Multiplexación y transfección de plasmión de ADN de alto rendimiento mediante tecnología de nanodispensación acústica
En este artículo
Resumen
Este protocolo describe la transfección de plásmido de alto rendimiento de células de mamíferos en una placa de 384 pocillos utilizando tecnología de eyección de gotas acústicas. La dispensación y multiplexación de ADN, que consume mucho tiempo y propensa a errores, pero también la dosificación del reactivo de transfección, son impulsadas por software y realizadas por un dispositivo de nanodispensador. Las células son sembradas en estos pozos precargados.
Resumen
La transfección celular, indispensable para muchos estudios biológicos, requiere controlar muchos parámetros para un logro preciso y exitoso. La mayoría de las veces se realiza a bajo rendimiento, además es lento y propenso a errores, aún más cuando multiplexa varios plásmidos. Hemos desarrollado un método fácil, rápido y preciso para realizar la transfección celular en un diseño de placa de 384 pocillos utilizando la tecnología de eyección de gotas acústicas (ADE). El dispositivo de nanodispensador utilizado en este estudio se basa en esta tecnología y permite la entrega precisa de nanovolúmenes a alta velocidad desde una placa de pozo de origen a una placa de destino. Puede dispensar y multiplexar ADN y reactivo de transfección de acuerdo con una hoja de cálculo prediseñada. Aquí presentamos un protocolo óptimo para realizar la transfección de plásmido de alto rendimiento basada en ADE que permite alcanzar una eficiencia de hasta el 90% y una cotransfección de casi el 100% en experimentos de cotransfección. Ampliamos el trabajo inicial proponiendo una macro basada en hojas de cálculo fácil de usar, capaz de administrar hasta cuatro plásmidos/pozos de una biblioteca que contiene hasta 1.536 plásmidos diferentes, y una aplicación de guía de pipeteo basada en tabletas. La macro diseña las plantillas necesarias de las placas de origen y genera los archivos listos para usar para la aplicación basada en nanodispensador y tableta. El protocolo de transfección de cuatro pasos implica i) un diluyente dispense con un manipulador de líquidos clásico, ii) distribución y multiplexación de plásmidos, iii) un reactivo de transfección dispensando por el nanodispensador, y iv) chapado celular en los pozos precargados. El control basado en software descrito de multiplexación y transfección de plástidos ADE permite incluso a los no especialistas en el campo realizar una transfección celular confiable de una manera rápida y segura. Este método permite una rápida identificación de la configuración óptima para un tipo de celda determinado y se puede transponer a enfoques manuales y de mayor escala. El protocolo facilita aplicaciones, como la proteína ORFeome humana (conjunto de marcos de lectura abiertos [ORF] en un genoma) o la validación de la función génica basada en CRISPR-Cas9, en estrategias de cribado no agrupadas.
Introducción
El método presentado aquí describe en detalle cómo realizar multiplexación y transfección de plásmido de ADN en células de mamíferos a alto rendimiento utilizando un nanodispensador líquido de base acústica en una placa de 384 pocillos, incluso para no especialistas en el campo. Este método publicado recientemente1 permite realizar hasta 384 condiciones independientes de multiplexación y transfección de ADN plásmido en un experimento, en menos de 1 h. Los experimentos individuales o de cotransfección tuvieron éxito, alcanzando un cotransfección dentro de la población de células transtrinfectadas. Este protocolo facilita la transfección porque la mayoría de los pasos tediosos, lentos y propensos a errores ahora son controlados por software (consulte la figura 1 para obtener una visión general). Se han hecho más esfuerzos para desarrollar herramientas específicas para mejorar la facilidad de uso evitando al mismo tiempo errores humanos durante el proceso general y para promover la transfección exitosa incluso para los no especialistas en la materia. El protocolo descrito incluye una hoja de cálculo macro "fácil de usar" que hemos desarrollado con el fin de gestionar 384 condiciones de transfección independientes con posibilidades de multiplexación de hasta cuatro plásmidos en cada pozo. La macro genera automáticamente plantillas de las placas de origen para cargar el volumen de plásmido de ADN esperado desde el inicio de las soluciones de stock y los archivos necesarios para conducir el software nanodispensador sobre el diseño experimental que se ha introducido. Como la dosificación manual del ADN en una placa fuente de 384 pocillos es tediosa y propensa a errores, también desarrollamos una aplicación dedicada a la tableta para guiar al usuario mientras dispensamos la solución de ADN de acuerdo con la plantilla.
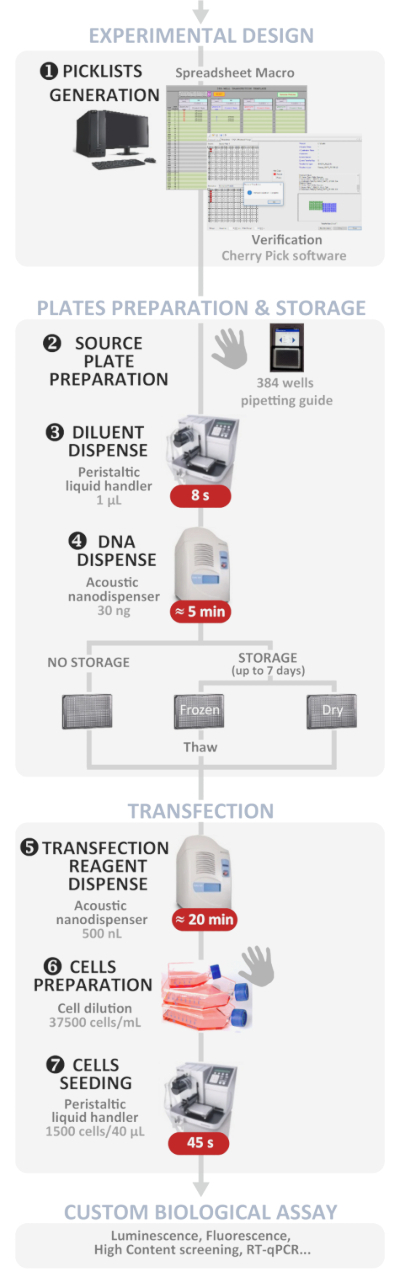
Figura 1: Flujo de trabajo experimental. Representación esquemática del protocolo automatizado óptimo de transfección inversa de alto rendimiento (desde el diseño experimental hasta el ensayo biológico personalizado). Los pasos manuales se indican mediante el símbolo de la mano y la hora aproximada de cada paso se escribe en un cuadro rojo. Haga clic aquí para ver una versión más grande de esta figura.
Muchos experimentos basados en células comienzan con la transfección de ADN de plásmido, e incluso si muchos reactivos dedicados han sido y todavía se están desarrollando para mejorar la eficiencia de la transfección y / o facilitar el procedimiento, queda mucho por hacer2,3 , 4. La transfección celular de plásmido de ADN implica varios pasos para alcanzar una alta eficiencia, como una toma compleja inicial, escape endosomal y transporte citoplasmático al núcleo5,6. Además de la precipitación de calcio o técnicas físicas como la electroporación o la microinyección utilizando dispositivos dedicados7, los métodos químicos modernos se han centrado en mejorar la entrega de células de ADN mientras se reduce la toxicidad celular8, 9. El uso de lípidos o polímeros catiónicos que forman complejos liposomas y, más recientemente, sistemas de química polimérica no liposómica ha hecho que la transfección sea más fácil y eficiente10. A pesar de estos avances, la transfección celular todavía requiere habilidades específicas para ser realizadas con precisión, ya que la mayoría de estos protocolos de transfección física o química requieren que los científicos preparen manualmente cada condición de reacción de transfección de ADN, por lo tanto deteriorando el rendimiento. Para evitar este problema, se han desarrollado protocolos de transfección inversa utilizando reactivos de transfección química11,12,13,lo que permite al usuario probar o combinar varios plásmidos de una manera más rápida. En estos protocolos, se forman complejos de ácido nucleico con reactivos de transfección antes de sembrar las células en los complejos. Sin embargo, estos protocolos inversos siguen estando limitados por el manejo manual de las soluciones de ADN y por la combinación de cada una de las condiciones independientes. Aunque es factible realizarlos en un formato de placa de 96 pocillos, la preparación del ADN y los dispensadores serán tediosos, y es probable que haya errores. Cuando se requieren diferentes cantidades de varios plásmidos de ADN y se multiplexan entre sí, la transfección celular se vuelve aún más difícil de lograr y consume más tiempo, y los errores humanos se vuelven bastante inevitables. Escalar hasta el formato de placa de 384 pocillos en un enfoque de transfección inversa, a pesar de las pocas condiciones de transfección de ADN multiplexado, se convierte en un desafío imposible debido a las siguientes razones. i) Las cantidades de ADN, el reactivo de transfección o los volúmenes de mezcla de reacción que se deben manejar son inferiores a 1 l para cada poca. ii) La multiplexación de plásmidos para 384 condiciones independientes se vuelve extremadamente complicada. La entrega en cada uno de los 384 pozos también es iii) muy lento y iv) propenso a errores. De hecho, la dispensación de la solución correcta en los pozos esperados es difícil de gestionar porque los volúmenes bajos ya dispensados no permiten la supervisión visual entre los pozos vacíos y ya llenos. v) Por último, existe un alto riesgo de secado de la mezcla por evaporación antes de que se añadan las células debido al tiempo necesario para realizar los pasos de dosificación necesarios. En resumen, el factor limitante para configurar ensayos de transfección de plaños de ADN de alto rendimiento parece ser la miniaturización del ensayo, lo que implica multiplexación y gestión de bajo volumen que ya no se puede manejar manualmente, pero que tampoco se pueden lograr en un forma fiable por los manipuladores de líquidos peristáticos clásicos.
Como prueba de dificultad para automatizar tales ensayos y obtener un alto rendimiento, sólo unos pocos intentos de automatizar la transfección se han publicado hasta ahora: un formato de placa de 96 pocillos utilizando un dispositivo comercial de manipulación de líquidos y precipitación de fosfato cálcico14 y, más recientemente, un reactivo lipoplex, y un chip microfluídico que permite 280 transfecciones independientes15 pero que requieren habilidades especializadas en este campo. Otro método, la acoustopforesis, que permitía la levitación de líquidos y que conducía a la manipulación y mezcla de fluidos, se utilizó para realizar la transfección de ADN en formatos de placa de 24 a 96 pocillos16. Aunque es factible, este enfoque sufre de un rendimiento extremadamente bajo, ya que la mezcla de células con mezcla de transfección de ADN requiere una incubación de 60 s para cada punto antes de la sembración. Esto implica una duración de al menos 96 minutos para una placa completa de 96 pocillos. Además, este protocolo está lejos de ser susceptible de la audiencia general de los biólogos, ya que este trabajo se realizó con un dispositivo interno diseñado y fabricado que actualmente no está disponible en el mercado. Por el contrario, en los últimos años, una tecnología de dosificación basada en acústica basada en software fácil de usar ha surgido con dispositivos dispensadores de nanovolúmenes. Utilizando energía acústica enfocada, estos dispositivos permiten la expulsión estrechamente controlada de pequeños volúmenes de líquido de 2,5 nL a 500 nL desde una placa de origen hasta un destinode 17. Esta tecnología, llamada eyección de gotas acústicas (ADE), tiene numerosas ventajas: es totalmente automatizada, sin contacto, sin puntas, precisa, precisa y altamente reproducible, y tiene un alto rendimiento18. Primero dedicado a la entrega de soluciones de dimetilsulfóxido (DMSO), los ajustes se han mejorado para dispensar tampones acuosos19. Los nanodispensadores acústicos, entonces, parecen adecuados para los protocolos de transfección de células inversas y podrían eludir la mayoría de las limitaciones manuales antes mencionadas. Como no se describieron previamente los intentos de transfección de plásmidos utilizando esta tecnología, recientemente evaluamos la idoneidad de un sistema de dosificación basado en acústica para realizar la transfección de células inversas.
Aprovechando el rendimiento del nanodispensador y la facilidad de uso, optimizamos un protocolo de transfección inversa para las células HeLa mediante pruebas cruzadas de varios parámetros que pueden influir en la transfección del ADN en una placa única de 384 pocillos, a saber, la cantidad total de ADN y concentración inicial del ADN fuente, volumen diluyente, reactivo de transfección y número de células diseminados. El protocolo desarrollado elude las limitaciones manuales de transfección celular antes descritas y presenta varias ventajas sobre otros intentos de transfección automatizados. En primer lugar, se miniaturiza, lo que permite un reactivo de transfección rentable al ahorrar preparaciones de plásmido de ADN y reactivo de transfección. En segundo lugar, es mucho más alto rendimiento y reproducible que el protocolo manual (incluso para principiantes), ya que la transfección de toda una placa de 384 pocillos se puede lograr en menos de 1 h. Por último, está impulsado por software, permitiendo el control de la cantidad de ADN dispensado y la multiplexación de varios plásmidos. De hecho, gracias al software nanodispenser (Tablade Materiales),el usuario puede elaborar un plan de estudio para controlar los volúmenes a dispensar desde una placa de pozo de origen definida a una placa de destino.
El protocolo presentado aquí está destinado principalmente a aquellos que tienen acceso a un nanodispensador y les gustaría establecer experimentos de transfección a alto rendimiento, pero también para aquellos que quieren optimizar rápidamente sus parámetros de transfección para un tipo de celda dado por aplicando este protocolo para probar varios parámetros a un alto rendimiento. De hecho, hemos demostrado que los parámetros optimizados identificados con este protocolo a nanoescala se pueden transponer a experimentos de transfección manuales y a mayor escala. Por último, dado que el reactivo de transfección utilizado en el presente protocolo permite la transfección de ADN o siRNA según el fabricante, el protocolo también es de interés para aquellos que tienen como objetivo realizar enfoques de matriz para la sobreexpresión genética o derribo. Las placas de destino precargadas con ADN se pueden conservar hasta 7 días antes de su uso en un ensayo de transfección sin pérdida de eficacia, que es otra ventaja del siguiente protocolo para este tipo de aplicación.
Protocolo
1. Preparaciones anticipadas
- Preparación de los programas de manipulación de líquidos peristálticos
NOTA: Para los pasos de dosificación de diluyentes y células del protocolo, se debe preparar un programa dedicado, teniendo en cuenta la altura del cabezal dispensador a la placa utilizada y la intención del paso.- Para el paso de dispensación de diluyente de 1 l, monte un cassette de 1 l y prepare un programa con los ajustes descritos en los pasos 1.1.1.1 y 1.1.1.2.
- Ajuste el parámetro de caudal a Alto para obtener el mejor rendimiento, ya que no se espera ningún daño material biológico en este paso. Ajustar la altura de la dosificación a 9,6 mm (según la placa de cultivo celular utilizada, Figura suplementaria 1) para permitir que la caída de 1 L toque la parte inferior de los pozos durante la dispensación.
NOTA: Este paso es crucial para evitar la retención de las gotas en el cabezal dispensador hasta alcanzar un volumen suficiente para caer. - Ajuste la altura clara de la placa a 14,4 mm para permitir un desplazamiento libre del cabezal dispensador sobre la placa después de dispensar cada fila. Controle visualmente los ajustes adecuados de la altura de la cabeza del controlador de líquido peristáltico: asegúrese de que no se conserven gotas en las puntas de dosificación durante la dosificación y compruebe que la cabeza es lo suficientemente alta como para permitir el desplazamiento de la cabeza después de dispensar cada fila.
NOTA: Evitar la retención de caídas es un parámetro crucial, ya que perjudicará la precisión del volumen de la dispensación.
- Ajuste el parámetro de caudal a Alto para obtener el mejor rendimiento, ya que no se espera ningún daño material biológico en este paso. Ajustar la altura de la dosificación a 9,6 mm (según la placa de cultivo celular utilizada, Figura suplementaria 1) para permitir que la caída de 1 L toque la parte inferior de los pozos durante la dispensación.
- Para la dosificación de la suspensión de celda de 40 l, monte un cassette de 10 ml y prepare un programa con los ajustes descritos en los pasos 1.1.2.1-1.1.2.2.
- Ajuste el parámetro de caudal a Bajo para dispensar celdas con una velocidad baja para evitar promover posibles daños a las células por tensión cortante y alto impacto en la parte inferior de los pozos. Ajuste la altura de dispensación a 11,43 mm (según la placa de cultivo celular utilizada, Figura suplementaria 1), lo suficientemente alta como para reducir el impacto celular en la parte inferior de los pozos durante el proceso de dosificación, pero lo suficientemente baja como para evitar la retención de las gotas en el cabeza dispensadora. Ajuste la altura clara de la placa a 16 mm para permitir el desplazamiento libre del cabezal dispensador sobre la placa después de dispensar cada fila.
- Controle visualmente los ajustes adecuados de la altura de la cabeza del controlador de líquido peristáltico: asegúrese de que no se conserven gotas en las puntas de dosificación durante la dosificación y compruebe que la cabeza es lo suficientemente alta como para permitir el desplazamiento de la cabeza después de dispensar cada fila.
NOTA: Evitar la retención de caídas es un parámetro crucial, ya que conducirá a la dispensación de un número de celda poco fiable.
- Para el paso de dispensación de diluyente de 1 l, monte un cassette de 1 l y prepare un programa con los ajustes descritos en los pasos 1.1.1.1 y 1.1.1.2.
- Preparación del plásmido de ADN (protocolo clásico de extracción miniprep)
- Cultivar una cepa de bacterias DH5 transformada en medio LB suplementada con 125 g/ml de antibiótico de selección de ampicilina (Tablade materiales)durante la noche a 37 oC y bajo agitación suave (200 rpm) en un agitador orbital (Tabla de materiales).
- Cosecha 2 mL del cultivo, peletlas por centrifugación durante 5 min a 6.000 x g, y descarta el sobrenadante.
- Resuspenda el gránulo de celda con 250 ml de tampón de resuspensión que contenga RNase A (Tablade materiales). Añadir 250 l de tampón de lisis e incubar durante 5 minutos a temperatura ambiente, de acuerdo con las instrucciones del fabricante.
- Detenga la reacción de lisis añadiendo 300 s de búfer de neutralización (Tablade materiales)y vortexting en breve. Centrifugar los tubos durante 5 min a 11.000 x g.
- Coloque una nueva minicolumna de plásmido (Tablade Materiales)en un tubo de recogida de 2 ml y decantele el sobrenadante en la columna centrifugando durante 1 min a 11.000 x g.
- Deseche el flujo y vuelva a colocar la minicolumna en el tubo de recogida.
- Lavar la minicolumna de plásmido con 500 ml de tampón de lavado opcional (Tabla deMateriales)y centrífuga durante 1 min a 11.000 x g, de acuerdo con las instrucciones del fabricante.
- Deseche el flujo y vuelva a colocar la minicolumna plásmido en el tubo de recogida.
- Añadir 700 l de tampón de lavado (Tablade materiales)complementado con etanol y centrífuga durante 1 min a 11.000 x g,de acuerdo con las instrucciones del fabricante.
- Deseche el flujo y centrifugar la minicolumna plásmido y su tubo de recogida 1 veces más durante 2 min a 11.000 x g para secar la membrana de sílice.
- Colocar la minicolumna de plásmido seco en un nuevo tubo de 1,5 ml y añadir 30 ml de agua destilada precalentada a 60 oC, incubarla durante 2 minutos a temperatura ambiente, y luego, centrifugela durante 1 min a 11.000 x g.
- Deseche la minicolumna plásmido y mantenga el eluido que contiene el plásmido de ADN purificado.
- Mida la concentración de ADN del ADN eludado utilizando un espectrofotómetro de microvolúmenes (Tablade materiales).
- Encienda el espectrofotómetro y elija los ajustes de medición de ADN.
- Elevar el brazo de muestreo del espectrofotómetro y la pipeta 1 l de agua en el pedestal de medición para realizar una calibración en blanco.
- Baje el brazo de muestreo, inicie la medición en blanco y espere a que se complete.
- Levante el brazo de muestreo y limpie la muestra de los pedestales superior e inferior.
- Pipetear 1 l de la solución de ADN en el pedestal inferior para medirlo.
- Baje el brazo de muestreo, inicie la medición de la concentración de ADN y espere a que se complete.
- Levante el brazo de muestreo y limpie la muestra de los pedestales superior e inferior.
- Para mediciones adicionales de la concentración de ADN, repita los pasos 1.2.13.5-1.2.13.7.
- Una vez finalizadas las mediciones, guarde las soluciones de ADN a 4oC hasta su uso.
2. Diseño experimental y generación de las listas de selección para impulsar los dispensadores basados en ADE
NOTA: Se desarrolló una macro de hoja de cálculo "fácil de usar" dedicada para administrar cantidades de ADN y mezclar hasta cuatro plásmidos en un formato de placa de 384 pocillos. Basado en el diseño experimental introducido, esta macro genera los archivos necesarios para conducir el protocolo de transfección de ADN basado en ADE por nanodispenser. Para generar estos archivos, varios campos tienen que ser llenados en la hoja de plantilla tal y como se muestra en de la figura2.
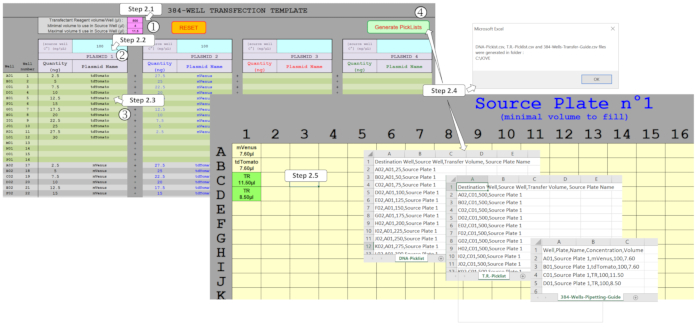
Figura 2 : Generación de las listas de selección para impulsar la dispensación de ADE mediante la macro de hoja de cálculo. Hay que rellenar varios parámetros, a saber ,( 1) el reactivo de transfección (TR) y los volúmenes mínimos/máximos que se utilizarán en la placa de origen, (2) las concentraciones iniciales de plásmido que se dispensarán en la placa de origen, y (3) el diseño de placa completa, incluyendo las cantidades de plásmido esperadas y multiplexación en cada uno de los 384 pozos. (4) La activación Generar listas de selección permite verificar los diferentes campos y, una vez rellenados correctamente, se generan automáticamente listas de selección para la dispensación de ADN y TR y la plantilla de placa de origen necesaria. Haga clic aquí para ver una versión más grande de esta figura.
- Introduzca los parámetros del protocolo nanodispenser en los campos rosados. Establezca el valor de mezcla del reactivo de transfección (TR) en 500 nL. Fije el valor mínimo del volumen en los pocillos de la placa de origen a 4 l. Ajuste el volumen máximo en los pocillos de la placa de origen a 11,25 l.
NOTA: El nanodispensador utilizado aquí sólo puede transferir un máximo de 500 nL en una ejecución de ADE. Estos campos rosados se rellenan previamente con los valores recomendados, pero se pueden modificar según las necesidades del usuario. - Introduzca las concentraciones iniciales de ADN de 100 ng/L en los campos azules correspondientes al ADN subyacente.
NOTA: Este valor es la concentración óptima definida previamente, pero, sin embargo, se puede modificar para diferentes necesidades del usuario. - Introduzca la cantidad de ADN deseada en los campos gris/verde. Introduzca los importes y los nombres plásmidos de los 384 pozos, asegurando la misma ortografía si se utiliza el mismo plásmido en varios pozos.
- Genere el diseño de la placa de origen, los archivos de listas de selección y el archivo de guía de pipeteo. Haga clic en Generar listas de selección para permitir que la macro genere los archivos DNA-Picklist.csv, T.R.-Picklist.csv y 384-Wells-Pipetting-Guide.csv a partir de los datos recopilados en la hoja correspondiente. Si se solicita, corrija los valores de celda rellenos de naranja, ya que indica errores o volúmenes que el nanodispensador no puede controlar.
- Imprima las plantillas desde la hoja Placa de origen. Se indican los nombres plásmidos y el volumen mínimo para rellenar los pozos. Del mismo modo, los volúmenes de mezcla de reactivos de transfección que a continuación tendrán que rellenarse en los siguientes pozos se indican como TR y se resaltan en verde.
3. Preparación de la placa de origen de ADN utilizando la aplicación de guía de pipeteo de 384 pocillos
- Diluir el plásmido de ADN almacenado desde el paso 1.2.14 hasta 100 ng/-L utilizando agua destilada.
- Calibre la rejilla de 384 pocillos a las dimensiones de la placa: abra la aplicación de guía de pipeteo de 384 pocillos en una tableta (Figura3). Coloque la placa de origen en la cuadrícula en la pantalla inferior, y en el menú de calibración superior izquierdo, haga clic en + o - (o utilice el cursor rojo) para mejorar o reducir el tamaño de la rejilla y los pozos con el fin de ajustar los pozos verdes a los cuatro pozos de esquina de la placa .

Figura 3 : Uso de la aplicación de guía de pipeteo de 384 pozos. (1) Calibración de la rejilla de 384 pocillos al tamaño de la placa; (2) ) Montaje de un adaptador de placa impreso en 3D universal en la tableta mediante cinta adhesiva de doble cara; (3) Colocación de la placa en el adaptador; (4) Desplazamiento de la rejilla para centrarla en la placa montada. (5) Bloqueo del paso de calibración. (6) Apertura del archivo 384 wells pipetting guide.csv. (7) Dada la lista de archivos, la aplicación indicará el nombre esperado de la placa fuente, el reactivo (ADN o reactivo de transfección), la concentración y el volumen a dispensar en los pozos de destino, que se iluminarán uno por uno. (8) Los botones de flecha izquierda y derecha permiten al usuario seguir la guía de pipeteo para dispensar fácilmente los reactivos de acuerdo con las plantillas de placa de origen de macro de hoja de cálculo. Haga clic aquí para ver una versión más grande de esta figura.
- Con cinta adhesiva de doble cara, monte el adaptador de placa impresa en 3D en la pantalla para evitar movimientos de la placa de origen durante la dosificación. Si es necesario, mueva la rejilla calibrada utilizando las flechas de rotación y los botones Arriba/Abajo/Derecha/Izquierda para ajustar la cuadrícula de la pantalla a la posición de la placa. Una vez que la rejilla y los tamaños del pozo estén correctamente calibrados y localizados, marque la casilla Bloquear calibración.
- Haga clic en ARCHIVO y abra el archivo 384 wells pipetting guide.csv. Siga las instrucciones de la pantalla para dispensar manualmente el volumen indicado del plásmido indicado en la concentración indicada en el pozo blanco resaltado correspondiente al destino objetivo adecuado de la placa esperada. Utilice - o + flechas para volver o más en el proceso de dosificación de ADN. Deje de dispensar al llegar a la primera solución de reactivo de transfección que se va a cargar.
- Una vez finalizadas las dispensaciones de ADN, retire la placa fuente del adaptador. Si se tienen que llenar varias placas de origen, coloque una nueva placa de origen en el adaptador y siga las instrucciones de dosificación. Una vez finalizada la dispensación de ADN, centrifugar la(s) placa(s) fuente(s) llena(s) de ADN (a 1.500 x g durante 2 min) para asegurar una nivelación adecuada del líquido y eliminar las burbujas que conducen a la inexactitud en las transferencias basadas en ADE.
4. Dispensación de diluyente peristáltica a base de manipulador de líquidos de 1 l en la placa de destino
NOTA: Realice los pasos 4.1-4.5 en un armario de seguridad biológica.
- Desinfectar el cabezal de cassette de 1 l rociándolo con un desinfectante por pulverización (Tablade materiales)y permitir que esta solución entre en el soporte de la punta. Absorba el desinfectante remanente en papel absorbente. Monte el cassette de 1 l en el dispositivo de manipulador de líquidos peristálticos. Encienda el dispositivo y asegúrese de que el ajuste del tipo de cassette es correcto (1 L), así como el formato de la placa (384 pocillos).
- Desinfectar todo el lumen del tubo: inserte el organizador del tubo (sosteniendo los ocho tubos juntos) en un recipiente estéril y llénelo con 5 ml de 70% de alcohol. Usando la función de cebado del manipulador de líquidos peristálticos, primero enjuague el alcohol en el tubo y luego enjuáguelo pasando 5 ml de agua destilada y 5 ml de medio libre de suero (medio de águila modificado de Dulbecco [DMEM] complementado con 100 U/ml penicilina-estreptomicina; Tabla de Material), rellenando sucesivamente el mismo recipiente. Asegúrese de que ninguna de las puntas esté obstruida inspeccionando visualmente el flujo de líquido de todos ellos.
- Prepara el tubo con medio libre de suero llenando un nuevo recipiente estéril con 10 ml de medio sin suero precalentado y buceando en él el organizador del tubo. Pulse el botón principal del manipulador de líquidos peristálticos durante unos 10 s. Una vez más, asegúrese de que ninguna punta esté obstruida inspeccionando visualmente el flujo de líquido de todos ellos.
- Llene la placa con 1 l de diluyente. Coloque una placa de cultivo estéril de 384 pocillos (destino) en el soporte de la placa controladora de líquido peristáltico y retire su tapa.
- Ejecute el programa precalibrado para dispensar 1 l en cada pocal de la placa de 384 pocillos. El tiempo de dosificación es de aproximadamente 8 s. A continuación, reemplace la tapa de la placa de 384 pocillos.
NOTA: Alternativamente, este paso se puede manejar manualmente, en un gabinete de seguridad, utilizando un micropipeta multicanal.
5. Ejecución de una encuesta para controlar los volúmenes dosificados manualmente
NOTA: Para obtener más información, consulte la Figura 4.
- Ejecute el programa de nanodispensador, vaya a la pestaña de diagnóstico, marque la caja de salida de la placa de origen, cargue la placa de origen en el soporte de la placa y marque In para entrar en la placa. Cuando se le solicite, seleccione 384LDV_AQ_B2 para establecer el nanodispensador en el modo de dosificación del búfer acuoso y pulse Ok.
- Seleccione Encuesta en el menú Varios y haga clic en Iniciar. Seleccione los pozos precargados para analizar y haga clic en el botón Ir. Compruebe que los volúmenes medidos coincidan con los esperados y asegúrese de que no se hayan cargado pozos con volúmenes de más de 12 ml, ya que esto evitará las transferencias.
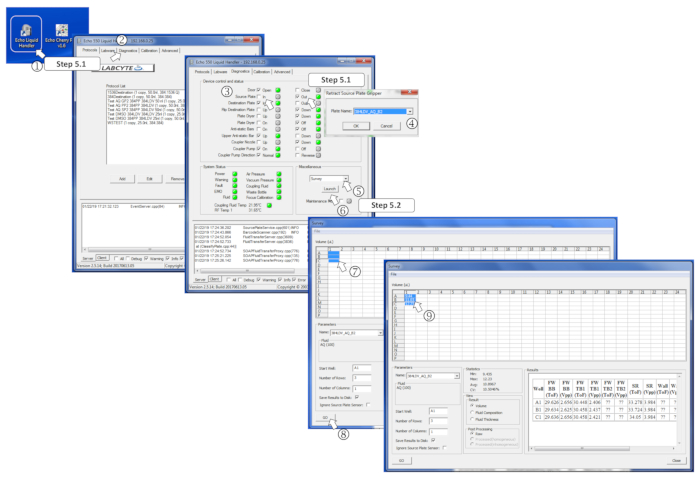
Figura 4 : Definición de los parámetros del software de topografía. (1) Inicie el programa nanodispenser. (2) Abra la pestaña Diagnóstico. (3) Inserte la placa de origen marcando Hacia fuera para la placa de origen y, a continuación, en. (4) Defina el tipo de placa de origen en el menú cuando se le solicite. (5) En el cuadro Varios, seleccione Encuesta en el menú desplegable. 6) Inicie el programa de encuestas haciendo clic en Iniciar. (7) Seleccione los pozos precargados para medir. (8) Iniciar el análisis haciendo clic en Ir. (9) Una vez realizada la encuesta, los volúmenes medidos se escriben en los pozos seleccionados correspondientes. Haga clic aquí para ver una versión más grande de esta figura.
6. Dispensación de ADN impulsada por ADE en la placa de destino
- Ejecute el software de lista de selección, establezca los tipos de placa de origen y destino de 384 pocillos en 384_LDV y Greiner 384PS_781096, respectivamente (Figura5). Establezca el dispositivo en modo de dosificación de búfer acuoso seleccionando 384LDV_AQ_B2 y desmarque "optimizar el rendimiento de transferencia".

Figura 5 : Rendimiento de las dispensaciones basadas en listas de selección. (1) Inicie el software nanodispenser. En la pestaña Protocolo, seleccione (2) el formato de placa de muestra, (3) el tipo de placa de destino y (4) desmarque "optimizar el rendimiento de transferencia". (5) Seleccione la pestaña Lista de selección. (6) Haga clic en Importar y seleccione el archivo *.csv adecuado (DNA-PickList o T.R.-Picklist). (7) Una vez seleccionado, haga clic en Importar. (8) Haga clic en Reproducir y guarde el protocolo. (9) Realice una simulación de dispensación haciendo clic en Simularo (10) Iniciar la dispensación programada haciendo clic en Ejecutar. Haga clic aquí para ver una versión más grande de esta figura.
- Seleccione la pestaña "Lista de selección", haga clic en Importar, seleccione el archivo DNA-Picklist.csv. Haga clic en Reproducir y guarde el protocolo. Haga clic en Simular para realizar una simulación de las dispensaciones programadas para asegurarse de que la lista de selección coincide con el diseño experimental esperado. Una vez completado, haga clic en Cerrar.
- Haga clic en Reproduciry, a continuación, en Ejecutar, para iniciar el programa de dosificación: cuando se le pregunte, inserte la placa de origen solicitada (soluciones de ADN rellenadas manualmente) y la placa de destino (rellenada con diluyente) en el nanodispensador.
NOTA: El tiempo de dosificación es de aproximadamente 5-20 min para una placa completa de 384 pocillos, dependiendo de los volúmenes seleccionados y el número total de dispensaciones en el diseño experimental. - Alternativamente, detenga el protocolo aquí, ya que las placas llenas de diluyente y ADN pueden manejar almacenamiento seco o congelado durante un máximo de 7 días. Para el almacenamiento en seco, deje que las placas se sequen en el banco a temperatura ambiente y luego guárdelas de la misma manera. Descongelar y centrifugar (a 1.500 x g durante 2 min) placas almacenadas congeladas antes de su uso en un paso de transfección (sección 7).
7. Dispensación de reactivos de transfección impulsados por ADE
- En un gabinete de bioseguridad, diluir extemporáneamente el reactivo de transfección lipopolyplex en medio libre de suero a una concentración final de 1x. Vortex y dispensar inmediatamente esta mezcla de reactivos de transfección de acuerdo con la(s) placa(s) fuente(s) predefinida(s) diseñada(s) por la macro y utilizando la aplicación precalibrada de guía de pipeteo de 384 pocillos como se describe en el paso 3.4.
NOTA: No centrifugar la placa de origen una vez que esté cargada con el reactivo, ya que no se observa ningún transfecto después de la centrifugación. - Ejecute el programa de nanodispensadores para realizar una "encuesta" como se describe en la sección 5, con el fin de controlar los volúmenes de todos los pozos TR llenados manualmente de las placas de origen para evitar errores de dosificación debidos a volúmenes superiores a 12 ol.
- Haga clic en Restablecer para borrar la lista de muestras de la lista de selección de ADN en el software de lista de selección y compruebe que los parámetros del dispositivo todavía están configurados en búferes acuosos y en los tipos de placa de origen y destino utilizados, como en el paso 6.1.
- Haga clic en Importar y elija el archivo TR-Picklist.csv. Haga clic en Reproducir y guarde el protocolo si se le solicita, y (esto es opcionalpero, pero se recomienda encarecidamente) realizar una simulación de las dispensaciones programadas de la mezcla de reactivos de transfección para asegurar el diseño adecuado de las dispensaciones haciendo clic en el Botón Simular. Una vez completado, haga clic en Cerrar.
- Haga clic en Reproduciry, a continuación, en el botón Ejecutar para iniciar el programa de dosificación: según lo solicitado, coloque la(s) placa(s) de origen (TR-mezcla rellenada) y la placa de destino (diluyente- y lleno de ADN) en el nanodispensador.
NOTA: El tiempo de dosificación es inferior a 20 minutos para una placa completa de 384 pocillos al dispensar 500 nL de mezcla TR. - Incubar 15-30 min a temperatura ambiente después de añadir el TR al ADN según lo indicado por el protocolo del fabricante.
8. Dispensación de células peristáltica basada en manipulador de líquidos
- Prepare el manipulador de líquidos peristálticos para la dosificación de células. Desinfecte un cabezal de casete de 10 ml rociándolo con Aniospray Surf 29 Desinfectante y absorbiendo el remanente en papel. Monte el cassette en el dispositivo de manipulador de líquidos peristálticos, cambie el ajuste del tipo de cassette a 10 l y asegúrese de que el formato de la placa esté ajustado a 384 pozos.
- Desinfecte el tubo de casete de 10 l como se describió anteriormente en el paso 4.2. Sumergir al organizador del tubo en un recipiente estéril y lavar el tubo con 5 ml de 70% de alcohol, luego con 5 ml de agua destilada, y finalmente, con 5 ml de medio libre de suero, rellenado sucesivamente en el mismo recipiente y hasta que cada tubo esté vacío.
- Prepare la suspensión celular para dispensar. A partir de un plato de cultivo B10 de células HeLa confluente, lave las células 1x con 1 solución salina con búfer de fosfato (PBS) y luego disocia las células con tripina/EDTA durante 5 min a 37 oC.
- Verificar la disociación celular bajo un microscopio y detener la acción de trippsina/EDTA añadiendo 10 ml de medio completo (DMEM complementado con 10% de suero bovino fetal y 100 U/ml de penicilina-estreptomicina; ver la Tabla deMateriales) en el plato de cultivo. Cosecha células en un tubo de 50 ml y cuenta las células bajo el microscopio, usando una célula Malassez o un contador de células automático.
- Preparar al menos 25 ml de suspensión celular HeLa a una concentración de 37.500 células/ml en un medio completo (es decir, 1.500 células/40 l) para una placa completa de 384 pocillos, para asegurar el cebado del tubo y una dosificación de 40 l/pozo.
- Para dispensar las células, llene un nuevo recipiente estéril con la suspensión de la célula preparada y revuelva para evitar la sedimentación que conduce a la inexactitud en la densidad celular de la dispensación. Inserte el organizador del tubo en esta solución y pulse el botón Prime hasta que la suspensión de la celda comience a vaciarse del cabezal dispensador. Asegúrese de que ninguna de las puntas esté obstruida inspeccionando visualmente el flujo de líquido de todos ellos, y asegúrese de que cada tubo esté cargado con suspensión celular.
- Cargue la placa de destino de 384 pocillos llena de ADN y TR en el soporte de la placa controladora de líquido peristáltico y retire su tapa. Ejecute el programa precalibrado para dispensar 40 l de la suspensión celular en la placa completa de 384 pocillos (es decir, 1.500 celdas/pozo). El tiempo de dosificación es de aproximadamente 8 s. Reemplace la tapa de la placa de 384 pocillos.
NOTA: Alternativamente, la suspensión celular de 40 l se puede dispensar manualmente utilizando un micropipeta multicanal.
9. Ensayo biológico personalizado (monitoreo de la eficiencia de la transfección celular)
NOTA: Siguiendo los ajustes experimentales y la intención del experimento, utilice los métodos necesarios para la luminiscencia, la fluorescencia, el cribado de alto contenido y la reacción en cadena cuantitativa de la polimerasa de transcripción inversa (RT-qPCR). En esta sección del protocolo, la eficiencia de la transfección celular se evalúa mediante microscopía de fluorescencia automatizada y análisis de imágenes.
- Incubar la placa a 37oC con 5% de CO2 en una atmósfera saturada de agua y hasta la expresión adecuada de proteínas.
NOTA: Aquí, se utiliza un tiempo de incubación de 48 h para que las células DeLa supervisen la eficiencia de la transfección, utilizando plásmidos que expresan tdTomato y mVenus. - Retire el medio de cultivo 48 h después de la transfección invirtiendo la placa, añada 30 ol/pozo de formalina del 10% utilizando el manipulador de líquido peristáltico (cassette de 10 ol) e incubar durante 15 minutos a temperatura ambiente.
- Retire la formalina invirtiendo la placa; luego, incubar las células durante 15 minutos a temperatura ambiente con 0,1 ng/mL Hoechst diluido en 1 solución PBS.
- Lavar las células 3 veces durante 15 minutos con 80 ml de 1pbS ajustado a pH a 8 para recuperar la señal de alta fluorescencia perdida por el pH de 6,9 del paso de incubación de la solución de formalina.
- Usando un microscopio fluorescente automatizado, adquiera imágenes de dos o tres canales fluorescentes (Hoechst, tdTomato y mVenus) secuencialmente con objetivos 10x y un conjunto de filtros de emisión adecuado (4o,6-diamidino-2-fenilindolo [DAPI], dsRed y fluoresceína isotiocianato [FITC], respectivamente).
- Para evaluar la eficiencia de la transfección, utilice el software de análisis de imágenes para determinar las eficiencias de transfección mediante el análisis de scripts basado en la tinción de núcleos.
Resultados
fPara determinar si la tecnología ADE podría utilizarse para un protocolo automatizado de transfección inversa, monitoreamos la eficiencia de la transfección celular mediante microscopía de fluorescencia, utilizando un plásmido de expresión tdTomato fluorescente rojo. En primer lugar con el objetivo de determinar los mejores parámetros de transfección, diferentes volúmenes de diluyente y cantidades totales de ADN fueron probados en crucigé. El volumen diluyente se utilizó para...
Discusión
El establecimiento y la optimización de un método preciso de transfección de alto rendimiento para una línea celular determinada requieren que los científicos sigan algunos parámetros clave descritos en esta sección. Recomendamos encarecidamente comenzar con los valores recomendados en todo el protocolo, ya que estos ajustes optimizados para las células HeLa también demostraron ser eficientes para las células HEK. Sin embargo, como los mejores parámetros pueden depender de las líneas celulares y los reactivos...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores revelaron un recibo del siguiente apoyo financiero para la investigación, autoría y/o publicación de este artículo: Inserm, Lille University, Lille Pasteur Institute, Conseil Régional du Nord, y PRIM-HCV1 y 2 (Péle de Recherche Interdisciplinaire sur le Médicament), Agence Nationale de la Recherche (ANR-10-EQPX-04-01), el Feder (12001407 (D-AL) Equipex Imaginex BioMed) y la Comunidad Europea (ERC-STG INTRACELLTB no 260901). Los autores desean dar las gracias al Dr. S. Moureu, al Dr. B. Villemagne, al Dr. R. Ferru-Clément y al Dr. H. Groult por su revisión crítica y correcciones del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| 384LDV Microplate | Labcyte | LP-0200 | |
| 384-well Microplate μClear Black | Greiner | 781906 | |
| Ampicilin | Sigma | A9393-5G | Selection antibiotic for bacteria transformed with ampicilin expressing vector |
| Android Tablet | Samsung | Galaxy Note 8 | used to guide the user while the source plate manual dispense |
| Aniospray Surf 29 | Anios | 2421073 | disinfectant to clean the MicroFlo head |
| Columbus software | Perkin Elmer | image analysis software | |
| Dulbecco's Modified Eagle Medium (DMEM), high glucose, GlutaMAX Supplement, pyruvate | Thermo Fisher Scientific | 10566032 | cell culture medium |
| Echo Cherry Pick 1.5.3 software | Labcyte | Software enabling ADE-based dispenses by the Echo550 device from a *.csv file; nanodispenser software | |
| Echo550 | Labcyte | ADE-based dispenser | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | to add in cell culture medium |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | to fix cell |
| HeLa cells | ATCC | HeLa (ATCC® CCL-2™) | |
| Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | 10 mg/mL Solution in Water |
| INCell Analyzer 6000 | GE Healthcare | 29043323 | automated laser-based confocal imaging platform |
| LB medium | Thermoischer Scientific LB Broth Base (Lennox L Broth Base)®, powder | 12780052 | culture medium for bacteria growth |
| Lysis Buffer (A2) | Macherey-Nagel | 740912.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| MicroFlo 10µL cassette | Biotek Instruments Inc | 7170013 | to use with the Microflo Dispenser |
| MicroFlo 1μL cassette | Biotek Instruments Inc | 7170012 | to use with the Microflo Dispenser |
| MicroFlo Dispenser | Biotek Instruments Inc | 7171000 | peristaltic pump-based liquid handler device |
| Microvolume spectrophotometer | Denovix | DS-11 Spectrophotometer | Measure the DNA concentration of samples |
| mVenus plasmid | mVenus cDNA was cloned by enzymatic restriction digestion and ligation in Age1/BsrG1 sites of the tdTomato-N1 plasmid | Vector type: Mammalian Expression, Fusion Protein: mVenus | |
| Neutralization Buffer (A3) | Macherey-Nagel | 740913.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| NucleoSpin Plasmid kit | Macherey-Nagel | 740588.50 | used to prepare plasmid from bacterial culture |
| Optimal-Modified Eagle Medium (Opti-MEM) Medium | Thermo Fisher Scientific | 31985070 | |
| optional Wash bufferWash Buffer (A4) | Macherey-Nagel | 740914.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| orbital shaker | incubated large capacity shaker | 444-7084 | Used to grow bacteria under gentle agitation and 37°C |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | 10,000 U/mL |
| Phosphate-Buffered Saline | Thermo Fisher Scientific | 10010001 | |
| Plasmid mini-columns | Macherey-Nagel | 740499.250 | Silica membrane mini-column to prepare plasmid from bacterial culture |
| Resuspension Buffer (A1) | Macherey-Nagel | 740911.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| RNAse A | Macherey-Nagel | 740505 | Enzyme from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| tdTomato-N1 plasmid | Addgene | Plasmid #54642 | Vector type: Mammalian Expression, Fusion Protein: tdTomato |
| TransIT-X2 Dynamic Delivery System | Mirus Bio | MIR 6000 | |
| Wash Buffer (AW) | Macherey-Nagel | 740916.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| 3D printer | Creality | CR10S | used to print the plate adapter |
| Blender Software | https://www.blender.org/ Free software under GNU General Public License (GPL). | version 2.79b | used to design the plate adapter |
Referencias
- Colin, B., Deprez, B., Couturier, C. High-Throughput DNA Plasmid Transfection Using Acoustic Droplet Ejection Technology. SLAS Discovery: Advancing Life Sciences R & D. , 2472555218803064 (2018).
- Mirus Bio. . Optimising Transfection Performance. , (2019).
- Thermo Fisher Scientific. . Factors Influencing Transfection Efficiency | Thermo Fisher Scientific - FR. , (2019).
- Boussif, O., et al. A versatile vector for gene and oligonucleotide transfer into cells in culture and in vivo: polyethylenimine. Proceedings of the National Academy of Sciences of the United States of America. 92 (16), 7297-7301 (1995).
- Figueroa, E., et al. A mechanistic investigation exploring the differential transfection efficiencies between the easy-to-transfect SK-BR3 and difficult-to-transfect CT26 cell lines. Journal of Nanobiotechnology. 15 (1), 36 (2017).
- Kirchenbuechler, I., Kirchenbuechler, D., Elbaum, M. Correlation between cationic lipid-based transfection and cell division. Experimental Cell Research. 345 (1), 1-5 (2016).
- Zhang, Z., Qiu, S., Zhang, X., Chen, W. Optimized DNA electroporation for primary human T cell engineering. BMC Biotechnology. 18 (1), 4 (2018).
- Cao, D., et al. Transfection activity and the mechanism of pDNA-complexes based on the hybrid of low-generation PAMAM and branched PEI-1.8k. Molecular bioSystems. 9 (12), 3175-3186 (2013).
- Bos, A. B., et al. Development of a semi-automated high throughput transient transfection system. Journal of Biotechnology. 180, 10-16 (2014).
- Colosimo, A., et al. Transfer and expression of foreign genes in mammalian cells. BioTechniques. 29 (2), 314-318 (2000).
- Villa-Diaz, L. G., Garcia-Perez, J. L., Krebsbach, P. H. Enhanced transfection efficiency of human embryonic stem cells by the incorporation of DNA liposomes in extracellular matrix. Stem Cells and Development. 19 (12), 1949-1957 (2010).
- Sabatini, D. M. . Reverse transfection method. , WO2001020015A1 (2001).
- Raymond, C., et al. A simplified polyethylenimine-mediated transfection process for large-scale and high-throughput applications. Methods (San Diego, CA). 55 (1), 44-51 (2011).
- Junquera, E., Aicart, E. Recent progress in gene therapy to deliver nucleic acids with multivalent cationic vectors. Advances in Colloid and Interface Science. 233, 161-175 (2016).
- Woodruff, K., Maerkl, S. J. A High-Throughput Microfluidic Platform for Mammalian Cell Transfection and Culturing. Scientific Reports. 6, 23937 (2016).
- Vasileiou, T., Foresti, D., Bayram, A., Poulikakos, D., Ferrari, A. Toward Contactless Biology: Acoustophoretic DNA Transfection. Scientific Reports. 6, 20023 (2016).
- Hadimioglu, B., Stearns, R., Ellson, R. Moving Liquids with Sound: The Physics of Acoustic Droplet Ejection for Robust Laboratory Automation in Life Sciences. Journal of Laboratory Automation. 21 (1), 4-18 (2016).
- Grant, R. J., et al. Achieving accurate compound concentration in cell-based screening: validation of acoustic droplet ejection technology. Journal of Biomolecular Screening. 14 (5), 452-459 (2009).
- Sackmann, E. K., et al. Technologies That Enable Accurate and Precise Nano- to Milliliter-Scale Liquid Dispensing of Aqueous Reagents Using Acoustic Droplet Ejection. Journal of Laboratory Automation. 21 (1), 166-177 (2016).
- Day, R. N., Davidson, M. W. The fluorescent protein palette: tools for cellular imaging. Chemical Society Reviews. 38 (10), 2887-2921 (2009).
- Zielinski, D., Gordon, A., Zaks, B. L., Erlich, Y. iPipet: sample handling using a tablet. Nature Methods. 11 (8), 784-785 (2014).
- Brunner, S., et al. Cell cycle dependence of gene transfer by lipoplex, polyplex and recombinant adenovirus. Gene Therapy. 7 (5), 401-407 (2000).
- Nii, T., et al. Single-Cell-State Culture of Human Pluripotent Stem Cells Increases Transfection Efficiency. BioResearch Open Access. 5 (1), 127-136 (2016).
- Noonan, D. J., Henry, K., Twaroski, M. L. A High-Throughput Mammalian Cell-Based Transient Transfection Assay. Signal Transduction Protocols. 284, 051-066 (2004).
- . . Transfection | TransIT Transfection Reagents | Mirus Bio. , (2015).
- American Type Culture Collection. . General protocol for transfection of stem cells, primary cells, and continuous cell lines with ATCC TransfeX Transfection Reagent. , (2017).
- American Type Culture Collection. . Transfection Reagents for Nucleic Acid Transfer into ATCC Cells. , (2017).
- de Los Milagros Bassani Molinas, M., Beer, C., Hesse, F., Wirth, M., Wagner, R. Optimizing the transient transfection process of HEK-293 suspension cells for protein production by nucleotide ratio monitoring. Cytotechnology. 66 (3), 493-514 (2014).
- Promega. . FuGENE® 6 Transfection Reagent. , (2019).
- Olden, B. R., Cheng, Y., Yu, J. L., Pun, S. H. Cationic polymers for non-viral gene delivery to human T cells. Journal of Controlled Release: Official Journal of the Controlled Release Society. 282, 140-147 (2018).
- Park, E., Cho, H. B., Takimoto, K. Effective gene delivery into adipose-derived stem cells: transfection of cells in suspension with the use of a nuclear localization signal peptide-conjugated polyethylenimine. Cytotherapy. 17 (5), 536-542 (2015).
- Wood, R. W., Loomis, A. L. The physical and biological effects of high-frequency sound-waves of great intensity. The London, Edinburgh, and Dublin Philosophical Magazine and Journal of Science. 4 (22), 417-436 (1927).
- Mamat, U., et al. Eliminating Endotoxin at the Source - A Novel Competent Cell Line with Modified Lipopolysaccharide for Low-Endotoxin Plasmid Production. , (2014).
- Ivanova, N. V., Kuzmina, M. L. Protocols for dry DNA storage and shipment at room temperature. Molecular Ecology Resources. 13 (5), 890-898 (2013).
- Lesnick, J., Lejeune-Dodge, A., Ruppert, N., Jarman, C. . High-Precision Cell Dispensing with the Labcyte Echo® Liquid Handler. , (2017).
- Yang, X., et al. A public genome-scale lentiviral expression library of human ORFs. Nature Methods. 8 (8), 659-661 (2011).
- Peng, J., Zhou, Y., Zhu, S., Wei, W. High-throughput screens in mammalian cells using the CRISPR-Cas9 system. The FEBS journal. 282 (11), 2089-2096 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados