Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Examen de la dinámica de la adhesión celular y la propagación de células epiteliales en la fibronectina durante el estrés oxidativo
En este artículo
Resumen
Este método es útil para cuantificar la dinámica temprana de la adhesión celular y la propagación de células dependientes del anclaje en la fibronectina. Además, este ensayo se puede utilizar para investigar los efectos de la homeostasis redox alterada en la propagación celular y/ o vías de señalización intracelular relacionadas con la adhesión celular.
Resumen
La adhesión y propagación de las células a la matriz extracelular (ECM) son procesos celulares esenciales durante el desarrollo del organismo y para la homeostasis de los tejidos adultos. Curiosamente, el estrés oxidativo puede alterar estos procesos, contribuyendo así a la fisiopatología de enfermedades como el cáncer metastásico. Por lo tanto, comprender los mecanismos de cómo las células se unen y se propagan en el ECM durante las perturbaciones en el estado de redox puede proporcionar información sobre los estados normales y de la enfermedad. A continuación se describe un protocolo escalonado que utiliza un ensayo basado en inmunofluorescencia para cuantificar específicamente la adhesión celular y la propagación de células fibroblastas inmortalizadas en fibronectina (FN) in vitro. Brevemente, las células dependientes del anclaje se mantienen en suspensión y se exponen al inhibidor de la quinasa ATM Ku55933 para inducir estrés oxidativo. Las celdas se chapan en la superficie recubierta de FN y se les permite fijar se adhieren durante períodos de tiempo predeterminados. Las células que permanecen unidas se fijan y se etiquetan con marcadores de adhesión a base de fluorescencia (por ejemplo, paxillina) y propagación (por ejemplo, F-actina). La adquisición y el análisis de datos se realizan utilizando equipos de laboratorio comúnmente disponibles, incluido un microscopio de epifluorescencia y software de Fiji disponible libremente. Este procedimiento es muy versátil y se puede modificar para una variedad de líneas celulares, proteínas ECM, o inhibidores con el fin de examinar una amplia gama de preguntas biológicas.
Introducción
Las adherencias de matriz celular (es decir, adherencias focales) son complejos proteicos multimoleculares grandes y dinámicos que median la adhesión y la propagación celular. Estos procesos son críticos para el desarrollo de tejidos, el mantenimiento y la función fisiológica. Las adherencias focales se componen de receptores ligados a la membrana, como integrinas, así como proteínas de andamios que vinculan la actina citoesquelética con la matriz extracelular (ECM)1. Estos complejos son capaces de responder a las señales fisioquímicas presentes en el entorno extracelular a través de la activación de varias vías de transducción de señalización. Como tal, las adherencias focales sirven como centros de señalización para propagar señales mecánicas extracelulares en una serie de procesos celulares, incluyendo migración dirigida, regulación del ciclo celular, diferenciación y supervivencia1,2. Un grupo de moléculas de señalización que regulan e interactúan con adherencias focales incluye miembros de la familia Rho de pequeñas GTPases. Rho GTPases son proteínas clave que regulan la migración celular y la dinámica de adhesión a través de su activación espaciotemporal específica3. No es sorprendente, la desregulación de la función de la proteína Rho se ha implicado en un número de patologías humanas como metástasis, angiogénesis, y otros. De particular interés, el estado celular de redox desempeña un papel predominante en la modulación de la migración y adhesión celular. Se ha demostrado que las alteraciones en la homeostasis redox, como los aumentos de las especies reactivas de oxígeno (ROS), regulan la actividad de la proteína Rho, así como la adhesión, en varios tipos de células y enfermedades humanas4,5,6 ,7,8. Por ejemplo, las personas que sufren del trastorno neurológico ataxia-telangiectasia (A-T), que es causada por una mutación en la reparación del daño del ADN serina/threonina quinasa A-T-mutada (ATM), tienen un mayor riesgo de cáncer metastásico9, 10. La pérdida de actividad de la quinasa ATM en estos pacientes y líneas celulares, ya sea a través de mutación genética o inhibición química, resulta en altos niveles de estrés oxidativo debido a la disfunción de la vía del fosfato de pentosa7,11, 12. Además, estudios recientes del laboratorio han puesto de relieve un papel fisiopatológico de ROS en A-T alterando la dinámica citoesquelética (es decir, la adhesión y la propagación) como resultado directo de la activación de la familia Rho GTPases in vitro5. En últimainstancia, estas alteraciones en la dinámica citoesquelética causada por la activación de la familia Rho pueden conducir al mayor riesgo de cáncer metastásico observado en pacientes con A-T5,13. Por lo tanto, comprender la interacción entre las interacciones entre la matriz celular durante el estrés oxidativo puede proporcionar información sobre la regulación de la adhesión y la propagación. Estos estudios también pueden sentar las bases para más investigaciones sobre un posible papel para la familia Rho GTPases en estos procesos de señalización.
Descrito aquí es un protocolo para estudiar la dinámica celular temprana del ensamblaje de adhesión y la propagación durante el estrés oxidativo causado por la inhibición de la actividad de la quinasa ATM. Este ensayo se basa en el mecanismo bien caracterizado de adhesión de las células dependientes del anclaje a la fibronectina proteica ECM (FN). Cuando las células mantenidas en suspensión se chapan en FN, varias Rho GTPases coordinan el control de la remodelación citoesquelética actina3,14. Los cambios morfológicos se observan a medida que las células cambian de redonda y circular en apariencia a aplanadas y expandidas. Concomitante con estas observaciones es el desarrollo de numerosas adherencias de matriz con el ECM. Estos cambios se atribuyen a la activación bifásica de RhoA con Rac1 durante la primera hora a medida que las células se adhieren y se extienden 15,16.
Se han utilizado una variedad de métodos para examinar la morfología y la dinámica de adhesión, así como la propagación celular. Sin embargo, estos métodos se basan en sofisticados sistemas de fluorescencia de reflexión interna total (TIRF) a largo plazo, imágenes en vivo o sistemas de microscopía confocal. Por lo tanto, los usuarios deben tener acceso a equipos y software especializados. Además, el tiempo de configuración requerido por estos sistemas de bioimágenes hace que la captura de eventos de adhesión temprana sea un reto, especialmente cuando se prueban múltiples inhibidores o condiciones de tratamiento simultáneamente.
Los métodos detallados, aquí, proporcionan una forma directa, económica y cuantitativa de evaluar los parámetros que rigen el ensamblaje de adhesión y la propagación in vitro. El protocolo se realiza utilizando equipos de laboratorio comúnmente disponibles, como un microscopio de epifluorescencia y una cámara CCD. Este ensayo implica la aplicación de células dependientes del anclaje a una superficie recubierta de FN después de un período de estrés oxidativo causado por la inhibición química de la actividad de la quinasa ATM, que se ha demostrado previamente5. Después del revestimiento, las celdas se pueden unir y adherir durante períodos de tiempo especificados. Las células no conectadas se lavan, mientras que las células adheridas se fijan y etiquetan con anticuerpos basados en fluorescencia a marcadores de adhesión (por ejemplo, paxillina) y que se propagan (por ejemplo, F-actina)2,5. Estas proteínas se visualizan y registran mediante un microscopio de epifluorescencia. El análisis de datos posterior se realiza utilizando el software de Fiji disponible libremente. Además, este método se puede adaptar para examinar la dinámica de adhesión en una amplia gama de condiciones, incluyendo diferentes proteínas ECM, tratamiento con diversos oxidantes /condiciones de cultivo celular o una variedad de líneas celulares dependientes del anclaje para abordar una amplia gama de preguntas biológicas.
Protocolo
1. Preparativos
NOTA: El protocolo descrito a continuación se ha optimizado para su uso con células REF52 y fibroblastos humanos ATM+/+ o ATM-/-. Otros tipos de celda pueden requerir una optimización adicional como se describe en las notas y las secciones de solución de problemas a continuación.
- Haga 500 ml de medio de cultivo celular completo para células REF52. A 500 ml de glucosa alta que contiene el medio modificado de Dulbecco Eagle (DMEM) añadir 10% FBS, 2 mM L-glutamina, y 100 unidades/ml penicilina-estreptomicina.
- Preparar una solución de 25 g/ml de fibronectina (FN) añadiendo 300 ml de solución de FN de 1 mg/ml a 12 ml de solución salina tamponada de fosfato estéril 1x (PBS), pH 7,4. Mezcla bien.
- Preparar una solución de albúmina sérica bovina (dlBSA) deslipidada al 0,5% (p/v) (es decir, sin ácidos grasos) en el medio de cultivo celular DMEM libre de suero. Añadir 0,5 g de dlBSA a 100 ml de medio DMEM libre de suero. Mezcle bien la solución, pero no vórtice. Filtrar estérilmente la solución en un nuevo recipiente estéril, utilizando un filtro de jeringa de 0,22 m antes de su uso. Conservar a 4oC.
- Hacer una solución de paraformaldehído del 3,7% disolviendo 3,7 g de paraformaldehído en 100 ml de 1x PBS. Use calor suave y revolviendo para obtener la solución del paraformaldehído.
NOTA: La solución de paraformaldehído es sensible a la luz y debe protegerse de la luz. Es bueno hasta una semana cuando se almacena a 4 oC.
ADVERTENCIA: Paraformaldehído es tóxico, inflamable, corrosivo y un peligro para la salud. Revise la hoja de datos de seguridad del material para ver si hay paraformaldehído antes de su uso. Utilice el equipo de protección personal adecuado cuando manipule, incluyendo protector ocular, protector facial, respirador de partículas de cara completa, guantes y capa de laboratorio. - Preparar la solución de permeabilización que contenga un tensioactivo no iónico al 0,2% en 1x PBS (v/v). Para 100 ml, agregue lentamente 0,2 ml de Triton X-100 a 100 ml de 1x PBS, mientras revuelve.
- Realizar el tampón de bloqueo de inmunofluorescencia que contenga 2,5% de BSA, 5% de suero de cabra y 0,05% de surfactante no iónico (p/v/v) disuelto en 1 solución de PBS. Para 100 ml, añadir 5 ml de suero de cabra, 2,5 g de BSA y 0,05 ml de Triton X-100 en 95 ml 1x PBS, mientras se agita.
- Cultivar células REF52 en un medio de cultivo completo DMEM en una placa tratada con cultivo celular de 10 cm2 (o cualquier otro tamaño de recipiente) en una incubadora de cultivo celular a 37 oC y 5% de CO2.
2. Recubrimiento de placas de cultivo celular con la matriz extracelular de fibronectina
NOTA: Realice esta sección utilizando técnica aséptica y reactivos estériles en una campana de flujo laminar certificada por BSL-2. Refiera al cuadro 1A para una visión general de los pasos clave antes de comenzar.
- Usando una placa de 24 pocillos certificada por un cultivo de tejido, coloque un cubreobjetos de vidrio (12-Cir-1) en cada pocal. Etiquete la placa de acuerdo con la Figura 1B.
- Pipetear 500 l de la solución de 25 g/ml FN a cada pocto de una placa de 24 pocillos.
- Pipetear la solución sobre cada cubreobjetos unas cuantas veces para asegurar un recubrimiento uniforme y una inmersión completa. Vuelva a colocar la tapa en la placa.
- Incubar la placa en una incubadora de cultivo celular a 37oC y 5%CO2 durante 1 h.
NOTA: Alternativamente, incubar durante la noche a 4oC. - Después de 1 h, retire la placa de la incubadora y aspire la solución FN de los pozos.
- Lavar los pozos tres veces con 500 ml de 1 x PBS. Aspirar el lavado final de 1x PBS.
- Bloquee los pozos con 500 ml de solución de dlBSA al 0,5% durante un mínimo de 15 min a 37 oC y 5% deCO2.
- Aspirar la solución dlBSA antes de las células de chapado en el paso 3 a continuación.
NOTA: Si almacena las placas, añada 500 ml de 1 PBS a cada cubreobjetos después de la aspiración de la solución dlBSA. Las placas se pueden mantener a 4 oC durante un máximo de una semana.
3. Preparación de células dependientes del anclaje para el ensayo de adhesión
NOTA: Realice esta sección utilizando técnica aséptica y reactivos estériles en una campana de flujo laminar certificada por BSL-2.
- Al menos 30 minutos antes del revestimiento celular, precaliente las siguientes soluciones: Medio completo de DMEM, solución dlBSA, 1x PBS, solución de trippsina-EDTA al 0,5% y suero neutralizante de trippsina (TNS) en un baño de agua de 37oC.
- Comenzando con una monocapa confluente de células REF52 en un plato de 10 cm2, lave las células dos veces con 6 ml de PBS caliente 1x. El suero muero de hambre las células durante al menos 1 h (dependiendo del tipo de célula) en 6 ml de solución de dlBSA caliente a 37 oC y 5% deCO2.
- Lave las células con 6 ml de PBS calentado 1x, aspire pbS y agregue 1,5 ml de solución tibia 0,5% de tripina-EDTA.
- Coloque las células en una incubadora de cultivo celular a 37 oC y 5% deCO2 durante 2 min.
- Observe las células bajo un microscopio de luz para asegurarse de que el desprendimiento está completo. Si las células siguen siendo adherentes después de tocar la placa en la parte superior del banco, vuelva a la incubadora de 37 oC durante 2 minutos adicionales.
- Pipetear 1,5 ml de solución neutralizante de trippsina tibia (TNS) al plato para detener la trippsinización y recoger las células separadas. Pipetear la solución hacia arriba y hacia abajo sobre la parte inferior de la placa numerosas veces para eliminar todas las células adherentes restantes. Si las células parecen grumosas, tritura relinve aún más la suspensión celular pipeteando suavemente hacia arriba y hacia abajo sobre la parte posterior del plato.
- Cuente las células usando la exclusión azul de trypan y un hemocitómetro bajo un microscopio de luz. Alternativamente, utilice un contador de celdas automatizado.
- Retire una cantidad apropiada de células para crear una suspensión de células de 1.0 - 3.0 x 104 células/ml en 5 ml de dlBSA en un tubo cónico de 15 ml.
- Células centrífugas a 300 x g durante 5 min utilizando un rotor de ángulo fijo en una centrífuga de mesa.
- Aspirar el sobrenadante del pellet celular y resuspender las células en un total de 7 ml de solución de dlBSA caliente. No permita que las células sean demasiado confluentes sobre el revestimiento, pero se distribuyan uniformemente con pocas células que se tocan entre sí.
- Divida uniformemente la suspensión celular en dos tubos cónicos de 15 ml, uno para el solo control del vehículo (DMSO) y otro para Ku55933 (inhibidor de la quinasa ATM, oxidante)5. Asegúrese de que cada tubo contenga 3,5 ml de la suspensión celular.
- Usando un rotador de tubo, gire los tubos a 37 oC durante 90-120 min en una incubadora de cultivo celular.
- 30 min antes del enchapado, añadir una concentración final de 10 M Ku55933 y DMSO (1:1,000) a cada tubo respectivo. Vuelva a colocar la suspensión de la celda en el rotador durante el tiempo restante.
- Inmediatamente antes de enchapar las células, recupere la placa de 24 pocillos de la incubadora y aspirar la solución dlBSA.
- Después de rotar la suspensión celular durante 90-120 min, retire 500 ml de suspensión celular de cada grupo de tratamiento y agregue a un cubreobjetos recubiertos de FN en la placa de 24 pocillos del paso 2 como se ilustra(Figura 1B). Devolver la placa a la incubadora de cultivo celular de 37 oC y 5% de CO2 y la suspensión celular a la rotación.
- Después de enchapar la suspensión de la celda en el FN cubierto-covers-coverslips, deje que las células se adhieran durante el período de tiempo deseado (por ejemplo, 10 min, 15 min, 20 min, 30 min) y luego proceda inmediatamente al paso 4.
4. Fijación celular y tinción de anticuerpos para inmunofluorescencia
NOTA: Los siguientes pasos se realizan en condiciones no estériles y a temperatura ambiente a menos que se indique lo contrario.
- Después de que haya pasado el tiempo deseado para la adhesión, aspirar la solución celular de cada cubreobjetos en la placa.
- Usando los lados del pozo, dispensar suavemente 500 l de solución de paraformaldehído 3.7% en cada cubrepito y esperar 10-15 min.
- Retire la solución paraformaldehído y lave cada cubreobjetos con 500 ml de 1 PBS durante un total de dos veces.
NOTA: Deseche los residuos de paraformaldehído de manera responsable, de acuerdo con el plan de salud y seguridad ambiental de la institución. - Aspirar el PBS, y permeabilizar las células en cada cubreportada con 500 éL de 0.2% Triton X-100 en 1x PBS (v/v) durante 10-15 min a temperatura ambiente.
- Lave cada cubreobjetos con 500 ml de 1 pbS tres veces.
- Bloquear las células en cada cubreobjetos con 500 ml de tampón de bloqueo de inmunofluorescencia que contiene 5% de suero de cabra, 2,5% de BSA y 0,05% de Tritón X-100 disuelto en una solución de 1 x PBS durante 30-60 minutos.
- Diluir el anticuerpo anti-paxillina primario (1:250) en el tampón de bloqueo. Mezclar bien y añadir 200 sL de la solución de anticuerpos a cada cubreobjetos. Incubar a temperatura ambiente durante al menos 1 h.
NOTA: Alternativamente, la solución primaria de anticuerpos se puede incubar durante la noche a 4 oC. Existen muchos marcadores de adhesión focal comunes que podrían utilizarse para tinserar complejos de adhesión y posterior análisis de FA. Estos incluyen anticuerpos contra las siguientes proteínas: subunidades de integrina (-1, 5 o V), talino ovinculina 2. - Aspirar la solución de anticuerpos y lavar cada cubreobjetos con 500 ml de 1 PBS tres veces durante 10 minutos cada uno. Proteja las muestras de la luz desde este punto hacia adelante.
- Diluir la sonda de faloiderina F-actina conjugada con el tinte rojo fluorescente Alexa 594 (1:1000) y el anticuerpo secundario fluorescente 488 fluorescente (1:400) en la misma solución tampón de bloqueo. Mezclar bien y añadir 200 sL de la solución de anticuerpos a cada cubreobjetos durante 30 min.
NOTA: También se pueden utilizar anticuerpos secundarios conjugados fluorescentes de otras especies. Sin embargo, el uso de anticuerpos de otras especies requerirá la modificación del suero tampón de bloqueo. - Aspirar la solución de anticuerpos y lavar cada cubreobjetos con 500 ml de 1 PBS tres veces durante 10 minutos cada uno.
- Aspirar el 1x PBS y enjuagar una vez con 500 s de dIH2O.
- Monte los cubreobjetos en las guías del microscopio utilizando un medio de montaje anti-fade que contenga DAPI.
- Deje que los portaobjetos del microscopio se ajusten durante la noche en la oscuridad a temperatura ambiente.
- Almacene las diapositivas del microscopio en la oscuridad a 4 oC para el almacenamiento a largo plazo y hasta la toma de imágenes.
NOTA: Imagen utilizando técnicas de inmunofluorescencia estándar. Se recomienda utilizar una lente objetivo de inmersión en aceite 60x de alta potencia para garantizar una resolución suficiente para observar las adherencias focales y los volantes periféricos en los bordes de las células. Adquirir imágenes de 20-30 células en múltiples campos de visión para cada capa bajo cada condición de tratamiento y tiempo. A partir de réplicas combinadas, esto debe producir al menos 60 células para realizar el análisis estadístico. Guardar y exportar imágenes de fluorescencia como un archivo . TIFF con una resolución mínima de 300 ppp.
5. Cuantificación de fibras de tensión, circularidad celular y formación de adhesión focal
NOTA: Los siguientes análisis de imágenes se realizan utilizando la última versión del paquete de procesamiento de imágenes de código abierto Fiji Is Just Image J (Fiji), que se puede descargar de forma gratuita al (http://fiji.sc/).
- Procesamiento general de imágenes
NOTA: Todas las imágenes deberán estar preparadas para análisis computacionales realizando los pasos 5.1.1-5.1.5 a continuación(Figura 2). Después, se pueden seleccionar cualquiera o todos los procedimientos de cuantificación posteriores.- Abra el archivo . Imagen de fluorescencia TIFF con Fiji. Asegúrese de que las imágenes sean de 8 bits y escala de grises.
- Seleccione Imagen-Ajustar-Ventana/nivel y seleccione Automático (Figura 2A).
- Seleccione Procesar-Restar fondo para restar la fluorescencia de fondo. Marque Paraboloides deslizantes y seleccione la opción de un radio de bola rodante de 50 píxeles(figura 2B).
NOTA: Para verificar el tamaño adecuado para el radio de bola rodante, seleccione la herramienta Línea y dibuje un radio en la adhesión más grande de la imagen. Seleccione Medir para comprobar la longitud de la línea dibujada. Si el valor del radio es demasiado grande, las entidades que incluyen adherencias se perderán en la imagen. Si el radio es demasiado pequeño, dará lugar a artefactos en la imagen procesada debido al ruido de fondo. - Seleccione Imagen-Ajustar- Brillo/Contraste para comprobar la intensidad de la adhesión sobre el fondo. Ajuste si es necesario.
NOTA: Para optimizar el brillo/contraste y evitar saturar la señal, utilice la herramienta de búsqueda de la imagen para examinar su histograma y ajustar el brillo/contraste. - Seleccione los siguientes parámetros en Analizar-Establecer medidas:área, valor medio de gris, descriptores de forma y densidad integrada.
- Análisis de formación de fibra de estrés
NOTA: Las fibras de tensión se pueden cuantificar de varias maneras dependiendo del fenotipo.- Cuente el número de células con fibras de tensión como un porcentaje sobre el número total de células. Este análisis es mejor si hay diferencias visuales en el número de fibras de tensión formadas en diferentes condiciones experimentales.
- Cuente el número de fibras de tensión que transversalmente la célula. Este análisis permite la comparación del número de fibras de tensión formadas por célula.
- Mida la intensidad total de fluorescencia dada por la tinción de faloide (p. ej., F-actina) por célula17,18. Este método resaltará aumentos/disminuciones drásticas en la intensidad de la fluorescencia debido a la tinción de F-actina.
- Establezca los parámetros de medición en el paso 5.1.5 anterior.
- Seleccione la herramienta a mano alzada en la barra de herramientas de Fiji y rastree manualmente las celdas de interés. Seleccione Analizar-Medida. Aparecerá una nueva ventana que muestra los parámetros de medición seleccionados.
- Seleccione la herramienta a mano alzada en la barra de herramientas de Fiji y trace manualmente un espacio vacío sin celdas presentes. Seleccione Analizar-Medida. Esta medida servirá como fluorescencia de fondo.
- Utilice la ecuación siguiente para determinar el total de fluorescencia de F-actina por célula:
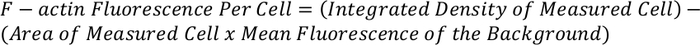
NOTA: La medición resultante se puede normalizar y comparar con otras células para dar fluorescencia de F-actina por célula.
- Análisis de circularidad celular
NOTA: También se puede registrar información sobre el área de la celda (un indicador de la difusión de la célula a lo largo del tiempo), así como la circularidad. Esta medición se da como una relación entre 0 y 1 como una forma de cuantificar las células que son alargadas a redondas, respectivamente.- Seleccione la herramienta a mano alzada en la barra de herramientas de Fiji y trace una celda individual. Seleccione Imagen-Medida y registre el área de celda y las medidas perimetrales para cada celda. Repita este procedimiento para cada celda.
NOTA: En la función Establecer medidas, la circularidad se proporciona como la medida De los descriptores de forma (paso 5.1.5). - Cuente manualmente ruffling o protuberancias enriquecidas con actina por celda como se muestra en la Figura 3 y la Figura 4.
- Seleccione la herramienta a mano alzada en la barra de herramientas de Fiji y trace una celda individual. Seleccione Imagen-Medida y registre el área de celda y las medidas perimetrales para cada celda. Repita este procedimiento para cada celda.
- Análisis de adhesión focal
NOTA: Antes de realizar un análisis de adhesión focal, instale el plugin Mexican Hat Filter en la última versión de Fiji. El siguiente protocolo ha sido modificado a partir de estudios anteriores19,20,21.- Seleccione Procesar-Mejorar contraste local (Clahe) utilizando un tamaño de bloque de 19, bins de histograma 256 y una pendiente máxima de 6, sin máscara y no rápida. (Figura 2C)
- Seleccione Process-Filters- Gaussian Blur with a Sigma (Radius) of 2.0 para filtrar la imagen(Figura 2D).
- Seleccione Plugins-Filtro de sombrero mexicano (Mhf) con un radio de 2.0(Figura 2E).
- Ejecute Umbral y seleccione Fondo oscuro y Sobre/Bajo usando Huang o Isodata como método de umbral. Seleccione Umbral automático.
NOTA: Este paso garantiza que se resalten las adherencias, pero también distintas entre sí. - Seleccione Analizar-Analizar partículas con los siguientes parámetros seleccionados: tamaño 20, píxeles infinitos y circularidad0,00-0,99. Compruebe los contornos para garantizar la detección y separación adecuadas de las adherencias focales.
NOTA: Estos resultados producen la descripción del número, el área y la forma de las adherencias focales individuales.
Resultados
Un esquema general de la configuración experimental
La Figura 1 representa el esquema general para el protocolo de adhesión y propagación celular que comienza con la inanición sérica de células REF52 y termina con el análisis computacional de las imágenes de fluorescencia adquiridas. Los pasos clave del protocolo se ilustran en la línea de tiempo. Cabe destacar que el paso 2 del protocolo describe la preparación de los cubreobjetos recubiertos con FN, que ...
Discusión
El protocolo descrito aquí es una forma versátil y económica de examinar rápidamente una serie de tipos de células dependientes del anclaje para la remodelación dinámica del citoesqueleto durante la propagación celular. En particular, este método examina cuantitativamente la fibra de tensión y la formación de adherencia focal durante el estrés oxidativo cuando las células se adhieren a FN(Figura 1A). Además, estos fenotipos celulares pueden sugerir un papel regulador para los m...
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
Los autores agradecen a los Doctores Scott R. Hutton y Meghan S. Blackledge por la revisión crítica del manuscrito. Este trabajo fue financiado por los Programas de Investigación y Patrocinados de la Universidad de High Point (MCS) y el Programa de Biotecnología de la Universidad Estatal de Carolina del Norte (MCS).
Materiales
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA (1x) | Gibco by Life Technologies | 25300-054 | cell dissociation |
| 10 cm2 dishes | Cell Treat | 229620 | sterile, tissue culture treated |
| 15 mL conical tubes | Fisher Scientific | 05-539-5 | sterile |
| 1X Phosphate Buffered Saline | Corning Cellgro | 21-031-CV | PBS, sterile, free of Mg2+ and Ca2+ |
| 24-well cell culture treated plates | Fisher Scientific | 07-200-740 | sterile, tissue culture treated |
| 4°C refrigerator | Fisher Scientific | ||
| Mouse IgG anti-paxillin primary antibody (clone 165) | BD Transduction Laboratories | 610620 | marker of focal adhesions |
| Aspirator | Argos | EV310 | |
| Biosafety cabinet | Nuair | NU-477-400 | Class II, Type A, series 5 |
| Delipidated Bovine Serum Albumin (Fatty Acid Free) Powder | Fisher Scientific | BP9704-100 | dlBSA |
| Dimethyl Sulfoxide | Fisher Scientific | BP231-100 | organic solvent to dissolve Ku55933 |
| Dulbecco's Modified Eagle Media, High Glucose | Fisher Scientific | 11965092 | REF52 base cell culture medium |
| Fetal bovine serum | Fisher Scientific | 16000044 | certified, cell culture medium supplement |
| Fiji | National Institutes of Health | http://fiji.sc/ | image analysis program |
| Filter syringe | Fisher Scientific | 6900-2502 | 0.2 µM, sterile |
| Glass coverslips (12-Cir-1.5) | Fisher Scientific | 12-545-81 | autoclave in foil to sterilize |
| Goat anti-mouse IgG secondary antibody Alexa Fluor 488 | Invitrogen | A11001 | fluorescent secondary antibody, light sensitive |
| Goat Serum | Gibco by Life Technologies | 16210-064 | component of blocking solution for immunofluorescence |
| Hemocytometer | Fisher Scientific | 22-600-107 | for cell counting |
| Human Plasma Fibronectin | Gibco by Life Technologies | 33016-015 | FN |
| IX73 Fluorescence Inverted Microscope | Olympus | microscope to visualize fluorescence, cell morphology, counting and dissociation | |
| Ku55933 | Sigma-Aldrich | SML1109-25MG | ATM kinase inhibitor, inducer of reactive oxygen species |
| L-glutamine | Fisher Scientific | 25-030-081 | cell culture medium supplement |
| Monochrome CMOS 16 bit camera | Optimos | ||
| Paraformaldehyde | Sigma-Aldrich | P6148-500G | PFA, fixative for immunofluorescence |
| Penicillin-streptomycin | Fisher Scientific | 15-140-122 | P/S, antibiotic solution for culture medium |
| Alexa Fluor 594 phalloidin (F-actin probe) | Invitrogen | A12381 | marker of F-actin, light sensitive |
| ProLong Gold Anti-fade reagent with DAPI | Invitrogen | P36941 | cover slip mounting media including nuclear dye DAPI, light sensitive |
| REF52 cells | Graham, D.M. et. al. Journal of Cell Biology 2018 | ||
| Stir plate with heat control | Corning Incorporated | PC-420D | |
| Syringe | BD Biosciences | 309653 | 60 mL syringe |
| Tissue culture incubator | Nuair | ||
| Triton X-100 | Fisher Scientific | BP151-500 | detergent used to permeabilize cell membranes |
| Trypan Blue Solution | Fisher Scientific | 15-250-061 | for cell counting |
| Trypsin Neutralizing Solution (1x) | Gibco by Life Technologies | R-002-100 | TNS, neutralizes trypsin instead of fetal bovine serum |
| tube rotator | Fisher Scientific | 11-676-341 | |
| water bath | Fisher Scientific | FSGPD02 |
Referencias
- Geiger, B., Bershadsky, A., Pankov, R., Yamada, K. M. Transmembrane crosstalk between the extracellular matrix--cytoskeleton crosstalk. Nature Reviews: Molecular Cell Biology. 2 (11), 793-805 (2001).
- Geiger, B., Yamada, K. M. Molecular architecture and function of matrix adhesions. Cold Spring Harbor Perspectives in Biology. 3 (5), (2011).
- Lawson, C. D., Burridge, K. The on-off relationship of Rho and Rac during integrin-mediated adhesion and cell migration. Small GTPases. 5, e27958 (2014).
- Heo, J., Campbell, S. L. Mechanism of redox-mediated guanine nucleotide exchange on redox-active Rho GTPases. Journal of Biological Chemistry. 280 (35), 31003-31010 (2005).
- Tolbert, C. E., Beck, M. V., Kilmer, C. E., Srougi, M. C. Loss of ATM positively regulates Rac1 activity and cellular migration through oxidative stress. Biochemical and Biophysical Research Communications. 508 (4), 1155-1161 (2019).
- Hobbs, G. A., et al. Redox regulation of Rac1 by thiol oxidation. Free Radical Biology and Medicine. 79, 237-250 (2015).
- Zhang, Y., et al. Mitochondrial redox sensing by the kinase ATM maintains cellular antioxidant capacity. Science Signaling. 11 (538), (2018).
- Hobbs, G. A., Zhou, B., Cox, A. D., Campbell, S. L. Rho GTPases, oxidation, and cell redox control. Small GTPases. 5, e28579 (2014).
- Shiloh, Y., Ziv, Y. The ATM protein kinase: regulating the cellular response to genotoxic stress, and more. Nature Reviews: Molecular Cell Biology. 14 (4), 197-210 (2013).
- Lang, L., et al. ATM-Mediated Phosphorylation of Cortactin Involved in Actin Polymerization Promotes Breast Cancer Cells Migration and Invasion. Cellular Physiology and Biochemistry. 51 (6), 2972-2988 (2018).
- Peter, Y., et al. Elevated Cu/Zn-SOD exacerbates radiation sensitivity and hematopoietic abnormalities of Atm-deficient mice. European Molecular Biology Organization Journal. 20 (7), 1538-1546 (2001).
- Takao, N., Li, Y., Yamamoto, K. Protective roles for ATM in cellular response to oxidative stress. Federation of European Biochemical Societies Letters. 472 (1), 133-136 (2000).
- Jansen, S., Gosens, R., Wieland, T., Schmidt, M. Paving the Rho in cancer metastasis: Rho GTPases and beyond. Pharmacology & Therapeutics. 183, 1-21 (2018).
- Berrier, A. L., Martinez, R., Bokoch, G. M., LaFlamme, S. E. The integrin beta tail is required and sufficient to regulate adhesion signaling to Rac1. Journal of Cell Science. 115 (Pt 22), 4285-4291 (2002).
- Arthur, W. T., Petch, L. A., Burridge, K. Integrin engagement suppresses RhoA activity via a c-Src-dependent mechanism. Current Biology. 10 (12), 719-722 (2000).
- Arthur, W. T., Burridge, K. RhoA inactivation by p190RhoGAP regulates cell spreading and migration by promoting membrane protrusion and polarity. Molecular Biology of the Cell. 12 (9), 2711-2720 (2001).
- Chandra, S., Kalaivani, R., Kumar, M., Srinivasan, N., Sarkar, D. P. Sendai virus recruits cellular villin to remodel actin cytoskeleton during fusion with hepatocytes. Molecular Biology of the Cell. 28 (26), 3801-3814 (2017).
- Fitzpatrick, M. . Measuring Cell Fluorescence Using ImageJ. , (2014).
- Berginski, M. E., Vitriol, E. A., Hahn, K. M., Gomez, S. M. High-resolution quantification of focal adhesion spatiotemporal dynamics in living cells. PLoS One. 6 (7), e22025 (2011).
- Horzum, U., Ozdil, B., Pesen-Okvur, D. Step-by-step quantitative analysis of focal adhesions. MethodsX. 1, 56-59 (2014).
- Elosegui-Artola, A., et al. Image analysis for the quantitative comparison of stress fibers and focal adhesions. PLoS One. 9 (9), e107393 (2014).
- Meller, J., Vidali, L., Schwartz, M. A. Endogenous RhoG is dispensable for integrin-mediated cell spreading but contributes to Rac-independent migration. Journal of Cell Science. 121 (Pt 12), 1981-1989 (2008).
- Donaldson, J. G. Immunofluorescence Staining. Current Protocols in Cell Biology. 69 (43), 1-7 (2015).
- Burry, R. W. Controls for immunocytochemistry: an update. Journal of Histochemistry and Cytochemistry. 59 (1), 6-12 (2011).
- Grashoff, C., et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 466 (7303), 263-266 (2010).
- Kumar, A., et al. Correction: Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 214 (2), 231 (2016).
- Kumar, A., et al. Talin tension sensor reveals novel features of focal adhesion force transmission and mechanosensitivity. Journal of Cell Biology. 213 (3), 371-383 (2016).
- Friedrichs, J., Helenius, J., Muller, D. J. Quantifying cellular adhesion to extracellular matrix components by single-cell force spectroscopy. Nature Protocols. 5 (7), 1353-1361 (2010).
- Brown, M. A., et al. The use of mild trypsinization conditions in the detachment of endothelial cells to promote subsequent endothelialization on synthetic surfaces. Biomaterials. 28 (27), 3928-3935 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados