Method Article
Una plataforma integrada de espectroscopia y espectrometría de masas de Raman para estudiar la admisión, el metabolismo y los efectos de fármacos de una sola célula
En este artículo
Resumen
Este protocolo presenta una plataforma integrada de espectroscopia de masas (MS) de Raman que es capaz de lograr una resolución de una sola célula. La espectroscopia de Raman se puede utilizar para estudiar la respuesta celular a los medicamentos, mientras que la EM se puede utilizar para el análisis específico y cuantitativo de la toma de drogas y el metabolismo.
Resumen
Se sabe que las células son inherentemente heterogéneas en sus respuestas a las drogas. Por lo tanto, es esencial que la heterogeneidad de una sola célula se tenga en cuenta en los estudios de descubrimiento de fármacos. Esto se puede lograr midiendo con precisión la plétora de interacciones celulares entre una célula y un medicamento a nivel de una sola célula (es decir, la toma de drogas, metabolismo, y el efecto). Este artículo describe una plataforma de espectroscopia ramópica de una sola célula y espectrometría de masas (MS) para monitorear los cambios metabólicos de las células en respuesta a los medicamentos. Usando esta plataforma, los cambios metabólicos en respuesta a la droga se pueden medir por espectroscopia Raman, mientras que el fármaco y su metabolito se pueden cuantificar utilizando espectrometría de masas en la misma célula. Los resultados sugieren que es posible acceder a información sobre la toma de drogas, metabolismo, y la respuesta a un nivel de una sola célula.
Introducción
Las células responden de manera diferente a los cambios en su microambiente a nivel de una sola célula, un fenómeno llamado heterogeneidad celular1. A pesar de esto, los estudios actuales de descubrimiento de fármacos se basan en mediciones medias de las poblaciones celulares, que ofuscan información sobre subpoblaciones potenciales, así como variaciones de una sola célula2. Esta información que falta puede explicar por qué algunas células son más susceptibles a los medicamentos, mientras que otras son resistentes. Curiosamente, la falta de información de una sola célula sobre la respuesta a los medicamentos es una posible razón para el fracaso de los ensayos clínicos de fase II de fármacos3. Por lo tanto, para abordar este problema, las interacciones celulares con el medicamento (es decir, la admisión, el metabolismo y la respuesta) deben medirse a nivel de una sola célula.
Para lograr esto, hemos diseñado un sistema único en el que las células individuales vivas se examinan utilizando espectroscopia Raman sin etiquetas y luego se caracterizan aún más usando espectrometría demasas 4. La espectroscopia Raman proporciona una huella molecular del estado celular, un espectro complejo resultante de las contribuciones de muchas moléculas dentro de la célula. A pesar de esta complejidad, se puede considerar que las huellas dactilares de Raman reflejan la estructura y el metabolismo de toda una célula5,6. La espectroscopia de Raman sobresale en la medición de los estados celulares de una manera no invasiva y relativamente alta, lo que la hace útil para la detección y evaluación de la respuesta de medicamentos a nivel de una sola célula.
Por el contrario, la EM proporciona la sensibilidad y selectividad necesarias para medir la toma de fármacos a nivel de una sola célula. Dado que la EM es destructiva (la muestra [célula] se consume normalmente durante el análisis), integrarla con espectroscopia Raman no destructiva y sin etiquetas puede proporcionar un sistema sensible y de alto rendimiento. Esta plataforma combinada es capaz de proporcionar más información sobre la toma de drogas, metabolismo, y efectos a nivel de una sola célula.
Este manuscrito aclara un protocolo utilizado para estudiar las interacciones celulares con medicamentos a nivel de una sola célula utilizando cultivos in vitro mediante el uso de una plataforma Raman-MS integrada. Para ello, se utilizan como modelo células de carcinoma hepatocelular (HepG2) y tamoxifeno. Células HepG2 fueron elegidos porque toman tamoxifeno y metabolizan la droga, y se ven afectados simultáneamente debido a sus efectos hepatotóxicos. En este manuscrito se utilizan dos estados: células tratadas con drogas frente a células no tratadas (control).
Protocolo
1. Cultivo celular
- Células de cultivo de interés en un medio de cultivo adecuado. Se puede añadir penicilina-estreptomicina para evitar la contaminación. En el caso de las células HepG2, las células de cultivo de un medio de cultivo que contiene el medio de águila modificado de Dulbecco (DMEM) se complementan con un 10% de suero bovino fetal (FBS) y un 0,1% de penicilina-estreptomicina. Para facilitar las mediciones de espectroscopia de Raman, las células se pueden cultivar en un plato con fondo de vidrio recubierto de gelatina del 0,1%o en diapositivas de cuarzo.
- Incubar células durante 2 días a 37oC y 5%co2 en una incubadora humidificada.
- Sincronizar cultivos celulares para alcanzar el 70% de confluencia.
- Las células de subcultivo en una placa de rejilla de vidrio-inferior de 35 mm o diapositivas de cuarzo utilizando el mismo medio a una densidad de semillas de 0.7 x 106,luego incuban a 37 oC durante 24 h.
NOTA: Los platos o toboganes de cultivo se pueden pre-recubrir con solución de recubrimiento de colágeno o gelatina con una relación de superficie de cultivo de 5 ug/cm2 para permitirles fijarlos, asegurando su supervivencia durante la medición.
2. Tratamiento farmacológico
- Retire los cultivos celulares de la incubadora y lave 2 veces con el tampón de PBS precalentado (37 oC).
NOTA: Es óptimo eliminar las células para el tratamiento farmacológico con una confluencia del 50%-60%. - Divida las células en subgrupos tratados con drogas y no tratados en platos de cultivo de 35 mm.
- Mezclar la droga de elección con los medios de cultivo. Por ejemplo, disolver tamoxifeno en dimetil sulfóxido (DMSO) y mezclar con los medios de cultivo para obtener un volumen final de 2 ml y concentración de tamoxifeno de 10 m. Este será el grupo tratado con drogas.
- Mezclar un volumen correspondiente de disolvente (DMSO) en el medio como un control para estudiar los efectos de DMSO. Este será el grupo de control.
- Incubar ambos grupos en 2 ml de los medios espigados preparados en los pasos 2.3-2.4 para 24 h. La confluencia esperada después de la incubación debe ser del 70%-80%.
3. Imágenes espectrales de Raman y procesamiento espectral
NOTA: Aunque los sistemas de espectroscopia Raman están disponibles comercialmente, el sistema de espectroscopia Raman utilizado aquí es un microscopio confocal de escaneo de línea casero descrito anteriormente7,8. En resumen, este sistema está equipado con un láser de estado sólido bombeado por diodo de 532 nm. La luz láser se forma en un plano utilizando una lente cilíndrica, que permite la medición de 400 espectros en una sola exposición. Los espectros de Raman se grabaron utilizando una cámara CCD enfriada montada en un policromator que utiliza una rejilla de 1.200 ranuras/mm para maximizar la resolución espectral de la región de huellas dactilares (de 500-1.800 cm-1). Esta área espectral contiene una alta densidad de frecuencias específicas de las moléculas que genera dispersión de Raman. También se utiliza una lente de inmersión en agua (NA a 0,95). La resolución espacial de este sistema es de 300 nm y la resolución espectral es de 1 cm-1. Para garantizar la supervivencia celular durante el experimento, se utiliza una microcámara fijada en una etapa motorizada del microscopio.
- Antes de las mediciones espectrales, verifique la alineación de la óptica. Se puede utilizar un agujero de 50 m para verificar que el agujero y la posición del láser coincidan exactamente. Introduzca la ranura del espectrofotómetro cuando se estremece tanto como sea posible.
- Utilice etanol para calibrar el espectrofotómetro antes de cada experimento. Para ello, coloque EtOH en un plato con fondo de vidrio, mida el espectro a una intensidad de láser determinada (medida en la muestra) durante 1 s y asocie el pico a longitudes de onda conocidas7.
- Minimice la intensidad del láser en la muestra a 2,4 mW/m2 para que las células sobrevivan a la exposición al láser.
- Poner la microcámara a 5% de CO2 y 37 oC.
- Una vez que el sistema de microscopio esté listo, retire las células de la incubadora y enjuague inmediatamente las células 2 veces con el tampón de PBS caliente (37 oC), luego agregue 2 ml de PBS calentado (37 oC) o DMEM para resuspender las células.
NOTA: Tanto los medios basados en PBS como los basados en FluoroBrite DMEM son opciones suficientes para las mediciones de espectroscopia de Raman porque producen una señal de fondo mínima. - Agregue 10 ml de agua a la lente objetivo de inmersión en agua y coloque delicadamente la placa celular con fondo de vidrio en la etapa del microscopio.
- Mida cada celda enfocando la línea láser. Un tiempo de exposición de 15 s por célula es suficiente aquí para obtener una sección transversal de una célula con una señal Raman clara. Un espejo galvano permite escanear una célula o un grupo de células dentro de varias docenas de minutos.
NOTA: Una mayor resolución de las imágenes espectrales completas de las células requiere más tiempo y puede provocar fotodaños. Aquí, las células se midieron usando una exposición de una sola línea para obtener una sección transversal de cada célula. Este enfoque es un buen equilibrio para aumentar el rendimiento y obtener información suficiente para discriminar las células, al mismo tiempo que se garantiza la viabilidad celular al limitar el daño fotográfico.
4. Preprocesamiento de datos espectrales y análisis multivariados
NOTA: El preprocesamiento es un paso necesario antes del análisis adicional para eliminar las variaciones técnicas no deseadas dentro de los datos espectrales. Debido a la diversidad de los métodos y software, no se puede proporcionar una lista exhaustiva, y hay muchos comentarios útiles que se encuentran en la literatura7,8. En esta sección, describimos brevemente el enfoque utilizado para analizar e interpretar los datos espectrales de Raman obtenidos de células individuales vivas.
- Extraiga y preprocesar imágenes Raman para eliminar posibles interferencias de rayos cósmicos.
NOTA: El eje espectral de los espectros obtenidos durante diferentes días/semanas/meses puede incluir algunas variaciones debido a pequeñas variaciones técnicas durante la calibración con etanol. Esto afectará fuertemente a los análisis multivariados subsiguientes y a las comparaciones estadísticas. En el caso de que los experimentos se realicen durante diferentes semanas/meses, se esperan pequeñas variaciones ópticas. En este caso, los datos deben interpolarse para corregir los desplazamientos espectrales eventuales de los datos entre experimentos. La interpolación mediante spline cúbica se utiliza aquí. Después de este paso, todos los ejes de espectros deben estar alineados. Se considera un rango de 500-1,800 cm-1 para su posterior análisis. - Extraiga datos espectrales de celdas y fondo (ausencia de celdas) de cada imagen utilizando un algoritmo casero. Restar la señal de fondo de la señal de la celda. A continuación, promediar los espectros de los píxeles restantes, que deben corresponder a una sola celda. Los siguientes pasos se realizan utilizando los espectros 2D de las células.
- Realice una corrección de línea base utilizando el ModPoly9 o cualquier otro algoritmo que se estima que se ajuste lo suficiente. Recortar el rango espectral a 600-1,700 cm-1 para seleccionar la región de la huella digital y asegurarse de que no hay efectos de borde no deseados en los espectros debido a un mal ajuste polinómico.
- Realizar un paso de normalización como la normalización vectorial (la intensidad en cada número de onda se divide por la norma l2 global o el valor singular máximo de un espectro) para normalizar la intensidad espectral10,aunque se pueden considerar otras normalizaciones.
- Prepare un conjunto de datos con la etiqueta adecuada para cada clase o condición.
NOTA: Se pueden perforar análisis espectrales comparativos para explorar la naturaleza de las posibles diferencias entre la clase/condiciones de las células (por ejemplo, restando el espectro medio del grupo de control a otros grupos para identificar regiones de interés [como biomarcadores potenciales]). Los cálculos de puntuaciones ANOVA y Fisher también se pueden realizar10. - Para identificar las células tratadas y no tratadas en función de las entidades espectrales, se pueden aplicar análisis multivariados. Los datos espectrales normalizados se deben utilizar como conjunto de datos de entrenamiento y se debe usar un conjunto de datos desconocido (sin etiqueta) de un experimento de réplica como datos de prueba, si es posible.
NOTA: El análisis discriminatorio realizado en algún vector de un análisis de componentes principales (PCA-DA10),la proyección en puntuaciones latentes seguida de el análisis discriminante (PLS-DA) y las máquinas vectoriales de soporte (SVM)11 son modelos que se utilizan a menudo en el campo, y cada uno presenta diferentes consideraciones estadísticas. En consecuencia, se debe realizar el preprocesamiento de los datos. - Utilice el aprendizaje automático que se ajuste a los objetivos experimentales. Aquí, se construye una proyección sobre el modelo de estructura latente (PLS) utilizando la región de huellas dactilares espectrales de los espectros Raman (600-1,710 cm-1)11,12. Centrar los datos según sea necesario. Para la validación cruzada del modelo, se pueden aplicar diferentes técnicas.
NOTA: Aquí se aplica una validación ciega veneciana con 10 divisiones. La complejidad del modelo (número de componentes o variable latente) se debe probar para que el mejor modelo minimice el valor de error cuadrado medio raíz (RMSE). Se encontró que tres vectores latentes (LV) proporcionaban la mejor discriminación con nuestro conjunto de datos. - Identificar qué picos espectrales de Raman contribuyen a la discriminación de las células (por ejemplo, trazando la puntuación de importancia variable en la proyección [VIP] para cada número de onda de Raman o la magnitud del coeficiente de regresión).
NOTA: La puntuación VIP de una variable se calcula como una suma ponderada de las correlaciones cuadradas entre los componentes PLS-DA y la variable original. Los detalles sobre PLS y VIP scores algoritmo se pueden encontrar en la literatura11,12.
5. Configuración y procedimientos de muestreo de una sola célula
- Fije el sistema de muestreo celular en el microscopio Raman como se muestra en la Figura 1. Conecte el micromanipulador 3D al soporte capilar de vidrio que está unido a una jeringa vacía para la succión de muestras aplicando presión negativa(Figura 1).
- Ajuste el microscopio a un campo de aumento alto (40x) para observar la punta del capilar de vidrio y asegurarse de que no esté roto. Controle la posición del capilar de vidrio utilizando el micromanipulador (x-, y-, z-axes). Asegúrese de que la punta capilar esté centrada en el campo de visión, luego mueva el capilar hacia arriba en el eje z para dar espacio libre para el plato de cultivo más adelante.
NOTA: El micromuestreo de las células se realiza mediante el capilar de vidrio para células con diámetros entre 10-15 m. Se recomienda un capilar con un tamaño de agujero de 5 m. Si el tamaño del agujero es demasiado pequeño, la punta capilar será enchufada por la célula, y si es demasiado grande, la sensibilidad de las mediciones posteriores de la EP puede verse comprometida. - Coloque la placa/plato de muestra en el escenario del microscopio, ajuste la ampliación y el enfoque, seleccione la celda de destino en el plato de la rejilla y muévala al centro de visión. Luego, baje cuidadosamente el capilar de vidrio usando micromanipulador (eje z) hasta que la punta entre en foco.
NOTA: Asegúrese de no mover el capilar en los ejes X e Y hasta que el capilar esté enfocado. - En observación microscópica, toque la célula única objetivo con la punta capilar y, a continuación, comience a aplicar presión negativa utilizando la jeringa para atrapar la célula dentro de la punta capilar. Grabe este procedimiento tomando una foto o un video para comprobar el tiempo y la ubicación aspirada de la celda con precisión, si es necesario.
- Mueva el capilar hacia arriba en el eje z. A continuación, separar el capilar del soporte capilar utilizando fórceps en preparación para el análisis de EM.
6. Mediciones de espectrometría de masas
- Calibre la precisión de masa del instrumento MS de acuerdo con las recomendaciones del fabricante. Después de la calibración, asegúrese de que el error de masa no sea superior a 3 ppm.
- Optimice el instrumento MS a los parámetros que mejor se adapten al analito de interés.
NOTA: En el caso del tamoxifeno y el análisis 4-OHT, los parámetros del instrumento se establecen en lo siguiente: temperatura capilar de entrada: 400 oC, voltaje de pulverización: 1500 V, objetivo de control de ganancia automática (AGC): 5.00E+06, nivel de RF De lente S: 90%, rango SIM: 347-397 m/z, tiempo de inyección máximo SIM: 200 ms, Resolución SIM: 140.000 FWHM, rango MS/MS: 50-400 m/z, mS/MS AGC objetivo: 2.00E+05, MS/MS tiempo máximo de inyección: 100 ms, resolución MS/MS: 17.500 FWHM, ventana de aislamiento MS/MS: 1 m/z, MS/MS energía de colisión normalizada (NCE 35): - Configure un método de adquisición automática con una duración de 5 min para el modo SIM para lograr una cuantificación relativa, y otro método MS/MS para la identificación positiva del fármaco y su metabolito. Los parámetros del método de adquisición deben establecerse en los valores optimizados mencionados en el paso 6.2.
- Prepare el disolvente de ionización bajo una campana de humos. La composición del disolvente depende del analito de interés. Aquí, el disolvente orgánico utilizado consiste en 80% MeOH, 10% DMSO, y 0.1% ácido fórmico.
- Mezclar un estándar interno adecuado con el disolvente orgánico antes de las mediciones. En este experimento, 5.31 nM de d5-tamoxifeno se utiliza como un estándar interno.
- Para evitar falsos positivos, aspirar los medios que rodean las células tratadas con el fármaco utilizando un capilar de tamaño de diámetro de 1 m con observación microscópica constante para evitar el muestreo de cualquier parte celular.
- Añadir 2 l del disolvente de ionización al extremo ancho del capilar que contiene el medio utilizando una pipeta unida a las puntas del cargador. A continuación, analizar los medios muestreados por EM, comprobar la presencia del analito de interés (normalmente, no debe ser detectable).
- Añadir el disolvente de ionización de 2 l al capilar que contiene la célula, fijar el capilar a un adaptador de nanoelectrospray (nano-ESI) conectado a un espectrómetro de masas adecuado e iniciar el método de adquisición automática.
7. Procesamiento y análisis de datos de espectrometría de masas
NOTA: Cualquier software adecuado se puede utilizar para realizar análisis de datos. Sin embargo, si los investigadores desean realizar el análisis de datos utilizando un software que no es proporcionado por el proveedor de MS, entonces los datos sin procesar deben convertirse del formato de proveedor propietario a un formato abierto o como un archivo de texto primero (que se hizo aquí).
- Normalice los datos dividiendo el área pico del analito de interés por el del estándar interno de la misma exploración MS. A continuación, el registro transforma las proporciones de pico para reducir la asimetría.
- Trazar la intensidad normalizada de la droga o su metabolito como una gráfica de caja o curva de densidad para visualizar la distribución a través de células individuales. Aquí, se utiliza el software estadístico R, junto con el paquete ggplot2.
- Calcular la proporción de fármaco sin metabolizar a fármaco no metabolizado dividiendo la abundancia del metabolito del fármaco por la de la molécula madre no metabolizada en cada célula (es decir, 4-OHT y tamoxifeno, respectivamente.
NOTA: Se puede estudiar la correlación entre las variaciones de los picos específicos de interés de Raman y las variaciones en los picos de EM de la droga o sus metabolitos. Esta es una adición a la posible correlación entre la droga en sí y su metabolito en células individuales. Esto se puede hacer calculando el coeficiente de correlación de Pearson utilizando una prueba de dos colas. También deben considerarse enfoques integradores más avanzados.
Resultados
El análisis de una sola célula de las interacciones farmacológicas (aceptación, metabolismo y efectos) es esencial para descubrir cualquier subpoblación oculta o farmacorresistente, así como para comprender los efectos de la heterogeneidad celular. En este protocolo, se utilizaron dos técnicas complementarias para medir las interacciones antes mencionadas en células individuales: espectroscopia de Raman y Em. Espectrometría Raman identifica rápidamente las células afectadas por fármacos basados en biomarcadores espectrales de la respuesta del fármaco. La EP se utiliza para controlar la ingesta y el metabolismo de la droga de una manera selectiva y semicuantitativa. Las células fueron examinadas primero por espectroscopia Deraman y luego muestreadas individualmente para su análisis por EM.
En la Figura 2se muestra un análisis comparativo del espectro medio de cada condición (con y sin tratamiento farmacológico). El espectro promediado de las dos condiciones difiere claramente en varios picos, que fueron previamente identificados y asignados a los compuestos moleculares2. En particular, los picos de 1000 cm- (asignado a compuestos aromáticos como la fenilalanina y la tirosina) muestran fuertes diferencias. La importancia de la diferencia estadística debe evaluarse mediante nuevos análisis multivariados.
El conjunto de datos se utilizó entonces para entrenar un modelo de PLS (pasos 4.5-4.8) destinado a distinguir los dos tratamientos celulares (con el fármaco: n a 290, sin fármaco: n a 115). La capacidad predictiva de clasificar las células cultivadas en presencia de tamoxifeno alcanzó una sensibilidad del 100% y una especificidad del 72% en los datos de prueba (desconocidas del modelo entrenado validado). La sensibilidad es una medida de los verdaderos positivos que se identifican correctamente por el modelo, mientras que la especificidad es una medida de los negativos reales que se identifican por el modelo. Los modelos alternativos como las SMS, los LDA y las redes neuronales pueden proporcionar resultados similares o mejores, aunque no se ha realizado una comparación completa en este estudio.
Sobre la base del modelo PLS, se calcularon las puntuaciones VIP, que representan la importancia de las longitudes de onda (desplazamientos de Raman) en la discriminación de las condiciones experimentales(Figura 3). Es importante destacar que los picos más altos de los perfiles VIP correspondieron a picos de Raman para los que se vieron fuertes diferencias entre los dos tratamientos. Esto confirmó las diferencias moleculares específicas entre las células tratadas y las no tratadas. En consecuencia, los investigadores pueden identificar posibles biomarcadores espectrales que reflejan la respuesta de las células individuales al tratamiento farmacológico. Estos biomarcadores se pueden probar más a fondo para verificar su relevancia biológica y generalización en diversas condiciones y líneas celulares.
Un sistema vivo de espectrometría de masas de una sola célula (LSC-MS) fue capaz de detectar tanto el fármaco como sus metabolitos en células HepG2 únicas tratadas con medicamentos que se midieron previamente mediante espectroscopia de Raman. Además, se puede utilizar una EM en tándem para confirmar la estructura de ambas moléculas. Después de la identificación positiva, la abundancia relativa de la droga y sus metabolitos se midieron en cada célula y se compararon con los picos de fondo en las células no tratadas. Se observó una fuerte variación en la abundancia de tamoxifeno, y este fenómeno fue aún más pronunciado en el caso de su metabolito, 4-OHT(Figura 4). También se estudió la relación entre la abundancia de tamoxifeno y sus metabolitos, en la que se encontró una correlación positiva significativa entre los dos (r a 0,54, p a 0,0001, n a 31).

Figura 1: Sistema de selección celular montado en una etapa del microscopio. Haga clic aquí para ver una versión más grande de esta figura.
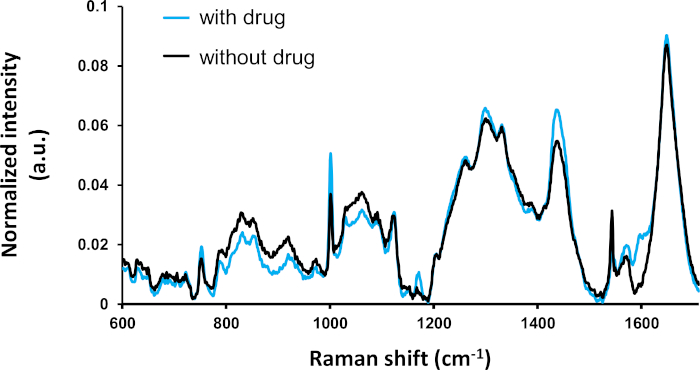
Figura 2: Espectro promediado de las células tratadas con medicamentos (con tamoxifeno: n a 295) y células no tratadas (sin tamoxifeno: n a 115). Los picos de Raman se pueden identificar a partir de la literatura. La mayoría de las fuertes diferencias espectrales son estadísticamente significativas (ANOVA, p a 0,5) como se describió anteriormente4. Esta cifra ha sido modificada de una publicación anterior4. Haga clic aquí para ver una versión más grande de esta figura.
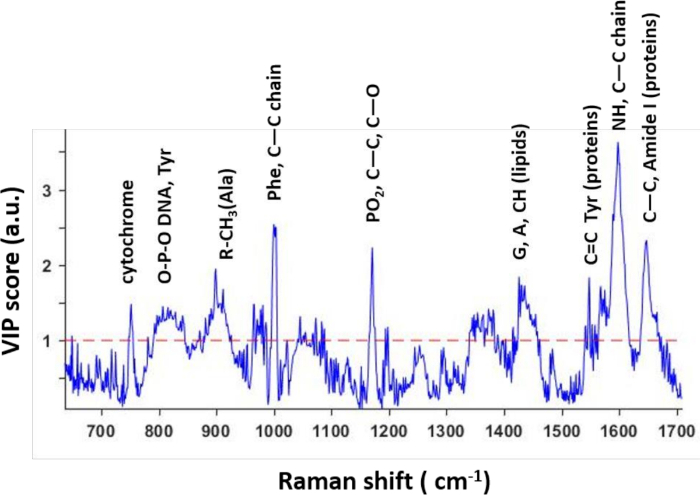
Figura 3: Puntuaciones VIP extraídas del modelo PLS predictivo. Las puntuaciones VIP reflejan las longitudes de onda que contribuyen a distinguir entre las dos clases del modelo. La mayoría de los picos corresponden a moléculas específicas que se observan como biomarcadores espectrales de efectos farmacológicos en células tratadas con fármacos. Esta cifra ha sido modificada de una publicación anterior4. Haga clic aquí para ver una versión más grande de esta figura.
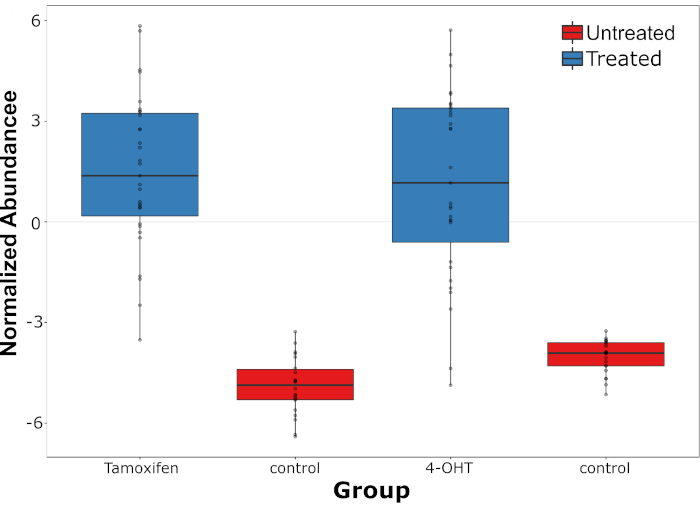
Figura 4: Distribución de la abundancia de tamoxifeno y su metabolito. Distribución de la abundancia de tamoxifeno y su metabolito, 4-OHT (medido a nivel de una sola célula) en comparación con los picos endógenos en las células no tratadas (control). Esta cifra ha sido modificada de una publicación anterior4. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En este manuscrito, se eligió un caso simple en el que las células HepG2 estaban expuestas (o no) al tamoxifeno. Se demuestra que la capacidad de un sistema de espectroscopia y espectrometría de masas de Raman monitorea los efectos del tamoxifeno en las células. La espectroscopia de Raman permitió la identificación de biomarcadores potenciales que reflejaban una respuesta general de células individuales a la exposición a fármacos. Se observó cierta heterogeneidad entre células individuales, lo que sugiere que algunas células no respondieron a la exposición a los medicamentos. Por otro lado, LSC-MS fue capaz de realizar un análisis específico de la droga y su metabolito a nivel de una sola célula, en el que se observó un alto grado de heterogeneidad en la droga y su abundancia de metabolitos. Esta heterogeneidad ayuda a explicar por qué algunas células se ven afectadas por la droga, mientras que otras aparentemente no lo son, a pesar de las células que provienen de una población supuestamente uniforme12.
Entre aspectos particulares de esta técnica que requieren atención, es importante evaluar la calidad de la configuración del microscopio y el procesamiento de la señal para garantizar la reproducibilidad de los datos. Si el preprocesamiento de los espectros se realiza cuidadosamente, las variaciones de la señal deben maximizarse al máximo local de cada pico. Por el contrario, la línea base y el borde de los espectros deben superponerse entre las condiciones celulares probadas. Otro aspecto importante es el modelo multivariado utilizado para investigar las diferencias entre los tratamientos. Uno debe evaluar cuidadosamente los modelos y los parámetros del modelo para asegurar un análisis preciso y preciso. Una ventaja del modelo PLS, a diferencia de las redes neuronales, es que permite el acceso a los pesos asociados con cada longitud de onda (desplazamientos Raman) que distinguen mejor las condiciones probadas por el modelo.
A pesar de la espectroscopia de Raman que discrimina con éxito la respuesta del fármaco, cabe destacar que esta técnica es limitada en su uso para proporcionar interpretación biológica. Esto se debe principalmente a la complejidad de la señal espectral, que abarca una mezcla de miles de moléculas. Por lo tanto, se requiere una investigación adicional para evaluar las variaciones sistemáticas entre las intensidades espectrales de Raman y las variaciones en las concentraciones de fármacos. Además, se requieren estudios similares de otras líneas celulares para evaluar la generalización de biomarcadores espectrales asociados con el tamoxifeno.
Además, puede ser de interés realizar mediciones de tejidos vivos para evaluar la farmacodinámica y estudiar cómo los medicamentos penetran y fluyen dentro de cada célula. Además, cabe señalar que el paso de muestreo en LSC-MS depende en gran medida de la habilidad del operador. Parámetros como la resolución espacial, la posición de la celda dentro del capilar después del muestreo y la resistencia al rendimiento dependen toquemente del operador, lo que limita la adopción a gran escala de LSC-MS. Aunque, los sistemas de muestreo automatizados pueden aliviar este problema. Además, mientras que LSC-MS sobresale en el muestreo de células adherentes o flotantes en sus estados nativos, funciona más mal en el muestreo de células incrustadas en secciones de tejido. Esto se debe a la tendencia de la punta capilar de muestreo a romperse si la densidad de la muestra es alta. Por lo tanto, otro enfoque como la sonda única puede ser más adecuado en tales casos14,15.
Puesto que las células utilizadas aquí se muestrean en condiciones ambientales con una preparación mínima de la muestra, LSC-MS se puede integrar fácilmente con otras tecnologías, como lo demuestra su integración con Raman en este protocolo. Otra integración similar con la holografía 3D ha permitido lograr la cuantificación absoluta de metabolitos celulares en el nivel subcelular16. Además, la integración con la citometría de flujo ha permitido el descubrimiento de biomarcadores metabólicos en células tumorales circulantes únicas de pacientes con cáncer de neuroblastoma17,18.
En el futuro, debido al creciente interés reciente en combinar conjuntos de datos de las modalidades de imagen19, también puede ser de interés estudiar las variaciones sistemáticas entre las señales Raman y los resultados de la espectrometría de masas (así como otros métodos de ómica) mediante el uso de enfoques computacionales integradores. Curiosamente, ya hemos encontrado varias correlaciones lineales débiles pero significativas entre las intensidades de los picos de Raman identificados por las puntuaciones VIP y la abundancia de tamoxifeno o su metabolito a nivel de una sola célula identificado por MS4. Estos datos pueden sugerir una relación metabólica entre los perfiles de EM y los espectros de Raman y la posibilidad de predecir estos valores.
Divulgaciones
Los autores no declaran conflictos de intereses.
Agradecimientos
Los autores agradecen a Toshio Yanagida por su apoyo y los fondos colaborativos internos rikEN atribuidos al Dr. Arno Germond.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.1% penicillin-streptomycin | Nacalai Tesque | 09367-34 | |
| 35mm glass bottom grid dish | Matsunami | ||
| 4-Hydroxy Tamoxifen standard | Sigma-Aldrich | 94873 | |
| 532 nm diode pumped solid-state laser | Ventus, Laser Quantum | ||
| BIOS-L101T-S motorized microscope stage | OptoSigma | ||
| CT-2 cellomics coated sampling capillaries | HUMANIX | ||
| d5-Tamoxifen standard | Cambridge Isotope Laboratories | ||
| Dimethyl sulfoxide LC-MS grade | Nacalai Tesque | D8418 | |
| Dulbecco's Modified Eagle's medium | Sigma-Aldrich | D5796 | |
| Eppendorf GELoader tips | Eppendorf | ||
| fetal bovine serum | Hyclone laboratories | SH3006603 | |
| FluoroBrite DMEM | Thermo Fisher Scientific | ||
| Formic acid LC-MS grade | Sigma-Aldrich | 33015 | |
| HepG2 cell line (RCB1886) | RIKEN cell bank center | RCB1886 | |
| MC0-19A1C Incubator | Sanyo Electric Co. | MC0-19A1C | |
| Methanol LC-MS grade | Sigma-Aldrich | 1060352500 | |
| MMO-203 3-D Micromanipulator | Narshige | MMO-203 | |
| NA:0.95, UPL40 water-immersion Olympus objective lens | Olympus | ||
| Nanoflex nano-ESI adaptor | Thermo Fisher Scientific | ES071 | |
| On-stage incubator | ibidi | ||
| Pierce LTQ Velos ESI calibration solution | Thermo Fisher Scientific | 88323 | |
| PIXIS BR400 cooled CCD camera | Princeton Instruments | ||
| Q-Exactive Orbitrap | Thermo Fisher Scientific | ||
| Rat-tail collagen coating solution | Cell Applications Inc. | ||
| Tamoxifen standard | Sigma-Aldrich | 85256 |
Referencias
- Altschuler, S. J., et al. Cellular heterogeneity: do differences make a difference?. Cell. 141 (4), 559-563 (2010).
- Ali, A., et al. Single-cell metabolomics by mass spectrometry: Advances, challenges, and future applications. TrAC Trends in Analytical Chemistry. , (2019).
- Bunnage, M., et al. Target validation using chemical probes. Nature Chemical Biology. 9 (4), 195-199 (2013).
- Ali, A., et al. Single-Cell Screening of Tamoxifen Abundance and Effect Using Mass Spectrometry and Raman-Spectroscopy. Analytical Chemistry. 91 (4), 2710-2718 (2019).
- Wu, H., et al. In vivo lipidomics using single-cell Raman spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 108 (9), 3809-3814 (2011).
- Okada, M., et al. Label-free Raman observation of cytochrome c dynamics during apoptosis. Proceedings of the National Academy of Sciences of the United States of America. 109 (1), 28-32 (2012).
- Palonpon, A. F., et al. Raman and SERS microscopy for molecular imaging of live cells. Nature Protocols. 8 (4), 677-692 (2013).
- Butler, H. J., et al. Using Raman spectroscopy to characterize biological materials. Nature Protocols. 11 (4), 664-687 (2016).
- Mark, H., Workman, J. . Chemomtrics in Spectroscopy. , (2018).
- Lieber, C. A., Mahadevan-Jansen, A. Automated method for subtraction of fluorescence from biological Raman spectra. Applied Spectroscopy. 57, 1363-1367 (2003).
- Germond, A., et al. Raman spectral signature reflects transcriptomic features of antibiotic resistance in Escherichia coli. Communications Biology. 1, 85 (2018).
- Wold, S., et al. Partial Least Squares Projections to Latent Structures (PLS) in Chemistry. Encyclopedia of Computational Chemistry. , (2002).
- Chong, I. G., Jun, C. H. Performance of some variable selection methods when multicollinearity is present. Chemometrics and Intelligent Laboratory Systems. 78, (2005).
- Inde, Z., Dixon, S. J. The impact of non-genetic heterogeneity on cancer cell death. Critical Reviews in Biochemistry and Molecular Biology. 53 (1), 99-114 (2018).
- Pan, N., et al. The single-probe: a miniaturized multifunctional device for single cell mass spectrometry analysis. Analytical chemistry. 86 (19), 9376-9380 (2014).
- Rao, W., et al. Applications of the Single-probe: Mass Spectrometry Imaging and Single Cell Analysis under Ambient Conditions. Journal of Visualized Experiments. (112), e53911 (2016).
- Ali, A., et al. Quantitative Live Single-cell Mass Spectrometry with Spatial Evaluation by Three-Dimensional Holographic and Tomographic Laser Microscopy. Analytical Sciences: the International Journal of the Japan Society for Analytical Chemistry. 32 (2), 125-127 (2016).
- Abouleila, Y., et al. Live single cell mass spectrometry reveals cancer-specific metabolic profiles of circulating tumor cells. Cancer Science. 110, 697-706 (2018).
- Hiyama, E., et al. Direct Lipido-Metabolomics of Single Floating Cells for Analysis of Circulating Tumor Cells by Live Single-cell Mass Spectrometry. Analytical Sciences: the International Journal of the Japan Society for Analytical Chemistry. 31 (12), 1215-1217 (2015).
- Ryabchykov, O., et al. Fusion of MALDI Spectrometric Imaging and Raman Spectroscopic Data for the Analysis of Biological Samples. Frontiers in Chemistry. 6, 257 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados