Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Evaluación de la oxidación celular utilizando una proteína fluorescente verde sensible al redox específica del compartimento subcelular
En este artículo
Resumen
Este protocolo describe la evaluación del estado redox subcelular específico del compartimento dentro de la célula. Una sonda fluorescente sensible al redox permite un análisis ratiométrico conveniente en las células intactas.
Resumen
La medición del equilibrio de oxidación/reducción intracelular proporciona una visión general del estado redox fisiológico y/o fisiopatológico de un organismo. Los tioles son especialmente importantes para iluminar el estado redox de las células a través de sus proporciones reducidas de ditiool y disulfuro oxidado. Las proteínas fluorescentes que contienen cisteína de diseño abren una nueva era para los biosensores sensibles al redox. Una de ellas, la proteína fluorescente verde sensible al redox (roGFP), se puede introducir fácilmente en las células con transducción adenoviral, lo que permite evaluar el estado redox de los compartimentos subcelulares sin interrumpir los procesos celulares. Las ciisteínas reducidas y las cistinas oxidadas de roGFP tienen máximas de excitación a 488 nm y 405 nm, respectivamente, con emisión a 525 nm. La evaluación de las proporciones de estas formas reducidas y oxidadas permite el cálculo conveniente del equilibrio redox dentro de la célula. En este artículo del método, se utilizaron células de cáncer de mama de triple negativo humano inmortalizados (MDA-MB-231) para evaluar el estado de redox dentro de la célula viva. Los pasos del protocolo incluyen la transducción de línea celular MDA-MB-231 con adenovirus para expresar roGFP citosólico, el tratamiento con H2O2y la evaluación de la relación de cisteína y cistina con citometría de flujo y microscopía de fluorescencia.
Introducción
El estrés oxidativo fue definido en 1985 por Helmut Sies como "una alteración en el equilibrio prooxidante-antioxidante a favor de la primera"1, y se ha llevado a cabo una plétora de investigación para obtener enfermedad-, nutrición-, y el envejecimiento-específico estado redox de los organismos1,2,3. Desde entonces, la comprensión del estrés oxidativo se ha vuelto más amplia. Probar las hipótesis del uso de antioxidantes contra enfermedades y/o envejecimiento ha demostrado que el estrés oxidativo no sólo causa daño, sino que también tiene otras funciones en las células. Además, los científicos han demostrado que los radicales libres desempeñan un papel importante en la transducción de señales2. Todos estos estudios refuerzan la importancia de determinar los cambios en la relación reducción-oxidación (redox) de las macromoléculas. La actividad enzimática, antioxidantes y/o oxidantes, y los productos de oxidación se pueden evaluar con varios métodos. Entre estos, los métodos que determinan la oxidación del tiol son posiblemente los más utilizados porque informan sobre el equilibrio entre antioxidantes y proxóxidos en las células, así como los organismos4. Específicamente, las relaciones entre el disulfuro de glutatión (GSH)/glutatión (GSSG) y/o cisteína (CyS)/cistina (CySS) se utilizan como biomarcadores para controlar el estado de redox de los organismos2.
Los métodos utilizados para el ensayo del equilibrio entre prooxidantes y antioxidantes dependen principalmente de los niveles de proteínas reducidas/oxidadas o moléculas pequeñas dentro de las células. Las manchas occidentales y la espectrometría de masas se utilizan para evaluar ampliamente las proporciones de macromoléculas reducidas/oxidadas (proteínas, lípidos, etc.), y las proporciones GSH/GSSG se pueden evaluar con espectrofotometría5. Una característica común de estos métodos es la perturbación física del sistema por lelisis celular y/o homogeneización tisular. Estos análisis también se vuelven desafiantes cuando es necesario medir el estado de oxidación de diferentes compartimentos celulares. Todas estas perturbaciones causan artefactos en el entorno de ensayo.
Las proteínas fluorescentes sensibles a redox abrieron una era ventajosa para evaluar el equilibrio redox sin causar una perturbación en las células6. Pueden apuntar a diferentes compartimentos intracelulares, permitiendo la cuantificación de actividades específicas del compartimiento (por ejemplo, el ensayo del estado redox de las mitocondrias y el citosol) para investigar la interferencia entre los orgánulos celulares. Meyer y sus colegas6revisan la proteína fluorescente amarilla (YFP), la proteína fluorescente verde (GFP) y las proteínas HyPeR. Entre estas proteínas, GFP sensible al redox (roGFP) es único debido a las diferentes lecturas fluorescentes de sus residuos CyS (por ejemplo, 488 nm/em. 525 nm) y CySS (por ejemplo, 405 nm/525 nm), que permite el análisis ratiométrico, a diferencia de otras proteínas sensibles al redox como YFP7,,8. La salida ratiométrica es valiosa porque contrarresta las diferencias entre los niveles de expresión, las sensibilidades de detección y el fotoblanqueo8. Los compartimentos subcelulares de las células (citosol, mitocondrias, núcleo) u diferentes organismos (bacterias, así como células de mamíferos) pueden ser objetivo mediante la modificación de roGFP7,9,10.
Los ensayos roGFP se llevan a cabo utilizando técnicas de imagen fluorescente, especialmente para experimentos de visualización en tiempo real. Los análisis citométricos de flujo de roGMP también son posibles para experimentos con puntos de tiempo predeterminados. El artículo actual describe tanto el uso de la microscopía fluorescente como la citometría de flujo para realizar una evaluación ratiométrica del estado de redox en las células de los mamíferos que sobreexpresan el roGFP (dirigido al citosol) a través de la transducción adenoviral.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: Este protocolo se optimizó para celdas MDA-MB-231 confluentes del 70% al 80%. Para otras líneas celulares, se debe reoptimizar el número de células y la multiplicidad de infección (MOI).
1. Preparación de células (día 1)
- Mantener la línea celular MDA-MB-231 en matraces de 75 cm2 con 10 ml del medio águila modificado de Dulbecco (DMEM) complementado con un 10% de suero bovino fetal (FBS) a 37 oC en una atmósfera humidificada de 5% CO2.
NOTA: DMEM complementado con 10% FBS, 37 oC, y una atmósfera humidificada 5% CO2 se utilizan para todas las incubaciones de apego y tratamiento a lo largo de todo el protocolo. - Prepare las células MDA-MB-231 para el experimento.
- Aspirar el medio dentro del matraz, separar las células con 2 ml de solución de trippsina-EDTA al 0,25% durante 2 min, e inactivar la actividad de tripsina con 6 ml de medio completo (DMEM con 10% FBS). Centrifugar las células a 150 x g durante 5 min. Aspirar el sobrenadante y suspender las células en 5 ml de medio completo.
- Mezcla una suspensión celular de igual volumen y un 0,4% de color azul trypan. Tome 10 l de esta mezcla y cuente las células con el contador de células automatizado.
NOTA: También se puede utilizar un contador Coulter o un hemociclometro para el recuento de células. - Siembra las células en una placa de 6 pozos para análisis de citometría de flujo y siembra 150.000 células en 1 ml de medio por pozo. Espere 16 h para el accesorio de celda.
- Siembra las células en un portaobjetos de cámara de 4 pozos para imágenes fluorescentes y siembra 25.000 células en 0,5 ml de medio por pozo. Espere 16 h para el accesorio de celda.
NOTA: Los pozos de control de semillas además de los pozos de tratamiento. Utilice uno de los pozos de control para determinar el número de celda (opcional: si el período de unión para las celdas es más corto que el tiempo de duplicación, se puede suponer que el número de celda es el mismo que la densidad de siembra) y el otro para un control no infectado (0 MOI).
2. Transducción adenoviral roGFP (días 2 y 3)
ADVERTENCIA: Los adenovirus pueden causar enfermedades. Mientras transduzca las células, utilice puntas filtradas y descontamine las puntas, las pipetas Pasteur y los tubos de microcentrífuga con 10% de lejía.
NOTA: Este protocolo se demostró con roGFP específico de citosol, pero otros compartimentos celulares (por ejemplo, mitocondrias o espacio intermembrano mitocondrial) pueden ser dirigidos con este mismo protocolo.
- Generar una curva de dosis-respuesta para que el MOI obtenga la mayor eficiencia de transducción calculando el volumen de adenovirus (ml) necesario para cada valor MOI para la línea celular MDA-MB-231 (Tabla 1):
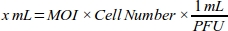
NOTA: El título funcional de cada lote de stock adenoviral, que se expresa como unidad de conformado de placa (PFU) por ml, es proporcionado por la empresa. El MOI óptimo para la transducción difiere entre los tipos de células. Para la mayoría de las células de mamíferos, el rango óptimo de MOI está entre 10 y 300. De acuerdo con la respuesta celular, los valores de MOI deben ser recalculados (por ejemplo, el rango de MOI debe reducirse si las células tienen respuesta citotóxica, o el rango debe aumentarse si las células tienen baja eficiencia de transducción). - Haga una dilución 1:100 de 6 x 1010 PFU/ml solución roGFP adenoviral con medio de cultivo celular (DMEM con 10% FBS) para pipeteo confiable.
- Pipetear y añadir 0,0125 ml (12,5 l), 0,025 ml (25 l), 0,05 ml (50 ml) de dilución roGFP adenoviral en cada pocóver de la placa de 6 pozos para transducir las 150.000 células con 50, 100 y 200 MOI respectivamente para el análisis de citometría de flujo (Tabla 1).
- Pipetear y añadir 0,0042 ml (4,2 l) de dilución roGFP adenoviral en los pocillos deslizantes de 4 cámaras para transducir 25.000 células con 100 MOI para imágenes de fluorescencia (Tabla 1).
NOTA: Se debe utilizar una cantidad mínima de medio en los pozos para garantizar la mayor interacción entre la construcción adenoviral roGFP y las células. El contenido sérico del medio de cultivo puede necesitar ser disminuido para diferentes líneas celulares porque los altos niveles de suero pueden afectar negativamente la eficiencia de la transducción en algunos tipos celulares. - Incubar células durante 16–24 h bajo las condiciones de mantenimiento celular. Al día siguiente (día 3), cambiar el medio de cultivo medio a celular (DMEM con 10% FBS) para permitir la recuperación celular durante 24 h adicionales. Visualizar células bajo un microscopio para evaluar su morfología; las células pueden expresar roGFP incluso si tienen cambios morfológicos.
NOTA: En el día 3, las células deben comenzar a expresar roGFP; por lo tanto, la eficiencia de la transducción puede controlarse mediante microscopía de fluorescencia (filtros con ex. 488/em. 525). Para obtener resultados consistentes en el ensayo, tenga en cuenta y documente los cambios morfológicos bajo el microscopio de contraste de fase y observe la morfología mientras evalúa la eficiencia de la transducción. - Construir una curva de respuesta a la dosis utilizando las muestras de 50, 100 y 200 MOI preparadas en el paso 2.3 y sus resultados de eficiencia de transducción obtenidos del análisis de citometría de flujo (pasos 3.1 y 4.1). Evalúe la eficiencia óptima de la transducción con la documentación de los cambios morfológicos (paso 2.5) y la curva dosis-respuesta del MOI.
NOTA: Aunque más del 98% de la población celular en 100 MOI y 200 MOI express roGFP (ver resultados representativos), 200 MOI grupo mostró cambios sustanciales en la morfología celular de las células MDA-MB-231. En consecuencia, se determinó que el MOI más eficaz para las células MDA-MB-231 era de 100 MOI. - Después de MOI óptimo (aquí, 100 MOI) fue elegido para la línea celular MDA-MB-231, llevar a cabo un experimento con materiales de prueba (10 m H2O2 y su vehículo 0.1% agua desionizada).
- Preparar y sembrar las células de acuerdo con la sección 1. Utilizando el volumen de transducción adenoviral para 100 MOI calculado en el paso 2.1, repita los pasos 2.2 a 2.4 para 100 transducción adenoviral MOI de células. A continuación, incubar la placa y los portaobjetos de la cámara de acuerdo con el paso 2.5.
3. Adquisición del saldo CyS/CySS
- Citometría de flujo (día 4)
- En el día 4, incubar las células del paso 2.7.1 con 10 m H2O2 durante 1 h.
NOTA: Se utilizaron 10 m H2O2 como sustancia de ensayo y se utilizó agua desionizada al 0,1% como tratamiento vehiculoso en este protocolo. Otros agentes oxidantes se pueden utilizar como controles positivos aquí. - Aspirar los medios de la placa de 6 pozos, reemplazarlo con 750 l de solución de trippsina-EDTA al 0,25% y esperar 2 minutos para que las células se desprendan. Inactivar la trippsina con 2 ml de medio completo (DMEM con 10% FBS) y recoger el volumen en tubos cónicos de 15 ml.
- Centrifugar los tubos a 150 x g durante 5 min a 4oC. Deseche el sobrenadante y suspenda las células en 500 l de solución salina tamponada con fosfato (PBS).
- Repita el paso 3.1.3
- Filtrar las suspensiones celulares en tubos compatibles con citometría de flujo utilizando malla de 40 m. Mantenga los tubos sobre hielo y lejos de la luz y siga el paso 4.1 para el análisis de datos.
- En el día 4, incubar las células del paso 2.7.1 con 10 m H2O2 durante 1 h.
- Imágenes microscópicas (día 4)
- En el día 4, tratar las células con 10 m H2O2, adquirir imágenes inmediatamente (punto de tiempo 0) y 1 h después del tratamiento y seguir el paso 4.2 para el análisis de datos.
4. Análisis de datos
- Cuantificación de la citometría de flujo
- Establecer el método de citometría de flujo para 3 análisis diferentes a través del software de adquisición de muestras (ver Tabla de Materiales):dispersión hacia adelante (FCS) en eje x y dispersión lateral (SSC) en el eje Y para evaluar el tamaño de la célula y la complejidad de las células (SSC se puede utilizar para la identificación aproximada de células muertas y vivas); ex. 488 nm/em. Filtro de paso de banda de 525 nm (isotiocianato de fluoresceína [FITC]) en el eje X y SSC en el eje Y para evaluar CyS-roGFP; ex. 405 nm/em. Filtro de paso de banda de 525 nm (Violeta brillante 510 [BV510]) en el eje X y SSC en el eje Y para evaluar CySS-roGFP.
- Adquiera 0 MOI control y visualice celdas con software de adquisición de muestras. Repita este paso para las muestras restantes (50, 100, 200 grupos de MOI y más tarde 10 M H2O2 células tratadas y células tratadas con vehículos). Guarde los archivos para el análisis de datos.
- Abra el software deanálisis de datos (consulte Tabla de materiales ) y abra el archivo de ejemplo MOI 0. Evaluar la población celular de interés (Puerta 1). Configure las siguientes gatings para minimizar la fluorescencia de fondo para ex. 488 nm/em. 525 nm (puerta 2) y ex. 405 nm/em. Filtros de paso de banda de 525 nm (puerta 3) con las celdas de control no infectadas (0 MOI).
- Abra 50, 100 y 200 archivos de muestra MOI dentro del software de análisis de datos para evaluar la curva de dosis-respuesta. Analice las intensidades medias de fluorescencia con las puertas 2 y 3 para cada muestra. Repita este paso para las muestras de prueba (células tratadas con 10 m H2O2 y células tratadas con vehículos).
- Calcule la relación media de intensidad fluorescente entre las formas oxidadas frente a las reducidas de roGFP con la siguiente ecuación.

- Evaluación de la imagen
- Utilice un microscopio que contenga filtros de fluorescencia para CyS-roGFP y CySS-roGFP (por ejemplo, 488 nm/em. 525 nm y ex. 405 nm/em. filtros de 525 nm, respectivamente).
- En cada pozo de la diapositiva de la cámara, elija 4 áreas aleatorias para adquirir imágenes, utilizando el objetivo 4x para visualizar áreas más grandes.
NOTA: El objetivo 20x también se puede utilizar para las visualizaciones de imágenes. - Abra la imagen con el software ImageJ11. Aplicar Análisis ? Mida los comandos para cada imagen y utilice la ecuación en el paso 4.1.5 para cuantificar los datos.
NOTA: La cuantificación de las imágenes es ratiométrica; por lo tanto, el protocolo no incluye la resta de fondo. Sin embargo, para poder comparar imágenes, brillo, contraste y saturación debe ser el mismo para cada imagen. La significación estadística se evaluó con el análisis unidireccional de la varianza (ANOVA) y la prueba post hoc de Tukey.
Access restricted. Please log in or start a trial to view this content.
Resultados
El estado redox de CyS/CySS se ensaya fácilmente con roGPPs transducidos. La sonda fluorescente cuantifica la relación entre las formas reducidas y oxidadas (longitudes de onda de excitación 488 nm y 405 nm, respectivamente). Los datos de fluorescencia se pueden obtener mediante citometría de flujo y microscopía.
Un gran número de células se pueden adquirir de manera consistente y conveniente utilizando la citometría de flujo. El análisis consta de 3 pasos principales: 1) seleccionar ...
Access restricted. Please log in or start a trial to view this content.
Discusión
El equilibrio tiol/disulfuro en un organismo refleja el estado redox de las células. Los organismos vivos tienen glutatión, cisteína, tioles proteicos y tioles de bajo peso molecular, todos los cuales se ven afectados por el nivel de oxidación y se hacen eco del estado redox de las células4. Los roGRP diseñados permiten la cuantificación no disruptiva del equilibrio tiol/disulfuro a través de sus residuos CyS7. La propiedad ratiométrica de roGFP proporciona medicio...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El adenovirus constructor y recombinante para expresar roGFP específico de citosol en células se generó en el laboratorio de Paul T. Schumacker, PhD, Freiberg School of Medicine, Northwestern University y ViraQuest Inc., respectivamente. Este estudio fue apoyado por el Centro de Estudios de Respuesta Anfitriona a la Terapia del Cáncer subvención P20GM109005 a través del NIH National Institute of General Medical Sciences Centers of Biomedical Research Excellence (COBRE NIGMS), National Institute of General Medical Sciences Systems Pharmacology and Toxicology Training Program grant T32 GM106999, UAMS Foundation/Medical Research Endowment Award AWD00053956, UAMS Year-End Chancellor's Awards AWD00053484. La instalación del núcleo de citometría de flujo fue apoyada en parte por el Centro de Patogénesis Microbiana y Respuestas Inflamatorias anfitrionas otorgadaS P20GM103625 a través del COBRE NIGMS. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente las opiniones oficiales de los NIH. ATA fue apoyada por la beca 2214-A del Consejo de Investigación Científica y Tecnológica de Turquía (TUBITAK).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco by Life Sciences | 25200-056 | Cell culture |
| 4-well chamber slide | Thermo Scientific | 154526 | Cell seeding material for fluorescent imaging |
| 5 ml tubes with cell strainer cap | Falcon | 352235 | Single cell suspension tube for flow cytometry analysis |
| 6-well plate | Corning | 353046 | Cell seeding material for flow cytometry analysis |
| 15 ml conical tubes | MidSci | C15B | Cell culture |
| 75 cm2 ventilated cap tissue culture flasks | Corning | 4306414 | Cell culture |
| Adenoviral cytosol specific roGFP | ViraQuest | VQAd roGFP | roGFP construct kindly provided by Dr. Schumaker |
| Class II, Type A2 Safety Hood Cabinet | Thermo Scientific | 1300 Series A2 | Cell culture |
| Countess automated cell counter | Invitrogen | C10227 | Cell counting |
| Countess cell counter chamber slides | Invitrogen | C10283 | Cell counting |
| DMEM | Gibco by Life Sciences | 11995-065 | Cell culture |
| FBS | Atlanta Biologicals | S11150 | Cell culture |
| Filtered pipette tips, sterile, 20 µl | Fisherbrand | 02-717-161 | Cell culture |
| Filtered pipette tips, sterile, 1000 µl | Fisherbrand | 02-717-166 | Cell culture |
| Flow Cytometer | BD Biosciences | LSRFortessa | Instrument equipped with FITC and BV510 bandpass filters for flow cytometry analyses |
| Fluorescent Microscope | Advanced Microscopy Group (AMG) | Evos FL | Fluorescent imaging |
| Hydrogen Peroxide 30% | Fisher Scientific | H325-100 | Positive control |
| Light Cube, Custom | Life Sciences | CUB0037 | Fluorescent imaging of roGFP expressing cells (ex 405 nm) |
| Light Cube, GFP | Thermo Scientific | AMEP4651 | Fluorescent imaging of roGFP expressing cells (ex 488 nm) |
| MDA-MB-231 | American Tissue Culture Collection | HTB-26 | Human epithelial breast cancer cell line |
| Microcentrifuge tubes, 2 ml | Grenier Bio-One | 623201 | Cell culture |
| PBS | Gibco by Life Sciences | 10010-023 | Cell culture |
| Pipet controller | Drummond | Hood Mate Model 360 | Cell culture |
| Serologycal pipet, 1 ml | Fisherbrand | 13-678-11B | Cell culture |
| Serologycal pipet, 5 ml | Fisherbrand | 13-678-11D | Cell culture |
| Serologycal pipet, 10 ml | Fisherbrand | 13-678-11E | Cell culture |
| Tissue Culture Incubator | Thermo Scientific | HERACell 150i | CO2 incubator for cell culture |
| Trypan blue stain 0.4% | Invitrogen | T10282 | Cell counting |
Referencias
- Sies, H. Oxidative stress: A concept in redox biology and medicine. Redox Biology. 4, 180-183 (2015).
- Jones, D. P. Redefining Oxidative Stress. Antioxidants & Redox Signalling. 8 (9-10), (2006).
- Pizzino, G., et al. Oxidative Stress: Harms and Benefits for Human Health. Oxidative Medicine and Cellular Longevity. 20117, 8416763(2017).
- Go, Y. M., Jones, D. P. Thiol/disulfide redox states in signaling and sensing. Critical Reviews in Biochemistry and Molecular Biology. 48 (2), 173-191 (2013).
- Hansen, J. M., Go, Y., Jones, D. P. Nuclear and Mitochondrial Compartmentation of Oxidative Stress and Redox Signaling. Annual Review of Pharmacology and Toxicology. 46 (1), 215-234 (2006).
- Meyer, A. J., Dick, T. P. Fluorescent protein-based redox probes. Antioxidants and Redox Signaling. 13 (5), 621-650 (2010).
- Dooley, C. T., et al. Imaging dynamic redox changes in mammalian cells with green fluorescent protein indicators. Journal of Biological Chemistry. 279 (21), 22284-22293 (2004).
- Björnberg, O., Østergaard, H., Winther, J. R. Measuring intracellular redox conditions using GFP-based sensors. Antioxidants and Redox Signaling. 8 (3-4), 354-361 (2006).
- Bhaskar, A., et al. Reengineering Redox Sensitive GFP to Measure Mycothiol Redox Potential of Mycobacterium tuberculosis during Infection. PLoS Pathogens. 10 (1), 1003902(2014).
- Loor, G., et al. Mitochondrial oxidant stress triggers cell death in simulated ischemia-reperfusion. Biochimica et Biophysica Acta - Molecular Cell Research. 1813 (7), 1382-1394 (2011).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Loor, G., et al. Menadione triggers cell death through ROS-dependent mechanisms involving PARP activation without requiring apoptosis. Free Radical Biology and Medicine. 49 (12), 1925-1936 (2010).
- Esposito, S., et al. Redox-sensitive GFP to monitor oxidative stress in neurodegenerative diseases. Reviews in the Neurosciences. 28 (2), 133-144 (2017).
- Meyer, A. J., et al. Redox-sensitive GFP in Arabidopsis thaliana is a quantitative biosensor for the redox potential of the cellular glutathione redox buffer. Plant Journal. 52 (5), 973-986 (2007).
- Galvan, D. L., et al. Real-time in vivo mitochondrial redox assessment confirms enhanced mitochondrial reactive oxygen species in diabetic nephropathy. Kidney International. 92 (5), 1282-1287 (2017).
- Swain, L., Nanadikar, M. S., Borowik, S., Zieseniss, A., Katschinski, D. M. Transgenic organisms meet redox bioimaging: One step closer to physiology. Antioxidants and Redox Signaling. 29 (6), 603-612 (2018).
- Gutscher, M., et al. Proximity-based protein thiol oxidation by H2O2-scavenging peroxidases. Journal of Biological Chemistry. 284 (46), 31532-31540 (2009).
- Morgan, B., Sobotta, M. C., Dick, T. P. Measuring EGSH and H2O2 with roGFP2-based redox probes. Free Radical Biology and Medicine. 51 (11), 1943-1951 (2011).
- Dey, S., Sidor, A., O'Rourke, B. Compartment-specific control of reactive oxygen species scavenging by antioxidant pathway enzymes. Journal of Biological Chemistry. 291 (21), 11185-11197 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados