Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
3D Cell-Printed Hypoxic Cancer-on-a-Chip Para Recapitular la Progresión Patológica Del Cáncer Sólido
* Estos autores han contribuido por igual
En este artículo
Resumen
La hipoxia es un sello distintivo del microambiente tumoral y desempeña un papel crucial en la progresión del cáncer. Este artículo describe el proceso de la fabricación de un cáncer-en-una-viruta hipóxico basado en tecnología de célula-impresión 3D para recapitular una patología hipoxia-relacionada del cáncer.
Resumen
El microambiente del cáncer tiene un impacto significativo en la progresión de la enfermedad. Particularmente, la hipoxia es el conductor dominante de la supervivencia, de la invasión, y de la quimiorresistencia del cáncer. Aunque varios modelos ines vitro se hayan desarrollado para estudiar patología hipoxia-relacionada del cáncer, la interacción compleja del microambiente del cáncer observado in vivo no se ha reproducido todavía debido a la carencia del control espacial exacto. En cambio, se han propuesto enfoques de biofabricación 3D para crear sistemas microfisiológicos para una mejor emulación de la ecología del cáncer y una evaluación precisa del tratamiento contra el cáncer. Adjunto, proponemos un acercamiento de la célula-impresión 3D para fabricar un cáncer-en-un-viruta hipóxico. Los componentes hipoxia-que inducían en la viruta fueron determinados basados en una simulación de computadora de la distribución del oxígeno. los anillos concéntricos del Cáncer-tejido conectador fueron impresos usando los bioinks que contenían las células del glioblastoma y las células endoteliales para recapitular un tipo de cáncer sólido. La viruta resultante realizó hipoxia central y agravó malignidad en cáncer con la formación de marcadores patofisiológicos representativos. En general, se espera que el enfoque propuesto para crear un sistema microfisiológico sólido-cáncer-mimético cierre la brecha entre los modelos in vivo e in vitro para la investigación del cáncer.
Introducción
El microambiente del cáncer es un factor crítico que impulsa la progresión del cáncer. Los componentes múltiples, incluyendo señales bioquímicas, biofísicas, y celulares, determinan las características patológicas del cáncer. Entre éstos, la hipoxia se asocia fuertemente a supervivencia, a la proliferación, y a la invasión del cáncer1. Debido al crecimiento y a la división ilimitados de células cancerosas, los alimentos y el oxígeno se agotan continuamente, y se genera un gradiente hipóxico. En condiciones de bajo oxígeno, las células activan el factor de transcripción inducible por hipoxia (HIF)-cascada molecular asociada. Este proceso induce un núcleo necrótico, desencadena cambios metabólicos e inicia hiperplasia de vasos sanguíneos y metástasis2,3. Posteriormente, la hipoxia en las células cancerosas causa la destrucción de los tejidos normales vecinos. Además, la hipoxia se asocia fuertemente a la resistencia terapéutica de tumores sólidos de maneras multifactoriales. La hipoxia puede impedir gravemente la radioterapia, ya que la radiosensibilidad es limitada debido a las especies reactivas de oxígeno1,4. Además, disminuye los niveles de pH de los microambientes de cáncer, lo que disminuye la acumulación de fármacos1. Por lo tanto, la reproducción de las características patológicas relacionadas con la hipoxia in vitro es una estrategia prometedora para los resultados científicos y preclínicos.
Modelar un microambiente específico del cáncer es esencial para comprender el desarrollo del cáncer y explorar los tratamientos apropiados. Aunque los modelos animales han sido ampliamente utilizados debido a su fuerte relevancia fisiológica, existen cuestiones relacionadas con las diferencias de especies y problemas éticos5. Además, aunque los modelos 2D y 3D convencionales permiten la manipulación y la proyección de imagen en tiempo real de células cancerosas para un análisis profundizado, su complejidad arquitectónica y celular no se puede recapitular completamente. Por ejemplo, los modelos esferoides de cáncer han sido ampliamente utilizados, ya que la agregación de células cancerosas en un esferoide puede generar naturalmente hipoxia en el núcleo. Además, se ha producido un gran número de esferoides celulares de tamaño uniforme utilizando sistemas de múltiples pozos basados en plástico o silicona6,7. Sin embargo, la menor flexibilidad con respecto a la captura de la estructura heterogénea exacta de los tejidos cancerosos con plataformas convencionales ha requerido el establecimiento de una tecnología avanzada de biofabricación para construir una plataforma altamente biomimética para mejorar la investigación del cáncer8.
Los sistemas microfisiológicos 3D (MPS) son herramientas útiles para recapitular la geometría compleja y la progresión patológica de las células cancerosas9. Mientras que las células cancerosas detectan el gradiente bioquímico de factores de crecimiento y de chemokines y la heterogeneidad mecánica reproducida en el sistema, las características importantes del desarrollo del cáncer se pueden investigar in vitro. Por ejemplo, la viabilidad del cáncer, la malignidad metastática, y la resistencia a los medicamentos dependiendo de las concentraciones diversas del oxígeno se ha estudiado usandompss 10,11. A pesar de los avances recientes, la generación de condiciones hipóxicas de modelos in vitro se basa en procedimientos de fabricación complejos, incluida la conexión con bombas de gas físicas. Por lo tanto, se necesitan métodos simples y flexibles para construir microambientes específicos del cáncer.
La tecnología de impresión celular 3D ha ganado considerable atención debido a su control preciso de la disposición espacial de los biomateriales para recapitular arquitecturas biológicas nativas12. En particular, esta tecnología supera las limitaciones existentes de los modelos de hipoxia 3D debido a su alta controlabilidad y viabilidad para construir las características espaciales del microambiente del cáncer. La impresión 3D también facilita la fabricación asistida por computadora a través de un proceso capa por capa, proporcionando así una construcción rápida, precisa y reproducible de geometrías complejas para imitar arquitecturas de tejidos reales. Además de las ventajas de las estrategias de fabricación existentes para los MPS 3D, las características fisiopatológicas de la progresión del cáncer se pueden reproducir modelando los componentes bioquímicos, celulares y biofísicos13,14.
Aquí, presentamos una estrategia de impresión celular 3D para un cáncer hipóxico en un chip para recapitular la heterogeneidad de un cáncer sólido(Figura 1)15. Los parámetros de fabricación se determinaron mediante una simulación computacional de la formación de hipoxia central en el sistema. los anillos concéntricos del Cáncer-tejido conectador fueron impresos usando los bioinks del colágeno que contenían las células del glioblastoma y las células endoteliales para emular la patofisiología del glioblastoma, un tipo de cáncer sólido. La formación de un gradiente radial del oxígeno agravó malignidad del cáncer, indicando agresividad consolidada. Además, indicamos perspectivas futuras para las aplicaciones del chip a modelos preclínicos específicos del paciente. Se espera que el enfoque propuesto para crear un sistema microfisiológico sólido-cáncer-mimético cierre la brecha entre los modelos in vivo e in vitro de cáncer.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Simulación por computadora de la formación de gradiente de oxígeno
-
Generación de un modelo de geometría 3D para la impresión de cáncer hipóxico en un chip
- Ejecute un software CAD 3D.
- Esboce el modelo de geometría del cáncer hipóxico en un chip. Pulse en Esbozar (Sketch) y seleccione el plano deseado para dibujar la geometría. Consulte el dibujo (Figura 2A) para la escala de detalle de cada pieza.
- Establezca el grosor de la geometría pulsando en Característica-Protrusión Jefe/Base. Introduzca el espesor deseado (consulte la Figura 2A) en el cuadro vacío y seleccione el icono de verificación verde para formar la geometría 3D.
NOTA: La dimensión del cáncer-en-un-chip se define en base a los volúmenes deseados de medios e hidrogel. En el presente experimento, los volúmenes deseados de medios e hidrogel fueron de aproximadamente 1.500 μL y 500 μL, respectivamente, sobre la base de las experiencias prácticas previas para la resolución de la bioimpresora basada en extrusión. - Guarde el archivo de geometría como un formato de archivo CAD 3D (.prt o .stl).
-
Determinación de la densidad celular para la inducción del núcleo hipóxico
- Ejecute un programa de simulación de difusión física.
- Haga clic en LiveLink y seleccione el programa CAD utilizado. Haga clic en Sincronizar para importar la geometría del cáncer hipóxico en un chip en el programa de simulación. A medida que el espacio interior de la cámara se llenará con un medio de cultivo en un entorno experimental real, el oxígeno se difundirá a través del espacio interior de la cámara y la construcción celular, que se compondrán de hidrogeles cargados de células.
NOTA: Consulte el estudio anterior para obtener detalles sobre los parámetros físicos15. - Defina la geometría 3D importada como un volumen de control del espacio en el que el oxígeno se difunde, y las células consumen oxígeno (Figura 2B).
- Ejecutar un análisis informático para el análisis de difusión de gases siguiendo una guía del usuario y métodos previamenteestablecidos16,17.
- A partir de los resultados del análisis informático, exporte los datos de concentración de oxígeno estimados a través de la sección transversal A-A' en cada punto de tiempo siguiendo la guía del usuario. La ecuación gobernante se basa en la primera ley de Fick, expresada en Eq. (1) (Figura 2C).
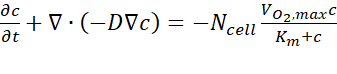
donde c es la concentración, D es el coeficiente de difusión de oxígeno, Ncélula es la densidad de las células,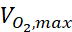 es la tasa máxima de absorción de oxígeno, y Km es la constante de Michaelis-Menten. Las constantes se aplicaron como se describe en una publicación anterior15.
es la tasa máxima de absorción de oxígeno, y Km es la constante de Michaelis-Menten. Las constantes se aplicaron como se describe en una publicación anterior15.
NOTA: Cada punto de tiempo significa un punto de paso para observar el cambio de difusión de oxígeno con el tiempo. - Evalúe si el nivel mínimo de oxígeno alcanza un umbral de hipoxia y repita el proceso de análisis informático con un incremento o decremento de la densidad celular.
NOTA: Definir que el gradiente de hipoxia se forma en el constructo si el nivel de oxígeno del 80% en el área de hidrogel es inferior a 0,02 mM después de 24 h. - Confirme el número de células necesarias para generar el gradiente de oxígeno que induce hipoxia en la región central a partir de la primera ley de Fick en el paso 1.2.5 y los resultados de la simulación del paso 1.2.6.
Nota : en este protocolo, número de celda era 2 × 106 celdas/cada construcción.
2. Cultivo celular de células cancerosas y células del estroma
-
Preparación de medios de cultivo celular para evitar el estrés fisiológico
- Para las células U-87 MG (línea celular inmortalizada de glioblastoma humano), coloque 12 ml de medio Eagle modificado de Dulbecco de alta glucosa que contenga 10% de suero bovino fetal, 100 U/mL de penicilina y 100 μg/mL de estreptomicina en un matraz de cultivo celular T-75 en una incubadora humidificada de 37 °C, 5% de CO2 durante 30 min para minimizar los efectos térmicos y alcalinos del medio en las células.
NOTA: El glioblastoma fue elegido como un tipo de cáncer sólido porque tiene características agresivas en un ambiente hipóxico. Se pueden aplicar otros tipos de cáncer a este modelo. - Para las células endoteliales de la vena umbilical humana (HUVECs), coloque 12 ml de medio de crecimiento de células endoteliales en un matraz de cultivo celular T-75 en una incubadora humidificada al 5% de CO2 a 37 °C durante 30 min.
NOTA: HuveCs fueron elegidos porque es una de las líneas celulares endoteliales más representativas. Varios tipos de células del estroma también se pueden aplicar a este modelo.
- Para las células U-87 MG (línea celular inmortalizada de glioblastoma humano), coloque 12 ml de medio Eagle modificado de Dulbecco de alta glucosa que contenga 10% de suero bovino fetal, 100 U/mL de penicilina y 100 μg/mL de estreptomicina en un matraz de cultivo celular T-75 en una incubadora humidificada de 37 °C, 5% de CO2 durante 30 min para minimizar los efectos térmicos y alcalinos del medio en las células.
-
Descongelación rápida de las células cancerosas criopreservadas y de las células del estroma y su mantenimiento
- Mueva los crioviales que contienen 5 x 105 células de MG U-87 y HUVECs del contenedor de nitrógeno líquido a un gabinete de flujo laminar. Afloje inmediatamente y vuelva a apretar la tapa para liberar la presión interna.
- Coloque suavemente las células criopreservadas en un baño de agua a 37 °C durante 2 min, manteniendo la tapa fuera del agua. Enjuague los viales con etanol al 70% bajo flujo laminar para evitar la contaminación.
- Transferir las células descongeladas a los matraces que contengan los medios de cultivo celular preparados descritos en el paso 2.1 y colocar los matraces que contienen células en una incubadora humidificada al 5% de CO2 a 37 °C para la recuperación celular.
- Actualice el medio de cultivo celular cada 2 días y mantenga el crecimiento celular.
- Después de 24 h de descongelación, substituya el medio de cultivo celular para evitar la citotoxicidad del sulfóxido de dimetilo (DMSO), que se utilizó para la congelación celular. Utilice HUVECs, que ha sufrido menos de 6 pasajes.
3. Preparación de la solución de colágeno pre-gel
-
Solubilización de la esponja de colágeno con ácido clorhídrico 0,1 N (HCl)
- Prepare una solución de 0,1 N HCl y filtre con un filtro de jeringa de 0,2 μm.
- Para 3 mL de una solución pre-gel de colágeno neutralizado al 1% (p/v), prepare esponjas de colágeno cortadas en 5 x 5 mm2 piezas y con un peso de 30 mg.
- Transfiera las piezas de colágeno cortadas a un vial de vidrio estéril de 10 ml.
NOTA: Preparar 1,5 veces el volumen del hidrogel de colágeno requerido, teniendo en cuenta la pérdida del hidrogel debido a la característica pegajosa de la solución de colágeno. - Añadir 2,4 ml de HCl de 0,1 N en el vial de vidrio que contiene colágeno e incubarlo en el balancín a 15 rpm y 4 °C durante 3 días.
NOTA: El volumen de la solución de HCl de 0,1 N fue de cuatro quintas partes del volumen final de hidrogel de colágeno requerido. En este caso, se prepararon 3 mL de colágeno. - Después de la digestión, tamice las partículas de colágeno no didigesadas usando un colador celular de 40 μm. Guarde la solución ácida de colágeno a 4 °C y úse dentro de los 7 días.
-
Ajuste del pH para la solución de pre-gel de colágeno neutralizado al 1%
- Centrifugar la solución ácida de colágeno a 1224 x g durante 5 min a 4 °C.
- Añadir 30 μL de solución de color rojo fenol como indicador de pH a una concentración final de 1% (v/v) y 300 μL de tampón salino tamponado con fosfato (PBS) 10x a una concentración final del 10% (v/v) en la solución de colágeno pre-gel.
- Neutralizar el pH a 7 con hidróxido de sodio de 1 N (NaOH), verificando el cambio de color.
NOTA: Con base en la fórmula, los lunares H+=molaridadH+x volumenH+=mols OH-=molaridadOH-x volumenOH-,añadimos 240 μL de NaOH. - Añadir agua destilada para obtener un volumen total de 3 mL.
- Después del ajuste del pH, guarde la solución de pre-gel de colágeno neutralizado al 1% (p/v) a 4 °C y úse dentro de los 3 días.
NOTA: Para comprobar previamente la gelificación de la solución de colágeno neutralizado pre-gel, haga gotitas de colágeno de 50 μL en un plato pequeño usando una pipeta de desplazamiento positivo e incubarlas en una incubadora de 37 °C durante 1 h. Consulte los tres métodos siguientes para verificar la reticulación de las gotitas de colágeno. - Compruebe si el color del colágeno ha cambiado a blanco opaco de color transparente.
- Incline el recipiente y compruebe si el colágeno está adherido a la parte inferior del recipiente.
- Vierta 1x PBS en las gotitas y verifique si la construcción de colágeno no está rota en la solución.
4.3D impresión de la barrera permeable al gas
-
Impresión 3D de un molde de poli (acetato de etileno-vinilo) (PEVA) de sacrificio
- Genere la geometría 3D del molde PEVA de sacrificio definido en el paso 1 utilizando un software CAD 3D (Figura 3A).
NOTA: La geometría 3D y la escala de modelo detallada, incluidas las dimensiones, unidades y tipos de línea, se mostraron en la Figura 2A. - Convierta el archivo CAD 3D en un formato de archivo STL haciendo clic en | Guardar tipo de archivo como STL. Además, haga clic en opción | Formulario de salida como ASCII para la generación de código G.
- Haga clic en | de archivos Abra el archivo STL y seleccione el archivo STL guardado para importar el archivo STL generado. Haga clic en Slice model of STL-CAD exchanger para generar automáticamente el código G del molde de PEVA de sacrificio(Figura 3B,C).
NOTA: La ruta de impresión se genera con la conexión de puntos intersecados entre la figura fundamental del archivo STL y el plano de corte (es decir, capa). Básicamente, la figura fundamental de un fragmento en un archivo STL es un triángulo que contiene las coordenadas 3D. Después de obtener los puntos intersecados entre el triángulo y la capa, se genera un código G para la impresión conectando cada punto sin un trazado superpuesto en una capa18. Cualquier algoritmo de generación de código G a bordo del software se puede utilizar para generar rutas de impresión para la fabricación del chip. - Preparar un adhesivo estéril y un portaobjetos de histología hidrofílica.
NOTA: El vidrio hidrofílico de la diapositiva es crítico para la unión permanente del polydimethylsiloxane (PDMS) en el cristal y la adherencia de las construcciones del colágeno que encapsulan las células cancerosas y las células stromal. - Imprima el molde de SACRIFICIO PEVA en la corredera con una boquilla de precisión de 50 G a una presión neumática de 500 kPa a 110 °C.
NOTA: El ancho de línea se ve afectado por la velocidad de avance, el medidor de boquilla y la temperatura del material. Se utilizó la boquilla de 50 G y se aplicó una velocidad de alimentación de 400 para generar un ancho de línea de 500 μm para la pared de sacrificio. El calibrador de la boquilla, la presión neumática y la velocidad de alimentación se definen con resultados prácticos19. La pared de sacrificio debe ser lo suficientemente gruesa como para sostener la solución PDMS, que es el siguiente paso de fabricación.
- Genere la geometría 3D del molde PEVA de sacrificio definido en el paso 1 utilizando un software CAD 3D (Figura 3A).
-
Barrera de fundición de polidimetilsiloxano (PDMS)
- Mezclar 6 mL de elastómero base PDMS y 0,6 mL de agente de curado homogéneamente durante 5 min en un depósito de plástico. Esto puede fabricar 6 cáncer-en-virutas hipóxicas, teniendo en cuenta la pérdida debido a la característica pegajosa de PDMS.
- Cargue la solución de PDMS mezclada en una jeringa desechable de 10 ml y coloque la cabeza de la jeringa con una punta de dispensación cónica de plástico de 20 G.
- Llene el molde de PEVA de sacrificio con la solución de PDMS mezclada en la jeringa. El PDMS mezclado llenará el molde de PEVA de sacrificio con una superficie convexa. La altura de la barrera PDMS será mayor que la del molde PEVA.
- Cure la barrera PDMS en un horno a 40 °C durante más de 36 h para evitar la fusión de PEVA. No aumente la temperatura a más de 88 °C, que es la temperatura de fusión de PEVA.
- Suelte el molde de PEVA de sacrificio con un par de pinzas de precisión y esterilice la barrera permeable al gas a 120 °C en un autoclave.
5. Preparación de bio-tintas de colágeno encapsuladas en células
-
Desprendimiento de las células cancerosas preparadas y las células del estroma
NOTA: Teniendo en cuenta la viabilidad de la célula, todo el proceso de impresión debe completarse tan pronto como sea posible después de separar las celdas.- Lave el cáncer y las células del estroma con 10 mL de 1x PBS usando una pipeta serológica; tratar con 2 mL de ácido tripsina-etilendiaminotetraacético al 0,25% utilizando una pipeta e incubarlos durante 3 min a 37 °C.
- Neutralizar las células tripsinizadas con 3 mL de medios de cultivo celular; recoger las suspensiones de las células en tubos cónicos de 15 mL y centrífuga a 516 x g durante 5 min a 20 °C.
- Aspirar el sobrenadante lentamente; resuspend las pelotillas de la célula en medios del cultivo celular de 5 mL y cuente el número de células usando un hemocytometer.
- Transferir 5 x 106 células de cada tipo de célula en nuevos tubos cónicos de 15 mL y centrifugarlos a 516 x g durante 5 min a 20 °C.
- Aspirar el sobrenadante y colocarlo sobre hielo húmedo.
-
Mezcla de cada tipo de célula con la solución de pre-gel de colágeno neutralizado al 1%
NOTA: Para evitar la solidificación térmica de la solución pre-gel de colágeno neutralizado al 1%, este proceso debe realizarse sobre hielo húmedo.- Resuspend cada tipo de pellet celular recogido en el paso 5.1.4 con 20 μL de medio de cultivo celular cada uno.
- Añadir 1 mL de la solución pre-gel de colágeno neutralizado al 1% en cada una de las suspensiones celulares resuspended y mezclarlas homogéneamente usando una pipeta de desplazamiento positivo. La concentración final de cada tipo de célula será de 5 x 106 células/mL.
- Transfiera los bioinks de colágeno encapsulados en células en jeringas desechables de 3 ml usando una pipeta desechable positiva y guarde las jeringas a 4 °C hasta la impresión celular 3D.
6.3D la célula-impresión de los anillos concéntricos del cáncer-tejido conectador
-
Impresión celular 3D de bioinks de colágeno que encapsulan las células cancerosas y las células del estroma
- Genere la geometría 3D de los anillos concéntricos del cáncer-estroma definidos en el paso 1.2 usando un software 3D cad.
NOTA: Las dimensiones de los anillos concéntricos del estroma del cáncer se definen vía parámetros simulados. Las dimensiones finales del parámetro de dimensión se muestran en la Figura 3A. - Convierta el archivo CAD 3D en un formato de archivo STL y genere un código G de los anillos concéntricos de cáncer-estroma utilizando un intercambiador STL-CAD.
Nota : consulte la nota en el paso 4.1.2 para el algoritmo de generación de código G. - Cargue los bioinks de colágeno encapsulados en células contenidos en jeringas desechables de 3 ml en la cabeza de la impresora 3D y ajuste la temperatura de la cabeza y la placa a 15 °C.
NOTA: Si la temperatura del cabezal y la placa de la impresora alcanza más de 37 °C, el bioink se reticulación y ya no imprime. - Cargue la ruta de impresión generada en el software de control de la impresora 3D.
- Al hacer clic en el botón Inicio, imprima los bioinks de colágeno que encapsulan las células cancerosas y las células del estroma en la barrera permeable al gas siguiendo el código G cargado con una aguja de plástico de 18 G a una presión neumática de aproximadamente 20 kPa a 15 °C.
- Al final de cada operación de impresión, coloque manualmente una cubierta de vidrio esterilizada de 22 mm x 50 mm en la parte superior de la barrera permeable al gas para generar el gradiente hipóxico.
NOTA: Comparar dos grupos en función de la presencia de cubierta de vidrio (GR+) y ausencia (GR-) de que para verificar la generación del gradiente hipóxico. - Después de generar tres cáncer hipóxico en chips, transfiera los chips a una incubadora a 37 °C durante 1 h para reticenar los bioinks de colágeno.
- Genere la geometría 3D de los anillos concéntricos del cáncer-estroma definidos en el paso 1.2 usando un software 3D cad.
-
Finalización del proceso de fabricación y mantenimiento del cáncer hipóxico en un chip
- Después de completar todos los procesos de impresión celular 3D del cáncer hipóxico en un chip, frote suavemente las gafas de cubierta en la parte superior de las barreras permeables al gas con el raspador de células para una unión estrecha (Figura 4A,B).
NOTA: El vidrio de la cubierta y la barrera permeable al gas se ensamblan a través de la unión hidrofóbica sin pegamentos químicos, simplemente raspando la parte unida entre el vidrio de la cubierta y la barrera PDMS. - Introduzca 1,5 mL de medio de crecimiento de células endoteliales en cada chip. Para evitar el desprendimiento de la construcción del cáncer, introduzca el medio de cultivo celular de un lado del chip. Incline el chip para permitir que el medio de cultivo celular fluya usando una pipeta.
- Actualice los medios de cultivo celular todos los días durante una semana. Utilice una pipeta para aspirar el medio de cultivo celular; no utilice una bomba de presión.
- Después de completar todos los procesos de impresión celular 3D del cáncer hipóxico en un chip, frote suavemente las gafas de cubierta en la parte superior de las barreras permeables al gas con el raspador de células para una unión estrecha (Figura 4A,B).
7. Evaluación de la viabilidad de las células postimpresión
-
Preparación de muestras y tratamiento con calceína AM y solución EthD-1
- Caliente 1x PBS en un baño de agua a 37 °C.
- Preparar la solución de ensayo añadiendo 0,75 μL de calceína acetoximetil (calceína AM) y 3 μL de etidio homodímero (EthD-1) a 1,5 mL de PBS precalentado.
- Aspirar cuidadosamente todos los medios del chip usando una pipeta.
- Lave la construcción del cáncer con PBS preguerra. Llene 1,5 mL de PBS en el chip con una pipeta y déjelo reposar durante 10 minutos a temperatura ambiente. Para evitar la deformación de la construcción del cáncer, introduzca 1x PBS de un lado de las virutas e incline las virutas para permitir que 1x PBS fluya.
- Aspirar el PBS desde el chip; tratar la solución de ensayo de 1,5 mL e incubar el chip a 37 °C durante 20 min utilizando una lámina para protegerla de la luz. Utilice una pipeta para aspirar 1x PBS; no utilice una bomba de succión.
-
Imágenes de la viabilidad celular utilizando un microscopio de fluorescencia
- Ver y capturar las células etiquetadas utilizando un microscopio de fluorescencia (Figura 4C).
NOTA: Calcein AM marca las células vivas con fluorescencia verde (longitud de onda ~ 488 nm). EthD-1 representa la señal de células muertas con fluorescencia roja (longitud de onda ~ 594 nm). - Cuente el número de células vivas y muertas utilizando un software de imágenes, un programa de procesamiento de imágenes de código abierto, y calcule la viabilidad con los números.
- Ver y capturar las células etiquetadas utilizando un microscopio de fluorescencia (Figura 4C).
8. Inmunofluorescencia para validar la formación de hipoxia central y su efecto sobre la malignidad del cáncer
-
Fijación, permeabilización y bloqueo de la construcción del cáncer
- Prepare 1x PBS, paraformadehído (PFA) del 4%, tritón X-100 del 0,1% (v/v) y al 2% (p/v) de albúmina de suero bovino (BSA) en temperatura ambiente.
- Aspire cuidadosamente todos los medios del chip usando una pipeta y enjuague el chip tres veces con 1x PBS. Para evitar la deformación de la construcción del cáncer, introduzca 1x PBS de un lado de las virutas e incline las virutas para permitir que 1x PBS fluya. Entre cada paso de lavado, deje reposar el chip con 1x PBS durante 5 min para eliminar las soluciones residuales.
NOTA: 1x PBS fue aspirado usando una pipeta, no una bomba de presión. - Añadir 500 μL de 4% de PFA a la construcción del cáncer en el chip usando una pipeta; déjelo durante 15 minutos y lave tres veces con 1x PBS para fijar las células en la construcción del cáncer.
- Tratar la construcción del cáncer con 500 μL de Triton X-100 al 0,1% utilizando una pipeta a temperatura ambiente durante 5 min y lavar tres veces con 1x PBS para solubilizar y permeabilizar la membrana celular.
- Tratar el cáncer de construcción con 500 μL de BSA al 2% utilizando una pipeta a temperatura ambiente durante 1 h para bloquear los epítopos reactivos.
NOTA: Cubra el chip con película de parafina para evitar la evaporación. - Después de 1 h, lave el chip tres veces con 1x PBS.
-
Tratamiento con el anticuerpo primario, el anticuerpo secundario, y DAPI y la proyección de imagen de la estructura usando un microscopio confocal.
- Preparar anticuerpos de control de isotipo y el cóctel de anticuerpos primarios diluyendo los anticuerpos en 1x PBS a cada concentración de trabajo deseada.
NOTA: Los detalles específicos de los anticuerpos se enumeran en la Tabla de Materiales. Se deben utilizar las mismas concentraciones de trabajo de anticuerpos de control de isotipo que los anticuerpos primarios. - Aspire cuidadosamente todo 1x PBS del chip usando una pipeta y trate el chip con una solución de anticuerpo primario de 200 μL a 4 °C durante la noche. Cubra las virutas con la película de la parafina para prevenir la evaporación.
- Aspirar la solución de anticuerpos primarios y lavar el chip tres veces con 1x PBS.
- Diluir anticuerpos secundarios y DAPI en 1x PBS a la concentración de trabajo deseada.
NOTA: Un anticuerpo secundario conjugado con fluorescencia verde se utiliza en este caso en una proporción de 1:200. Dapi se utilizó en una proporción de 1:1000. - Aspire cuidadosamente todo 1x PBS del chip usando una pipeta y trate el chip con una solución de anticuerpos secundarios-DAPI de 200 μL a 4 °C durante 3 h. Cubra el chip con película de parafina para evitar la evaporación y luego envuélvalo con papel de aluminio para evitar el fotoblanqueo.
- Aspire la solución secundaria de anticuerpos-DAPI y lave el chip tres veces con 1x PBS.
- Después de terminar el paso de tinción, transfiera la construcción del cáncer a un plato confocal agarrándose suavemente con fórceps.
- Visualizar y capturar las células etiquetadas utilizando un microscopio confocal (Figura 5).
NOTA: La longitud de onda del microscopio confocal se ajustó, dependiendo del tipo de marcadores fluorescentes. Los detalles específicos de los anticuerpos se enumeran en la Tabla de Materiales. Para detectar eficientemente la posición celular, sería mejor observar los núcleos teñidos dapi de la construcción al principio. Las longitudes de onda de excitación/emisión de detección de las señales fluorescentes fueron 358/461 nm (DAPI, Azul), 494/517 nm (Verde) y 590/617 nm (Rojo). Los aumentos fueron 4x, 10x y 20x, ajustados de menor a mayor.
- Preparar anticuerpos de control de isotipo y el cóctel de anticuerpos primarios diluyendo los anticuerpos en 1x PBS a cada concentración de trabajo deseada.
9. Análisis estadístico
-
Conteo de celdas con programa de procesamiento de imágenes
- Ejecute un programa de procesamiento de imágenes para contar el número de celdas vivas y muertas.
- Abra los archivos de imagen fluorescente. Haga clic en | de archivos Abra e importe las imágenes TIFF.
- Convierta las imágenes en imágenes en escala de grises de 16 bits. Haga clic en | de imagen Tipo | Escala de grises de 16 bits.
- Ajuste el umbral haciendo clic en | de imagen Ajustar | Umbral y, a continuación, seleccione el color de las celdas para que sea negro.
- Corte las celdas fusionadas haciendo clic en Procesar | | binario Cuenca hidrográfica para un recuento preciso de celdas.
- Cuente el número de celdas haciendo clic en Analizar y luego en Analizar partículas tres veces; calcular el promedio y presentar los datos como la media ± error estándar.
NOTA: Los marcadores de inmunofluorescencia se analizaron mediante la comparación de la intensidad de fluorescencia.
Access restricted. Please log in or start a trial to view this content.
Resultados
El cáncer hipóxico en un chip se desarrolló utilizando tecnología de impresión celular 3D asistida por computadora para recapitular la hipoxia y la patología relacionada con el cáncer (Figura 1). El transporte y el consumo de oxígeno se simularon utilizando el modelo de geometría 3D. El chip fue diseñado en forma de anillos concéntricos para imitar la difusión y depleción radial de oxígeno, en los tejidos cancerosos(Figura 2...
Access restricted. Please log in or start a trial to view this content.
Discusión
En este estudio, describimos el proceso de fabricación de un cáncer-en-un-viruta hipóxico basado en tecnología de la célula-impresión 3D. La formación del gradiente hipóxico en el chip diseñado se predijo a través de simulaciones por computadora. El ambiente que puede inducir un gradiente hipóxico heterogéneo se reprodujo a través de una estrategia simple que combina la barrera permeable al gas impresa en 3D y la cubierta de vidrio. Las características patológicas hipoxia-relacionadas del glioblastoma, inc...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen revelaciones.
Agradecimientos
Esta investigación fue apoyada por la Fundación Nacional de Investigación de Corea (NRF) financiada por el Ministerio de Educación (No. 2020R1A6A1A03047902 y NRF-2018H1A2A1062091) y el gobierno de Corea (MSIT) (No. NRF-2019R1C1C1009606 y NRF-2019R1A3A3005437).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Cells | |||
| Human umbilical vein endothelial cells | Promocell | C-12200 | |
| U-87 MG cells | ATCC | ATCC HTB-14 | |
| Disposable | |||
| 0.2 μm syringe filter | Sartorius | 16534-K | |
| 10 mL disposable syringe | Jung Rim | 10ml 21G32 | |
| 10 mL glass vial | Hubena | A0039 | |
| 10 mL Serological pipette tip | SPL lifescience | 91010 | |
| 15 mL conical tube | SPL lifescience | 50015 | |
| 18G plastic needle | Musashi engineering | PN-18G-B | |
| 20G plastic tapered dispense tip | Musashi engineering | TPND-20G-U | |
| 22x50 glass cover | MARIENFIELD | 0101142 | |
| 25 mL Serological pipette tip | SPL lifescience | 90125 | |
| 3 mL disposable syringes | HENKE-JET | 4020-X00V0 | |
| 40 µm cell strainer | Falcon | 352360 | |
| 5 mL Serological pipette tip | SPL lifescience | 91005 | |
| 50 mL conical tube | SPL lifescience | 50050 | |
| 50 mL Serological pipette tip | SPL lifescience | 90150 | |
| 50N precision nozzle | Musashi engineering | HN-0.5ND | |
| Aluminum foil | SINKWANG | ||
| Capillary tips | Gilson | CP1000 | |
| Cell-scrapper | SPL lifescience | 90030 | |
| Confocal dish | SPL lifescience | 200350 | |
| Parafilm | Bemis | PM996 | |
| Pre-coated histology slide | MATSUNAMI | MAS-11 | |
| Reservoir | SPL lifescience | 23050 | |
| T-75 cell culture flask | SPL lifescience | 70075 | |
| Equipment | |||
| 3DX printer | T&R Biofab | ||
| Autoclave | JEIOTECH | AC-12 | |
| Centrifuger | Cyrozen | 1580MGR | |
| Confocal laser microscopy | Olympus Life Science | FV 1000 | |
| Fluorescence microscope | FISHER SCEINTIFIC | O221S366 | |
| Forcep | Korea Ace Scientific | HC.203-30 | |
| Hand tally counter | KTRIO | ||
| Hemocytometer | MARIENFIELD | 0650030 | |
| Incubator | Panasonic | MCO-170AIC | |
| Laminar flow cabinet | DAECHUNG SCIENCE | CB-BMMS C-001 | |
| Metal syringe | IWASHITA engineering | SUS BARREL 10CC | |
| Operating Scissors | Hirose | HC.13-122 | |
| Oven | JEIOTECH | OF-12, H070023 | |
| Positive displacement pipette | GILSON | NJ05652 | |
| Refrigerator | SAMSUNG | CRFD-1141 | |
| Voltex Mixer | DAIHAN scientific | VM-10 | |
| Water bath | DAIHAN SCIENTIFIC | WB-11 | |
| Water purifier | WASSER LAB | DI-GR | |
| Materials | |||
| 0.25 % Trypsin-EDTA | Gibco | 25200-072 | |
| 10x PBS | Intron | IBS-BP007a | |
| 4% Paraformaldehyde | Biosesang | ||
| 70% Ethanol | Daejung | 4018-4410 | |
| Anti-CD31 antibody | Abcam | ab28364 | |
| Anti-HIF-1 alpha antibody | Abcam | ab16066 | |
| Anti-SHMT2/SHMT antibody | Abcam | ab88664 | |
| Anti-SOX2 antibody | Abcam | ab75485 | |
| Bovine Serum Albumin | Thermo scientific | J10857-22 | |
| Collagen from porcine skin | Dalim tissen | PC-001-1g | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Thermofisher | D1306 | |
| Endothelial Cell Growth Medium-2 | Promocell | C22011 | |
| Fetal bovine serum | Gibco | 12483-020 | |
| Goat anti-Mouse IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 488 | Theromofisher | A-11001 | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Alexa Fluor 594 | Theromofisher | A-11012 | |
| High-glucose Dulbecco’s Modified Eagle Medium(DMEM) | Hyclone | SH30243-0 | |
| Hydrochloric acid | Sigma-Aldrich | 311413-100ML | |
| Live/dead assay kit | Invitrogen | L3224 | |
| Mouse IgG1, kappa monoclonal [15-6E10A7] - Isotype Control | Abcam | ab170190 | |
| Penicillin/streptomycin | Gibco | 15140-122 | |
| Phenol red solution | Sigma-Aldrich | P0290-100ML | |
| Poly(ethylene-vinyl acetate) | Poly science | 06108-500 | |
| Polydimethylsiloxane | Dowhitech | sylgard 184 | |
| Rabbit IgG, polyclonal - Isotype Control | Abcam | ab37415 | |
| Sodium hydroxide solution | Samchun | S0610 | |
| Triton X-100 | Biosesang | TRI020-500-50 | |
| Trypan Blue | Sigma-Aldrich | T8154 | |
| Software | |||
| COMSOL Multiphysics 3.5a | COMSOL AB | ||
| IMS beamer | in-house software | ||
| SolidWorks Package | Dassault Systems SolidWorks Corporation |
Referencias
- Jing, X., et al. Role of hypoxia in cancer therapy by regulating the tumor microenvironment. Molecular Cancer. 18 (1), 157(2019).
- Al Tameemi, W., Dale, T. P., Al-Jumaily, R. M. K., Forsyth, N. R. Hypoxia-modified cancer cell metabolism. Frontiers in Cell and Developmental Biology. 7, 4(2019).
- Petrova, V., Annicchiarico-Petruzzelli, M., Melino, G., Amelio, I. The hypoxic tumour microenvironment. Oncogenesis. 7 (1), 1-13 (2018).
- Hockel, M., Vaupel, P. Tumor hypoxia: definitions and current clinical, biologic, and molecular aspects. Journal of the National Cancer Institute. 93 (4), 266-276 (2001).
- Kim, H., Lin, Q., Glazer, P. M., Yun, Z. The hypoxic tumor microenvironment in vivo selects the cancer stem cell fate of breast cancer cells. Breast Cancer Research. 20 (1), 16(2018).
- Jeong, G. S., Lee, J., Yoon, J., Chung, S., Lee, S. -H. Viscoelastic lithography for fabricating self-organizing soft micro-honeycomb structures with ultra-high aspect ratios. Nature Communications. 7 (1), 1-9 (2016).
- Razian, G., Yu, Y., Ungrin, M. Production of large numbers of size-controlled tumor spheroids using microwell plates. Journal of Visualized Experiments:JoVE. (81), e50665(2013).
- Nunes, A. S., Barros, A. S., Costa, E. C., Moreira, A. F., Correia, I. J. 3D tumor spheroids as in vitro models to mimic in vivo human solid tumors resistance to therapeutic drugs. Biotechnology and Bioengineering. 116 (1), 206-226 (2019).
- Wan, L., Neumann, C., LeDuc, P. Tumor-on-a-chip for integrating a 3D tumor microenvironment: chemical and mechanical factors. Lab on a Chip. 20 (5), 873-888 (2020).
- Nam, H., Funamoto, K., Jeon, J. S. Cancer cell migration and cancer drug screening in oxygen tension gradient chip. Biomicrofluidics. 14 (4), 044107(2020).
- Palacio-Castañeda, V., Kooijman, L., Venzac, B., Verdurmen, W. P., Le Gac, S. Metabolic switching of tumor cells under hypoxic conditions in a tumor-on-a-chip model. Micromachines. 11 (4), 382(2020).
- Ronaldson-Bouchard, K., Vunjak-Novakovic, G. Organs-on-a-chip: a fast track for engineered human tissues in drug development. Cell Stem Cell. 22 (3), 310-324 (2018).
- Mi, S., Du, Z., Xu, Y., Sun, W. The crossing and integration between microfluidic technology and 3D printing for organ-on-chips. Journal of Materials Chemistry B. 6 (39), 6191-6206 (2018).
- Yi, H. -G., Lee, H., Cho, D. -W. 3D printing of organs-on-chips. Bioengineering. 4 (1), 10(2017).
- Yi, H. -G., et al. A bioprinted human-glioblastoma-on-a-chip for the identification of patient-specific responses to chemoradiotherapy. Nature Biomedical Engineering. 3 (7), 509-519 (2019).
- Kang, T. -Y., Hong, J. M., Jung, J. W., Yoo, J. J., Cho, D. -W. Design and assessment of a microfluidic network system for oxygen transport in engineered tissue. Langmuir. 29 (2), 701-709 (2013).
- Woo Jung, J., et al. Evaluation of the effective diffusivity of a freeform fabricated scaffold using computational simulation. Journal of Biomechanical Engineering. 135 (8), (2013).
- Brown, A. C., De Beer, D. Development of a stereolithography (STL) slicing and G-code generation algorithm for an entry level 3-D printer. 2013 Africon (IEEE). , 1-5 (2013).
- Shim, J. -H., Lee, J. -S., Kim, J. Y., Cho, D. -W. Bioprinting of a mechanically enhanced three-dimensional dual cell-laden construct for osteochondral tissue engineering using a multi-head tissue/organ building system. Journal of Micromechanics and Microengineering. 22 (8), 085014(2012).
- Gillispie, G., et al. Assessment methodologies for extrusion-based bioink printability. Biofabrication. 12 (2), 022003(2020).
- Kim, B. S., Das, S., Jang, J., Cho, D. -W. Decellularized extracellular matrix-based bioinks for engineering tissue-and organ-specific microenvironments. Chemical Reviews. 120 (19), 10608-10661 (2020).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados