Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes en vivo de alto rendimiento de microcolonias para medir la heterogeneidad en crecimiento y expresión génica
En este artículo
Resumen
Los fenotipos de crecimiento de levadura se miden con precisión a través de imágenes de lapso de tiempo altamente paralelas de células inmovilizadas que crecen en microcolonías. Simultáneamente, se puede monitorear la tolerancia al estrés, la expresión proteica y la localización de proteínas, generando conjuntos de datos integrados para estudiar cómo las diferencias ambientales y genéticas, así como la heterogeneidad de la expresión génica entre las células isogénicas, modulan el crecimiento.
Resumen
Las mediciones precisas de la heterogeneidad entre cepas y dentro de la cepa en las tasas de crecimiento microbiano son esenciales para comprender los insumos genéticos y ambientales en la tolerancia al estrés, la patogenicidad y otros componentes clave de la aptitud física. Este manuscrito describe un ensayo basado en microscopios que rastrea aproximadamente 10 microcoloníassaccharomyces cerevisiae por experimento. Después de la toma automatizada de imágenes de lapso de tiempo de levadura inmovilizadas en una placa multipobajo, las tasas de crecimiento de microcolonía se analizan fácilmente con software de análisis de imágenes personalizado. Para cada microcolonía, también se puede controlar la expresión y localización de proteínas fluorescentes y la supervivencia del estrés agudo. Este ensayo permite una estimación precisa de las tasas de crecimiento promedio de las cepas, así como una medición integral de la heterogeneidad en el crecimiento, la expresión génica y la tolerancia al estrés dentro de las poblaciones clonales.
Introducción
Los fenotipos de crecimiento contribuyen críticamente a la aptitud de la levadura. La selección natural puede distinguir eficientemente entre linajes con tasas de crecimiento diferentes por la inversa del tamaño efectivo de la población, que puede superar los 108 individuos1. Además, la variabilidad de las tasas de crecimiento entre los individuos dentro de una población es un parámetro relevante para la evolución, ya que puede servir como base para estrategias de supervivencia como la cobertura de apuestas2,3,4,5,6. Por lo tanto, los ensayos que permiten mediciones altamente precisas de fenotipos de crecimiento y sus distribuciones son fundamentales para el estudio de microorganismos. El ensayo de crecimiento de microcolonía descrito aquí puede generar mediciones individuales de la tasa de crecimiento para ~105 microcolonies por experimento. Por lo tanto, este ensayo proporciona un poderoso protocolo para estudiar la genética evolutiva y la genómica de la levadura. Se presta particularmente bien para probar cómo se genera, mantiene y contribuye a la variabilidad dentro de las poblaciones de células individuales genéticamente idénticas, y contribuye a la aptitud de la población7,8,9,10.
El método descrito aquí (Figura 1) utiliza imágenes de campo brillante capturadas periódicamente y de baja ampliación de células que crecen en medios líquidos en una placa inferior de vidrio de 96 o 384 pozos para rastrear el crecimiento en microcolonías. Las células se adhieren a la lectina concanavalina A, que recubre la parte inferior de la placa del microscopio, y forman colonias bidimensionales. Debido a que las microcolonías crecen en una monocapa, el área de microcolonía está altamente correlacionada con el número de celda7. Por lo tanto, se pueden generar estimaciones precisas de la tasa de crecimiento de la microcolonía y el tiempo de retraso con software de análisis de imágenes personalizado que realiza un seguimiento de la tasa de cambio del área de cada microcolonía. Además, la configuración experimental puede monitorear las abundancias e incluso las localizaciones subcelulares de proteínas etiquetadas fluorescentemente expresadas en estas microcolonías. El procesamiento descendente de los datos de este ensayo de crecimiento de microcolonía se puede lograr mediante análisis personalizado o mediante software de análisis de imágenes existente, como Procesamiento de imágenes fácilmente (PIE)11, un algoritmo para el reconocimiento robusto del área de colonias y un análisis de crecimiento de alto rendimiento a partir de imágenes de bajo aumento y campo brillante, que está disponible a través de GitHub12.
Debido a que las estimaciones de la tasa de crecimiento derivadas del ensayo de crecimiento de microcolonía se generan a partir de un gran número de mediciones de una sola colonia, son extremadamente precisas, con errores estándar varios órdenes de magnitud más pequeños que las propias estimaciones para un experimento de tamaño razonable. Por lo tanto, el poder del ensayo para detectar diferencias de ritmo de crecimiento entre diferentes genotipos, tratamientos o condiciones ambientales es alto. El formato multiwell-plate permite comparar numerosas combinaciones diferentes de entornos y genotipos en un solo experimento. Si las cepas expresan constitutivamente diferentes marcadores fluorescentes, pueden mezclarse en el mismo pozo y distinguirse por un análisis de imagen posterior, lo que podría aumentar aún más la potencia al permitir una normalización de los datos bien por pozo.
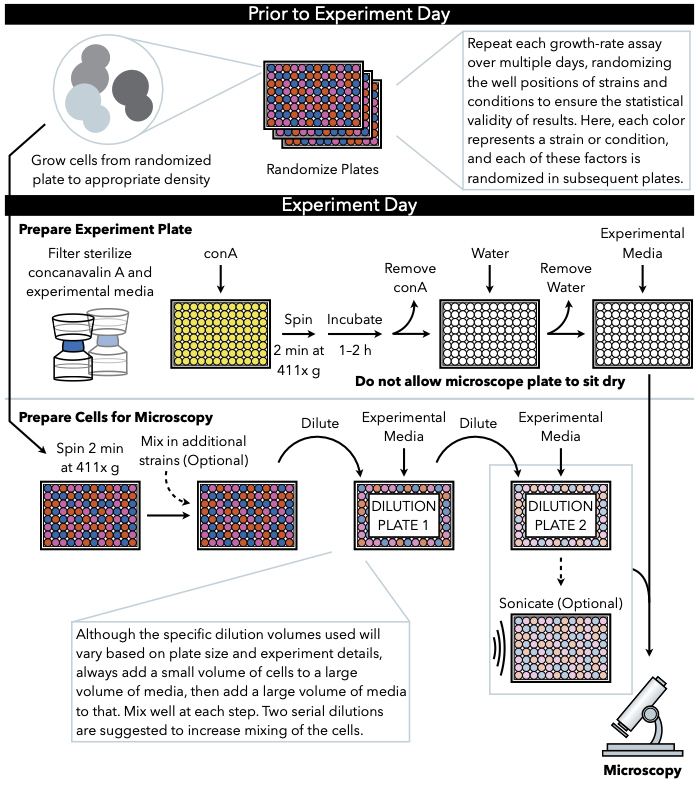
Figura 1: Representación esquemática del protocolo. Este protocolo sigue dos pasos principales, que son la preparación de la placa experimental y la preparación de las células para la imagen. La aleatorización de las placas y el crecimiento de las células deben llevarse a cabo antes y antes del día del experimento. La mezcla repetida de células en cada paso durante la dilución es imperativa en los pasos hasta el chapado, y por lo tanto se recomienda primero preparar la placa experimental para que esté lista para el chapado inmediatamente después de la finalización de la dilución celular. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Preparación de placas aleatorizadas (antes del día del experimento)
- Planifique las cepas y condiciones a probar con el ensayo de crecimiento. En este punto, asigne aleatoriamente cepas y condiciones a cualquier pozo.
NOTA: Al considerar la configuración de la placa, es recomendable incluir más de una réplica por cepa y condición de crecimiento en una sola placa para tener en cuenta el ruido bien relacionado en las mediciones. Consulte Discusión para obtener más detalles. - Aleatoriza computacionalmente la ubicación de cada cepa y condición ambiental para las réplicas de placas que se ejecutarán en diferentes días.
- Cultivar todas las células que se utilizarán en el experimento a la saturación en extracto de levadura-peptona-dextrosa (YEPD; 2% glucosa) medio en un agitador a 30 °C (o cualquier otra temperatura apropiada).
- Cree las placas de stock aleatorias manualmente o con un robot de manipulación de líquidos. Agregue 10 μL de las células saturadas designadas a cada pozo de una placa estéril de cultivo de tejido de fondo en U. Si se probarán varias cepas en un solo pozo, no las combine en este momento; esta combinación se realizará justo antes de las diluciones celulares el día del experimento para asegurar que todas las cepas estén en las concentraciones correctas cuando estén chapadas como células fundadoras de microcolonía.
- Añadir 10 μL de 30% de glicerol a cada pozo de cada plato. Pipetear hacia arriba y hacia abajo para que las células y el glicerol se mezclen bien.
- Selle cada placa con una cubierta de papel de aluminio y congele inmediatamente a -70 °C hasta que esté lista para usar.
NOTA: Es importante crear todas las placas aleatorizadas el mismo día, y congelarlas, de modo que las condiciones previas al crecimiento de las células en cada placa habrán sido idénticas y no generarán variación técnica en el ensayo de tasa de crecimiento.
2. Pre-Crecimiento de levadura
NOTA: Normalmente, esto comienza antes del día del experimento y depende en gran medida de la pregunta experimental. Consulte Discusión para obtener más información.
- Retire una placa de caldo (levadura de 10 μL, 10 μL de glicerol por pozo) del congelador -70 °C y agregue 180 μL del medio que se utilizará para el experimento. Si el experimento se llevará a cabo utilizando medios que limitan los nutrientes, no prefaere la levadura a la saturación en los medios que limitan los nutrientes, ya que puede ocurrir esporádica de levadura. En su lugar, pre-crecer en medios no limitantes.
- Cultivar levadura mientras tiembla a 30 °C. Considere si se debe ejecutar el ensayo a partir de celdas en fase de registro o en fase estacionaria para determinar si será necesario diluir las células varias veces antes del experimento. Si se espera que las cepas o condiciones de levadura en el ensayo tengan tasas de crecimiento significativamente diferentes, entonces será necesario un período de pre-crecimiento de dos días para que todas las diferentes condiciones lleguen a la fase estacionaria.
3. Configuración del microscopio
- Preparación de placas de microscopio
- Asegúrese de que la incubadora de microscopios esté encendida y calente la cámara del microscopio a la temperatura de crecimiento deseada para las condiciones experimentales. Para experimentos estándar utilizando células cerevisias Saccharomyces, la incubadora debe calentar la cámara del microscopio a 30 °C para asegurarse de que las condiciones de crecimiento de las células serán correctas durante el ensayo de la tasa de crecimiento.
- Desinfecte la mesa de trabajo, las pipetas y otras herramientas con un 70% de etanol. Recupera una placa de microscopio y colóquela en el banco encima de una toallita sin pelusas y estáticas.
NOTA: Nunca toque la parte inferior de la placa del microscopio, incluso con guantes puestos, y siempre coloque la placa del microscopio en la parte superior de una toallita sin pelusas y estáticas cada vez que toque cualquier superficie. Esto evita que las manchas o arañazos impidan las mediciones de la tasa de crecimiento una vez que se está realizando el experimento. - Descongelar 5 ml de solución de 5x concanavalina A, diluir a 1x con agua y esterilizar a través de una jeringa equipada con un filtro de 0,2-μm.
- Filtre esterilizar todos los demás líquidos que se utilizarán en el ensayo con un filtro de 0,2 μm, incluidos los medios experimentales, para eliminar los cristales o desechos que puedan haberse materializado en las soluciones. La presencia de cristales reduciría la calidad de las imágenes de microscopía.
- Pipete 200 μL de solución de concanavalina A en cada pozo de la placa del microscopio.
- Centrífuga la placa durante 2 minutos a 411 x gravedad (g) con una toallita libre de pelusas y estáticas debajo de la placa, para asegurarse de que la solución concanavalina A cubre uniformemente la parte inferior de cada pozo y que no hay burbujas de aire.
- Cubra el plato con su tapa y déjelo reposar durante 1-2 h. El tiempo preciso en que se sienta la placa es flexible, pero es importante ser consistente entre diferentes carreras del experimento.
- Retire toda la solución de concanavalina A de la placa ya sea por succión o descargando con fuerza en el fregadero o un receptáculo. Tenga cuidado de no tocar la parte de vidrio de la placa. Es aceptable si algunas gotas de solución de concanavalina A permanecen en los pozos.
- Lave los pozos de placas del microscopio añadiendo 400 μL de agua estéril. Retire el agua como se hizo con la concanavalina A en el paso anterior. No deje que el plato se seque.
- Agregue inmediatamente 185 μL de soporte experimental de crecimiento en la placa. Se añadirán 15 μL de células correctamente diluidas a esta placa.
- Dilución de células de levadura
NOTA: Los pasos siguientes describen una dilución de levadura de un cultivo saturado (aproximadamente 108 células/ml) 400 veces para lograr una concentración de 250.000 células/ml, de las cuales 15 μL se diluirán en 400 μL en la placa inferior de vidrio, dando un número final de aproximadamente 4000 células por pozo en una placa de 96 pozos. Si se utiliza una placa de 384 pozos, el número final de células por pozo debe ser de aproximadamente 700 y las diluciones deben ajustarse en consecuencia. Esta relación debe ajustarse para las células recogidas en fase de registro, creciendo en medios de pre-crecimiento más ricos o más pobres, o de diferentes cepas. La densidad final de las células por pozo debe reducirse al ejecutar el ensayo de la tasa de crecimiento durante períodos de tiempo superiores a 10 h.- Configure dos placas de cultivo de 96 pozos para diluciones en serie: etiqueta como placa 1 y 2, y agregue 90 μL de medios de crecimiento experimental (es decir, los medios en los que crecerá la levadura en el microscopio) a cada placa de dilución serie.
NOTA: Independientemente de qué dilución final se utilice, se recomiendan al menos dos diluciones en serie de células, en cada una de las cuales un pequeño volumen de levadura se canaliza en un mayor volumen de medios experimentales y luego se mezcla un gran volumen de medios experimentales vigorosamente con un pipete (como en los pasos 3.2.5 y 3.2.6 a continuación). - Recuperar la placa de las células del pre-crecimiento y centrifugar la placa durante 2 min a 411 x g.
NOTA: Es muy importante no contaminar diferentes pozos en la placa. El propósito de este paso de centrifugación antes de quitar la cubierta de papel de aluminio de las placas es asegurarse de que las gotas llenas de levadura de un pozo no vuelen fuera de la lámina y terminen en otros pozos. Tenga cuidado de nunca inclinar o agitar las placas para evitar que la levadura entre en contacto con la cubierta de la lámina después de la centrifugación. - Pelar cuidadosamente la lámina y las células resuspend mediante células vigorosamente pipeteadas con un pipete establecido en aproximadamente la mitad del volumen total en la placa mientras se mueve el pipete alrededor del pozo para mezclar. Compruebe que todas las celdas han sido resuspended desde la parte inferior de los pozos.
- Si se utilizarán múltiples cepas dentro de pozos individuales, las cepas deben mezclarse en este momento en la relación necesaria para el experimento. Si se utilizará una cepa de referencia para generar mediciones de la tasa de crecimiento, la relación de referencia a la tensión de prueba debe ser 1:1.
- Pipete 10 μL de levadura de medios de crecimiento a la placa de dilución 1. Añadir 100 μL de medios de crecimiento experimentales a cada pozo a un volumen final de 200 μL por pozo. Pipete hacia arriba y hacia abajo vigorosamente.
- Pipete 10 μL de levadura de la placa 1 a la placa 2. Agregue 100 μL de medios de crecimiento experimentales a cada pozo, y pipetee hacia arriba y hacia abajo vigorosamente para mezclar.
NOTA: Estos pasos de dilución son críticos para ayudar a separar los racimos de levadura que están unidos al final de la etapa previa al crecimiento y asegurar que aproximadamente igual número de células de levadura terminen en cada pozo. Tener un número constante de levadura en cada pozo ayuda a eliminar el ruido experimental y los sesgos en las mediciones de la tasa de crecimiento (véanse los resultados representativos).
- Configure dos placas de cultivo de 96 pozos para diluciones en serie: etiqueta como placa 1 y 2, y agregue 90 μL de medios de crecimiento experimental (es decir, los medios en los que crecerá la levadura en el microscopio) a cada placa de dilución serie.
- sonicación
NOTA: La sonicación es opcional,y sólo debe realizarse para cepas de levadura que tienen una alta propensión a adherirse entre sí (por ejemplo, algunas cepas silvestres). Para las cepas de laboratorio, la sonicación generalmente no es necesaria y puede omitirse procediendo al paso 3.4.- Desinfecte una cabeza de sonicator de 96 pines con un 70% de etanol colocándola en una placa de 96 pozos llena de 70% de etanol y seca con una toallita sin pelusas y estáticas.
- Establecer un programa de sonicación que sea lo suficientemente fuerte como para romper las células de levadura floculadas, pero no mata las células ni causa respuestas de estrés elevadas. Es posible que se requieran algunas pruebas para identificar el mejor programa de sonicación para un experimento determinado. El programa de sonicación utilizado en este experimento es: amplitud = 10, tiempo de proceso = 10 s, pulse-on = 1 s, pulse-off = 1 s. Es probable que este programa exacto no sea aplicable a todos los sonicadores, por lo que se sugieren pruebas antes del día del experimento.
- Mezcle la levadura en la placa de dilución en serie 2 una vez más pipeteando hacia arriba y hacia abajo vigorosamente cinco veces.
- Coloque la placa de dilución 2 en la plataforma y fíjela con los pasadores del sonicator en la suspensión celular, pero sin tocar la parte inferior de la placa. Ejecute el programa de sonicación con la protección del oído adecuada.
- Después de que el programa se ejecute, limpie la cabeza del sonicator con 70% de etanol y luego con agua, y luego proceda inmediatamente a la preparación de placas de microscopio para que las células no flocculen de nuevo.
- Preparar placa para microscopio:
- Pipete 15 μL de levadura de la placa de dilución en serie 2 en la placa del microscopio a un volumen de 200 μL. Agregue 200 μL de soporte de crecimiento experimental a cada pozo a un volumen final de 400 μL por pozo, y pipetee hacia arriba y hacia abajo vigorosamente para mezclar.
- Cubra la placa con una membrana transpirable. Es importante sellar bien la placa con esta membrana, por ejemplo utilizando un rodillo de goma.
- Para adherir las células de levadura a la concanavalina A en la superficie de vidrio, centrífuga la placa con una toallita sin pelusas y estática debajo de ella durante 2 minutos a 411 x g.
- En el microscopio, limpie la parte superior e inferior de la placa con una toallita sin pelusas y estáticas, y soplar aire comprimido en la placa para deshacerse de los desechos.
- Coloque la placa en el microscopio, asegurándose de que esté nivelada y de que el pozo A1 esté en la esquina superior izquierda.
4. Mediciones de la tasa de crecimiento de microscopía de lapso de tiempo
NOTA: Durante la microscopía de lapso de tiempo, las siguientes características se controlan por computadora: posición x, y y z, persianas y filtros de fluorescencia. Un sistema de enfoque automático basado en hardware es óptimo para evitar la deriva del plano focal durante las imágenes de lapso de tiempo. Como alternativa, se puede utilizar un bucle de enfoque automático basado en software. Para mantener la humedad en la cámara del microscopio, se recomienda mantener un vaso de precipitados con agua purificada en la cámara durante toda la duración del experimento.
- Cree una lista de posiciones (x,y) en la imagen, de modo que cada pozo de placa de microscopio esté completamente imagen. Evite las imágenes superpuestas para que ninguna celda se analice varias veces.
- Imagen en luminoso con iluminación diascópica (DIA) a una ampliación de 15x. Ajuste la exposición a ~5 ms.
- Acerque la imagen digitalmente para que las celdas sean claramente visibles. Utilice las perillas de enfoque para identificar el enfoque ideal para el experimento en los cuatro pozos en la esquina de la placa y en un pozo en el centro de la placa. Concéntrese de tal manera que obtenga el máximo contraste de las células.
- Establezca la posición z (o posición de enfoque automático) para que el experimento sea un promedio de las posiciones z/autofoco identificadas para cada uno de estos pozos. Si la placa del microscopio está bien hecha y el fondo de vidrio no tiene defectos, las posiciones de enfoque ideales deben ser similares para cada pozo.
NOTA: Al analizar imágenes con la canalización de análisis de imágenes De procesamiento fácilmente (PIE)11,12, es útil que las células estén ligeramente desenfocadas en el microscopio para que haya un borde oscuro fuera de la célula y un interior de color claro, lo que ayuda en el reconocimiento preciso de colonias y estimaciones de tamaño. - Si utiliza cepas fluorescentes, identifique los canales y las exposiciones con las que se debe realizar la imagen, asegurándose de que no haya píxeles sobreexpuestos. Al establecer el tiempo de exposición para los canales fluorescentes, apague el modo de "captura en vivo" en el microscopio para evitar exponer las células a la excitación por fluorescencia durante largos períodos de tiempo, ya que esto puede fotobleach las células y causar estrés.
- Configure la adquisición de secuencias de tiempo para capturar imágenes en el intervalo de tiempo deseado durante el tiempo deseado.
- Ejecuta el experimento.
Resultados
La novedad de este protocolo es que la tasa de crecimiento se puede calcular para las células individuales dentro de una población mediante el seguimiento de su crecimiento en microcolonías a través de imágenes de lapso de tiempo (Figura 2A). Debido a que las microcolonías crecen durante muchas horas de manera plana debido a la presencia de concanavalina A, sus áreas pueden ser rastreadas a lo largo del experimento, y un ajuste lineal al cambio en el tronco natural de la zona con el t...
Discusión
El protocolo descrito aquí es un ensayo versátil que permite monitorear simultáneamente el crecimiento celular y la expresión génica a nivel de microcolonías individuales. La combinación de estas dos modalidades produce conocimientos biológicos únicos. Por ejemplo, trabajos anteriores han utilizado este ensayo para mostrar una correlación negativa entre la expresión del gen TSL1 y la tasa de crecimiento de microcolonía en células de tipo salvaje isogénico midiendo ambas simultáneamente
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Naomi Ziv, Sasha Levy y Shuang Li por sus contribuciones al desarrollo de este protocolo, David Gresham por equipos compartidos y Marissa Knoll por su ayuda con la producción de video. Este trabajo fue apoyado por la subvención R35GM118170 de los Institutos Nacionales de Salud.
Materiales
| Name | Company | Catalog Number | Comments |
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are recommended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
Referencias
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genetics. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genetics. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genetics. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization--a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados