Method Article
Un marco computacional basado en el movimiento de cordones para el análisis tridimensional de la heterogeneidad del material de biopelícula
En este artículo
Resumen
Describimos un método para analizar y cuantificar el patrón de movimiento de perlas de carboxilato de 1 μm a través de biopelículas bacterianas heterogéneas. La comparación de los patrones de movimiento se puede utilizar para cuantificar las diferencias en las propiedades de los materiales de las biopelículas.
Resumen
Se han observado diferencias en las propiedades materiales de las biopelículas bacterianas en biopelículas de diferentes especies bacterianas, dentro de la misma especie en diferentes condiciones de crecimiento y después del tratamiento con moléculas modificadoras de la matriz. Para cuantificar mejor las propiedades del material de las biopelículas 3D, se desarrolló y aplicó un flujo de trabajo experimental y computacional para examinar las diferencias entre las biopelículas de Enterococcus faecalis, Salmonella enterica serotipo Typhimurium y Escherichia coli , así como el papel de los rizos amiloides en la confirmación de la rigidez de las biopelículas de Enterobacteriaceae. La dinámica espacio-temporal de las perlas de carboxilato de 1 μm en biopelículas se rastreó en biopelículas 3D de 20 μm durante 20 minutos. Las pilas de imágenes 4D se procesaron utilizando el plugin Mosaic en ImageJ para producir datos de trayectoria 3D del movimiento del cordón. Estos datos de trayectoria se analizaron con una caja de herramientas Bead Evaluator recientemente desarrollada, donde los datos de movimiento, incluida la vida útil de la trayectoria, las velocidades de la cuenta, las densidades de las celdas a lo largo de las trayectorias y la información del cuadro delimitador se calcularon y almacenaron en archivos csv. Este artículo presenta el flujo de trabajo desde la configuración experimental y el registro de imágenes hasta el cálculo y análisis de la trayectoria del cordón. La estructura de las biopelículas que contienen curli dio lugar a interacciones más estables con las perlas y menos movimiento de las perlas que en las biopelículas con mutaciones rizadas y enterocócicas. El movimiento del cordón no pareció depender en gran medida de la densidad celular al medir la velocidad del cordón y el volumen del cuadro delimitador de la trayectoria, lo que apoya la hipótesis de que otras propiedades materiales de las biopelículas controlan la dinámica del cordón. Esta técnica es ampliamente aplicable para cuantificar las diferencias en las biopelículas de diferentes composiciones de matriz, así como en las biopelículas antes y después de los tratamientos modificadores de la matriz.
Introducción
Las biopelículas bacterianas son ubicuas como parte de la microbiota humana e interactúan continuamente con las moléculas. Estas moléculas varían en tamaño desde antibióticos de 1 nm y bacterias de 1 a 3 mm hasta partículas más grandes de fibra en el tracto gastrointestinal. La composición de las biopelículas de una o varias especies afecta a las propiedades del material y, por lo tanto, al patrón de movimiento de las partículas a través de las biopelículas 1,2,3,4,5. Un ejemplo son los amiloides bacterianos, que tienen una estructura de lámina beta cruzada fibrilar conservada6. El curli amiloide se expresa en bacterias entéricas como Escherichia coli y Salmonella enterica serotipo Typhimurium, y se han detectado genes en muchos otros filos bacterianos7. Varias propiedades materiales de las biopelículas se ven afectadas por el rizo 8,9. Curli interactúa directamente con otros componentes de la matriz como el ADN extracelular (ADNe) y la celulosa 10,11. El curli rodea las células y afecta a la rigidez de la membrana celular12 y a las propiedades viscoelásticas generales del biofilm13. Curli media el aumento de la resistencia a la tracción al unirse a la fibronectina, lo que resulta en un aumento en la fuerte fijación de la superficie de vidrio14. Los bacteriófagos entrantes se unen a los rizos y limitan la invasión de fagos en las biopelículas15.
Cuando se utilizan portaobjetos de pocillos recubiertos multiprueba para analizar aproximadamente 20 μm de espesor de Enterococcus faecalis, E. coli y S. Biofilms de Typhimurium mediante microscopía confocal, claras diferencias entre E. coli, S. Se pudo observar Typhimurium10,16 y biopelículas de E. faecalis (estudio actual). Si bien las biopelículas de las especies de Enterobacteriaceae tenían un alto nivel de rigidez y las áreas con baja densidad celular eran fáciles de obtener imágenes, la obtención de imágenes claras y de alta resolución de las biopelículas de E. faecalis utilizando el promedio de líneas y marcos requirió la aplicación de presión al portaobjetos para inducir una tensión superficial suficiente para la estabilidad celular durante el proceso de imagen. Los amiloides bacterianos, como los rizos, forman estructuras muy ordenadas, lo que sugiere que pueden ser relativamente rígidos. Esto motivó la hipótesis de que el curli amiloide podría estar induciendo rigidez en E. coli y S. Biofilms de Typhimurium. No hubo evidencia clara de que E. faecalis estuviera expresando amiloides en las condiciones estudiadas. Recientemente se demostró que la proteína Esp, un gen de la pilina asociado con cepas más patógenas de E. faecalis, produce estructuras amiloides18; sin embargo, mediante búsquedas de blastn y blastp, este gen no se detectó en la cepa OG1RF de tipo comensal de E. faecalis utilizada en estos estudios. La feromona cOB1, producida por OG1RF, puede formar estructuras similares a las amiloides19. Sin embargo, con las condiciones de crecimiento de la biopelícula dadas y los métodos de detección de amiloide utilizados anteriormente para S. No se pudo detectar la tinción de amiloide por typhimurium10 en E. faecalis, los amiloides OG1RF (datos no mostrados). Se desarrolló una nueva técnica de imagen en cuatro dimensiones (4D) para comparar las propiedades generales del material entre los viscosos E. faecalis, E. coli y S. Typhimurium, así como para determinar la contribución del amiloide a las biopelículas de Enterobacteriaceae utilizando mutantes amiloides de S. Typhimurium y E. coli.
En el pasado, las perlas fluorescentes se utilizaron con éxito para analizar las propiedades del material de las biopelículas en dos dimensiones (2D) utilizando la microrreología 20,21,22,23,24,25. Esto se puede aplicar a una biopelícula tridimensional mediante el estudio de cortes ópticos 2D a varias profundidades en la biopelícula26. La técnica actual se desarrolló para rastrear perlas de microescala de 1 μm en 3D a lo largo del tiempo para su uso en el modelado 4D. Parte de la justificación fue el concepto general de utilizar el modelado 4D para comprender el movimiento de los plásmidos a través de las comunidades de microbiota gastrointestinal. Se utilizaron perlas de carboxilato cargadas fluorescentemente con un diámetro de 1 μm, ya que corresponden bien, en cuanto a tamaño y carga, a E. faecalis, el organismo modelo elegido para el movimiento y mantenimiento de plásmidos27, 28. Se desarrolló un ensayo 4D para cuantificar las propiedades físicas de las biopelículas (Figura 1A). En la metodología diseñada, se añadieron perlas a las biopelículas y sus trayectorias espacio-temporales se registraron a través de biopelículas de 10-20 μm de espesor en el transcurso de 10-20 minutos. A continuación, se cuantificaron las trayectorias de las cuentas en 3D en términos de longitud de la trayectoria, velocidad de la cuenta, volumen del cuadro delimitador de la trayectoria (cuadro mínimo que contiene la trayectoria) y densidad celular del cuadro delimitador utilizando una caja de herramientas recientemente desarrollada. El siguiente protocolo se puede emplear para generar datos de imágenes 4D de bacterias y biopelículas que contienen cordones, para preprocesar los datos con ImageJ29 y el complemento Mosaic, y para analizar las trayectorias de los cordones con una caja de herramientas Bead Evaluator.
Esta técnica tiene múltiples aplicaciones para examinar las propiedades de los materiales, así como para rastrear el movimiento de partículas y bacterias en tres dimensiones. Por ejemplo, se utilizó una primera versión de esta técnica para caracterizar el efecto de los anticuerpos monoclonales dirigidos contra el curli sobre la integridad estructural de las biopelículas16. La versión completa tiene múltiples herramientas para proporcionar un análisis más detallado de las propiedades del material de la biopelícula y se sigue utilizando para examinar los efectos del tratamiento con anticuerpos monoclonales en las biopelículas. Las partículas de diferentes cargas se pueden utilizar para examinar las propiedades de carga del material de las biopelículas y el movimiento de las partículas a través de biopelículas con diferentes composiciones de matriz. Esto podría usarse para comparar los resultados de la microrreología 2D que revelan las propiedades del material responsable del movimiento de las perlas que observamos en biopelículas que no estaban bajo flujo. Esta técnica también podría utilizarse en biopelículas de especies mixtas con regiones de diferente composición de biopelículas. Las biopelículas se pueden obtener imágenes en vivo bajo flujo en dispositivos microfluídicos y celdas de flujo para examinar los cambios en las propiedades del material entre las biopelículas estáticas y de flujo, así como el efecto del flujo en el movimiento de las partículas. Las técnicas también se pueden aplicar a bacterias marcadas con fluorescencia para caracterizar el movimiento de bacterias exógenas a través de una comunidad de biopelículas. Usando tres colores, las bacterias donantes marcadas con fluorescencia, las bacterias receptoras marcadas con fluorescencia y los plásmidos marcados con fluorescencia podrían usarse para rastrear el movimiento, el acoplamiento y la transferencia de plásmidos.
Protocolo
1. Preparación del biofilm
NOTA: Las biopelículas para análisis se pueden cultivar utilizando cualquier método que permita que la biopelícula se forme en una superficie de vidrio óptico. La estructura de la biopelícula debe adherirse a la superficie óptica lo suficiente como para que la estructura no se interrumpa durante los pasos de lavado y/o montaje del protocolo. A continuación se describe la técnica para placas de fondo óptico de 96 pocillos y cubreobjetos de vidrio de 12 mm en placas de 24 pocillos. Otras opciones incluyen placas inferiores ópticas de diferentes tamaños y cámaras de flujo óptico con y sin flujo.
- Configuración de la biopelícula
- Agregue el medio de crecimiento bacteriano a los pocillos del plato. Para este estudio, para E. faecalis, agregue 2 mL de Todd-Hewitt (TH) a una placa de 24 pocillos y agregue 0.4 mL de TH a las placas de 96 pocillos de fondo óptico. Para S. Typhimurium, E. coli y los mutantes isógenas csgAB curli, añaden 0,7 mL de caldo Luria sin sal (LB) a los pocillos. Si utiliza cámaras ópticas inferiores, continúe con el paso 1.2.
- Coloque cubreobjetos ópticos de vidrio # 1.5 de 12 mm en una placa de Petri y cúbralos con etanol.
- Con unas pinzas, retira el cubreobjetos y usa una llama para prender fuego al alcohol restante. Deja que el alcohol se queme. Solo usa la llama para prenderles fuego; No sostenga el cubreobjetos en la llama porque se agrietará. Deje que el cubreobjetos se enfríe durante 10-20 segundos antes de colocarlo en el pozo para evitar que se agriete.
- Coloque el cubreobjetos en el pocillo que contiene el medio en un ángulo para evitar que se coloque encima del medio. No agregue el cubreobjetos a un pozo seco y luego agregue medio porque esto hará que el cubreobjetos se pegue al fondo del pozo.
- Utilice una punta de pipeta estéril para empujar con cuidado el cubreobjetos hasta el fondo del pocillo que contiene el medio.
NOTA: Recuerde hacer coincidir el grosor (# 1 o # 1.5) del cubreobjetos de vidrio óptico o la placa inferior óptica con el grosor de la óptica del microscopio confocal.
- Incubar la biopelícula en condiciones adecuadas para el crecimiento de la biopelícula. En estos estudios, se cultivaron biopelículas de E. faecalis como cultivos estáticos a 37 °C y biopelículas de E. coli aeróbicamente a 30 °C.
- En estos estudios, cultive biopelículas de E. faecalis durante 2 días con cambios medios por la mañana y temprano en la noche. Para evitar dañar o desprender la biopelícula, incline con cuidado la placa. Coloque la punta de la pipeta cerca del borde inferior del pocillo y extraiga lentamente el medio. Agregue el primer ml de medio fresco de la misma manera. Agregue el segundo mL lentamente cerca de la interfaz medio/pocillo.
- En estos estudios, utilice las condiciones de crecimiento para una producción óptima de rizos. Crecer S. Las biopelículas de Typhimurium a 28 °C durante 6-8 días se incubaron en una inclinación que permitió que la biopelícula se adhiriera alrededor de 2/3 del camino hacia arriba por el portaobjetos y luego creciera como una película en la interfaz aire-líquido. Esto se hizo sin un cambio medio. Para evitar que el medio se seque, coloque la placa de 24 pocillos en una cámara con una bandeja de agua.
2. Imágenes 4D
- Preparación de la montura del biofilm
- Diluir carmesí 1 μm carboxilato FluoSpheres perlas 1:50 en PBS (2 x 107 perlas en 1 mL de PBS). Si utiliza cámaras ópticas inferiores, agregue Syto9 a una dilución de 1 μL a 300 μL de preparación de perlas.
- (Opcional) Lave la biopelícula para eliminar los rastros del medio de crecimiento si el medio de crecimiento tiene autofluorescencia. En estos experimentos, lavar dos veces con 1 mL de PBS, inclinando cuidadosamente la placa, colocando la punta de la pipeta cerca del borde inferior del pocillo y extrayendo lentamente el medio. Agregue PBS colocando la punta cerca del borde inferior y llene lentamente el pozo. Utilice esta técnica en pozos ópticos de fondo, además de los cubreobjetos en una placa de 24 pocillos.
- Retire el medio o PBS. Agregue perlas carmesí diluidas preparadas en 2.1.1 a la biopelícula. En estos estudios, agregue 1 mL de perlas (2x107) a los cubreobjetos y 0,2 mL de perlas (4x106) y placas inferiores ópticas Syto9 a 96 pocillos.
- Incubar durante 1 minuto a temperatura ambiente para permitir la asociación de las perlas.
- Retire las perlas y lave suavemente la biopelícula una vez con PBS para eliminar las perlas no asociadas. En estos estudios, para el cubreobjetos, lavar suavemente con 1 mL de PBS, volver a llenar el pocillo con 1 mL de PBS y proceder a 2.1.6. Para las placas inferiores ópticas de 96 pocillos, lave suavemente la biopelícula con 0,2 mL de PBS y vuelva a llenar el pocillo con 0,2 mL de PBS. La cámara óptica inferior ya está lista para la toma de imágenes, así que continúe con el paso 2.2.
- (Si se usa un cubreobjetos) Agregue 1 μL de Syto9 (tinción de ADN fluorescente verde; diluido según las instrucciones del fabricante) en el centro de un pocillo en un portaobjetos multiprueba de 10 pocillos recubierto. Estos cubreobjetos recubiertos tienen profundidades de pozo de 23-25 μm.
- (Si se usa un cubreobjetos) Con pinzas esterilizadas con llama de alcohol, retire con cuidado el cubreobjetos del pocillo e inviértalo en el pocillo que contiene la gota Syto9. Dejar el cubreobjetos en 1 mL de PBS hace que sea más fácil de quitar del pocillo y ayuda a lavar las perlas no asociadas de la biopelícula.
- (Si se usa un cubreobjetos) Selle cuidadosamente el cubreobjetos con esmalte de uñas sin deslizar ni presionar el cubreobjetos, lo que puede causar una tensión superficial que detendrá el movimiento en biopelículas más viscosas y menos rígidas.
- (Si se usa un cubreobjetos) Deja que el esmalte de uñas se seque. Limpie cuidadosamente la superficie exterior del cubreobjetos con etanol al 70%. Limpie sin aplicar ninguna presión al cubreobjetos por las razones mencionadas anteriormente.
- Imágenes confocales
NOTA: En estos estudios, utilice un microscopio confocal de barrido láser de imágenes espectrales invertidas equipado con un sistema confocal TCS con el objetivo 63x. El visor se utilizará para generar un vídeo 4D (Figura 1). El biofilm 3D estará compuesto por cortes Z capturados en pasos de 0,5 μm a través de biofilms de 18-20 μm de grosor, generando 36-40 cortes Z. Cada biopelícula 3D tardará entre 50 y 60 segundos en capturarse. Las rebanadas juntas formarán un marco, que se puede visualizar como una biopelícula en 3D. Este proceso se repetirá 20 veces para generar el video de lapso de tiempo 4D para un tiempo de seguimiento total de 18-20 minutos.- Ajuste el visor para capturar fluoróforos. En estos estudios, excite Syto9 (tinción de ADN bacteriano) con un láser de 488 nm y mida la emisión de 495 a 540 nm (el Leica Sp5 es un microscopio de imágenes espectrales). Excite perlas carmesí (rojas) con un láser de 633 nm y mida la emisión de 650 nm a 700 nm. Estos ajustes de fluoróforos se pueden ajustar para capturar los fluoróforos deseados.
- Elija el modo de imagen xyzt.
- Identifique una región de la biopelícula con una mezcla de regiones de mayor y menor densidad para capturar las diferencias en las propiedades viscoelásticas en regiones gruesas y delgadas de la biopelícula.
- Establezca una pila Z de 18-20 μm de grosor. En una biopelícula montada en cubreobjetos, evite que la parte superior e inferior de la biopelícula toque el vidrio para evitar artefactos que atrapen cuentas (consulte la discusión).
- Ajuste la ganancia y el desplazamiento para utilizar todo el rango dinámico de la intensidad en el punto más brillante de la biopelícula. Esto minimiza la superposición de señales de las capas inferiores de la biopelícula.
- Ajuste el grosor de la rebanada a 0,5 μm. Esto permite obtener imágenes rápidas sin pérdida de información del cordón.
- Establezca la resolución en 512 x 512 (0,48 μm). Esto permite obtener imágenes rápidas y generar imágenes con suficiente resolución para ver la estructura de la biopelícula y los detalles del movimiento de las cuentas.
- Minimice el tiempo de obtención de imágenes.
- Configurado para capturar 20 acumulaciones.
- Guardar como .lif (o archivo confocal similar). La película 4D se puede generar en ImageJ.
3. Generación del video de biopelícula 4D con ImageJ
- Abra el archivo .lif en ImageJ con la siguiente configuración: Pila de vista con Hyperstack, Orden de pila XYCZT, modo de color: coloreado, escalado automático marcado. A continuación, pulse OK.
- Seleccione Imagen > color > Dividir canales.
- Seleccione Imagen > color > Fusionar canales > Crear compuesto > OK.
- Seleccione Complementos > Visor 3D > Canales, desmarque la > azul OK.
- Seleccione Editar > Mostrar contenido marcado > Mostrar cuadro delimitador marcado.
- Utilice el clic y la pulsación del ratón para seleccionar la imagen. A continuación, gire la imagen sin dejar de mantener pulsado el botón de clic. Gire la imagen para que la parte inferior de la biopelícula quede en la parte inferior y el ángulo permita visualizar las cuentas. A continuación, suelte la imagen.
- Presione el botón rojo Grabar en la parte inferior de la ventana para grabar un video.
- Guarde como archivo .avi usando jpeg como compresión.
4. Generación de datos de trayectoria
- Instale la herramienta de código abierto ImageJ (https://imagej.net/Fiji) y el complemento de seguimiento de partículas Mosaic (https://imagej.net/MOSAICsuite).
- Importe el archivo .lif que contiene los dos canales para perlas y bacterias. Divida los canales y guarde los archivos por separado.
- En ImageJ, almacene el tamaño del vóxel de la imagen (dimensiones x, y y z) y el tamaño del período de tiempo en un archivo de texto.
- En ImageJ, vaya a Plugins | Mosaico e inicie el Particle Tracker 2D/3D.
- Introduzca los siguientes parámetros: Radio: 3, Corte: 0,003, Per/Abs: 0,12, Rango de enlace: 2, Desplazamiento: 10,00 con la dinámica establecida en Brownian. Por favor, vea los resultados para conocer la justificación detrás de la elección de browniano en estos experimentos.
- Genere trayectorias y exporte la lista de trayectorias como archivos .csv.
5. Análisis de trayectorias
- Instalar VRL Studio (https://vrl-studio.mihosoft.eu)
- Descargar el proyecto Biofilm (https:// neurobox3d.github.io/Biofilm/) y lanzarlo en VRL Studio
- Cargue el archivo de trayectoria en ImportData
- Especifique el tamaño de píxel x, y y z (use ImageJ para localizar estos valores) en ProcessTrajectories.
- Especifique el intervalo de fotogramas en ComputeVelocity (use ImageJ para localizar este valor).
- Cargue el archivo tiff bacteriano (consulte el paso 3.2) en Comdensity.
- Establezca las rutas de salida para los datos de velocidad en SaveVelocityDataToFile y los datos de trayectoria en SaveTrajectoryDataToFile.
- Invoque SaveVelocityDataToFile y SaveTrajectoryDataToFile.
- Importe datos a Excel para su análisis. Estos datos incluirán las longitudes de las trayectorias, la vida útil de las trayectorias, las dimensiones y los volúmenes del cuadro delimitador de trayectorias, las velocidades medias y las varianzas de los cordones. El análisis calcula las variables ponderadas utilizando el canal de las bacterias marcadas con Syto9 para calcular las densidades celulares locales (dentro de los cuadros delimitadores de trayectoria dados). El análisis da como resultado el cálculo de velocidades y varianzas promedio ponderadas, así como promedios ponderados y varianzas de la varianza del cuadro delimitador.
Resultados
Este método se utilizó para probar la hipótesis de que el curli puede conferir rigidez a E. coli y S. Biopelículas de Typhimurium, que reducen el movimiento de las perlas durante los experimentos de microscopía confocal. La caja de herramientas actual se utilizó para comparar las propiedades del material de la cepa OG1RF de tipo comensal de Enterococcus faecalis con el serotipo Typhimurium de Salmonella enterica, E. coli y sus respectivos mutantes isogénicos curli (Figura 1B y Video Complementario 1, Video Suplementario 2, Video Suplementario 3, Video Suplementario 4, Video Suplementario 5, Video Suplementario 6). Las propiedades del material de la biopelícula podrían diferir potencialmente con respecto a la rigidez (por ejemplo, rizos unidos al ADNe) o interacciones electrostáticas e hidrofóbicas entre las perlas cargadas negativamente y las células de la biopelícula y los materiales de la matriz, así como la densidad celular.
Reproducibilidad
La caja de herramientas Biofilm se programó en Groovy30 y Java31 dentro de VRL-Studio32 , lo que permitió un diseño de flujo de trabajo modular con generación automática de interfaz de usuario (UI) de todos los componentes computacionales. Esto permitió un flujo de trabajo automatizado, eliminando el sesgo inducido involuntariamente por el experimentador al analizar los resultados.
Uso de MSD para confirmar el tipo de movimiento en las biopelículas
Para el análisis de trayectorias utilizando Particle Tracker 2D/3D, hay disponibles diferentes ajustes de dinámica para el análisis de diferentes tipos de movimiento de cordones. Para estos estudios, se eligió el entorno "movimiento browniano" (es decir, movimiento impulsado por difusión) ya que E. faecalis es una bacteria inmóvil, E. coli y Salmonella no expresan flagelos en biopelículas, y los experimentos se realizaron en un sistema cerrado en ausencia de flujo. Este ajuste podría validarse aún más mediante los desplazamientos cuadrados medios calculados (MSD) de las cuentas. Usando la definición 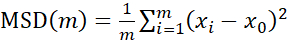 donde m es el número de segmentos de trayectoria, se puede calcular el cambio de la MSD en el transcurso de cada trayectoria. Las trayectorias lineales indican un movimiento difusivo del cordón (Figura 2A). Utilizando el ajuste de mínimos cuadrados cuadráticos, se calculó el patrón de movimiento promedio de todas las cuentas en la biopelícula, mostrando el orden lineal dominante y validando la difusión pasiva como la fuerza impulsora (Figura 2A-2F).
donde m es el número de segmentos de trayectoria, se puede calcular el cambio de la MSD en el transcurso de cada trayectoria. Las trayectorias lineales indican un movimiento difusivo del cordón (Figura 2A). Utilizando el ajuste de mínimos cuadrados cuadráticos, se calculó el patrón de movimiento promedio de todas las cuentas en la biopelícula, mostrando el orden lineal dominante y validando la difusión pasiva como la fuerza impulsora (Figura 2A-2F).
Análisis de cuadros delimitadores.
La toolbox utiliza ImageJ Mosaic y Particle Tracker 2D/3D para generar trayectorias (Paso 4) y, a continuación, utilizando la canalización automatizada de análisis de biopelícula, genera datos importantes sobre las trayectorias de las cuentas que se pueden utilizar para comparar las propiedades del material de biopelícula. El volumen de la caja delimitadora en μm3 se midió construyendo la caja mínima que contiene una trayectoria y midiendo su volumen (Figura 3).
Las biopelículas de E. faecalis tienen más movimiento de cordones con valores de cuadro delimitador de 1-6000μm3 (Figura 3B, 3C y 3D). Los resultados confirman que el movimiento observado en un cubreobjetos de vidrio montado sobre un portaobjetos recubierto con un pocillo recubierto de aproximadamente 25 μm (Figura 3C) frente a biopelículas cultivadas en el fondo de pocillos de vidrio óptico y fotografiadas directamente (Figura 3D) arroja resultados equivalentes con pocas diferencias. La única diferencia fue que cerca de la parte superior del cubreobjetos montado de las biopelículas de E. faecalis se podían registrar trayectorias estables con una vida útil superior a 10 minutos, pero al mismo tiempo pequeños cuadros delimitadores, mientras que en la placa inferior óptica se podía registrar un número selecto de cuentas con mayor movilidad. En conjunto, esto sugiere que el montaje del portaobjetos de vidrio puede haber cambiado la tensión superficial del sistema en la parte superior de la biopelícula contra el portaobjetos en el biofilm montado, lo que en última instancia disminuyó la movilidad de algunas perlas en regiones de biofilm menos viscosas (Figura 3B, 3C, 3D y 3I). Las trayectorias que entran en esta categoría resultaron ser un porcentaje muy pequeño y, incluso con este pequeño número de cuentas atrapadas, el MSD promedio de E. faecalis en un cubreobjetos montado fue ligeramente mayor que el MSD calculado a partir de una biopelícula de placa inferior óptica (Figura 3).
S. Las trayectorias de las cuentas de Typhimurium y E. coli tuvieron volúmenes de caja delimitadora más pequeños de 0-10 μm3 (Figura 3A, 3B, 3E y 3F), en comparación con los mutantes rizos isogénicos con cajas delimitadoras de 1-6000 μm3 para E. coli y 1-5000 μm3 para S. Typhimurium (Figura 3A, 3B, 3F y 3H), demostrando una mayor movilidad del cordón. Estos resultados sugirieron que la presencia del amiloide se correlacionaba con una mayor rigidez en las biopelículas y era consistente con la falta de movimiento notable de la biopelícula en los videos. Los volúmenes de los cuadros delimitadores fueron consistentemente pequeños (0-10 μm3) incluso en las regiones de baja densidad de la biopelícula. Esta observación es consistente con observaciones previas de que el curli puede estar presente en regiones de baja densidad celular del biofilm10.
No fue posible comparar el comportamiento de las biopelículas de Enterobacteriaceae en las placas inferiores ópticas debido a que crecen como películas en la interfaz aire-líquido (Paso 1.2.2). Cuando se utiliza un cubreobjetos, la película se adhiere al cubreobjetos en la interfaz y cuando se retira el cubreobjetos, la película se coloca sobre el cubreobjetos creando una sola superficie de imagen. En una placa inferior óptica cultivada de forma inclinada, se tomaron imágenes con líquido aún en el pozo. Esto significa que la película sigue flotando por encima del fondo óptico y hace que la película quede fuera de la profundidad de trabajo de un visor invertido como el Leica Sp5. La eliminación de suficiente medio para llevar la biopelícula a la profundidad de trabajo del microscopio hizo que la muestra se secara durante el proceso de obtención de imágenes de 20 minutos.
En general, los gráficos confirman las observaciones visuales en las películas suplementarias y son consistentes con las diferencias de MSD observadas (Figura 3I y 3J).
Vida útil de la trayectoria
La vida útil de la trayectoria se midió como el número de fotogramas consecutivos en los que se registró un cordón (Figura 3).
En las biopelículas de E. faecalis más viscosas, similares a fluidos, todas las perlas tuvieron una vida útil de menos de 10 minutos y la mayoría de las trayectorias oscilaron entre 2 y 5 minutos para las biopelículas de E. faecalis . Sin embargo, las perlas con una vida útil corta registrada pudieron localizarse mediante inspección visual en las biopelículas de E. faecalis durante la ventana de tiempo total de la imagen (Video suplementario 1 y 2). Por lo tanto, es posible que las perlas se muevan a lo largo de una trayectoria registrada, disociándose intermitentemente de la biopelícula y terminando una trayectoria, y volviéndose a asociar con la biopelícula, momento en el que se inicia una nueva trayectoria. En última instancia, esto conduciría a una vida útil corta bajo la presencia continua de perlas en la biopelícula. Es importante tener en cuenta que con esta técnica, la esperanza de vida de la trayectoria, especialmente en una biopelícula viscosa, tiende a subestimar el tiempo total que una cuenta está asociada con la biopelícula.
En el S. Las biopelículas de Typhimurium, que tenían volúmenes de caja delimitadora más pequeños, la mayoría de las cuentas (alrededor del 80%) tenían una larga vida útil de 16 a 20 fotogramas, lo que corresponde a aproximadamente 15 a 20 minutos en tiempo real (Figura 3A, 3G y 3H). A diferencia de estos, las biopelículas isogénicas mutantes de curli transportaban más perlas móviles con volúmenes de caja delimitadora que oscilaban entre 1-6000 μm3 (E. coli) y 1-5000 μm3 (S. Typhimurium) (Figura 3A, 3B, 3F y 3H). Sin embargo, a diferencia de las biopelículas de E. faecalis con trayectorias del >70% con volúmenes de caja delimitadora superiores a 10 μm3, las biopelículas de especies de Enterobacteriaceae registraron solo un 30% de trayectorias de cuentas con volúmenes de caja delimitadora superiores a 10 μm3. A pesar de que la vida útil general de la trayectoria del cordón fue menor en las biopelículas mutantes de rizo, algunas trayectorias reflejaron un movimiento sustancial del cordón y una larga vida útil de la trayectoria (Figura 3H). Esta observación podría indicar que esta variabilidad puede corresponder a diferentes propiedades del material de la biopelícula, como la viscoelasticidad, y/o cambios en la química de la superficie de las partículas, como la carga.
Análisis de longitudes y velocidades de trayectoria de cordones
La longitud de la trayectoria es una medida de la distancia recorrida por las cuentas en μm. Esta medición es consistente con la velocidad del movimiento del cordón en μm/s. De acuerdo con los volúmenes más grandes de la caja delimitadora, las perlas en las biopelículas de E. faecalis tenían trayectorias 10 veces más largas, 5-20 μm, frente a <4 μm en las biopelículas que contenían curli. Consistente con las trayectorias más cortas (Figura 4A). Las perlas de E. faecalis midieron velocidades hasta 15 veces más altas, con la mayoría de las perlas teniendo velocidades en el rango de 0,01-0,15 μm/s frente a velocidades <0,006 μm/s (Figura 4B). No obstante, las biopelículas mutantes de curli midieron velocidades generales más bajas y trayectorias más cortas en comparación con las biopelículas de E. faecalis , pero trayectorias más largas y velocidades más altas que las cepas parentales que contienen rizos (Figura 4A y 4B).
Cabe destacar el hecho de que la estructura fibrilar en forma de red de curli30 puede afectar la movilidad de forma anisotrópica, reduciendo el movimiento en el plano xy y permitiendo una mayor movilidad en la dirección z (Figura 2G). El gran grupo de trayectoria (aproximadamente 800) que pertenece a aproximadamente 50 cuentas únicas en biopelículas que contienen rizos sería consistente con las limitaciones del seguimiento de partículas en mosaico, contando cada una de estas cuentas que se mueven rápidamente como una sola cuenta en x, y y z. Será necesario realizar más investigaciones y desarrollar programas informáticos para confirmar esta observación.
Análisis de la dependencia del movimiento de las perlas de la densidad celular
La dependencia del movimiento de las perlas con la densidad celular se determinó mediante el uso de velocidades y varianzas promedio ponderadas, así como promedios/promedios ponderados y varianzas de los volúmenes de los cuadros delimitadores. El segundo canal de imagen para las bacterias marcadas con Syto9 se utilizó para calcular las densidades celulares locales en el cálculo de las velocidades ponderadas. La densidad celular se calculó promediando los datos del vóxel Syto9 sobre el cuadro delimitador de cada borde de trayectoria (Figura 5, derecha). Por lo tanto, la velocidad del cordón se puede ponderar mediante las densidades de celdas (locales) en los bordes. Existen múltiples tipos de tinciones que podrían usarse para visualizar bacterias, incluidas las tinciones para la pared celular, las membranas y el contenido de ADN. Para determinar la densidad celular, se eligió Syto9 porque proporciona la señal más consistente, independientemente del corte óptico Z que se esté visualizando. Las tinciones de envoltura (pared celular y membrana) darán una señal diferente dependiendo de la posición de la rebanada Z. Si el corte Z incluye la parte superior o inferior de la celda, la señal será más fuerte que si el corte Z pasa por el centro de la celda donde solo se tiñe el contorno de la celda.
Las trayectorias de los cordones desde el canal rojo se rastrearon durante 20 fotogramas, donde las trayectorias individuales tenían una vida útil mínima de 2 fotogramas y un máximo de 20 fotogramas con 19 segmentos de trayectoria que conectaban los fotogramas (Figura 5). Para estudiar la dependencia de la densidad celular de la movilidad de las perlas, se determinó la intensidad de GFP por vóxel (cada una de las mediciones individuales en la imagen de vóxel de 512x512). La densidad de celdas alrededor de cada segmento de la trayectoria del cordón se calculó como la densidad promediada localmente en el cuadro delimitador del segmento.
En el caso de algunas biopelículas, se pudo documentar una dependencia de la densidad estadísticamente significativa (Figura 6), sobre todo en el caso de las biopelículas de E. faecalis que se cultivaron en un cubreobjetos de vidrio y se invirtieron en un portaobjetos de varios pocillos (Figura 6A). Por el contrario, las biopelículas de E. faecalis que se cultivaron en el fondo de una placa de 96 pocillos (Figura 6B) no mostraron dependencia de la densidad. En conclusión, esto sugiere que las biopelículas altamente fluidas de E. faecalis podrían estar potencialmente ligeramente comprimidas debido al montaje en un portaobjetos de múltiples pocillos, lo que es consistente con una reducción en el número de perlas que se mueven más rápidamente y aquellas con pequeños volúmenes de caja delimitadora que están atrapadas en la parte superior de la biopelícula contra el portaobjetos de vidrio (Figura 3C vs Figura 3D). Tanto las biopelículas de Salmonella y E. coli (Figura 6C y 6D) como sus mutantes isogénicos (Figura 6E y 6F) mostraron una dependencia menor o nula de la densidad celular.
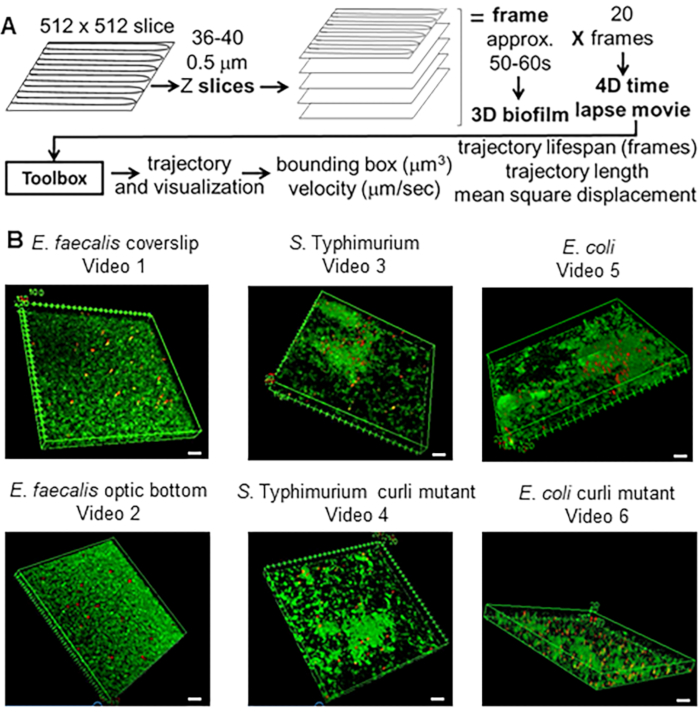
Figura 1. Proceso de obtención de imágenes y análisis (pasos 2-4) (A) Las biopelículas se obtienen como se describe en 2.2. Utilizando las biopelículas de las imágenes (ver 3) se generaron las trayectorias de las cuentas como se describe en 4. Utilizando las trayectorias, los datos relevantes se calcularon con la caja de herramientas de análisis (ver 5) (B) Las biopelículas se cultivaron como se describe en el paso 1 en cubreobjetos (E. faecalis, Video 1), S. Typhimurium (Video 3), E. coli (Video 5) y mutantes isogénicos curli (Video 4, Video 6) o en una placa de 96 pocillos con fondo óptico (E. faecalis bottom, Video 2). La barra de escala blanca es de 20 mm. Esta figura se reproduce con permiso de (31). Haga clic aquí para ver una versión más grande de esta figura.
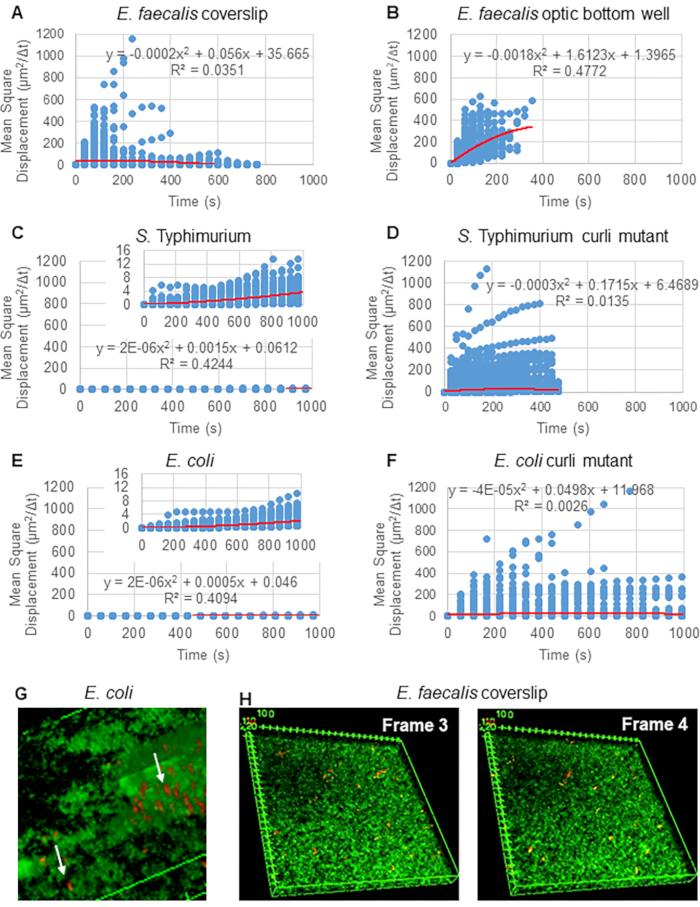
Figura 2. Se determinó que el movimiento de las cuentas en las biopelículas es de naturaleza difusiva (movimiento browniano, ver paso 5) (A-F) Los datos de MSD muestran un comportamiento lineal (línea roja) para validar el movimiento browniano. Las biopelículas se cultivaron de acuerdo con el paso 1 (G) Ejemplo de movimiento elíptico de las perlas observado en las biopelículas de E. coli y S. Typhimurium tomadas de un fotograma del ensayo de biopelícula 4D de E. coli (H) Ejemplo de grandes cambios en los patrones de las perlas entre los fotogramas 3 y 4 tomadas del pozo de fondo óptico de E. faecalis . Nótese que la biopelícula en sí misma provoca algo de flujo (Video 1 y 2), lo que hace parecer que los fotogramas 3 y 4 están orientados de manera diferente. Esta figura se reproduce con permiso de (31). Haga clic aquí para ver una versión más grande de esta figura.
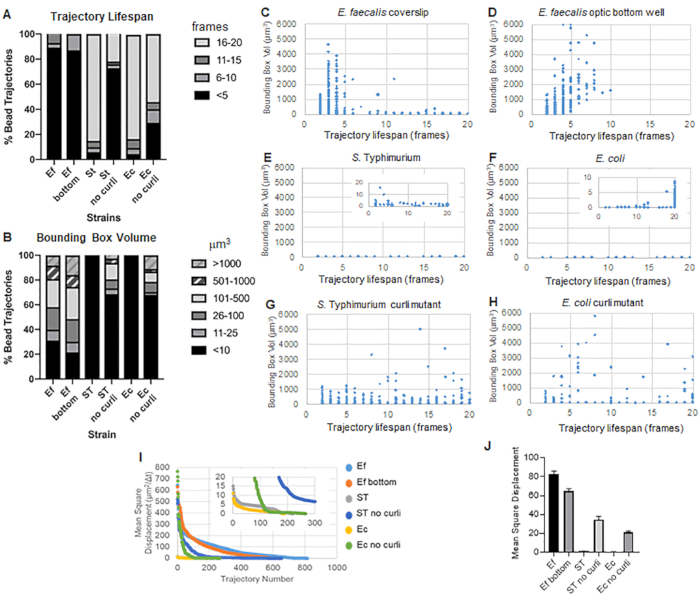
Figura 3. Análisis de diferencias de rigidez mediante cuadros delimitadores y longevidades de trayectoria. Las biopelículas se cultivaron como se describe en el paso 1 en cubreobjetos (E. faecalis, Video 1), S. Typhimurium (Video 3), E. coli (Video 5) y mutantes isogénicos curli (Video 4, Video 6) o en una placa de 96 pocillos con fondo óptico (E. faecalis bottom, Video 2). La vida útil de las trayectorias se presenta en % de las trayectorias totales de las cuentas (A) y los gráficos de dispersión (C-H), junto con los volúmenes de los cuadros delimitadores (calculados en el paso 5) (I) Comparación de los MSD de las cuentas en las diferentes biopelículas (H) Medias de los MSD de cada tipo de biopelícula. Las barras indican el intervalo de confianza del 95% de los datos. Esta figura se reproduce con permiso de (31). Haga clic aquí para ver una versión más grande de esta figura.
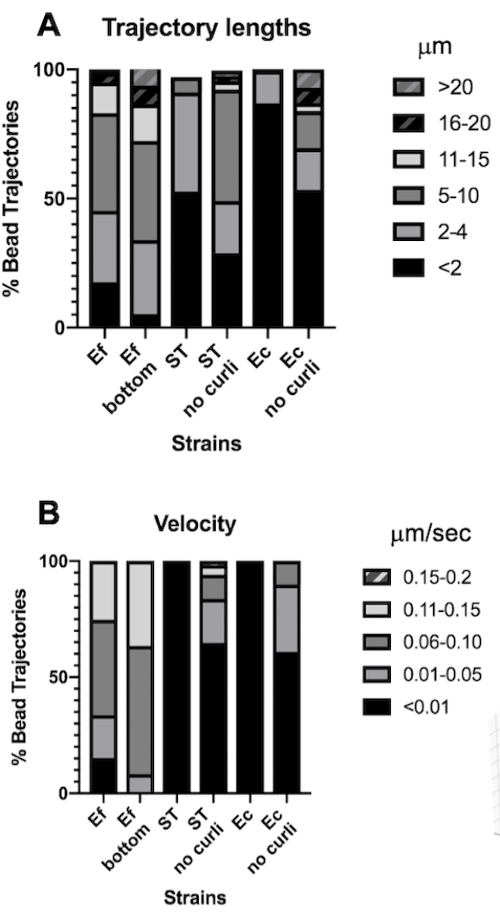
Figura 4. Análisis de diferencias en la longitud de la trayectoria y la velocidad del cordón. Las biopelículas se cultivaron como se describe en el paso 1. Las longitudes de las trayectorias se muestran en μm y se presentan como % de las trayectorias totales del cordón (A). La velocidad se muestra en μm/s y se presenta como % de las trayectorias totales del cordón (B). Esta figura se adapta con permiso de (31). Haga clic aquí para ver una versión más grande de esta figura.
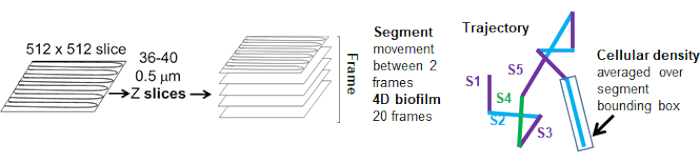
Figura 5. Esquema del análisis de segmentos de trayectoria. Esta figura se adapta con permiso de (31). Haga clic aquí para ver una versión más grande de esta figura.
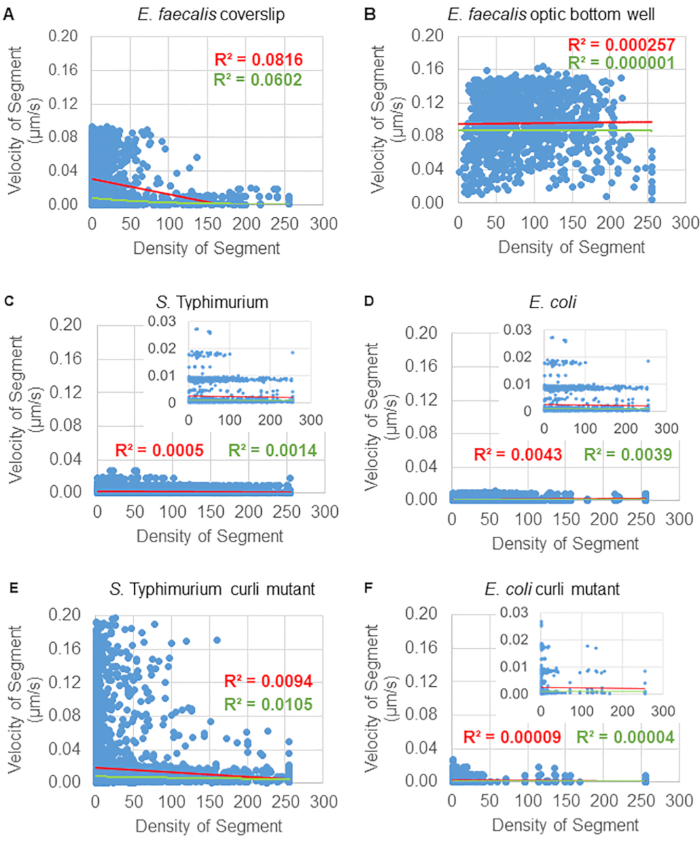
Figura 6. Estudio del efecto de la densidad de la biopelícula en la velocidad de la perla utilizando la tubería de análisis de biopelícula (paso 5). Los resultados indicaron que la velocidad del cordón no depende exclusivamente de la densidad celular. Las biopelículas se cultivaron como se describe en el paso 1. Las trayectorias se analizaron a escala de segmentos de trayectoria individuales (Figura 5). Para cada segmento, la velocidad del cordón en μm/s se representó en función de la densidad celular del cuadro delimitador (GFP promedio por vóxel dentro del cuadro delimitador). La línea roja muestra la regresión lineal y la línea verde el trazado de la regresión exponencial. Esta figura se reproduce con permiso de (31). Haga clic aquí para ver una versión más grande de esta figura.
Video complementario 1. Video 4D de la biopelícula OG1RF de E. faecalis de 24 horas cultivada en un cubreobjetos de vidrio óptico de 1,5 de espesor. El video de lapso de tiempo 4D se generó utilizando un microscopio con una resolución de 512x512. Se generó una serie Z de 40 imágenes mediante la obtención de imágenes de una región de aproximadamente 20 μm de espesor de una biopelícula en pasos de 0,5 μm. Cada serie Z era de un fotograma y requería entre 50 y 60 segundos para capturar. Se capturó una serie de 20 fotogramas contiguos para producir un vídeo en 4D. La reproducción de video es de aproximadamente 120x. El video es representativo de al menos 6 experimentos independientes. Haga clic aquí para descargar este video.
Video complementario 2. Video 4D de una biopelícula OG1RF de E. faecalis de 24 horas cultivada en una placa inferior óptica de 96 pocillos. El video de lapso de tiempo 4D se generó utilizando un microscopio con una resolución de 512x512. Se generó una serie Z de 40 imágenes mediante la obtención de imágenes de una región de aproximadamente 20 μm de espesor de una biopelícula en pasos de 0,5 μm. Cada serie Z era de un fotograma y requería entre 50 y 60 segundos para capturar. Se capturó una serie de 20 fotogramas contiguos para producir un vídeo en 4D. La reproducción de video es de aproximadamente 120x. El video es representativo de 3 experimentos independientes.Por favor, haga clic aquí para descargar esto vídeo.
Video complementario 3. Vídeo 4D de una biopelícula de Typhimurium ATCC 14028 del serotipo Salmonella enterica cultivada en cubreobjetos de vidrio óptico de 1,5 de espesor durante 6-7 días. El video de lapso de tiempo 4D se generó utilizando un microscopio con una resolución de 512x512. Se generó una serie Z de 40 imágenes mediante la obtención de imágenes de una región de aproximadamente 20 μm de espesor de una biopelícula en pasos de 0,5 μm. Cada serie Z era de un fotograma y requería entre 50 y 60 segundos para capturar. Se capturó una serie de 20 fotogramas contiguos para producir un vídeo en 4D. La reproducción de video es de aproximadamente 120x. El video es representativo de 3 experimentos independientes.Por favor, haga clic aquí para descargar esto vídeo.
Video complementario 4. Vídeo 4D del serotipo Salmonella enterica Typhimurium biofilms ATCC 14028 curli mutant (csgBA) se cultivaron en cubreobjetos de vidrio óptico de 1,5 espesores durante 6-7 días. El video de lapso de tiempo 4D se generó utilizando un microscopio con una resolución de 512x512. Se generó una serie Z de 40 imágenes mediante la obtención de imágenes de una región de aproximadamente 20 μm de espesor de una biopelícula en pasos de 0,5 μm. Cada serie Z era de un fotograma y requería entre 50 y 60 segundos para capturar. Se capturó una serie de 20 fotogramas contiguos para producir un vídeo en 4D. La reproducción de video es de aproximadamente 120x. El video es representativo de 3 experimentos independientes.Por favor, haga clic aquí para descargar esto vídeo.
Video complementario 5. Video 4D de E. coli UTI89 cultivada en cubreobjetos de vidrio óptico de 1,5 espesores durante 6-7 días. El video de lapso de tiempo 4D se generó utilizando un microscopio con una resolución de 512x512. Se generó una serie Z de 40 imágenes mediante la obtención de imágenes de una región de aproximadamente 20 μm de espesor de una biopelícula en pasos de 0,5 μm. Cada serie Z era de un fotograma y requería entre 50 y 60 segundos para capturar. Se capturó una serie de 20 fotogramas contiguos para producir un vídeo en 4D. La reproducción de video es de aproximadamente 120x. El video es representativo de 3 experimentos independientes. Por favor, haga clic aquí para descargar esto vídeo.
Video complementario 6. Video 4D E . coli UTI89 curli mutante (csgBA) cultivado en cubreobjetos de vidrio óptico de 1,5 espesores durante 6-7 días. El video de lapso de tiempo 4D se generó utilizando un microscopio con una resolución de 512x512. Se generó una serie Z de 40 imágenes mediante la obtención de imágenes de una región de aproximadamente 20 μm de espesor de una biopelícula en pasos de 0,5 μm. Cada serie Z era de un fotograma y requería entre 50 y 60 segundos para capturar. Se capturó una serie de 20 fotogramas contiguos para producir un vídeo en 4D. La reproducción de video es de aproximadamente 120x. El video es representativo de 3 experimentos independientes. Por favor, haga clic aquí para descargar esto vídeo.
Discusión
Pasos críticos y solución de problemas
El mayor reto de esta técnica es utilizar un cubreobjetos montado con una biopelícula muy viscosa como E. faecalis. El cubreobjetos debe colocarse con cuidado y precisión en el portaobjetos multipocillo sin necesidad de reposicionarlo. Durante el paso de sellado, se debe tener cuidado para evitar empujar hacia abajo el cubreobjetos o empujarlo/deslizarlo accidentalmente por la superficie del portaobjetos. Cualquier movimiento o presión puede crear tensión superficial y bloquear el movimiento de una biopelícula viscosa. Si es posible, la comparación de las propiedades del material de la biopelícula mediante la obtención de imágenes de una biopelícula en un pozo de fondo óptico con una montura de cubreobjetos permitirá la evaluación de la técnica. Cuando se realiza correctamente, una montura de cubreobjetos se asemeja mucho a una biopelícula en una placa inferior óptica para E. faecalis.
Además, cuando se utiliza un cubreobjetos montado, se deben evitar las imágenes de las interfaces de la biopelícula con el cubreobjetos en la parte inferior o el portaobjetos en la parte superior. Cuando se utiliza un visor invertido, con el cubreobjetos en la parte inferior, puede quedar atrapado en la base de la biopelícula contra el cubreobjetos. Estas perlas atraviesan la biopelícula y quedan atrapadas contra el cubreobjetos incluso después de un lavado suave. Tienen las coordenadas x, y y z de 0 y las coordenadas del cuadro delimitador de 0. Sin embargo, para ciertas aplicaciones, como el examen de la integridad de la biopelícula después del tratamiento, estos puntos de datos se pueden utilizar como herramienta. La capacidad de las perlas para penetrar a través de una región gruesa de la biopelícula hasta el fondo del cubreobjetos se puede utilizar para evaluar la integridad de la biopelícula después del tratamiento (manuscrito en preparación en colaboración con el laboratorio Tükel). En la parte superior de la biopelícula, en una biopelícula viscosa como E. faecalis , teníamos algunas evidencias de compactación impuesta por el cubreobjetos. Esto limitó el movimiento de algunas cuentas en la interfaz del portaobjetos de vidrio y puede haber introducido cierta dependencia de la densidad en el análisis del movimiento de las cuentas.
Los pasos de lavado fueron necesarios para las biopelículas porque el medio de crecimiento tiene una fuerte autofluorescencia en el canal verde. Elegimos utilizar el exceso de cuentas y eliminar las cuentas no asociadas mediante lavado para maximizar las cuentas asociadas y obtener la caracterización más precisa de las regiones observadas.
El número de cordones y lavados necesarios para obtener los conjuntos de datos deseados debe determinarse empíricamente. La presencia de demasiadas perlas en una biopelícula genera conjuntos de datos imposiblemente grandes que son difíciles de analizar. La presencia de muy pocas perlas no genera un muestreo exhaustivo de los entornos de biopelícula. Sin embargo, el control del número de perlas añadidas (2x107 perlas en 1 mL de PBS) y el uso de pasos de lavado, dieron como resultado un número relativamente constante de perlas (40-140) que se asocian con la biopelícula dependiendo de su estructura, disposición espacial y composición.
Al estudiar biopelículas con regiones viscosas y rígidas mixtas, las perlas pueden quedar atrapadas en las regiones rígidas con el tiempo. En este caso, la imagen debe iniciarse inmediatamente después de la adición de las cuentas. Esto a menudo no se puede lograr usando cubreobjetos, sino que requiere placas inferiores ópticas o celdas de flujo donde se pueden obtener imágenes inmediatamente después de la adición de las perlas y los pasos de lavado.
Modificaciones y aplicaciones futuras
Uso de dispositivos microfluídicos. En nuestros estudios, las condiciones óptimas establecidas para el estudio de las biopelículas de Enterobacteriaceae requirieron el crecimiento de la biopelícula como una película en la interfaz aire-líquido. Esto limitó el uso de placas inferiores ópticas y dispositivos microfluídicos en los estudios. Sin embargo, cuando las condiciones de formación de biopelículas lo permiten, las biopelículas pueden cultivarse en cámaras microfluídicas o celdas de flujo. A continuación, las biopelículas podrían lavarse e introducirse perlas a través del dispositivo microfluídico con una interrupción mínima de la biopelícula.
Adición de perlas durante el crecimiento de la biopelícula. Elegimos agregar el exceso de perlas a las biopelículas y luego eliminar las perlas no asociadas mediante un lavado suave para optimizar la cantidad de perlas presentes durante el análisis. En las biopelículas viscosas de E. faecalis , es posible que las perlas se disociaran y se volvieran a asociar durante el tiempo de imagen de 20 minutos. Si se agrega un número bajo de perlas en diferentes momentos durante el crecimiento de la biopelícula, podría ser posible atrapar las perlas en la biopelícula, lo que permite una caracterización más precisa del movimiento de la biopelícula en biopelículas más viscosas.
Elección de la región a la imagen. Para los estudios sobre las propiedades de los materiales, es mejor elegir regiones gruesas y delgadas de la biopelícula. Sin embargo, cuando se estudian los cambios en las propiedades del material de una biopelícula tratada, se pueden obtener imágenes de regiones confluentes gruesas para determinar los cambios en las propiedades viscoelásticas y la penetración del cordón en esas regiones. En este caso, la búsqueda de perlas que penetraron en la biopelícula y terminaron atrapadas contra el cubreobjetos es una medida útil de la interrupción de la biopelícula.
Imágenes bajo flujo. Usando celdas de flujo de vidrio óptico o dispositivos microfluídicos, se puede obtener una imagen del movimiento de perlas o bacterias en una biopelícula bajo flujo. Esto se puede hacer de diferentes maneras. Se puede hacer mediante la inyección de perlas en toda la cámara, seguida de una breve incubación para permitir la asociación de las perlas con la biopelícula. Las perlas no asociadas se pueden eliminar mediante lavado y se pueden obtener imágenes de la biopelícula con o sin flujo. Por el contrario, se puede introducir un pequeño número de perlas en un lado de la cámara y su movimiento a través y dentro de la biopelícula se puede rastrear bajo flujo. Al usar el flujo, deberá tenerse cuidado al elegir la configuración de seguimiento del cordón de mosaico (Paso 4.5). En los estudios actuales, el entorno dinámico fue browniano. Los cálculos de MSD confirmaron que era probable que el movimiento fuera difusivo, lo que hacía que Brownian fuera el escenario apropiado.
Tinción de matriz. En los estudios actuales, la tinción con Syto9 examina la densidad celular y no la densidad de la estructura de la biopelícula. Por ejemplo, es probable que la presencia de amiloides aumente la densidad del material de la matriz de la biopelícula. La dependencia del movimiento de la densidad de amiloide podría determinarse mediante el uso de tinciones de matriz fluorescente en lugar de Syto9.
Bacterias marcadas con fluorescencia. Las bacterias marcadas con fluorescencia se pueden utilizar para rastrear el movimiento de bacterias exógenas a través de biopelículas (por ejemplo, bacterias que contienen plásmidos). El desafío con las bacterias marcadas con fluorescencia, como los enterococos, es que forman cadenas simples, diplococcos y cortas, lo que complica la capacidad de rastrear con precisión las bacterias. Este proceso sería más fácil si las bacterias tienen una morfología unicelular.
Limitaciones
Limitaciones en la visualización de trayectorias y costuras.
Una limitación del método es la visualización de la trayectoria y la costura. Las trayectorias reconstruidas y analizadas constan de coordenadas de puntos x, y, z, donde los puntos subsiguientes definen la trayectoria lineal entre estos puntos. La visualización de estas trayectorias lineales por partes se puede lograr mediante varias herramientas. Un enfoque fue usar cuadernos de Python y Jupyter junto con los complementos de Python, Pandas y Matplotlib. Si bien fue posible visualizar las trayectorias individuales existentes en el artículo del Journal of Bacteriology donde se publicó originalmente esta técnica34, aún existen limitaciones significativas que se están abordando en futuras investigaciones.
Actualmente, el número de trayectorias reconstruidas es mayor que el número de cuentas en la biopelícula, lo que significa que múltiples trayectorias pueden corresponder a una cuenta. Esto puede deberse a una señal confocal débil en un fotograma en el que Mosaic terminará una trayectoria e iniciará una segunda. Esto puede registrarse como múltiples trayectorias más cortas para una cuenta, especialmente en biopelículas menos viscosas. Otra causa del gran número de trayectorias es la falta de costura de trayectorias. Especialmente en las biopelículas de pozos ópticos de fondo de E. faecalis , las perlas permanecen visualmente asociadas con la biopelícula durante la obtención de imágenes (Video complementario 2). Sin embargo, no hubo trayectorias de más de 10 y más del 90% de las trayectorias tenían una vida útil de 5 fotogramas o menos (Figura 3D). Si el software se utiliza para analizar solo trayectorias por encima de una longitud definida (por ejemplo, cuando se rastrean células que son capaces de transferir plásmidos), las trayectorias más cortas se pueden eliminar automáticamente del conjunto de datos. Sin embargo, hay otros propósitos para los que la costura de las trayectorias puede ser muy importante. Finalmente, la incapacidad de rastrear el movimiento rápido de las cuentas como una sola trayectoria podría resultar en más trayectorias en las biopelículas de Enterobacteriaceae debido al rápido movimiento en la dirección Z que da como resultado cuentas de forma elíptica (Figura 2G). La posibilidad de trayectorias de sutura interrumpidas por el movimiento anisotrópico rápido será importante para estudiar el efecto de la matriz de amiloide curli en Enterobacteriaceae.
Importancia
Se desarrolló un flujo de trabajo computacional para estudiar las trayectorias de las cuentas y comparar las propiedades de los materiales de las biopelículas 3D. El flujo de trabajo permite a los investigadores identificar parámetros críticos que se pueden utilizar en el modelado computacional de la dinámica de fluidos en biopelículas heterogéneas. Con la ayuda de este evaluador de cuentas de código abierto, se pudo estudiar el efecto del curli amiloide bacteriano en las propiedades del material, mostrando una mayor rigidez de la matriz de biopelícula debido al rizado. En un contexto más general, el evaluador se puede utilizar para estudiar los cambios en la estructura de la biopelícula inducidos por el tratamiento de la biopelícula o diferentes condiciones ambientales, como el flujo. Por ejemplo, la herramienta se está utilizando para analizar el efecto del tratamiento con anticuerpos monoclonales en la disrupción de las estructuras de biopelículas en colaboración con el laboratorio Tükel (LKSOM Temple University). La caja de herramientas del evaluador de cuentas es totalmente adaptable y ampliable de forma modular utilizando VRL-Studio para mejorar y ampliar aún más sus funciones.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El trabajo en los laboratorios GQ y BAB no recibió ninguna subvención específica de ninguna agencia de financiación en los sectores público, comercial o sin fines de lucro. Los autores agradecen a Isaac Klapper, Ph.D (Departamento de Matemáticas, Universidad de Temple) por su útil discusión y a Çagla Tükel (Departamento de Microbiología e Inmunología, Universidad de Temple) por su experiencia en Enterobacteriaceae en la publicación inicial de la contención de esta técnica.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-well plates, No. 1.5 Uncoated Coverslip, 5 mm Glass Diameter | MatTek | P96G1.55F | |
| Fisherbrand Cover Glasses: Circles | Fisher Scientific | 12-293-232P | 1.5 optic glass coverslip |
| Invitrogen Syto 9 Green Fluorescent Nucleic Acid Stain | Invitrogen | S34854 | |
| Molecular Probes FluoSpheres Carboxylate-modified Microspheres, 1 um, crimson fluorescent (625/645) | Molecular Probes | F8816 |
Referencias
- Huang, R., Li, M., Gregory, R. L. Bacterial interactions in dental biofilm. Virulence. 2, 435-444 (2011).
- Nadell, C. D., Drescher, K., Wingreen, N. S., Bassler, B. L. Extracellular matrix structure governs invasion resistance in bacterial biofilms. ISME Journal. 9, 1700-1709 (2015).
- Billings, N., Birjiniuk, A., Samad, T. S., Doyle, P. S., Ribbeck, K. Material properties of biofilms - key methods for understanding permeability and mechanics. Reports on Progress in Physics. 78, 036601 (2015).
- Araújo, G. R. d. e. S., Viana, N. B., Gómez, F., Pontes, B., Frases, S. The mechanical properties of microbial surfaces and biofilms. The Cell Surface. 5, 100028 (2019).
- Tallawi, M., Opitz, M., Lieleg, O. Modulation of the mechanical properties of bacterial biofilms in response to environmental challenges. Biomaterials Science. 5, 887-900 (2017).
- Tursi, S. A., Tükel, &. #. 1. 9. 9. ;. Curli-Containing Enteric Biofilms Inside and Out, Matrix Composition, Immune Recognition, and Disease Implications. Microbiology and Molecular Biology Reviews. 82, (2018).
- Dueholm, M. S., Albertsen, M., Otzen, D., Nielsen, P. H. Curli functional amyloid systems are phylogenetically widespread and display large diversity in operon and protein structure. PLoS One. 7 (12), 51274 (2012).
- Hung, C., et al. Escherichia coli biofilms have an organized and complex extracellular matrix structure. mBio. 4, 00645 (2013).
- Kikuchi, T., Mizunoe, Y., Takade, A., Naito, S., Yoshida, S. Curli fibers are required for development of biofilm architecture in Escherichia coli K-12 and enhance bacterial adherence to human uroepithelial cells. Microbiology and Immunology. 49, 875-884 (2005).
- Gallo, P. M., et al. Amyloid-DNA Composites of Bacterial Biofilms Stimulate Autoimmunity. Immunity. 42, 1171-1184 (2015).
- Serra, D. O., Richter, A. M., Hengge, R. Cellulose as an architectural element in spatially structured Escherichia coli biofilms. Journal of Bacteriology. 195, 5540-5554 (2013).
- Oh, Y. J. Characterization of curli A production on living bacterial surfaces by scanning probe microscopy. Biophysical Journal. 103, 1666-1671 (2012).
- Lembré, P., Di Martino, P., Vendrely, C. Amyloid peptides derived from CsgA and FapC modify the viscoelastic properties of biofilm model matrices. Biofouling. 30, 415-426 (2014).
- Oh, Y. J., et al. Curli mediate bacterial adhesion to fibronectin via tensile multiple bonds. Scientific Reports. 6, 33909 (2016).
- Vidakovic, L., Singh, P. K., Hartmann, R., Nadell, C. D., Drescher, K. Dynamic biofilm architecture confers individual and collective mechanisms of viral protection. Nature Microbiology. 3, 26-31 (2018).
- Tursi, S. A., et al. Salmonella Typhimurium biofilm disruption by a human antibody that binds a pan-amyloid epitope on curli. Nature Communications. 11, 1007 (2020).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15 (8), 1007978 (2019).
- Taglialegna, A., et al. The biofilm-associated surface protein Esp of Enterococcus faecalis forms amyloid-like fibers. Npj Biofilms and Microbiomes. 6, 15 (2020).
- Gour, S., Kumar, V., Rana, M., Yadav, J. K. Pheromone peptide cOB1 from native Enterococcus faecalis forms amyloid-like structures, A new paradigm for peptide pheromones. Journal of Peptide Science. 25, 3178 (2019).
- Stoodley, P., Debeer, D., Lewandowski, Z. Liquid flow in biofilm systems. Applied and Environmental Microbiology. 60, 2711-2716 (1994).
- Birjiniuk, A., et al. Single particle tracking reveals spatial and dynamic organization of the E. coli biofilm matrix. New Journal of Physics. 16, 085014 (2014).
- Chew, S. C., et al. Dynamic remodeling of microbial biofilms by functionally distinct exopolysaccharides. mBio. 5, 01536 (2014).
- Cao, H., et al. Revealing region-specific biofilm viscoelastic properties by means of a micro-rheological approach. Npj Biofilms and Microbiomes. 2, 5 (2016).
- Galy, O., et al. Mapping of bacterial biofilm local mechanics by magnetic microparticle actuation. Biophysical Journal. 103, 1400-1408 (2012).
- Rogers, S. S., vander Walle, C., Waigh, T. A. Microrheology of bacterial biofilms in vitro, Staphylococcus aureus and Pseudomonas aeruginosa. Langmuir. 24, 13549-13555 (2008).
- Hart, J. W., Waigh, T. A., Lu, J. R., Roberts, I. S. Microrheology and Spatial Heterogeneity of Staphylococcus aureus Biofilms Modulated by Hydrodynamic Shear and Biofilm-Degrading Enzymes. Langmuir. 35 (9), 3553-3561 (2019).
- van Merode, A. E. J., van der Mei, H. C., Busscher, H. J., Krom, B. P. Influence of culture heterogeneity in cell surface charge on adhesion and biofilm formation by Enterococcus faecalis. Journal of Bacteriology. 188, 2421-2426 (2006).
- Tariq, M., Bruijs, C., Kok, J., Krom, B. P. Link between Culture Zeta Potential Homogeneity and Ebp in Enterococcus faecalis. Applied and Environmental Microbiology. 78, 2282-2288 (2012).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ, 25 years of image analysis. Nature Methods. 9, 671-675 (2012).
- . Oracle.com Available from: https://docs.oracle.com/en/java (2021)
- Hoffer, M., Poliwoda, C., Wittum, G. Visual reflection library, a framework for declarative GUI programming on the Java platform. Computing and Visualization in Science. 16, 181-192 (2013).
- Perov, S., et al. Structural Insights into Curli CsgA Cross-β Fibril Architecture Inspire Repurposing of Anti-amyloid Compounds as Anti-biofilm Agents. PLOS Pathogens. 15, 1007978 (2019).
- Malhotra, K., et al. Development of a New Bead Movement-Based Computational Framework Shows that Bacterial Amyloid Curli Reduces Bead Mobility in Biofilms. Journal of Bacteriology. 202, 00253 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados