Method Article
Inmunopeptidomics: Aislamiento de péptidos asociados a MHC de clase I y II de ratón y humano para el análisis de espectrometría de masas
En este artículo
Resumen
Aquí, presentamos un protocolo para la purificación de complejos peptídicos MHC clase I y clase II a partir de líneas celulares de ratón y humanos que proporcionan datos inmunopeptidómicos de alta calidad. El protocolo se centra en la preparación de muestras utilizando anticuerpos disponibles comercialmente.
Resumen
La inmunopeptidómica es un campo emergente que alimenta y guía el desarrollo de vacunas e inmunoterapias. Más específicamente, se refiere a la ciencia de investigar la composición de péptidos presentados por moléculas de clase I y clase II de complejos de histocompatibilidad (MHC) utilizando plataformas tecnológicas de espectrometría de masas (MS). Entre todos los pasos en un flujo de trabajo de inmunopeptidómica basado en em, la preparación de muestras es de vital importancia para capturar datos de alta calidad de relevancia terapéutica. Aquí, se describen instrucciones paso a paso para aislar péptidos asociados a MHC clase I y II mediante la purificación de inmunoafinidad a partir de muestras de control de calidad, de ratón (EL4 y A20) y líneas celulares humanas (JY) más específicamente. Los diversos reactivos y anticuerpos específicos se describen a fondo para aislar los péptidos asociados a MHC de estas líneas celulares, incluidos los pasos para verificar la eficiencia de unión a las perlas del anticuerpo y la eficiencia de elución de los complejos de péptidos MHC de las perlas. El protocolo se puede utilizar para establecer y estandarizar un flujo de trabajo de inmunopeptidomica, así como para comparar nuevos protocolos. Además, el protocolo representa un gran punto de partida para cualquier no experto, además de fomentar la reproducibilidad intra e interlaboratorio del procedimiento de preparación de muestras en inmunopeptidómica.
Introducción
Durante la última década, el interés en investigar el repertorio de péptidos asociados a MHC ha superado al sector académico y ha llegado a las industrias biotecnológica y farmacéutica. De hecho, en el cáncer, el descubrimiento de neoantígenos específicos de tumores accionables representa un importante foco de investigación en el sector industrial para desarrollar inmunoterapias clínicas que conduzcan a una oncología personalizada1,2,3. Fundamentalmente, los péptidos asociados a MHC se presentan en todo el cuerpo, reflejan la etapa intracelular de la célula y son significativos en diversas condiciones de enfermedad como autoinmunidad, trasplante, enfermedades infecciosas, inflamación, cáncer y alergias1,4. Por lo tanto, los péptidos asociados a MHC, o ligandos de antígeno leucocitario humano (HLA) en humanos, son de gran interés médico y se conocen colectivamente como inmunopeptidome5.
La EM es un potente enfoque analítico para caracterizar el inmunopeptidome6,7, incluyendo el descubrimiento de neoantígenos tumorales específicos8,9,10,11. Un flujo de trabajo típico para realizar un experimento de inmunopeptidómica incluye tres pasos principales: 1) preparación de muestras para el aislamiento de péptidos asociados a MHC, 2) adquisición de datos por MS y 3) análisis de datos utilizando varias herramientas de software computacional12. La generación de muestras de alta calidad descritas en este protocolo visualizado es fundamental para el éxito de cualquier proyecto en inmunopeptidómica basada en EM. El protocolo que se describe a continuación se centra en aislar péptidos asociados a MHC de clase I y II de líneas celulares bien establecidas que son adecuadas para generar datos inmunopeptidómicos de alta calidad. Los resultados representativos de esas líneas celulares se muestran en el protocolo actual.
Protocolo
El protocolo proporcionado aquí fue adaptado de los protocolos establecidos13,14,15,16,17,18,19,20. El procedimiento general para la purificación de inmunoafinidad (IP) de los péptidos MHC de clase I y II se ilustra en la Figura 1. Consulte la Tabla de materiales para obtener detalles sobre las líneas celulares y los anticuerpos utilizados.
1. Acoplamiento de perlas con los anticuerpos (Día 1): Acoplamiento de anticuerpos a cuentas de sefalrosa activadas por CNBr
NOTA: Prepare nuevas soluciones para cada nuevo experimento. Consulte la Tabla de materiales y la Tabla suplementaria 1 para obtener la lista de recetas de reactivos y soluciones. Todos los pasos se llevan a cabo a temperatura ambiente (RT). Cuando utilice un nuevo anticuerpo, compruebe la eficiencia de unión recogiendo alícuotas clave (ver la indicación OPCIONAL) y realizando la tinción azul de Coomassie SDS-PAGE (Figura 2).
- Activación de las perlas de sefarosa CNBr
- Pesar 80 mg de perlas activadas con CNBr de sefalrosa por muestra y transferirlas a un tubo cónico de 15 ml.
- Para facilitar la resuspensión de cuentas secas, primero, agregue 5 ml de HCl de 1 mM y pipetee hacia arriba y hacia abajo 5 veces. Luego, llene el tubo cónico con 8.5 mL adicionales de 1 mM HCl.
- Gire a 20 rpm (revoluciones por minuto) durante 30 minutos a RT utilizando un dispositivo rotador. Centrifugar las perlas a 200 x g durante 2 min en RT y retirar el sobrenadante por aspiración.
- Agregue 500 μL de tampón de acoplamiento al pellet de perlas y transfiéralo a un nuevo tubo de centrífuga de 2.0 ml y manténgalo a un lado para el paso 1.2.2.
- Acoplamiento de anticuerpos a perlas activadas por CNBr
- Preparar el anticuerpo seleccionado para el aislamiento de los péptidos MHC de clase I o II en un nuevo tubo de microcentrífuga de 2,0 ml añadiendo 2 mg del anticuerpo (según la concentración especificada por el fabricante). Complete el volumen a 1 ml con la solución tampón de acoplamiento para obtener una concentración final de 2 mg / ml.
- Centrifugar las perlas del paso 1.1.4 a 200 x g durante 2 min en RT y luego retirar el sobrenadante por aspiración.
- Agregue la solución de anticuerpos al tubo de microcentrífuga de 2,0 ml que contiene las perlas activadas.
- (OPCIONAL) Tome una alícuota de 18 μL (INPUT), agregue 6 μL de 4x búfer SDS-PAGE y congele inmediatamente.
- Gire el tubo de la microcentrífuga del paso 1.2.3 a 20 rpm durante 120 min utilizando un dispositivo rotador. Centrifugar las perlas a 200 x g durante 2 min y luego retirar el sobrenadante.
- (OPCIONAL) Tome una alícuota de 18 μL del sobrenadante (anticuerpo no unido), agregue 6 μL de tampón 4x SDS-PAGE y congele inmediatamente.
- Bloqueo y lavado de perlas acopladas de anticuerpos
- Añadir 1 ml de glicina de 0,2 M al tubo de la microcentrífuga que contiene las perlas acopladas a anticuerpos del paso 1.2.5. Gire a 20 rpm durante 60 minutos a RT con un dispositivo rotador.
- Centrifugar las perlas a 200 x g durante 2 min, luego retirar el sobrenadante. Añadir 1 ml de PBS (solución salina tamponada con fosfato).
- Centrifugar las perlas a 200 x g durante 2 min, luego retirar el sobrenadante.
- (OPCIONAL) Tome una alícuota de 18 μL del sobrenadante (anticuerpo no unido, último lavado), agregue 6 μL de tampón 4x SDS-PAGE y congele inmediatamente.
- Agregue 1 ml de PBS a las cuentas en el paso 1.3.3.
- (OPCIONAL) Tome una alícuota de 18 μL de mezcla de perlas (perlas junto con anticuerpos), agregue 6 μL de tampón 4x SDS-PAGE y congele inmediatamente.
- Mantener a 4 °C hasta su uso el mismo día en el paso 2.5.
NOTA: Las cuentas se pueden preparar el día antes de la inmunocaptura, pero no se ha probado un almacenamiento más prolongado.
2. Lisis celular e inmunocaptura con perlas acopladas de anticuerpos (Día 1)
NOTA: El nivel de moléculas MHC varía de un tipo de célula a otra, y se sugiere la cuantificación de moléculas MHC/HLA por célula21 (Figura 4A). Recomendamos un mínimo de 1 x 108 celdas para cada IP. Este número de células corresponde a 6-10 mg de proteína de células JY, EL4 y A20 solubilizadas con un tampón de Chaps al 0,5%. Para preparar gránulos celulares para IP, las células deben cosecharse, centrifugarse y lavarse dos veces con 5 ml de PBS. Luego, los gránulos celulares se pueden almacenar en un tubo de microcentrífuga de 1,5 ml o en un tubo cónico de 15 ml a -80 °C hasta el momento de la IP. Tenga en cuenta que las IP se pueden realizar en gránulos de células recién cosechadas o congeladas.
- Para aislar los péptidos MHC de clase I o II, descongele un gránulo congelado de 1 x 108 células calentando la parte inferior del tubo con la palma de la mano. Añadir 500 μL de PBS al pellet y pipetear hacia arriba y hacia abajo hasta que la suspensión sea homogénea.
NOTA: Dependiendo del tipo de célula, el volumen de pellets de células puede variar sustancialmente. Si 500 μL de PBS no son suficientes para disolver el pellet celular, use más PBS hasta que las células se desagreguen fácilmente mientras pipetea hacia arriba y hacia abajo. - Mida el volumen total del pellet resuspendido en PBS y transfiéralo a un nuevo tubo de microcentrífuga de 2 ml. Dividir en más tubos si es necesario.
- Agregue un volumen de tampón de lisis celular (1% de tampón de chaps en PBS que contiene inhibidores de la proteasa, 1 pellet/10 ml de tampón) equivalente al volumen de pellet celular resuspendido en PBS medido en el paso anterior. La concentración final del tampón de lisis es de 0,5% Chaps.
- Gire a 10 rpm durante 60 min a 4 °C con un dispositivo rotador. Centrifugar el lisado celular a 18.000 x g durante 20 min a 4 °C con el freno completo y transferir el sobrenadante (que contiene los complejos MHC-péptidos) en un nuevo tubo de microcentrífuga de 2,0 ml.
- Recupere las perlas acopladas a anticuerpos del paso 1.3.7 por centrifugación a 200 x g durante 2 min y retire el sobrenadante.
- Transfiera el sobrenadante de lisado celular en el paso 2.4 a las perlas acopladas a anticuerpos e incube con rotación (10 rpm) durante 14-18 h (durante la noche) a 4 ° C utilizando un dispositivo rotador.
3. Elución de péptidos MHC (Día 2)
NOTA: La columna de polipropileno permite la elución de complejos MHC-péptidos mientras retiene las perlas en la columna. Para evaluar la proporción de complejos de péptidos MHC unidos a las perlas antes y después de la elución ácida con TFA al 1% (ácido trifluoroacético), es posible realizar un western blot con alícuotas tomadas en pasos clave durante el protocolo (ver la indicación OPCIONAL). Western blotting mostrado en la Figura 3 revela el enriquecimiento de complejos MHC-péptidos después de la elución ácida con 1% de TFA. La ausencia de señal en esta fracción indicaría que el paso de elución no fue exitoso. Nótese que la proporción de complejos de péptidos MHC no unidos a las perlas también se puede evaluar en paralelo mediante western blotting utilizando una alícuota descrita en el paso 3.1.6.
- Elución de complejos MHC-péptidos a partir de las perlas acopladas de anticuerpos
- Retire la tapa inferior de la columna de polipropileno, coloque la columna en el estante de columnas de polipropileno e instale un recipiente vacío debajo para recoger el flujo.
NOTA: Se requiere una columna por muestra. - Enjuague la columna de polipropileno con 10 ml de tampón A y déjela drenar por gravedad. Si la velocidad de flujo de la elución del líquido es demasiado lenta, corte aún más la punta inferior de la columna de polipropileno.
- Mida y recoja la mezcla de perlas-lisado (~2 ml) del paso 2.5 y transfiérala a la columna de polipropileno.
- (OPCIONAL) Tome una alícuota de 20 μL (o 1/100 del volumen total) para el western blotting y congele inmediatamente. Esta fracción corresponde al total de complejos de péptidos MHC incubados con las perlas.
- Deje que la mezcla líquida eluya por gravedad.
- (OPCIONAL) Recolecte y mida el flujo y tome una alícuota de 20 μL (o 1/100 del volumen total) para el western blotting y congele inmediatamente. Esta fracción representa los complejos residuales no unidos de péptidos MHC.
- Para recuperar la mezcla de perlas y lisado tanto como sea posible, enjuague el tubo del paso 3.1.3 con 1 ml de tampón A y transfiéralo a la columna de polipropileno.
- Lave las perlas retenidas en la columna de polipropileno agregando 10 ml de tampón A. Deje que el amortiguador de lavado eluya por gravedad.
- Repita el paso de lavado con 10 ml de tampón B, 10 ml de tampón A y luego 10 ml de tampón C.
- Retire la columna de polipropileno del bastidor y colóquela en la parte superior de un nuevo tubo de microcentrífuga de 2,0 ml. Sostenga la columna y el tubo juntos con la mano.
- Agregue 300 μL de TFA al 1% a la columna de polipropileno y mezcle las perlas mediante pipeteo hacia arriba y hacia abajo 5 veces.
NOTA: Las perlas se retendrán en la columna de polipropileno, y los péptidos unidos a MHC se eluirán en el tubo de microcentrífuga de 2.0 ml. - Transfiera el eluido en un nuevo tubo de microcentrífuga de 2,0 ml. Repita el paso 3.1.11 y agrupe los 2 eluidos (los eluidos que contienen los complejos de péptidos MHC deben corresponder a un total de 600 μL y se utilizarán en el paso 3.2.4).
- (OPCIONAL) Recoger una alícuota de 6 μL (o 1/100 del volumen total) para el western blotting y congelar inmediatamente. Esta fracción corresponde a los complejos MHC-péptidos eluidos de las perlas.
- Retire la tapa inferior de la columna de polipropileno, coloque la columna en el estante de columnas de polipropileno e instale un recipiente vacío debajo para recoger el flujo.
- Desalinización y elución de péptidos MHC
NOTA: Los pasos de desalinización y elución de péptidos MHC se pueden realizar instalando la columna C18 en un tubo de microcentrífuga de 2.0 mL. Para un mejor ajuste, instale los recocidos proporcionados por el fabricante entre la columna C18 y el tubo de microcentrífuga de 2,0 ml. En este protocolo, se utiliza una columna C18 con una capacidad de volumen de 5-200 μL (6-60 μg). Todos los pasos se llevan a cabo en RT.- Agregue 200 μL de metanol en la parte superior de la columna C18, luego centrífique a 1546 x g durante 3 min. Descarta el flujo.
- Agregue 200 μL de 80% ACN (acetonitrilo)/0.1%TFA en la parte superior de la columna C18, luego centrífuga a 1546 x g durante 3 min. Descarta el flujo.
- Agregue 200 μL de 0.1% TFA en la parte superior de la columna C18, luego centrífuga a 1546 x g durante 3 min. Descarta el flujo.
- Cargue 200 μL de los complejos de péptidos MHC desde el paso 3.1.12 en la parte superior de la columna C18. Centrifugar a 1546 x g durante 3 min y desechar el flujo.
- Repita el paso 3.2.4 dos veces hasta que se haya cargado el volumen completo. Tenga en cuenta que los péptidos MHC se retienen en la columna C18.
- Agregue 200 μL de 0.1% TFA a la columna C18, luego centrífuga a 1546 x g durante 3 min. Descarta el flujo.
- Transfiera la columna C18 a un nuevo tubo de microcentrífuga de 2,0 ml. Elute péptidos MHC de la columna C18 añadiendo 150 μL de 28%ACN/0,1%TFA.
- Centrífuga a 1546 x g durante 3 min.
- Transfiera el flujo a través en un nuevo tubo de microcentrífuga de 1,5 ml. Tenga cuidado de no descartar el flujo; contiene los péptidos aislados MHC clase I o II.
- Repita los pasos 3.2.7-3.2.9 dos veces para un volumen total de 450 μL.
- Congelar los 450 μL de eluido (péptidos MHC purificados de clase I o II) a -20 °C hasta que las muestras sean analizadas por LC-MSMS.
- Antes del análisis LC-MS/MS, los péptidos MHC purificados de clase I o II del paso 3.2.11 se evaporan a sequedad utilizando un concentrador de vacío con ajustes preestablecidos de 45 °C durante 2 h, nivel de vacío: 100 mTorr y rampa de vacío: 5.
NOTA: La evaporación de muestras congeladas es altamente eficiente. Los péptidos secos se pueden volver a congelar hasta el análisis.
4. Identificación de péptidos MHC clase I y II por LC-MS/MS
NOTA: Analice el inmunopeptidomo MHC clase I y II utilizando un Orbitrap de alto rendimiento y espectrómetros de masas de tiempo de vuelo cuadrupolo de alta resolución6. La siguiente información se proporciona solo como indicación, teniendo en cuenta que los diversos instrumentos de espectrometría de masas en tándem existentes funcionan de acuerdo con diferentes estándares operativos. A continuación se analiza un breve resumen de los pasos:
- Solubilizar las muestras secas (a partir del paso 3.2.12) en 50 μL de ácido fórmico (FA) al 4%.
- Cargue tres inyecciones de 16 μL para cada muestra y separe en una columna de fase inversa casera (150-μm i.d. por 250 mm de longitud, Júpiter 3 μm C18 300 Å) con un gradiente de 5%-30% ACN-0.1% FA y un caudal de 600 nL/min en un UHPLC de nanoflujo conectado a un MS.
- Adquirir cada espectro completo de MS a una resolución de 120000, un AGC de 4 x 105 con modo automático para el tiempo de inyección y espectros utilizando tandem-MS (MS-MS) en los iones precursores de carga multiplicada más abundantes para un máximo de 3 s.
NOTA: Los experimentos de Tandem-MS se realizan utilizando disociación de colisión (HCD) de mayor energía a una energía de colisión del 30%, una resolución de 30,000, un AGC de 1.5 x 105 y un tiempo de inyección de 300 ms. - Procese los archivos de datos de las tres inyecciones/muestra utilizando un software de análisis proteómico LC-MS/MS (por ejemplo, PEAKS X) utilizando las bases de datos de ratones y humanos (UniProtKB/Swiss-Prot (2019_09)).
- Seleccione 'Digestión enzimática no especificada' para el parámetro enzimático, y 10 ppm y 0,01 Da para las tolerancias de masa en iones precursores y fragmentos, respectivamente.
NOTA: Las modificaciones variables son la desamidación (NQ) y la oxidación (M). Todos los demás parámetros de búsqueda son los valores predeterminados. Las listas finales de péptidos se filtran utilizando ALC del 80% y con una tasa de descubrimiento falso (FDR) del 1% utilizando el software de análisis proteómico LC-MS/MS.
5. Visualización de datos inmunopeptidómicos
NOTA: La calidad de los datos inmunopeptidómicos generados por la EM puede ser evaluada de múltiples maneras, como se describió recientemente22,23. Para visualizar los datos y evaluar su calidad general, composición y especificidad MHC, se puede utilizar la herramienta de software MhcVizPipe (MVP).
- Siga todas las instrucciones y la documentación relacionada para instalar y ejecutar el software MVP disponible en el sitio web de Caron Lab GitHub24.
NOTA: MVP proporciona una visión rápida y consolidada de la calidad de la muestra, la composición y la especificidad de MHC. MVP paraleliza el uso de algoritmos inmunopeptidómicos bien establecidos (NetMHCpan25, NetMHCIIpan26 y GibbsCluster27) y genera informes organizados y fáciles de entender en formato HTML (HyperText Markup Language). Los informes son totalmente portátiles y se pueden ver en cualquier computadora con un navegador web moderno. Consulte Datos complementarios 1-4 para ver ejemplos de informes HTML.
Resultados
El flujo de trabajo general para aislar complejos de péptidos MHC para el análisis de inmunopeptidomas por EM se ilustra en la Figura 1. Se muestran resultados representativos para la verificación de la eficiencia de unión a perlas del anticuerpo (Figura 2) (utilizando el anticuerpo anti-H2Kb Y3) y la eficiencia de elución de los complejos de péptidos MHC de las perlas (Figura 3) (utilizando el anticuerpo anti-HLA-ABC W6/32). También se aplicaron ensayos de cuantificación basados en citometría de flujo21 para medir el número absoluto de moléculas MHC clase I por célula EL4 (H2Kb y H2Db) y célula JY (HLA-ABC), como se muestra en la Figura 4A.
La reproducibilidad intra e interindividual de los resultados utilizando el protocolo actual se muestra en la Figura 4B-E. Se muestran resultados representativos para los péptidos MHC de clase I identificados a partir de 1 x 108 células EL4 y 1 x 108 células JY. Los resultados se generaron a partir de dos miembros diferentes del laboratorio (Usuario 1 y Usuario 2). Para el usuario 1, el número medio de péptidos específicos de MHCI detectados en células EL4 y JY fue de 1341 y 6361, respectivamente; para el usuario 2, 1933 y 7238, respectivamente (Figura 4B,D). El coeficiente de variación (CV) promedio para el número de péptidos detectados en tres réplicas/experimentos biológicos diferentes varía de 1.9% a 11% (Figura 4B, D). Aunque los CV para el número de péptidos detectados en los tres experimentos diferentes fueron relativamente pequeños, la identidad de los péptidos varió considerablemente (Figura 4C, E). De hecho, los diagramas representativos de Venn muestran que la proporción de péptidos que se detectaron de manera reproducible en tres réplicas biológicas varió del 39% (usuario 2, células EL4) al 63% (usuario 1, células JY) (Figura 4C, E y Tabla suplementaria 2).
Los mapas de calor generados por el software MVP muestran la fuerza de unión MHC predicha de los péptidos identificados utilizando las herramientas de la suite NetMHCpan25,26,28. Dos opciones de rutina de GibbsCluster, que se denominan 'GibbsCluster no supervisado' y 'GibbsCluster específico de alelo', también son realizadas por MVP para extraer motivos de unión a péptidos MHC. Tenga en cuenta que MVP tiene limitaciones; su objetivo principal no es extraer motivos específicos de alelos y anotar péptidos de una manera altamente precisa, sino más bien proporcionar una vista de pájaro sobre la calidad general, la composición y la especificidad MHC de las muestras.
Para los péptidos MHC de clase I (H2Db y H2Kb) de ratón en células EL4 (Figura 5A; paneles superiores y datos complementarios 1), un mapa de calor representativo muestra que se predice que el 89% de todos los péptidos 8-12-mer detectados son aglutinantes fuertes (SB: NetMHCpan %Rank <0.5) o aglutinantes débiles (WB: NetMHCpan %Rank <2) para moléculas H2Db o H2Kb. Los grupos de secuencia generados a partir de los péptidos 8-12-mer están en concordancia con los logotipos reportados para H2Db (asparagina en P5 y leucina en P9) y H2Kb (fenilalanina en P5 y leucina en P8) (Figura 5)29. Vale la pena mencionar que un tercer motivo dominante (histidina en P7 y leucina en P9), así como motivos "artefactuales" adicionales se pueden observar utilizando el anticuerpo M1 (Datos complementarios 1). De hecho, se sabe que el anticuerpo M1 reacciona de forma cruzada con la molécula no clásica Qa2 y, por lo tanto, los péptidos asociados a Qa2 también son detectados por la EM (Datos complementarios 1). Aquí, para simplificar, la Figura 5 se centra en mostrar los motivos de unión peptídica bien establecidos para los dos alelos H2b clásicos (es decir, H2Db o H2Kb) expresados en células EL4.
Para los péptidos humanos HLA clase I (HLA-ABC) en células JY (Figura 5A; paneles inferiores y datos complementarios 2), un mapa de calor representativo muestra que se predice que el 97% de todos los péptidos 8-12-mer detectados son SB o WB para HLA-A * 0201, -B * 0702 o -C * 0702. Los péptidos se agruparon para visualizar los motivos de unión a péptidos para HLA-A* 0201 y -B * 0702. El motivo de unión para HLA-C*0702 no se muestra en la Figura 5A porque el alelo C*0702 tiene un nivel de expresión relativamente bajo. Por lo tanto, se aislaron e identificaron muy pocos péptidos C*0702 para generar un motivo C*0702 representativo. Tenga en cuenta que el motivo C*0702 se puede visualizar en otros estudios30,31,32 o desde el sitio web de NetMHCpan 4.1 Motif Viewer33.
Para los péptidos humanos HLA clase II (HLA-DR) en células JY (Figura 5B; paneles superiores y datos complementarios S3), un mapa de calor representativo muestra que se predice que el 70% de todos los péptidos 9-22-mer detectados son SB o WB para HLA-DRB1 * 0404 y -DRB1 * 1301. Se muestran los motivos de unión peptídica para estos dos alelos (Figura 5B). Tenga en cuenta que los motivos de unión a péptidos que se muestran aquí pueden no estar en completa concordancia con los logotipos recientemente informados para HLA-DRB1 * 0404 y -DRB1 * 130134. Esta discrepancia pone de relieve la incapacidad actual de MVP/NetMHCpan para anotar con precisión péptidos a alelos HLA clase II que están menos caracterizados, como HLA-DRB1*0404 y -DRB1*1301 expresados en células JY. Se puede encontrar información adicional sobre los motivos de unión a péptidos de clase II en otros estudios34,35 y en el sitio web NetMHCIIpan 4.0 Motif Viewer36.
Finalmente, para los péptidos MHC de clase II (H2-IAd y H2-IEd) de ratón en células A20 (Figura 5B; paneles inferiores y datos complementarios 4), un mapa de calor representativo muestra que se predice que el 87% de todos los péptidos 9-22-mer detectados son SB o WB para H2-IAd o H2-IEd. Los motivos de unión peptídica para estos dos alelos están en concordancia con los logos reportados37.
Los informes HTML completos generados por el software MVP para evaluar la calidad general y la especificidad MHC de las muestras están disponibles en los datos complementarios 1-4.
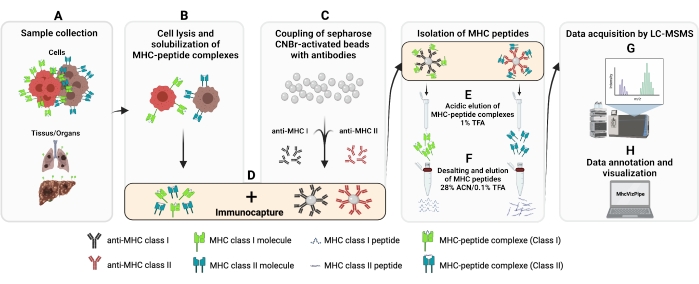
Figura 1: Esquema del procedimiento completo para el aislamiento de péptidos MHC clase I y II. (A-B)100 millones de células se granulan y lisan con un tampón de Chaps del 0,5%. (C) El lisado celular se centrifuga y el sobrenadante se agrega a las perlas de CNBr-sefarosa acopladas al anticuerpo deseado de antemano y (D) se incuba de 14 a 18 h a 4 °C. (E) Después de la inmunocaptura, las perlas se transfieren a una columna de polipropileno y se lavan, y los complejos de péptidos MHC se eluyen con una solución de TFA al 1%. (F) Los péptidos se desalan y eluyen utilizando una columna C18. (G) Posteriormente, los péptidos se secan a velocidad de vacío y se analizan mediante espectrometría de masas en tándem. (H) La calidad de los péptidos aislados MHC clase I y II puede evaluarse en función de los subtipos HLA utilizando el software MhcVizPipe disponible gratuitamente. Figura creada con BioRender.com (NT22ZL8QSL). Haga clic aquí para ver una versión más grande de esta figura.
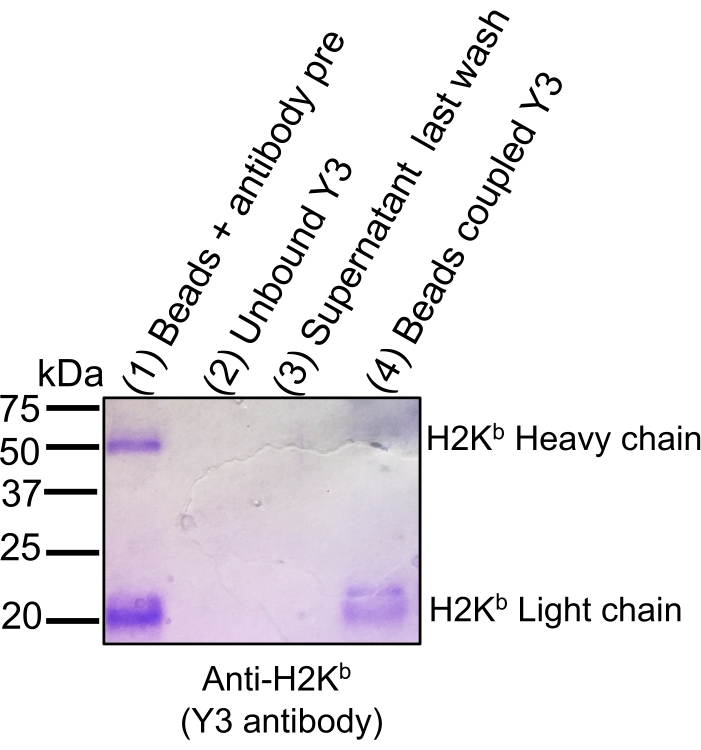
Figura 2: Tinción en gel de coomassie para rastrear la eficiencia de unión de anticuerpos a las perlas activadas por CNBr de sefalrosa. Las alícuotas de volúmenes equivalentes se cargaron en gel SDS-PAGE al 12% seguido de tinción azul de Coomassie: perlas + preacoplamiento de anticuerpos (1), anticuerpo no unido después del paso de acoplamiento (2), sobrenadante después del último lavado después del acoplamiento (3) y perlas acopladas con anticuerpos (4). La eficiencia de la unión se ilustra con una disminución significativa en la intensidad de tinción de la señal de las cadenas ligeras y pesadas de H2Kb cuando las perlas se unen covalentemente (Carril 4) a las perlas CNBr en comparación con el anticuerpo antes del acoplamiento (Carril 1). La figura ha sido reimpresa y adaptada de bioRxiv38. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Western blotting para el seguimiento de complejos de péptidos MHC después de la elución ácida de perlas activadas por CNBr acopladas a anticuerpos. Las alícuotas tomadas de los pasos indicados en el protocolo (1/100 del volumen total medido) se cargaron en un gel SDS-PAGE al 12% y se transfirieron a la membrana de nitrocelulosa: Perlas + lisado después de la inmunocaptura y el último lavado (A); sobrenadante de los complejos de último lavado (B) y MHC-péptido eluidos de las perlas (C). La fuerte señal de detección de los complejos MHC-péptido en (C) utilizando anticuerpos de cadena pesada anti-HLA-ABC (Abcam, #ab 70328, 1:5000) confirmó el aislamiento de complejos de péptidos MHC después de la elución ácida. Tenga en cuenta que es posible evaluar la proporción de complejos de péptidos MHC que no fueron capturados por las perlas acopladas a anticuerpos recolectando el flujo a través del paso 3.1.6. Se puede agregar una alícuota al western blot (no se muestra en este gel). Haga clic aquí para ver una versión más grande de esta figura.
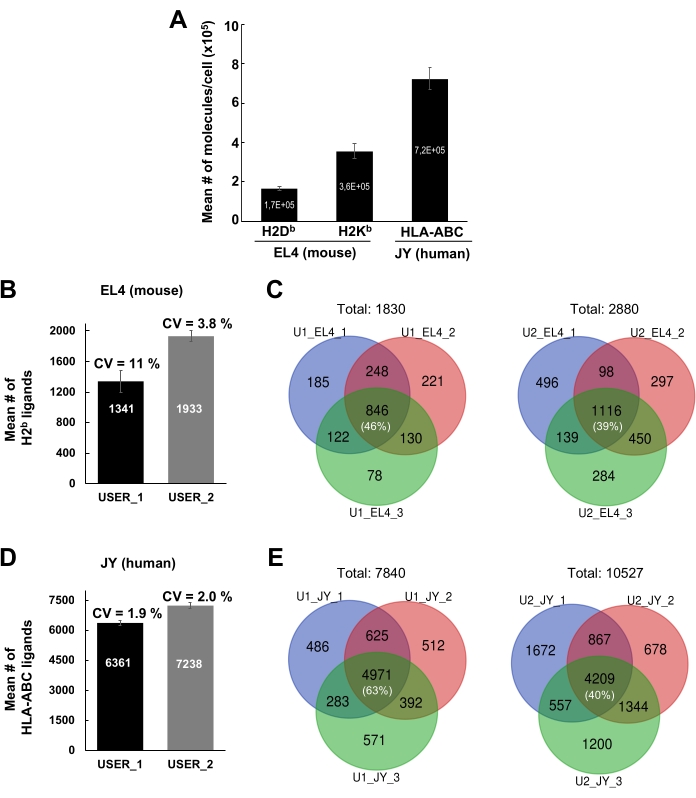
Figura 4: Identificación de péptidos MHC clase I de células JY y EL4. (A) Histograma que muestra el número absoluto de moléculas H2Db y H2Kb de la superficie celular por célula EL4 y de moléculas HLA-ABC por célula JY. La cuantificación se realizó mediante citometría de flujo. El error medio y estándar de la media se obtuvieron a partir de tres réplicas biológicas. (B, D) Histograma que muestra el número medio y la desviación estándar de la media de los péptidos MHC clase I identificados por dos usuarios independientes (USER_1 [U1] y USER_2 [U2]). Se muestra el número medio y la desviación estándar de la media de los péptidos MHC de clase I detectados en células EL4 (B) de ratón y células JY humanas (D). Se indican coeficientes de variación (CV) en tres réplicas biológicas independientes. (C, E) Diagramas de Venn que muestran el número de péptidos que fueron detectados reproduciblemente en células EL4 (C) y JY (E) por dos usuarios independientes (U1 y U2) a través de tres réplicas biológicas independientes. La Figura 4A ha sido reimpresa y adaptada de bioRxiv38. Haga clic aquí para ver una versión más grande de esta figura.
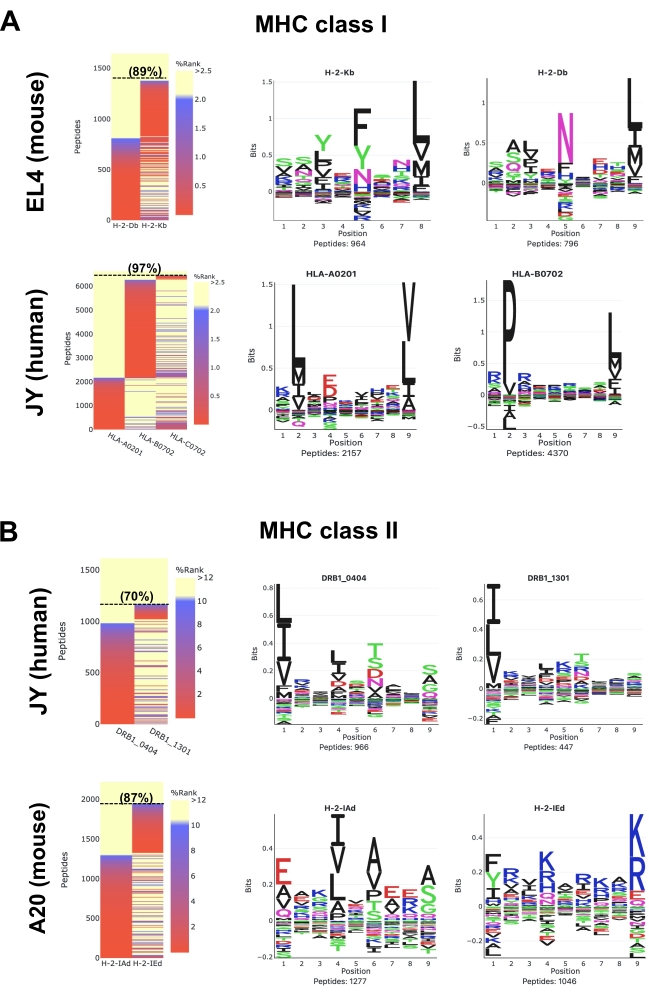
Figura 5: Visualización de datos inmunopeptidómicos utilizando la herramienta de software MVP. Análisis de datos de péptidos MHC de ratón y humanos (A) clase I y (B) MHC clase II. Se muestran mapas de calor de afinidad de unión representativos (paneles izquierdos) y motivos de unión peptídica (paneles derechos). Los colores de los mapas de calor representan la afinidad de unión MHC predicha por NetMHCpan 4.1 (%rank). Los aglutinantes fuertes son rojos (%rango <0.5), los aglutinantes débiles son azules (%rango <2) y los no aglutinantes son amarillos (%Rango >2). La proporción (%) de péptidos 8-12mer (A) y péptidos 9-22mer (B) que son SB o WB está indicada entre paréntesis. Los motivos de unión peptídica fueron generados por MVP utilizando la opción 'Gibbscluster específico de alelo'. Estos resultados representativos se obtuvieron a partir de 1 x 108 células y mediante el uso de los siguientes anticuerpos y líneas celulares: anticuerpos M1 y células EL4 para péptidos MHC clase I de ratón; Anticuerpo w6/32 y células JY para péptidos humanos MHC clase I; Anticuerpo m5 y células A20 para péptidos MHC clase II de ratón; Anticuerpo L243 y células JY para péptidos humanos MHC clase II. Consulte los Datos suplementarios 1-4 para acceder a los informes HTML completos generados por el software MVP. Haga clic aquí para ver una versión más grande de esta figura.
Datos complementarios 1-4: Haga clic aquí para descargar este archivo.
Datos complementarios 1: Informe HTML generado por el software MVP a partir de péptidos que se aislaron de 1 x 108 CÉLULASEL4 utilizando el anticuerpo M1. Se muestran tres réplicas biológicas. Este informe está relacionado con la Figura 4 y la Figura 5 y los péptidos representativos se enumeran en la Tabla suplementaria 2.
Datos complementarios 2: Informe HTML generado por el software MVP a partir de péptidos que se aislaron de 1 x 108JY células utilizando el anticuerpo W6/32. Se muestran tres replates biológicos. Este informe está relacionado con la Figura 4 y la Figura 5, y los péptidos representativos se enumeran en la Tabla suplementaria 2.
Datos complementarios 3: Informe HTML generado por el software MVP a partir de péptidos que se aislaron de 1 x 108JY células utilizando el anticuerpo L243. Este informe está relacionado con la Figura 5, y los péptidos representativos se enumeran en la Tabla Suplementaria 2.
Datos complementarios 4: Informe HTML generado por el software MVP a partir de péptidos que se aislaron de 1 x 108A20 células utilizando el anticuerpo M5. Este informe está relacionado con la Figura 5, y los péptidos representativos se enumeran en la Tabla Suplementaria 2.
Cuadro complementario 1: Lista de colchones. Se describen recetas para todos los búferes utilizados en el protocolo. Haga clic aquí para descargar esta tabla.
Tabla suplementaria 2: Listas representativas de péptidos asociados con H2Db/Kb, HLA-ABC, HLA-DR y H2-IAd/IEd. Esta tabla contiene las listas de péptidos representativos MHC clase I y II aislados de líneas celulares EL4 y A20 de ratón, respectivamente, y HLA clase I y II de línea celular JY humana. Estos datos han sido depositados en el ProteomeXchange (PXD028633). Haga clic aquí para descargar esta tabla.
DISPONIBILIDAD DE DATOS:
Los conjuntos de datos utilizados en este manuscrito se depositaron en el ProteomeXchange (http://proteomecentral.proteomexchange.org/cgi/GetDataset): PXD028633.
Discusión
Dos líneas celulares de ratón (EL4 y A20), una línea celular humana (JY) y cinco anticuerpos disponibles comercialmente [M1 (anti-H2Db/Kb), Y3 (anti-H2Kb), M5 (anti-H2-IAd/IEd), W6/32 (anti-HLA-ABC) y L243 (anti-HLA-DR)] fueron probados y validados en el contexto de este protocolo y proporcionan datos inmunopeptidómicos de alta calidad. Otros anticuerpos anti-HLA están disponibles (por ejemplo, anti-HLA-A2 BB7.2) pero no se probaron aquí. Tenga en cuenta que el anticuerpo W6/32 es ampliamente utilizado y el anticuerpo más establecido en el campo; permite el aislamiento de péptidos presentados por todas las moléculas HLA-ABC en humanos y fue previamente reportado por laboratorios expertos para trabajar a partir de diversas fuentes biológicas como tejidos frescos o congelados8,39, células mononucleares de sangre periférica y células mononucleares de médula ósea40, biopsias41, xenoinjertos41,42, autopsias43 y muestras plasmáticas44,45.
La preparación de soluciones frescas que se utilizan a lo largo del protocolo es crítica. En particular, el uso de soluciones ácidas frescas en botellas de vidrio es fundamental para evitar la contaminación posterior de las muestras analizadas por MS. Además, cuando el protocolo se realiza por primera vez y/o con un nuevo anticuerpo, es importante evaluar que el anticuerpo está realmente acoplado a las perlas de SEpharose CNBr utilizando un gel azul de Coomassie. Los pasos de lavado de las perlas acopladas a anticuerpos después de la inmunocaptura de los complejos de péptidos MHC también son críticos para evitar la contaminación de péptidos no MHC. Finalmente, se requiere un cuidado especial para no desechar los eluidos después de la elución de los complejos MHC-péptido con 1% TFA y la elución de los péptidos de la columna C18 con ACN28% / 0.1% FA.
Los protocolos existentes disponibles en la literatura describen pasos adicionales para purificar aún más los péptidos al final del procedimiento de aislamiento, por ejemplo, fraccionamiento de péptidos por diferentes métodos14,46 o ultrafiltración utilizando filtros de 10-30 kDa13,47. El protocolo actual no proporciona detalles sobre esos pasos adicionales y es suficiente para proporcionar datos inmunopeptidómicos de alta calidad. Sin embargo, tales pasos podrían ser considerados por no expertos para modificar y solucionar problemas para optimizar aún más el procedimiento de aislamiento de péptidos.
El tipo de perlas y el tipo de tampón de elución ácida que se utilizan para eluir los complejos MHC de las perlas también se pueden modificar para la solución de problemas13,14,15,16,17,18,19. En este sentido, las perlas activadas por sefarosa CNBr son generalmente un buen punto de partida, ya que son relativamente baratas además de mostrar flexibilidad en términos de unión con varios tipos de anticuerpos. En el protocolo actual, se demostró que las perlas activadas por sefarosa CNBr funcionan relativamente bien utilizando cinco anticuerpos diferentes disponibles comercialmente (es decir, M1, Y3, W6/32, L243 y M5). Además de las perlas activadas por sefarosa CNBr, las perlas de flujo rápido de proteína A o G o A / G de sefarosa 4 también son fáciles de manejar y, aunque relativamente más caras, pueden generar resultados similares. Otro factor a considerar es la afinidad del anticuerpo por la proteína A o G. Además, las cuentas magnéticas de sefarosa también son muy fáciles de usar, pero son relativamente caras. Independientemente del tipo de perlas seleccionadas por los no expertos, se recomienda recolectar alícuotas en los pasos críticos del protocolo y realizar geles SDS-PAGE teñidos de Coomassie azul para rastrear la eficiencia de unión del anticuerpo a las perlas, como se muestra en la Figura 2.
Otro factor importante que influye en el éxito del aislamiento de péptidos MHC se refiere al tipo de tampón de elución ácida utilizado para aislar los complejos MHC-péptidos de las perlas. Se han reportado diferentes tampones que incluyen 0.1%, 1% o 10% TFA, 0.2% FA y 10% ácido acético. 1% TFA fue el tampón de elución que funcionó para todos los anticuerpos probados. Este paso también podría rastrearse mediante western blotting contra las moléculas MHC utilizadas para capturar péptidos MHC, como se muestra en la Figura 3.
Todos los tampones que contienen acetonitrilo (ACN) y/o ácido trifluoroacético (TFA) son agresivos y pueden conducir a la contaminación de la muestra con pequeñas sustancias moleculares y poliméricas como plastificantes si están en contacto con plástico. Para evitar tales problemas, todas las soluciones que contienen un disolvente orgánico y / o TFA se preparan frescas diariamente y se almacenan en una botella de vidrio hasta su uso. La mayoría de los pasos se realizan en el tubo de plástico Protein LoBind. Estos tubos están diseñados específicamente para la proteómica y están hechos de polipropileno virgen de la más alta calidad, libre de biocidas, plastificantes y látex. También se producen con moldes optimizados y altamente pulidos sin el uso de agentes antideslizantes. Es importante tener en cuenta estas precauciones para permitir la generación de datos inmunopeptidómicos de alta calidad.
El anticuerpo es una limitación importante para el aislamiento de péptidos unidos a MHC. El anticuerpo W6/32 permite el aislamiento de péptidos presentados por todas las moléculas HLA-ABC de clase I en humanos y es el anticuerpo más utilizado y establecido en el campo. La tipificación HLA de alta resolución de líneas celulares humanas o bioespecímenes no caracterizados no es una necesidad tras la aplicación del anticuerpo W6/32, pero se recomienda para ciertas aplicaciones para facilitar la interpretación de los datos48. La información de tipificación HLA/MHC también se puede encontrar en recursos públicos para múltiples líneas celulares y modelos de ratón49. Además del anticuerpo W6/32, los otros cuatro anticuerpos (M1, M5, Y3 y L243) que se probaron y validaron en el contexto de este protocolo están disponibles comercialmente. Por otro lado, muchos otros anticuerpos que se informaron en estudios previos de inmunopeptidomica no han sido adoptados en gran medida por la comunidad y no están disponibles comercialmente o están disponibles a través del cultivo de líneas celulares de hibridoma, que es relativamente costoso.
Otra limitación importante para el aislamiento de péptidos unidos a MHC es la cantidad de material de partida requerido. La cantidad requerida es inversamente proporcional al nivel de expresión de las moléculas de MHC en la superficie celular, que puede cuantificarse mediante citometría de flujo (Figura 4A). Las células que muestran altos niveles de expresión de moléculas MHC (por ejemplo, células dendríticas y células hematopoyéticas en general) generalmente producen datos inmunopeptidómicos de alta calidad. Los laboratorios expertos utilizan desde tan solo 50 millones de células50, pero se recomiendan de 100 millones a 1.000 millones de células para los no expertos. También se notificó el uso de biopsia de tejido (<13 mg)41, xenoinjerto42,43, autopsia44 y plasma45,46 muestras, pero sigue siendo un desafío para los laboratorios no expertos. Además, el número total de péptidos asociados a MHC esperado está bien documentado para las líneas celulares establecidas (aquí, entre ~ 2000 y ~ 10000 péptidos dependiendo de la línea celular y el anticuerpo utilizado), pero las cantidades absolutas de péptidos presentados naturalmente que son eficientemente arrastrados hacia abajo por la técnica siguen siendo objeto de debate. De hecho, estudios previos estimaron que la eficiencia del procedimiento de aislamiento depende del péptido y puede ser tan baja como 0.5% -2%51. Otras limitaciones en inmunopeptidomics son la reproducibilidad de los métodos y la incapacidad de las herramientas de la suite NetMHCpan para anotar correctamente los péptidos a los alelos MHC que están menos caracterizados. A este respecto, se seguirá desarrollando y aplicando métodos de MS de adquisición independientes de los datos relativamente nuevos7,32,52, así como nuevos algoritmos de predicción de agrupación de péptidos y de unión a péptidos MHC31,34,53,54 se espera que mejore la reproducibilidad y la precisión de la anotación peptídica en inmunopeptidómica. La inmunopeptidómica se enfrenta a otras limitaciones con respecto a la adquisición de EM y el análisis computacional de péptidos asociados a MHC y se cubren en otros lugares1,6,55.
Si bien el aislamiento de péptidos asociados a HLA-ABC de muestras humanas utilizando el anticuerpo W6/32 está bien establecido y es ampliamente aplicado por muchos grupos de investigación, el aislamiento de péptidos asociados a MHC de clase I y II de ratón está relativamente menos establecido. Por lo tanto, se necesitan protocolos robustos para el aislamiento de ligandos MHC de ratón. Aquí, proporcionamos un protocolo optimizado para el aislamiento de péptidos MHC clase I y péptidos MHC clase II de dos líneas celulares de ratón de origen C57BL/6 y BALB/c, respectivamente. Específicamente, el protocolo permite el aislamiento de péptidos asociados a H2Kb y H2Db de clase I utilizando el anticuerpo M1, así como péptidos asociados a H2-IAd y H2-IEd de clase II utilizando el anticuerpo M5. Por lo tanto, la difusión y aplicación del protocolo actual debería facilitar la investigación inmunopeptidómica básica y traslacional en varios modelos de ratón.
El protocolo se puede utilizar para establecer y estandarizar un flujo de trabajo de inmunopeptidomica, así como para comparar nuevos protocolos. Por ejemplo, se puede adaptar y optimizar aún más para realizar el cribado de inmunopeptidómica en diversas matrices biológicas que van desde sangre / plasma hasta tejido fresco o congelado y FFPE (Formalin-Fixed Paraffin-Embedded). Además, el protocolo fomentará la reproducibilidad intra e interlaboratorio del procedimiento de preparación de muestras en inmunopeptidómica y, por lo tanto, se espera que encuentre una amplia aplicación en la investigación básica y clínica.
Divulgaciones
Los autores declaran que no tienen intereses financieros contrapuestos.
Agradecimientos
Agradecemos a Pierre Thibault, Eric Bonneil, Joël Lanoix, Caroline Côté (Instituto de Investigación en Inmunología y Cáncer, Universidad de Montreal) y Anthony Purcell (Universidad de Monash) por sus perspicaces comentarios. Este trabajo fue apoyado por fondos del Fonds de recherche du Québec - Santé (FRQS), la Fundación Cole, CHU Sainte-Justine y las Fundaciones Charles-Bruneau, la Fundación Canadiense para la Innovación, el Consejo Nacional de Investigación en Ciencias e Ingeniería (NSERC) (#RGPIN-2020-05232) y los Institutos Canadienses de Investigación en Salud (CIHR) (# 174924).
Materiales
| Name | Company | Catalog Number | Comments |
| A20 cell line | ATCC | TIB-208 | mouse B lymphoblast |
| Acetonitrile, LC/MS Grade | FisherScientific | A955-4 | |
| anti-Human HLA A, B, C (W6/32) - MHC class I | BioXcell | BE0079 | |
| anti-Human/Monkey HLA-DR (L243) - MHC class II | BioXcell | BE0308 | |
| anti-Mouse H2 (M1/42.3.9.8) - MHC class I | BioXcell | BE0077 | |
| anti-Mouse H2-IAd/IEd (M5/114) - MHC class II | BioXcell | BE00108 | |
| anti-Mouse H2Kb (Y3) - MHC class I | BioXcell | BE0172 | |
| BupH Phosphate Buffered Saline Packs (PBS) | ThermoFisher | 28372 | Pouch contents dissolved in a final volume of 500 mL deionized water (FisherScientific, W64) |
| CHAPS (3-[(3-cholamidopropyl)dimethylammonio]-1-propanesulfonate) | EMDMilipore | 220201-10MG | |
| CNBr-activated Sepharose | Cytivia | # 17-0430-01 | |
| EL4 cell line | ATCC | TIB-39 | mouse T lymphoblast |
| epTIPS LoRetention Tips, 1000 µL/Eppendorf | FisherScientific | 02-717-352 | Better results with low retention material |
| epTIPS LoRetention Tips, 200 µL/Eppendorf | FisherScientific | 02-717-351 | Better results with low retention material |
| Formic Acid, LC/MS Grade | FisherScientific | A117-50 | |
| Glycine | FisherScientific | RDCG0250500 | |
| Hydrochloric acid solution | FisherScientific | 60-007-11 | |
| JY cell line | Sigma Aldrich | 94022533-1VL | EBV-immortalised B cell lymphoblastoid line |
| Methanol, LC/MS Grade | FisherScientific | A456-4 | |
| Poly prep chromatography columns (polypropylene column) | Bio-Rad | 731-1550 | referred as polypropylene column in the protocol |
| Proteases inhibitor | ThermoFisher | A32963 | 1 pellet per 10 mL of cell lysis buffer |
| Qifikit | Dako | K007811-8 | |
| Sodium Bicarbonate | Amresco | # 0865-1kg | |
| Sodium Chloride | FisherScientific | MSX04201 | |
| Solid phase extraction disk, ultramicrospin column C18 | The nest group | SEMSS18V | capacity of 6–60 µg, max volume of 200 µL |
| Trifluoroacetic Acid (TFA), LC-MS Grade | FisherScientific | PI85183 | |
| Tris | FisherScientific | T395-500 | |
| Tris-HCl | FisherScientific | #10812846001 | |
| Tube LoBind 1.5 mL/Eppendorf | FisherScientific | E925000090 | Better results with low retention material |
| Tube LoBind 2 mL/Eppendorf | FisherScientific | 13-698-795 | Better results with low retention material |
| Water, LC/MS Grade | FisherScientific | W64 |
Referencias
- Vizcaíno, J. A., et al. The human immunopeptidome project: A roadmap to predict and treat immune diseases. Molecular & Cellular Proteomics. 19 (1), 31-49 (2019).
- Caron, E., Aebersold, R., Banaei-Esfahani, A., Chong, C., Bassani-Sternberg, M. A case for a human immuno-peptidome project consortium. Immunity. 47 (2), 203-208 (2017).
- Arnaud, M., Duchamp, M., Bobisse, S., Renaud, P., Coukos, G., Harari, A. Biotechnologies to tackle the challenge of neoantigen identification. Current Opinion in Biotechnology. 65, 52-59 (2020).
- Caron, E., et al. The MHC I immunopeptidome conveys to the cell surface an integrative view of cellular regulation. Molecular Systems Biology. 7 (1), 533 (2011).
- Istrail, S., et al. Comparative immunopeptidomics of humans and their pathogens. Proceedings of the National Academy of Sciences of the United States of America. 101 (36), 13268-13272 (2004).
- Caron, E., Kowalewski, D. J., Koh, C. C., Sturm, T., Schuster, H., Aebersold, R. Analysis of major histocompatibility complex (MHC) immunopeptidomes using mass spectrometry. Molecular & Cellular Proteomics. 14 (12), 3105-3117 (2015).
- Caron, E., et al. An open-source computational and data resource to analyze digital maps of immunopeptidomes. eLife. 4, 07661 (2015).
- Bassani-Sternberg, M., et al. Direct identification of clinically relevant neoepitopes presented on native human melanoma tissue by mass spectrometry. Nature Communications. 7 (1), 13404 (2016).
- Gubin, M. M., et al. Checkpoint blockade cancer immunotherapy targets tumour-specific mutant antigens. Nature. 515 (7528), 577 (2014).
- Yadav, M., et al. Predicting immunogenic tumour mutations by combining mass spectrometry and exome sequencing. Nature. 515 (7528), 572-576 (2015).
- Laumont, C. M., et al. Noncoding regions are the main source of targetable tumor-specific antigens. Science Translational Medicine. 10 (470), 5516 (2018).
- Lill, J. R., et al. Minimal information about an immuno-peptidomics experiment (MIAIPE). Proteomics. 18 (12), 1800110 (2018).
- Nelde, A., Kowalewski, D. J., Stevanović, S. Antigen processing, methods and protocols. Methods in molecular biology. 1988, 123-136 (2019).
- Purcell, A. W., Ramarathinam, S. H., Ternette, N. Mass spectrometry-based identification of MHC-bound peptides for immunopeptidomics. Nature Protocols. 14 (6), 1687-1707 (2019).
- Ebrahimi-Nik, H., et al. Mass spectrometry driven exploration reveals nuances of neoepitope-driven tumor rejection. JCI Insight. 5 (14), 129152 (2019).
- Schuster, H., et al. A tissue-based draft map of the murine MHC class I immunopeptidome. Scientific Data. 5, 180157 (2018).
- Ritz, D., Gloger, A., Weide, B., Garbe, C., Neri, D., Fugmann, T. High-sensitivity HLA class I peptidome analysis enables a precise definition of peptide motifs and the identification of peptides from cell lines and patients sera. PROTEOMICS. 16 (10), 1570-1580 (2016).
- Bassani-Sternberg, M. Mass spectrometry based immunopeptidomics for the discovery of cancer neoantigens. Methods Mol Biol. 1719, 209-221 (2018).
- Lanoix, J., et al. Comparison of the MHC I Immunopeptidome Repertoire of B-Cell Lymphoblasts Using Two Isolation Methods. Proteomics. 18 (12), 1700251 (2018).
- Kuznetsov, A., Voronina, A., Govorun, V., Arapidi, G. Critical review of existing MHC I immunopeptidome isolation methods. Molecules. 25 (22), 5409 (2020).
- Urlaub, D., Watzl, C. Coated latex beads as artificial cells for quantitative investigations of receptor/ligand interactions. Current Protocols in Immunology. 131 (1), 111 (2020).
- Ghosh, M., et al. Guidance document: Validation of a high-performance liquid chromatography-tandem mass spectrometry immunopeptidomics assay for the identification of HLA class I ligands suitable for pharmaceutical therapies*. Molecular & Cellular Proteomics. 19 (3), 432-443 (2020).
- Fritsche, J., et al. Pitfalls in HLA ligandomics - How to catch a li(e)gand. Molecular & Cellular Proteomics. 20, 100110 (2021).
- Caron Lab. GitHub Available from: https://github.com/CaronLab/MhcVizPipe (2021)
- Jurtz, V., Paul, S., Andreatta, M., Marcatili, P., Peters, B., Nielsen, M. NetMHCpan-4.0: Improved peptide-MHC Class I interaction predictions integrating eluted ligand and peptide binding affinity data. The Journal of Immunology. 199 (9), 3360-3368 (2017).
- Reynisson, B., Alvarez, B., Paul, S., Peters, B., Nielsen, M. NetMHCpan-4.1 and NetMHCIIpan-4.0: improved predictions of MHC antigen presentation by concurrent motif deconvolution and integration of MS MHC eluted ligand data. Nucleic Acids Research. 48, 449-454 (2020).
- Andreatta, M., Alvarez, B., Nielsen, M. GibbsCluster: unsupervised clustering and alignment of peptide sequences. Nucleic Acids Research. 45 (1), 458-463 (2017).
- Nielsen, M., et al. NetMHCpan, a method for quantitative predictions of peptide binding to any HLA-A and -B Locus protein of known sequence. PLoS ONE. 2 (8), 796 (2007).
- Fortier, M. -. H., et al. The MHC class I peptide repertoire is molded by the transcriptome. The Journal of Experimental Medicine. 205 (3), 595-610 (2008).
- Marco, M. D., Schuster, H., Backert, L., Ghosh, M., Rammensee, H. -. G., Stevanovic, S. Unveiling the peptide motifs of HLA-C and HLA-G from naturally presented peptides and generation of binding prediction matrices. The Journal of Immunology. 199 (8), 2639-2651 (2017).
- Sarkizova, S., et al. A large peptidome dataset improves HLA class I epitope prediction across most of the human population. Nature Biotechnology. 38 (2), 199-209 (2019).
- Pak, H., et al. . Sensitive immunopeptidomics by leveraging available large-scale multi-HLA spectral libraries, data-independent acquisition and MS/MS prediction. 20, 100080 (2021).
- Racle, J., et al. Robust prediction of HLA class II epitopes by deep motif deconvolution of immunopeptidomes. Nature Biotechnology. 37 (11), 1283-1286 (2019).
- Abelin, J. G., et al. Defining HLA-II ligand processing and binding rules with mass spectrometry enhances cancer epitope prediction. Immunity. 51 (4), 766-779 (2019).
- Sofron, A., Ritz, D., Neri, D., Fugmann, T. High-resolution analysis of the murine MHC class II immunopeptidome. European Journal of Immunology. 46 (2), 319-328 (2015).
- Kovalchik, K. A., et al. Immunopeptidomics for Dummies: Detailed Experimental Protocols and Rapid, User-Friendly Visualization of MHC I and II Ligand Datasets with MhcVizPipe. bioRxiv. , (2020).
- Schuster, H., et al. The immunopeptidomic landscape of ovarian carcinomas. Proceedings of the National Academy of Sciences. 114 (46), 9942-9951 (2017).
- Berlin, C., et al. Mapping the HLA ligandome landscape of acute myeloid leukemia: a targeted approach toward peptide-based immunotherapy. Leukemia. 29 (3), 1-13 (2014).
- Rijensky, N. M., et al. Identification of tumor antigens in the HLA peptidome of patient-derived xenograft tumors in mouse. Molecular & Cellular Proteomics. 19 (8), 1360-1374 (2020).
- Heather, J. M., et al. Murine xenograft bioreactors for human immunopeptidome discovery. Scientific Reports. 9 (1), 18558 (2019).
- Marcu, A., et al. HLA ligand atlas: A benign reference of HLA-presented peptides to improve T-cell-based cancer immunotherapy. Journal for Immunotherapy of Cancer. 9 (4), 002071 (2021).
- Shraibman, B., et al. Identification of tumor antigens among the HLA peptidomes of glioblastoma tumors and plasma. Molecular & Cellular Proteomics. 18 (6), 1255-1268 (2019).
- Bassani-Sternberg, M., Barnea, E., Beer, I., Avivi, I., Katz, T., Admon, A. Soluble plasma HLA peptidome as a potential source for cancer biomarkers. Proceedings of the National Academy of Sciences. 107 (44), 18769-18776 (2010).
- Demmers, L. C., Heck, A. J. R., Wu, W. Pre-fractionation extends, but also creates a bias in the detectable HLA class Ι ligandome. Journal of Proteome Research. 18 (4), 1634-1643 (2019).
- Kowalewski, D. J., Stevanović, S. Antigen processing,. Methods and Protocols. 960, 145-157 (2013).
- Bentley, G., et al. High-resolution, high-throughput HLA genotyping by next-generation sequencing. Tissue Antigens. 74 (5), 393-403 (2009).
- Boegel, S., Löwer, M., Bukur, T., Sahin, U., Castle, J. C. A catalog of HLA type, HLA expression, and neo-epitope candidates in human cancer cell lines. OncoImmunology. 3 (8), 954893 (2014).
- Klaeger, S., et al. Optimized liquid and gas phase fractionation increases HLA-peptidome coverage for primary cell and tissue samples. Molecular & Cellular Proteomics. 20, 100133 (2021).
- Hassan, C., et al. Accurate quantitation of MHC-bound peptides by application of isotopically labeled peptide MHC complexes. Journal of Proteomics. 109, 240-244 (2014).
- Ritz, D., Kinzi, J., Neri, D., Fugmann, T. Data-independent acquisition of HLA Class I peptidomes on the Q exactive mass spectrometer platform. Proteomics. 17 (19), (2017).
- O'Donnell, T. J., Rubinsteyn, A., Laserson, U. MHCflurry 2.0: Improved pan-allele prediction of MHC Class I-presented peptides by incorporating antigen processing. Cell Systems. 11 (1), 42-48 (2020).
- O'Donnell, T. J., Rubinsteyn, A., Bonsack, M., Riemer, A. B., Laserson, U., Hammerbacher, J. MHCflurry: Open-source Class I MHC binding affinity prediction. Cell Systems. 7 (1), 129-132 (2018).
- Faridi, P., Purcell, A. W., Croft, N. P. In immunopeptidomics we need a sniper instead of a shotgun. Proteomics. 18 (12), 1700464 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados