Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Explorando la interacción biomolecular entre la chaperona molecular Hsp90 y su proteína quinasa cliente Cdc37 utilizando tecnología de biodetección de efecto de campo
En este artículo
Resumen
La biodetección de efectos de campo (FEB) es una técnica sin etiquetas para detectar interacciones biomoleculares. Mide la corriente eléctrica a través del biosensor de grafeno al que se inmovilizan los objetivos de unión. Se utilizó la tecnología FEB para evaluar las interacciones biomoleculares entre Hsp90 y Cdc37 y se detectó una fuerte interacción entre las dos proteínas.
Resumen
Las interacciones biomoleculares desempeñan un papel versátil en numerosos procesos celulares al regular y coordinar eventos biológicos funcionalmente relevantes. Las biomoléculas como proteínas, carbohidratos, vitaminas, ácidos grasos, ácidos nucleicos y enzimas son componentes fundamentales de los seres vivos; se ensamblan en redes complejas en biosistemas para sincronizar una miríada de eventos de la vida. Las proteínas suelen utilizar redes complejas de interactomas para llevar a cabo sus funciones; por lo tanto, es obligatorio evaluar tales interacciones para desentrañar su importancia en las células tanto a nivel celular como de organismo. Hacia este objetivo, introducimos una tecnología rápidamente emergente, la biodetección de efecto de campo (FEB), para determinar interacciones biomoleculares específicas. FEB es una técnica de detección biomolecular confiable y de sobremesa, sin etiquetas para determinar interacciones específicas y utiliza biosensores electrónicos de alta calidad. La tecnología FEB puede monitorear las interacciones en el rango nanomolar debido a los nanomateriales biocompatibles utilizados en su superficie de biosensor. Como prueba de concepto, se dilucidó la interacción proteína-proteína (PPI) entre la proteína de choque térmico 90 (Hsp90) y el ciclo de división celular 37 (Cdc37). Hsp90 es una chaperona molecular dependiente de ATP que desempeña un papel esencial en el plegamiento, la estabilidad, la maduración y el control de calidad de muchas proteínas, regulando así múltiples funciones celulares vitales. Cdc37 se considera una chaperona molecular específica de la proteína quinasa, ya que reconoce y recluta específicamente proteínas quinasas para Hsp90 para regular sus vías de transducción de señales aguas abajo. Como tal, Cdc37 se considera un co-chaperone de Hsp90. La vía chaperona-quinasa (complejo Hsp90/Cdc37) está hiperactivada en múltiples neoplasias malignas que promueven el crecimiento celular; por lo tanto, es un objetivo potencial para la terapia contra el cáncer. El presente estudio demuestra la eficiencia de la tecnología FEB utilizando el sistema modelo Hsp90/Cdc37. FEB detectó un fuerte PPI entre las dos proteínas (valores de KD de 0,014 μM, 0,053 μM y 0,072 μM en tres experimentos independientes). En resumen, FEB es una plataforma de detección de PPI sin etiquetas y rentable, que ofrece mediciones rápidas y precisas.
Introducción
Interacciones biomoleculares:
Las proteínas son partes esenciales de los organismos y participan en numerosas vías moleculares como el metabolismo celular, la estructura celular, la señalización celular, las respuestas inmunes, la adhesión celular y más. Mientras que algunas proteínas realizan su(s) función(es) de forma independiente, la mayoría de las proteínas interactúan con otras proteínas utilizando una interfaz de unión para coordinar la actividad biológica adecuada1.
Las interacciones biomoleculares se pueden clasificar principalmente en función de las distintas características estructurales y funcionales de las proteínas involucradas2, por ejemplo, en función de las superficies de las proteínas, la estabilidad compleja o la persistencia de las interacciones3. La identificación de proteínas esenciales y su papel en las interacciones biomoleculares es vital para comprender los mecanismos bioquímicos a nivel molecular4. Actualmente, existen varios enfoques para detectar estas interacciones5: in vitro6, in silico7, en células vivas8, ex vivo9 e in vivo10, cada una con sus propias fortalezas y debilidades.
Los ensayos in vivo se realizan utilizando todo el animal como herramienta experimental11, ylos ensayos ex vivo se realizan en extractos de tejidos u órganos enteros (por ejemplo, corazón, cerebro, hígado) en un entorno externo controlado proporcionando alteraciones mínimas en condiciones naturales. La aplicación más común de los estudios in vivo y ex vivo es evaluar los efectos farmacocinéticos, farmacodinámicos y de toxicidad de los posibles agentes farmacológicos antes de los ensayos en humanos garantizando su seguridad y eficaciagenerales 12.
Las interacciones biomoleculares también se pueden detectar dentro de las células vivas. Las imágenes de células vivas nos permiten observar interacciones dinámicas a medida que ejecutan las reacciones de una vía bioquímica particular13. Además, las técnicas de detección, como la bioluminiscencia o la transferencia de energía de resonancia de fluorescencia, pueden proporcionar información sobre dónde y cuándo ocurren estas interacciones dentro de la célula14. Aunque la detección en células vivas ofrece detalles cruciales, estas metodologías de detección se basan en ópticas y etiquetas, que pueden no reflejar la biología nativa; también están menos controlados que los métodos in vitro y requieren experiencia especializada para realizar15.
Los métodos computacionales in silico se utilizan principalmente para el cribado a gran escala de moléculas diana antes de los experimentos in vitro . Los métodos de predicción computacional, las bases de datos basadas en computadora, el acoplamiento molecular, las relaciones cuantitativas estructura-actividad y otros enfoques de simulación de dinámica molecular se encuentran entre las herramientas in silico bien establecidas16. En comparación con las laboriosas técnicas experimentales, las herramientas in silico pueden hacer predicciones fácilmente con alta sensibilidad, pero con una precisión reducida en el rendimiento predictivo17.
Los ensayos in vitro se realizan con microorganismos o moléculas biológicas fuera de su contexto biológico estándar. Retratar las interacciones biomoleculares a través de métodos in vitro es fundamental para comprender las funciones de las proteínas y la biología detrás de la compleja red de funcionamiento celular. La metodología de ensayo preferida se elige de acuerdo con las propiedades intrínsecas de la proteína, los valores cinéticos y el modo e intensidad de las interacciones18,19.
La interacción Hsp90/Cdc37:
La vía chaperona-quinasa, que conecta Hsp90 y Cdc37, es una diana terapéutica prometedora en biología tumoral20. Hsp90 desempeña un papel central en el control del ciclo celular, el ensamblaje de proteínas, la supervivencia celular y las vías de señalización. Las proteínas que dependen de Hsp90 para sus funciones se entregan a Hsp90 para su complejación a través de una co-chaperona, como Cdc37. El complejo Hsp90/Cdc37 controla el plegamiento de la mayoría de las proteínas quinasas y sirve como centro para una multitud de redes de señalización intracelular21. Es una diana antitumoral prometedora debido a su elevada expresión en diversas neoplasias malignas, incluyendo leucemia mieloblástica aguda, mieloma múltiple y carcinoma hepatocelular22,23.
Técnicas de detección de interacción biomolecular in vitro de uso común
La coinmunoprecipitación (co-IP) es una técnica que se basa en la especificidad antígeno-anticuerpo para identificar interacciones biológicamente relevantes24. La principal desventaja de este método es su incapacidad para detectar interacciones de baja afinidad y valores cinéticos24. Se prefieren métodos biofísicos como la calorimetría de titulación isotérmica (ITC), la resonancia de plasmón de superficie (SPR), la interferometría de biocapa (BLI) y la tecnología FEB para determinar los valores cinéticos.
ItC es un método de detección biofísica basado en la determinación de la energía de unión junto con un análisis termodinámico completo para caracterizar las interacciones biomoleculares25. La principal ventaja de ITC es que no requiere ningún etiquetado o fijación de la proteína objetivo. Las principales dificultades encontradas por el ITC son la alta concentración de proteína objetivo requerida para un experimento y la dificultad para analizar complejos no covalentes debido a pequeñas entalpías de unión26. Tanto SPR como BLI son técnicas biofísicas sin etiquetas que se basan en la inmovilización de la molécula diana en la superficie del sensor, seguida de inyecciones posteriores del analito sobre el objetivo inmovilizado27,28. En SPR se miden alteraciones en el índice de refracción durante las interacciones biomoleculares27; en BLI, la interferencia en la luz reflejada se registra en tiempo real como un cambio en la longitud de onda en función del tiempo28. Tanto SPR como BLI comparten ventajas comunes de ofrecer capacidades de alta especificidad, sensibilidad y detección29. En ambos métodos, la proteína diana se inmoviliza en las superficies de los biosensores y, por lo tanto, puede haber cierta pérdida de la conformación nativa del objetivo, lo que dificulta la discriminación entre interacciones específicas vs. no específicas30. BLI utiliza costosos biosensores desechables de fibra óptica para inmovilizar el objetivo y, por lo tanto, es una técnica costosa31. En comparación con estas herramientas de detección biomolecular bien establecidas, la tecnología FEB ofrece una plataforma confiable y libre de etiquetas mediante el uso de bajas concentraciones nanomolares para la detección biomolecular en tiempo real con caracterización cinética. La tecnología FEB también supera los burbujeantes desafíos que enfrenta el ITC y es más rentable en comparación con SPR o BLI.
Los biosensores basados en transistores de efecto de campo (FET) son un campo emergente para detectar interacciones biomoleculares al ofrecer diversas aplicaciones biomédicas. En el sistema FET, los objetivos se inmovilizan a los chips del biosensor y las interacciones se detectan mediante cambios en la conductancia32. La característica única a considerar en el desarrollo de un biosensor electrónico eficiente son las propiedades fisicoquímicas como la naturaleza semiconductora y la estabilidad química del material de recubrimiento utilizado para fabricar la superficie del sensor33. Los materiales convencionales como el silicio utilizado para FET han limitado la sensibilidad de los sensores porque requiere capas de óxido intercaladas entre el canal del transistor y un entorno específico para un funcionamiento adecuado34. Además, los transistores de silicio son sensibles a ambientes con alto contenido de sal, lo que dificulta la medición de las interacciones biológicas en su entorno natural. El biosensor basado en grafeno se presenta como una alternativa, ya que ofrece una excelente estabilidad química y campo eléctrico. Dado que el grafeno es una sola capa atómica de carbono, es extremadamente sensible como semiconductor y químicamente compatible con soluciones biológicas; ambas cualidades son deseables para generar biosensores electrónicos compatibles35. El notable potencial de carga ultra alta de las biomoléculas ofrecidas por los biosensores recubiertos de grafeno conduce al desarrollo de la tecnología FEB de biosensores basados en grafeno.
Principio de la tecnología FEB: FEB es una técnica de detección biomolecular sin etiqueta que mide la corriente eléctrica a través del biosensor de grafeno al que se inmovilizan los objetivos de unión. Las interacciones entre la proteína inmovilizada y el analito dan lugar a alteraciones en la corriente que se monitorizan en tiempo real, lo que permite mediciones cinéticas precisas36.
Instrumentación: El sistema FEB comprende un chip sensor de transistor de efecto de campo de grafeno (gFET) y un lector electrónico que aplica un voltaje constante durante todo el experimento (Figura 1). El analito se aplica en solución a la proteína diana inmovilizada en la superficie del biosensor. Cuando se produce una interacción, se mide y registra una alteración en la corriente en tiempo real. A medida que aumenta la concentración de analitos, la fracción de analito unido también aumentará, causando mayores alteraciones en la corriente. Utilizando el software de análisis automatizado proporcionado con el instrumento (Tabla de Materiales), I-Response se mide y registra en términos de unidades de biodetección (BU)37. I-Response se define como la alteración en la corriente (I) a través del chip biosensor medida en tiempo real sobre la interacción del objetivo inmovilizado con el analito. El software de análisis automatizado FEB puede analizar tanto la I-Response como la C-Response a eventos de interacción dinámica, donde la C-Response registra las alteraciones en la capacitancia (C). Las variaciones tanto en la I-Response como en la C-Response corresponden directamente a la fracción de analito unido y pueden analizarse más a fondo para generar valores KD . La preferencia predeterminada del software de análisis automatizado es I-Response.
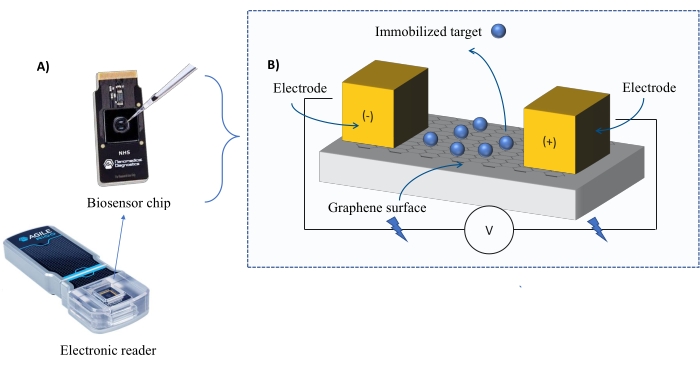
Figura 1: Descripción general de la configuración experimental. (A) Chip basado en grafeno y un lector electrónico. (B) Una visión general de los componentes del chip. El chip está unido a dos electrodos que suministran corriente al sistema. La superficie del chip está cubierta con grafeno, que cuando se activa puede unir al objetivo. Haga clic aquí para ver una versión más grande de esta figura.
Metodología:
Inicialmente, el chip biosensor activado se inserta en el dispositivo FEB (Figura 1) seguido de la ejecución de los pasos descritos a continuación: (1) Calibración: El experimento comienza con la calibración del sistema utilizando 1x solución salina tamponada con fosfato (PBS; pH = 7.4) para crear la respuesta de equilibrio basal. (2) Asociación: El analito se introduce en el chip, y el I-Response se monitorea hasta que se alcanza la saturación de unión. (3) Disociación: El analito se disocia usando 1x PBS. (4) Regeneración: Los restos del analito se eliminan usando 1x PBS. (5) Lavado: Se realizan un total de cinco lavados utilizando 1x PBS para la eliminación completa de los analitos unidos y no unidos del chip.
Análisis:
El análisis de datos se realiza utilizando el software totalmente automatizado proporcionado con el instrumento. El software de análisis automatizado genera un diagrama de ajuste de Hill con un valor KD . La gráfica de ajuste de Hill describe la asociación de un analito a la proteína objetivo en función de las concentraciones de analitos. La concentración a la que se alcanza una respuesta medio máxima es proporcional al valor KD . Un valor bajo de KD representa una alta afinidad de unión y viceversa.
Para validar los datos obtenidos del experimento FEB, las respuestas I se extraen de cada punto de lectura para cada concentración de analito utilizando el software de revisión / exportación de datos y se pueden exportar a otro software de análisis estadístico (consulte la Tabla de materiales) como se explica a continuación.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: Las proteínas recombinantes utilizadas en este estudio, Hsp90 y Cdc37, se obtuvieron comercialmente (ver Tabla de Materiales).
1. Activación del chip
NOTA: Todos los materiales que se utilizarán en el experimento se enumeran en la Tabla de materiales. Filtre todas las soluciones preparadas a través de un filtro estéril de 0,2 μm.
- Preparar la solución de 1-etil-3-(3-dimetilaminopropil) carbodiimida (EDC) añadiendo 2 mg de EDC a 2,5 ml de tampón de ácido sulfónico etano (MES) de 1 M de 2-(N-morfolino) (pH = 6,0) en un tubo de 15 ml. Preparar soluciones de N-hidroxisulfosuccinimida (sulfo-NHS) añadiendo 6 mg de sulfo-NHS a 2,5 ml de tampón MES de 1 M (pH = 6,0) en un tubo separado de 15 ml. Alícuota 50 μL de cada solución en tubos independientes y guárdela a -20 °C para su uso futuro.
- Mezclar volúmenes iguales de solución de sulfo-NHS de arena EDC (50 μL de EDC + 50 μL de sulfo-NHS) mediante pipeteo hacia arriba y hacia abajo (no vórtice).
NOTA: La solución mixta de EDC/sulfo-NHS debe utilizarse en un plazo de 30 minutos para mantener una reticulación efectiva para una correcta funcionalización del chip. - Coloque el chip biosensor (5,7 cm x 2,4 cm; ver Tabla de materiales) suministrado por la empresa en una placa de Petri de vidrio con una tapa ajustada. Se sugiere que todos los pasos de funcionalización involucrados en la activación del chip se realicen dentro de la placa de Petri. Aplique 50 μL de tampón MES de 1 M (pH = 6.0) al chip biosensor, incube durante 1 min a temperatura ambiente y luego aspire el tampón.
- Aplique 50 μL de solución EDC/sulfo-NHS inmediatamente al chip del sensor. Cubra la placa de Petri e incube durante 15 minutos a temperatura ambiente. Aspire la solución EDC/sulfo-NHS del chip.
- Enjuague el chip una vez con 50 μL de tampón MES de 1 M (pH = 6.0); aspirar el tampón MES.
2. Inmovilización de proteínas diana
- Enjuague el chip 2x con 50 μL de 1x PBS (pH = 7.4). Aspire el PBS del chip y agregue la molécula objetivo, Hsp90 (50 μL; 500 nM).
NOTA: La falta de coincidencia del búfer puede arruinar todo el experimento; por lo tanto, antes del experimento, es importante asegurarse de que la molécula objetivo esté en el mismo tampón que el utilizado para la calibración (por ejemplo, 1x PBS (pH = 7.4)). Si es necesario, realice un intercambio de tampón mediante diálisis durante la noche antes del experimento. En este experimento, se realizó diálisis durante la noche para ambas proteínas recombinantes, Hsp90 y Cdc37, contra 1x PBS (pH = 7.4; ver Tabla de Materiales) con un intercambio tampón adecuado a 4 °C. La concentración del material objetivo (Hsp90 en este caso) puede variar según los diferentes protocolos experimentales y la naturaleza de los materiales objetivo (proteína/péptido/ligandos). - Cubra la placa de Petri de vidrio e incube durante 30 minutos a temperatura ambiente. Aspire la solución que contiene la molécula diana y enjuague 3x con 50 μL de 1x PBS (pH = 7.4). Aspire la solución 1x PBS (pH = 7.4) del chip.
- Añadir 50 μL de solución Quench 1 (3,9 mM amino-PEG5-alcohol en 1x PBS (pH = 7,4)) al chip. Cubra la placa de Petri de vidrio e incube durante 15 min a temperatura ambiente. Aspire la solución Quench 1 del chip.
- Añadir 50 μL de solución Quench 2 (1 M de etanolamina (pH = 8,5)) al chip. Cubra la placa de Petri de vidrio e incube durante 15 minutos a temperatura ambiente. Aspire la solución Quench 2 del chip y enjuague el chip 5x usando 50 μL de 1x PBS, dejando la última gota de PBS en el sensor.
3. Preparación de muestras de analitos
- Preparar series de dilución de analitos para Cdc37 en el rango de concentración deseado. Para el primer experimento se utilizaron las siguientes concentraciones: 25 nM, 50 nM, 100 nM, 200 nM, 400 nM, 800 nM, 1.000 nM, 2.000 nM, 3.000 nM y 5.000 nM. Para el segundo experimento, se utilizó un conjunto diferente de concentraciones que van desde 0,4 nM a 200 nM.
- Diseñe el experimento para incluir al menos ocho concentraciones diferentes de analitos para obtener un valor KD confiable. Preparar las diferentes diluciones de la proteína de analito en el mismo tampón que el utilizado para la calibración y la proteína objetivo; aquí es 1x PBS (pH = 7.4).
4. Carga del chip biosensor activado en el dispositivo FEB
NOTA: El dispositivo FEB consiste en un lector con indicaciones de luz LED y un cartucho para insertar el chip biosensor.
- Después de la inmovilización de la proteína objetivo, inserte el chip activado en el cartucho del dispositivo, que está conectado a través de USB a una computadora. Después de la inserción del chip, se mostrará una luz LED verde en el lector que indica que el dispositivo FEB está listo para el experimento. Instale el software automatizado (consulte la Tabla de materiales) suministrado por la empresa en la computadora, a la que está conectado el dispositivo FEB, para monitorear el experimento paso a paso como se describe a continuación.
5. Ejecuta el experimento
- Presione el módulo Ejecutar experimento en el software automatizado y elija 10 puntos con regeneración o cualquier otro protocolo deseado. Complete los siguientes detalles: nombre del operador, nombre del experimento, fecha (por ejemplo, Yana, Hsp90 + Cdc37, 14.03.2021); tampón de regeneración (por ejemplo, tampón PBS); objetivo inmovilizado (por ejemplo, Hsp90); analito en solución (por ejemplo, Cdc37). Consulte la Figura suplementaria S1 para obtener más información.
- Pulse el botón Iniciar el experimento que se muestra en el software y siga las instrucciones que muestra el software automatizado como se describe a continuación.
NOTA: El software está totalmente automatizado, es fácil de usar y guía al usuario a lo largo del experimento paso a paso. Aparecerá una ventana emergente en la pantalla con instrucciones para continuar en cada paso del experimento. El software proporcionará instrucciones para cada paso repetitivo consecutivamente desde la calibración, la asociación de analitos, la disociación, la regeneración y el lavado (5x) para cada concentración de analitos a lo largo del experimento. - Realizar la calibración del instrumento. Para ello, aspire la solución de PBS restante del chip y aplique 50 μL de tampón de calibración (1x PBS; pH = 7.4). Pulse el botón Continuar y espere 5 minutos hasta que finalice el paso de calibración. El software muestra el punto final determinado para el paso de calibración (5 min) con una alarma de advertencia para el seguimiento.
- A continuación, realice una asociación de analitos. Para ello, aspire el tampón de calibración del chip y aplique 50 μL de la concentración de analito más baja (25 nM de Cdc37). Pulse el botón Continuar y espere 5 minutos hasta que finalice el paso de asociación. El software muestra el punto final para el paso de asociación (5 min) con una alarma de advertencia para continuar.
- Realizar una disociación de analitos. Para ello, aspire la solución de analito del chip y aplique 50 μL del tampón de disociación (1x PBS; pH = 7.4). Pulse el botón Continuar y espere 5 minutos hasta que finalice la duración del paso de disociación (5 min). El software muestra el punto final para el paso de disociación (5 min) con una alarma de advertencia para el seguimiento.
- A continuación, realice la regeneración del chip. Aspire la solución de disociación del chip y aplique 50 μL de tampón de regeneración (1x PBS; pH = 7.4). Pulse el botón Continuar y espere 30 s hasta que finalice la duración del paso de regeneración (30 s). El software muestra el punto final para el paso de regeneración (30 s) con una alarma de advertencia para el seguimiento.
- Por último, lava el chip. Aspire la solución de regeneración del chip y aplique 50 μL de tampón de lavado (1x PBS; pH = 7.4) al chip. Aspira la solución del chip y repite esto 5x. Deje la última gota de búfer de lavado en el chip y presione el botón Continuar y espere 30 s hasta que finalice la duración del paso de lavado en la pantalla del software.
NOTA: El software muestra el punto final para el paso de lavado (30 s) con una alarma de advertencia para continuar con el siguiente ciclo del experimento. - Repita los pasos para cada concentración de analito utilizada; los cinco pasos de calibración, asociación de analitos, disociación, regeneración y lavado (5x) constituyen un ciclo. Para el experimento que se muestra aquí, realizamos 10 ciclos para 10 concentraciones de analitos (que van desde 25 nM a 5,000 nM o 0.4 nM a 200 nM; Figura 2).
6. Análisis
- Presione el botón Análisis que se encuentra en la parte superior del software de análisis automatizado al final del experimento. Aparecerá una ventana de visualización que contiene todos los puntos experimentales. En la ventana, asegúrese de que las concentraciones de analitos utilizadas para el protocolo prescrito sean correctas.
- Pulse el botón Ejecutar análisis para generar el valor KD automáticamente. El software genera una gráfica de ajuste de Hill trazando las concentraciones de analitos contra las I-Respuestas correspondientes a partir de las cuales se calcula la constante de disociación en equilibrio, el valor KD .
- Exporte los datos sin procesar para su análisis en otro software de análisis estadístico utilizando el software de revisión/exportación de datos como se describe a continuación.
- Copie el archivo R1R creado automáticamente al final del experimento (por ejemplo, Hsp90 + Cdc37 14.03.2021) en una nueva carpeta en el escritorio. Abra el software de revisión/exportación de datos suministrado por la empresa (consulte la figura complementaria S2A).
- Haga clic en Procesamiento de datos > procesar archivos R1R > Aceptar en la pantalla de inicio del software de revisión / exportación de datos.
- Seleccione la carpeta que contiene los archivos R1R creados en el escritorio en el paso 6.3.1. y pulse el botón OK . Esto crea una copia de los datos originales de R1R, para revisar y editar los datos sin anular el archivo original.
- Presione el icono de procesamiento de datos que se ve en la pantalla de inicio del software de revisión / exportación de datos. Presione Cargar archivos R1R procesados > Aceptar. Seleccione la misma carpeta creada en el paso 6.3.1 que contiene los archivos R1R procesados. Pulse OK. En este paso, la carpeta que contiene los archivos del experimento está lista para ser revisada.
- En la pantalla de inicio del software de revisión/exportación de datos, pulse Análisis de datos. Seleccione Calibración > Calibrar archivos R1R editados > Aceptar (consulte la Figura suplementaria S2B). Este paso calibra todos los puntos de datos de acuerdo con el primer paso de calibración, para crear una línea de base.
- En la pantalla de inicio del software de revisión/exportación de datos, pulse Análisis de datos. Seleccione Revisar y editar archivos R1R > Aceptar. Revise los puntos de datos, elimine puntos o agregue pasos mediante el botón Eliminar/Agregar paso. Asegúrese de que todos los pasos estén en el lugar correcto, por ejemplo, el paso de calibración está en la línea de base, el paso de asociación está en el pico (consulte la Figura suplementaria S3) y, a continuación, presione el botón Guardar para guardar todos los cambios realizados.
- En la pantalla de inicio del software de revisión/exportación de datos, pulse Análisis de datos. Seleccione Analizar/Trazar datos, elija los pasos para exportar (consulte la Figura suplementaria S4). Agregue cada paso de asociación (por ejemplo, paso 2, paso 7, etc.) después de restar el paso de calibración (por ejemplo, paso 1, paso 6, etc.) a la lista de exportación antes de exportar los datos.
- Pulse Exportar estos datos. El software genera un archivo de hoja de cálculo que contiene la I-Response para cada punto de datos de concentración de analitos de cada transistor (si todos los transistores están funcionando, tendremos tres valores diferentes de I-response para cada punto de concentración). Utilice este archivo de hoja de cálculo para analizar más a fondo los datos en el software estadístico.
- Abra el software de análisis estadístico. Crear una tabla XY con tres valores Y; presione Crear. Los datos de esta tabla (valores X, Y) se copian del archivo de hoja de cálculo creado en el paso 6.3.8. El eje x corresponde a la concentración de analito (dependiendo de la concentración utilizada), y los tres valores Y corresponden a las I-Respuestas, obtenidas del software de Revisión de Datos/Exportación suministrado por la empresa.
- Pulse Analizar estos datos. Elija Análisis XY > regresión no lineal (ajuste de curva) > saturación de enlace > un sitio > total. El software analizará automáticamente los datos, generará el valor KD y creará un gráfico de los puntos de datos.
Access restricted. Please log in or start a trial to view this content.
Resultados
Resultados del experimento 1:
La proteína diana Hsp90 (500 nM) se inmovilizó en el chip siguiendo el protocolo de inmovilización objetivo descrito anteriormente. Para el primer experimento, se prepararon 10 concentraciones de la proteína analito, Cdc37, que van desde 25 nM a 5.000 nM, en base a los datos disponibles en la literatura (ver Tabla 1).
Los pasos del experimento pueden ser monitoreados en tiempo real siguiendo las alteraciones que ocurren en ...
Access restricted. Please log in or start a trial to view this content.
Discusión
En este estudio, se evaluó la viabilidad de utilizar la tecnología FEB (un enfoque de caracterización cinética en tiempo real) para determinar la interacción biomolecular entre Hsp90 y Cdc37. El experimento exploratorio inicial (primer experimento) sugirió que la elección de las concentraciones adecuadas de analito es una parte crítica del experimento y que el experimento debería diseñarse incluyendo puntos de concentración por encima y por debajo del valor de KD , que se predijeron en función de l...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran que no hay conflictos de intereses, financieros o de otro tipo.
Agradecimientos
Esta investigación fue apoyada por una subvención de la Biational Science Foundation (BSF) a S.K.S. y N.Q.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA | biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA | Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA | T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 | White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA | |
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 | White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA | Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 | Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA | Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA | Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA | Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA | |
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA | |
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA | |
| Statistical software | GraphPad, Prism | NA CAS number: NA | Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 | White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA | Tip 1-200 µl, in bulk, 1,000 pcs |
Referencias
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083(2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Cancer Research. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438(2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213(2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), San Diego, Calif. 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , Springer International Publishing. 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882(2014).
- Vu, C. -A., Chen, W. -Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214(2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434(2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era...? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados