Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Detección de alto rendimiento de aislados microbianos con impacto en la salud de Caenorhabditis elegans
* Estos autores han contribuido por igual
En este artículo
Resumen
Los microbios intestinales pueden afectar positiva o negativamente la salud de su huésped a través de mecanismos específicos o conservados. Caenorhabditis elegans es una plataforma conveniente para detectar tales microbios. El presente protocolo describe el cribado de alto rendimiento de 48 aislados bacterianos para determinar su impacto en la resistencia al estrés de los nematodos, utilizados como sustitutos de la salud de los gusanos.
Resumen
Con su pequeño tamaño, corta vida útil y genética fácil, Caenorhabditis elegans ofrece una plataforma conveniente para estudiar el impacto de los aislados microbianos en la fisiología del huésped. También fluoresce en azul cuando muere, proporcionando un medio conveniente para identificar la muerte. Esta propiedad se ha explotado para desarrollar ensayos de supervivencia de C. elegans (LFASS) de alto rendimiento y sin etiquetas. Estos implican el registro de fluorescencia de lapso de tiempo de poblaciones de gusanos colocadas en placas de múltiples pocillos, de las cuales se puede derivar el tiempo medio de muerte de la población. El presente estudio adopta el enfoque LFASS para detectar múltiples aislados microbianos a la vez para detectar los efectos sobre la susceptibilidad de C. elegans al calor severo y al estrés oxidativo. Aquí se informa de esta tubería de detección microbiana, que se puede utilizar en particular para preseleccionar probióticos, utilizando resistencia severa al estrés como un indicador de la salud del huésped. El protocolo describe cómo cultivar colecciones aisladas de microbiota intestinal de C. elegans y poblaciones de gusanos sincrónicos en matrices de pozos múltiples antes de combinarlos para los ensayos. El ejemplo proporcionado cubre la prueba de 47 aislados bacterianos y una cepa de control en dos cepas de gusanos, en dos ensayos de estrés en paralelo. Sin embargo, la tubería de enfoque es fácilmente escalable y aplicable a la selección de muchas otras modalidades. Por lo tanto, proporciona una configuración versátil para estudiar rápidamente un paisaje multiparamétrico de condiciones biológicas y bioquímicas que afectan la salud de C. elegans.
Introducción
El cuerpo humano alberga un estimado de 10-100 billones de células microbianas vivas (bacterias, hongos arquea), que se encuentran principalmente en el intestino, la piel y los ambientes de la mucosa1. En un estado saludable, estos proporcionan beneficios a su huésped, incluida la producción de vitaminas, la maduración del sistema inmunológico, la estimulación de las respuestas inmunes innatas y adaptativas a los patógenos, la regulación del metabolismo de las grasas, la modulación de las respuestas al estrés y más, con un impacto en el crecimiento y el desarrollo, el inicio de la enfermedad y el envejecimiento 2,3,4,5 . La microbiota intestinal también evoluciona considerablemente a lo largo de la vida. La evolución más drástica ocurre durante la infancia y la primera infancia6, pero también ocurren cambios significativos con la edad, incluyendo una disminución en la abundancia de Bifidobacterium y un aumento en las especies de Clostridium, Lactobacillus, Enterobacteriaceae y Enterococcus 7. El estilo de vida puede alterar aún más la composición microbiana intestinal que conduce a la disbiosis (pérdida de bacterias beneficiosas, crecimiento excesivo de bacterias oportunistas), lo que resulta en diversas patologías como la enfermedad inflamatoria intestinal, la diabetes y la obesidad5, pero también contribuye a las enfermedades de Alzheimer y Parkinson 8,9,10,11.
Esta comprensión ha contribuido críticamente a refinar el concepto del eje intestino-cerebro (GBA), donde las interacciones entre la fisiología intestinal (ahora incluyendo los microbios dentro de ella) y el sistema nervioso se consideran el principal regulador del metabolismo animal y las funciones fisiológicas12. Sin embargo, el papel preciso de la microbiota en la señalización intestino-cerebro y los mecanismos de acción asociados están lejos de ser completamente comprendidos13. Dado que la microbiota intestinal es un determinante clave del envejecimiento saludable, la forma en que las bacterias modulan el proceso de envejecimiento se ha convertido en un tema de intensa investigación y controversia 6,14,15.
Con la demostración de que el gusano redondo Caenorhabditis elegans alberga una microbiota intestinal de buena fe dominada, como en otras especies, por Bacteroidetes, Firmicutes y Actinobacteria 16,17,18,19,20, su rápido aumento como plataforma experimental para estudiar las interacciones comensales huésped-intestino21,22,23,24 ,25,26 ha ampliado significativamente nuestro arsenal de investigación26,27,28,29. En particular, los enfoques experimentales de alto rendimiento disponibles para que C. elegans estudien las interacciones gen-dieta, gen-fármaco, gen-patógeno, etc., se pueden adaptar para explorar rápidamente cómo los aislados bacterianos y los cócteles afectan la salud y el envejecimiento de C. elegans.
El presente protocolo describe una tubería experimental para examinar a la vez matrices de aislados o mezclas bacterianas colocadas en placas de múltiples pocillos para detectar efectos sobre la resistencia al estrés de C. elegans como un indicador de la salud, que puede usarse para identificar probióticos. Detalla cómo cultivar grandes poblaciones de gusanos y manejar matrices bacterianas en formatos de placas de 96 y 384 pocillos antes de procesar gusanos para el análisis automatizado de resistencia al estrés utilizando un lector de placas de fluorescencia (Figura 1). El enfoque se basa en ensayos automatizados de supervivencia sin etiquetas (LFASS)30 que explotan el fenómeno de la fluorescencia de muerte31, mediante el cual los gusanos moribundos producen una explosión de fluorescencia azul que puede usarse para determinar el momento de la muerte. La fluorescencia azul es emitida por ésteres glucosílicos de ácido antranílico almacenados en gránulos intestinales de C. elegans (un tipo de orgánulo relacionado con lisosomas), que estallan cuando se desencadena una cascada necrótica en el intestino del gusano al morir31.
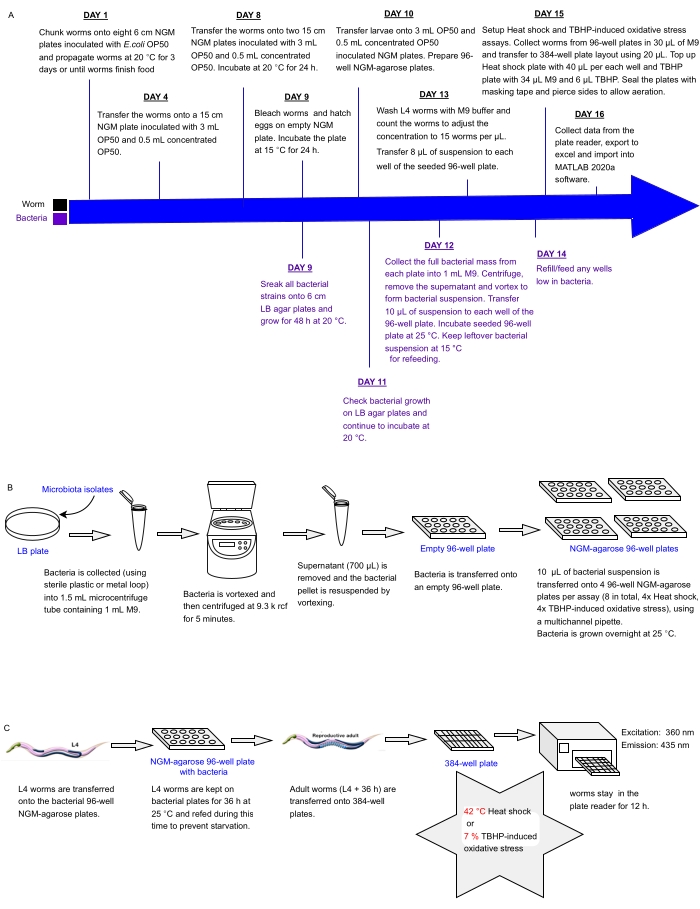
Figura 1: Flujo de trabajo experimental para el cribado de alto rendimiento de aislados bacterianos con impacto en la resistencia de C. elegans al estrés . (A) Línea de tiempo para el mantenimiento de gusanos y bacterias y la configuración del ensayo. (B) Configuración y manejo de la matriz de placas bacterianas de 96 pocillos. (C) Configuración de placa de tornillo sin fin de 384 pocillos. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Las dos cepas de C. elegans utilizadas en paralelo para el presente estudio fueron Bristol N2 wild type y HT1890: daf-16(mgDf50), que crecen a tasas similares. Sin embargo, el protocolo se puede replicar con cualquier combinación de dos cepas que tengan tasas de crecimiento similares. Tenga en cuenta que, al probar otras cepas en paralelo (por ejemplo, mutantes daf-2 de tipo salvaje y de crecimiento lento), se deben considerar diferentes tasas de crecimiento y, en consecuencia, se debe ajustar el protocolo. Las escalas de tiempo y las cantidades de gusanos y bacterias en el siguiente protocolo están optimizadas para pruebas paralelas de 48 aislados bacterianos en dos cepas de gusanos en dos ensayos LFASS en tetraplicados. Se necesitarán ajustes si se van a probar más condiciones en paralelo. Escherichia coli La cepa de la bacteria OP50 se obtuvo del Centro de Genética Caenorhabditis (CGC) de la Universidad de Minnesota. Los 48 aislados bacterianos se obtuvieron del laboratorio de Schulenburg y se mantuvieron en agar LB.
1. Cultivo de C. elegans en OP50 (Días 1 - 8)
NOTA: El enfoque actual tiene como objetivo cultivar C. elegans hermafroditas en un medio sólido en todas las etapas y evita cambios dietéticos innecesarios (es decir, utilizando cepas alternativas de E. coli de crecimiento más rápido como NA22 o medios de crecimiento más ricos como las placas de huevo) para permanecer lo más cerca posible de las condiciones de crecimiento estándar32,33 que todavía se usan ampliamente. La temperatura de crecimiento del gusano (aquí fijada en 15 °C) depende de la(s) cepa(s) de C. elegans utilizada(s) y puede ser necesario ajustarla (por ejemplo, para evitar o desencadenar la expresión de un fenotipo o biomarcador sensible a la temperatura). Para obtener información sobre la cría de lombrices, consulte la referencia33.
- Preparar ocho placas NGM de 6 cm de diámetro (10 mL de agar de medios de crecimiento de nematodos, NGM, Archivo Suplementario 1)32,33 por cepa de gusano y dejar secar durante 1 día a temperatura ambiente.
- Preparar un cultivo líquido saturado de la bacteria E. coli OP50 sembrando un solo clon bacteriano de una placa de agar caldo de lisogenia recién cultivado (agar LB, archivo suplementario 1) en 25 ml de medio OP50 (archivo suplementario 1) en un tubo cónico de 50 ml. Cultivar el cultivo durante la noche a 37 °C en una incubadora agitadora.
- Inocular las ocho placas NGM de 6 cm por cepa con 100 μL de cultivo líquido saturado de E. coli OP50 por placa y mantener las placas a 20 °C durante 2 días antes de su uso.
- Usando un bisturí, cortar y transferir un trozo cuadrado de agar de 0,5 cm con gusanos de una placa NGM recientemente hambrienta a cada una de las ocho placas NGM de 6 cm inoculadas e incubar estas placas a 20 °C durante 3 días (o hasta que los gusanos terminen la comida).
- Preparar cinco placas NGM de 15 cm por cepa de gusano (30 mL de medio NGM por placa) e inocular con 3 mL de OP50. Deje secar las placas antes de incubar a 37 °C durante la noche. Conservar las placas a 20 °C hasta su uso en pasos posteriores.
- Con una pipeta P-1000, añadir hasta 3 ml de tampón M9 estéril (archivo complementario 1) a las placas NGM de 6 cm (paso 1.1.) para resuspender los gusanos y recoger la solución de gusano de las ocho placas por cepa en un solo tubo cónico de 15 ml.
- Centrifugar a 142 x g durante 2 min a 4 °C. Retire con cuidado el sobrenadante con una pipeta P-5000 o una bomba de agua equipada con una pipeta o punta estéril Pasteur. Agregue 10 ml de tampón M9 estéril para lavar el pellet de lombriz. Repita 2x.
- Retirar el sobrenadante (tanto como sea posible) y transferir los gusanos a una placa NGM inoculada OP50 de 15 cm (Paso 1.5.) utilizando una pipeta. Añadir 0,5 ml de cultivo concentrado de OP50.
- Para hacer OP50 concentrado, inocular cada uno de los cuatro frascos de 1 L de LB con 2 ml de cultivo iniciador OP50 (preparado en el Paso 1.2.) y cultivar en una incubadora de agitación durante 6 h a 37 °C y 160 x g. Granular las bacterias a 3057 x g y 20 °C durante 15 min. Deseche el sobrenadante, resuspenda los gránulos bacterianos con 6 ml de medio OP50 y recoja en un tubo cónico estéril de 50 ml.
NOTA: Las bacterias pueden almacenarse a 4 °C durante un máximo de 1 semana.
- Cultivar cada cepa de gusano en una placa NGM de 15 cm de diámetro durante 3-4 días a 15 °C realimentando a los gusanos con 0,5 ml de OP50 concentrado diariamente.
- Una vez que los gusanos hayan terminado casi el alimento, recolectar y lavar en tampón M9 (Paso 1.6.1.), transferir cada cultivo de cepa de gusano a dos placas NGM de 15 cm (Paso 1.5.) y propagar gusanos a 20 °C hasta que ~ 95% de la población sean adultos grávidos (tomará aproximadamente 24 h para Bristol N2 de tipo salvaje).
NOTA: Los adultos grávidos se caracterizan por la presencia de huevos dentro del gusano, y la placa ideal también debe tener una abundancia de huevos no eclosionados puestos en la placa sin demasiadas larvas33.
- Una vez que los gusanos hayan terminado casi el alimento, recolectar y lavar en tampón M9 (Paso 1.6.1.), transferir cada cultivo de cepa de gusano a dos placas NGM de 15 cm (Paso 1.5.) y propagar gusanos a 20 °C hasta que ~ 95% de la población sean adultos grávidos (tomará aproximadamente 24 h para Bristol N2 de tipo salvaje).
2. Mantenimiento de las colecciones aisladas de microbiota intestinal (Día 9)
- Rayar los 48 aislados bacterianos en placas individuales de agar LB de 6 cm y crecer durante 48 h a 20 °C.
NOTA: Las bacterias se pueden cultivar a 25 ° C durante 24-36 h si es necesario antes, pero el crecimiento más largo de 20 ° C permite detectar contaminantes potenciales. - Sincronizar un gran número de C. elegans.
- Blanquear los gusanos adultos grávidos siguiendo el método estándar de preparación de huevos33 y transferir los huevos a dos placas NGM de 15 cm sin semillas durante 24 h a 15 °C para permitir que todas las larvas L1 eclosionen y crezcan sincrónicamente en los pasos siguientes.
PRECAUCIÓN: Tenga cuidado al manipular soluciones de lejía.
- Blanquear los gusanos adultos grávidos siguiendo el método estándar de preparación de huevos33 y transferir los huevos a dos placas NGM de 15 cm sin semillas durante 24 h a 15 °C para permitir que todas las larvas L1 eclosionen y crezcan sincrónicamente en los pasos siguientes.
3. Cultivo de grandes cultivos de C. elegans (Día 10)
- Una vez eclosionadas, recoger las larvas L1 (del Paso 2.2.1.) en 3-4 ml de M9 en un tubo cónico limpio de 15 ml. Pipetear cuatro gotas de 10 μL de solución de gusano en un portaobjetos o una placa y contar el número de gusanos en cada gota bajo un microscopio estereoscópico con un aumento de 16x. Determine la concentración de gusanos de la solución promediando el número de larvas de todas las gotas de solución de gusano. Multiplique este valor por el volumen restante y calcule el recuento total de gusanos para cada cepa.
NOTA: Se requieren 46,000-50,000 larvas L1 por cepa en esta etapa para luego llenar una placa de 384 pocillos o dos medias placas.- Para cada cepa, transferir todas las larvas L1 a dos placas NGM de 15 cm (23.000-25.000 L1 por placa) previamente inoculadas con 3 ml de OP50 (Paso 1.5.) y resembradas con 0,5 ml de OP50 concentrada.
- Incubar a 15 °C, rellenando con 0,5 ml de OP50 concentrado diariamente según sea necesario hasta que los gusanos alcancen la etapa L4.
NOTA: La etapa L4 se caracteriza por un intestino ligeramente más oscuro y un parche blanco en forma de media disco o media luna donde la vulva eventualmente formará32,33. - Prepare placas de 96 pocillos de NGM-agarosa siguiendo los pasos a continuación.
- Prepare ocho placas de NGM-agarosa de 96 pocillos llenando cada pocillo con 125 μL de NGM-agarosa (cuatro placas por ensayo).
NOTA: Se recomienda emplatar algunas placas adicionales si algunas se contaminan en los pasos posteriores. Se requerirán dos lectores de placas para ejecutar ensayos en paralelo, pero también pueden ejecutarse sucesivamente comenzando con el ensayo de estrés por calor, ya que puede ejecutarse durante tan solo 6 h. Para estas placas, el agar ceniza <4% se sustituye con agarosa (ver Tabla de materiales), lo que permite un secado más lento y uniforme a través de los tapones NGM y reduce la madriguera de gusanos para una mejor recuperación. - Asegúrese de que los pozos se llenen de manera uniforme y sin burbujas. Utilice un bloque de calor ajustado a 70 °C (con una transferencia de calor lenta a través del plástico de la placa de pocillos múltiples, la agarosa NGM solo puede calentarse hasta aproximadamente 55-60 °C) para evitar que la mezcla se solidifique durante el proceso. Para eliminar las burbujas dentro de los pozos, use una aguja estéril calentada por llama.
- Deje que las placas de 96 pocillos se fijen a temperatura ambiente en un ambiente estéril antes de invertirlas (tapar hacia abajo para evitar la condensación) y almacenar a 4 °C en una caja limpia hasta que sea necesario.
- Prepare ocho placas de NGM-agarosa de 96 pocillos llenando cada pocillo con 125 μL de NGM-agarosa (cuatro placas por ensayo).
- El día 11, verifique los gusanos del Paso 3.2., asegurándose de que no hayan aparecido contaminaciones y que los gusanos aún estén repletos.
- El día 12, verifique los gusanos del Paso 3.1., asegurándose de que no hayan aparecido contaminaciones y que los gusanos aún estén repletos. Además, verifique la etapa de desarrollo de los gusanos.
NOTA: El sexo/cepa y la etapa de desarrollo del gusano, como los gusanos L4 o L4 + 24 h utilizados, dependen de los tratamientos a los que se someten los gusanos. Aquí, los hermafroditas de tipo salvaje fueron expuestos a aislados bacterianos de L4 durante 36 h.
4. Preparación de colecciones aisladas de microbiota intestinal para realimentar gusanos
- Monitoree el crecimiento bacteriano en las placas de agar LB desde el Paso 2.1. y continuar incubando a 20 °C.
NOTA: Si bien no es ideal, en caso de que algunos clones no crezcan o revelen contaminaciones, las bacterias pueden volver a rayarse de las existencias limpias en placas de 6 cm LB y crecer a 25-28 ° C durante 24 h para estar listas para el experimento. - Definir un diseño de matriz de 96 pocillos para la recolección bacteriana que se está probando, facilitando la siembra sistemática de placas y el análisis de datos en los pasos posteriores (Tabla complementaria 1).
- Recoger la masa bacteriana de cada placa bacteriana de 6 cm (Paso 4.1.) y transferirla a un tubo de microcentrífuga de 1,5 ml marcado que contenga 1 ml de tampón M9. Realice esto utilizando un bucle de plástico estéril de 2 mm de diámetro de un solo uso o un bucle de metal de 5 mm de diámetro. Esterilice el bucle metálico entre las cepas bacterianas sumergiendo etanol al 100%, flameando y enfriando durante 5 s.
- Vortex los tubos de microcentrífuga hasta que los gránulos bacterianos estén completamente resuspendidos (dependiendo de la cepa bacteriana, esto puede tomar ~ 1-10 s).
- Girar hacia abajo a 9.300 x g durante 5 minutos a temperatura ambiente, eliminar 700 μL de sobrenadante y resuspender el pellet bacteriano mediante vórtice.
- Transfiera 200 μL de cada suspensión bacteriana a un solo pocillo de una placa vacía estéril de 96 pocillos de acuerdo con el diseño establecido en el paso 4.2.
- A partir de esta placa, inocular ocho placas de 96 pocillos de AGM-agarosa (preparadas en la etapa 3.3.) con 10 μL de solución bacteriana utilizando una pipeta multicanal e incubar con la tapa puesta a 25 °C durante 24 h. No selle las placas para permitir el secado de la placa y el crecimiento aeróbico bacteriano y para evitar el exceso de condensación.
- Selle la placa de suspensión de 96 pocillos preparada en el paso 4.6. con película de sellado adhesiva limpia (ver Tabla de materiales) y conservar a 15 °C durante un máximo de 5 días. Esto se utilizará para la realimentación de gusanos según sea necesario.
5. Configuración del choque térmico y ensayo oxidativo LFASS (días 13 - 14)
- Mirando las placas del Paso 3.5., evalúe la etapa de desarrollo de los gusanos. Una vez que el >90% de los gusanos hayan alcanzado L434, recoger los gusanos en hasta 10 ml de solución estéril M9 en tubos cónicos de 15 ml.
- Lave los gusanos extensamente (al menos 4 veces) girando hacia abajo a 142 x g durante 2 minutos a 4 °C, retirando el sobrenadante y agregando 10 ml de M9 estéril fresco entre cada lavado para eliminar la bacteria OP50. Resuspender el pellet de lombriz en 10 mL de M9.
- Transfiera 50 μL de solución de gusano a un tubo de unión superficial baja (ver Tabla de materiales) que contenga 950 μL de M9. Después de mezclar suavemente el contenido del tubo para evitar la sedimentación de gusanos, use rápidamente una punta de pipeta húmeda de bajo enlace para transferir 3-4 gotas separadas de 10 μL a un portaobjetos de vidrio o una placa NGM, y cuente el número de gusanos bajo un microscopio estereoscópico (consulte la Tabla de materiales) con un aumento de 16x. Promedie los recuentos de las 3-4 gotas y determine el número de gusanos por microlitro en la solución de gusano (ver Paso 3.1.).
- Ajuste la concentración de gusanos en el tubo de 10 ml para alcanzar ~120 gusanos en 8 μL. Si la solución preparada en el paso 5.2. no está lo suficientemente concentrado, gire los gusanos hacia abajo y elimine M9 en consecuencia para alcanzar 120 gusanos por 8 μL.
- Transfiera 8 μL de solución de gusano (~120 gusanos) a cada uno de los pocillos de las ocho placas de 96 pocillos de angm-agarosa del paso 4.7., utilizando una pipeta multicanal o una pipeta repetida. Asegúrese de usar puntas de retención baja para limitar la pérdida de gusanos. También podría ser necesario cortar los extremos de la punta para permitir que los gusanos adultos grandes limiten el estrés mecánico en los gusanos adultos.
NOTA: El ensayo requiere un mínimo de 30 gusanos vivos para funcionar de manera confiable, pero funciona mejor con aproximadamente 100 gusanos por pocillo. - Incubar las placas de 96 pocillos de gnanomasa de 96 pocillos sembradas por gusanos y bacterias a 25 °C durante 36 h.
- Revise las placas entre las 12 y las 24 h, asegurándose de que los gusanos permanezcan repletos en todo momento. Si es necesario volver a alimentar, resuspender las bacterias dentro de la placa de matriz bacteriana de 96 pocillos almacenada a 15 °C en la etapa 4.8., y añadir hasta 10 μL de la solución bacteriana correspondiente a las placas de 96 pocillos de agarosa NGM en las que los gusanos corran riesgo de inanición antes de que finalice el período de incubación de 36 h (los gusanos hambrientos producirán resultados muy diferentes, así que esto es muy importante).
NOTA: Los siguientes pasos deben llevarse a cabo el día 15. Antes de comenzar el ensayo, puede ser necesario optimizar la altura de lectura. La lectura óptima se logrará 20-50 μm por encima del fondo del pozo. Esto dependerá del modelo del lector de placas. Algunos ofrecen la posibilidad de un Z-scan, mientras que otros permiten la entrada manual de altura. Establezca la altura óptima en el nivel donde se detecta la señal de fluorescencia azul más alta (365 nm/430 nm). Algunos lectores de placas pueden funcionar a una altura fija optimizada para ensayos de células adherentes y pueden no ser ideales para ensayos LFASS. - Después de 36 h, dispensar 30 μL de M9 en cada pocillo de la placa de 96 pocillos.
NOTA: Para los ensayos de esfuerzo térmico, el lector de placas debe haber alcanzado la temperatura requerida para realizar el ensayo y es posible que deba encenderse con anticipación. El protocolo actual utiliza 42 ° C para maximizar la velocidad de muerte, pero el enfoque se aplica a otras temperaturas superiores a 30 ° C. - Transfiera los gusanos (aproximadamente 20 μL) a la placa de 384 pocillos de acuerdo con los diseños establecidos, utilizando puntas de baja retención (considere cortar el extremo de las puntas para permitir que los gusanos grandes reduzcan el estrés mecánico de los gusanos adultos).
NOTA: Para el presente estudio, se utilizan dos configuraciones diferentes de lector de placas para los dos ensayos descritos aquí (estrés térmico y estrés oxidativo) y, por lo tanto, las muestras destinadas a estos dos ensayos no deben chaparse en la misma placa de 384 pocillos. - Asegúrese de que los lectores de placas estén configurados correctamente (Tabla 1).
- Rellene las placas de 384 pocillos con más M9, con el objetivo de obtener un volumen final de 60 μL por pocillo. Para el ensayo de estrés térmico, agregue 40 μL de M9, y para el estrés oxidativo inducido por t-BHP, agregue 34 μl de M9 en 6 μl de t-BHP (consulte la Tabla de materiales).
- Comience el ensayo dentro de los 2 minutos posteriores a la adición de t-BHP (idealmente, todos los gusanos deben estar expuestos a t-BHP simultáneamente, la resolución del tiempo de ensayo es de 2 min). Si no es posible, utilice un temporizador para estimar el tiempo empleado en pipetear t-BHP antes del inicio del ensayo para permitir el ajuste posterior de la mediana de tiempo de muerte.
- Cierre las placas con su tapa transparente. Selle los bordes de las placas de 384 pocillos con cinta adhesiva (pegando cinta adhesiva sobre la placa y la tapa), asegurándose de que la cinta no pase por encima de la tapa o debajo de la placa. Corte la cinta entre la tapa y la placa a intervalos con un bisturí para permitir el intercambio de aire y minimizar la evaporación durante el ensayo.
- Inserte la placa en el lector de placas (consulte Tabla de materiales) e inicie la ejecución. Trate de excitar a 365 nm y detectar la emisión a 435 nm cada 2 min durante 6-12 h (Tabla 1).
NOTA: Normalmente, 6 h es suficiente para ensayos de estrés térmico a 42 °C y 8 h para ensayos de estrés oxidativo t-BHP al 7%.
6. Tratamiento de datos del lector de placas
- Guarde los datos de fluorescencia sin procesar del lector de placas como formatos .txt, .csv o .xls /.xlsx separados por comas o tabulaciones y, a continuación, conviértalos al formato xls /.xlsx. Dependiendo del formato de datos, reorganícelos para que coincidan con el diseño de hoja de Excel necesario para el análisis LFASS. Siga las instrucciones detalladas proporcionadas en la referencia30.
NOTA: Mientras que los datos se pueden analizar manualmente, normalizando cada serie temporal y buscando el momento en que la fluorescencia de muerte alcanza la mitad máximo, el análisis automatizado se puede llevar a cabo en Matlab ejecutando la rutina LFASS30. - Descargue e instale Matlab (versión 2014a o superior) y el paquete de software LFASS desde https://github.com/ABA80/LFASS. Siga las pautas y anotaciones proporcionadas en él.
NOTA: La Figura 1C ofrece una breve descripción del enfoque. Matlab es necesario para ejecutar la rutina LFASS. Alternativamente, el código de Matlab puede ser traducido a Oracle, excepto para la función de ajuste, que es propietaria. Las nuevas funciones de suavizado y sigmoide se pueden reescribir para permitir su uso en una plataforma totalmente de código abierto. - Entre análisis LFASS, mueva los datos y los resultados a una nueva ubicación, ya que el análisis LFASS procesará todos los archivos de la carpeta de datos y sobrescribirá los archivos de la carpeta Resultados.
7. Inspección de datos
- Abra el archivo de Excel y etiquete las filas de acuerdo con la posición del pozo en la placa de 384 pocillos. El archivo complementario 2 muestra un ejemplo del archivo Excel de los datos de fluorescencia sin procesar generados para el ensayo de choque térmico. Use la posición del pozo en la placa de 384 pocillos para etiquetar las cepas de gusanos y bacterias.
- Antes del análisis de Matlab, inspeccione visualmente los datos en Excel, trazando la intensidad de fluorescencia a lo largo del tiempo para un pozo representativo. Dependiendo del lector de placas utilizado, los datos pueden ser ruidosos, pero deben mostrar un pico claro. En particular:
- Determine un valor de fluorescencia por debajo del cual un pico no sería significativamente diferente del ruido (establecer dicho umbral en LFASS acelerará el análisis al excluir los pozos vacíos).
- Tenga en cuenta el punto de tiempo más temprano cuando las fluctuaciones de fluorescencia disminuyen antes de elevarse (los animales pueden golpear vigorosamente durante hasta 30 minutos, lo que lleva a lecturas de fluorescencia azul que fluctúan rápidamente).
NOTA: La conexión de pico puede mejorarse excluyendo estos puntos de tiempo tempranos de la ventana de conexión de curva. - Tenga en cuenta los puntos de tiempo entre los cuales se espera que caigan los valores de fluorescencia mínimos y máximos (observe varios pocillos para identificar estos rangos), ya que se utilizarán para el ajuste de curvas.
- Compruebe si las amplitudes de los picos de fluorescencia varían significativamente entre los pocillos, normalice los datos antes de realizar un análisis adicional utilizando la siguiente fórmula:
Pozo de fluorescencia normalizada n (t) = (Pozo de fluorescencian [t] - pozo de fluorescencia mínima [Dt]) / (pozo de fluorescencia máxima [Dt] - pozo de fluorescencia mínima [Dt])
donde "n" es el número de pozo actual, "t" es el punto de tiempo y "Dt" es la serie de puntos de tiempo para el ensayo.
8. Tratamiento de datos de LFASS
NOTA: Los detalles se proporcionan en https://github.com/ABA80/LFASS y en los materiales complementarios de la Referencia30.
- Cree dos subcarpetas dentro de la carpeta LFASS, una para los datos que se van a analizar y otra para los resultados, por ejemplo, "mis datos" y "resultados".
- Copie el archivo de datos excel del ensayo en la subcarpeta LFASS "mis datos" después de la inspección de datos.
- Inicie MATLAB, navegue hasta la carpeta LFASS , escriba y ejecute fitfolder en la ventana de comandos (Archivo complementario 3). A continuación, siga las instrucciones que aparecen en pantalla.
- Después de escribir "fitfolder", el sistema solicita el nombre de la carpeta en la que se encuentra el archivo de Excel, por ejemplo, 'mis datos'. Escriba el nombre de su carpeta de datos (en este ejemplo, "mis datos").
- Siga las instrucciones en pantalla, proporcionando los diversos parámetros solicitados.
- Introduzca "2" para el intervalo de tiempo entre mediciones sucesivas en el protocolo actual (especificar esto permite que los resultados se expresen en minutos en lugar de unidades de punto de tiempo).
NOTA: El intervalo de tiempo se puede modificar para realizar mediciones de fluorescencia con más o menos frecuencia (para disminuir o aumentar la resolución de tiempo) y también dependiendo de las capacidades del lector de placas (es decir, es posible que sea necesario aumentar el intervalo de tiempo para los lectores de placas que no pueden realizar mediciones lo suficientemente rápidas). Asegúrese de hacer coincidir siempre el intervalo de tiempo experimental con la rutina LFASS. - Asigne el umbral de tolerancia superior escribiendo "0.95" (esto se puede cambiar según sea necesario para mejorar el ajuste) y el umbral de tolerancia inferior escribiendo "0.05" (esto se puede cambiar según sea necesario para mejorar el ajuste) para restringir el ajuste sigmoide.
NOTA: Otros parámetros de tiempo se basan en notas de usuario de la inspección de datos (Paso 7.2.).
- Introduzca "2" para el intervalo de tiempo entre mediciones sucesivas en el protocolo actual (especificar esto permite que los resultados se expresen en minutos en lugar de unidades de punto de tiempo).
- Elija si desea mostrar o no curvas ajustadas y suavizadas escribiendo "y" para SÍ o "n" para NO cuando se le solicite. Para inspeccionar visualmente los ajustes convergentes, seleccione el primero.
NOTA: Este último es útil para visualizar todos los datos suavizados, pero generalmente no se selecciona porque genera demasiados gráficos emergentes. Después de esto, Matlab ejecutará la rutina LFASS, que puede tardar de 1 a 10 minutos si se procesan varios archivos de Excel de una sola vez. Las ventanas emergentes con curvas aparecerán de acuerdo con la selección en el Paso 8.6. El archivo complementario 4A muestra un ejemplo de una curva ajustada. - Elija si desea (1) analizar curvas identificadas como ruido o (2) reacondicionar curvas mal ajustadas con una opción [y/n]. Escriba y para aprobar y n para rechazar.
NOTA: Se recomienda aprobar el reacondicionamiento, especialmente si hay muchas curvas mal ajustadas o no ajustadas. Esto permitirá al usuario proporcionar parámetros de ajuste de curva personalizados para cada curva a medida que aparecen en la pantalla y solo solicitar límites anteriores y posteriores para el ajuste sigmoide. Se puede intentar tantas veces como sea necesario. - Una vez analizados los datos, cierre Matlab y abra la carpeta LFASS .
- Haga clic en la subcarpeta LFASS Mis resultados, ya que los archivos de resultados se guardan automáticamente en la carpeta Resultados como .txt.
NOTA: Matlab genera tres archivos .txt: "Batch-fitted.txt", "Batch and noise-fitted.txt" y "Refitted.txt". Los dos primeros se guardan como precaución en caso de un bloqueo de la computadora o un error del usuario durante el reajuste. El archivo que contiene el análisis completo más preciso es "Refitted.txt". - Abra el archivo Refitted.txt con Microsoft Excel y guárdelo como .xls para su posterior procesamiento. El archivo complementario 4B muestra un ejemplo de dicho archivo de resultados.
NOTA: Para cada pocillo (organizado en filas), se proporcionan tres valores en las columnas que dan estimaciones del tiempo medio de muerte de la población de gusanos: "Crudo": informa el tiempo de intersección en la mitad máximo del pico de datos experimentales; "Instalación por lotes": indica el tiempo de intersección en la mitad como máximo de la curva de instalación por lotes; "Reajustado": informa el tiempo de intersección en la mitad máxima de la curva reajustada. - Guarde el archivo en formato .xls como copia en un lugar seguro. De lo contrario, se correrá el riesgo de que los archivos se sobrescriban durante la próxima ejecución de la rutina LFASS.
NOTA: Los resultados se pueden procesar posteriormente para gráficos o análisis estadísticos.
Access restricted. Please log in or start a trial to view this content.
Resultados
Los ensayos LFASS proporcionan una detección robusta, de alto rendimiento y rápida de múltiples condiciones de prueba a la vez, como la detección de numerosos parámetros genéticos y de microbiota que contribuyen a la resistencia al estrés y al envejecimiento. Solo toma 2-3 semanas para que el experimento adquiera un extenso conjunto de datos de múltiples condiciones de prueba. Las poblaciones de gusanos adultos de tipo salvaje L4 + 36 h fueron expuestas a 42 °C de estrés térmico y 7% de estrés oxidat...
Access restricted. Please log in or start a trial to view this content.
Discusión
C. elegans ofrece muchas ventajas para examinar rápidamente múltiples parámetros experimentales a la vez, debido a su pequeño tamaño, transparencia, rápido desarrollo, corta vida útil, bajo costo y facilidad de manejo. Su genoma, plan corporal, sistema nervioso, intestino y microbioma considerablemente más simples, pero lo suficientemente complejos y similares a los humanos, lo convierten en un poderoso modelo preclínico, donde se puede obtener una visión mecanicista mientras se prueba la eficacia o to...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos al CGC Minnesota (Madison, EUA, NIH - P40 OD010440) por proporcionar cepas de gusanos y OP50 y al Pr. Hinrich Schulenburg (CAU, Kiel, Alemania) por proporcionar todos los aislados microbianos ambientales representados aquí. Este trabajo fue financiado por una subvención UKRI-BBSRC a AB (BB / S017127 / 1). JM está financiado por una beca de doctorado FHM de la Universidad de Lancaster.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 10 cm diameter plates (Non-vented) | Fisher Scientific | 10720052 | Venting is not necessary for bacterial cultures |
| 15 cm diameter plates (Vented) | Fisher Scientific | 168381 | |
| 384-well black, transparent flat bottom plates | Corning | 3712 or 3762 | Not essential to be sterile for fast stress assays |
| 6 cm diameter plates (Vented) | Fisher Scientific | 150288 | Venting is necessary for worm cultures to avoid hypoxia |
| 96-well transparent plates (Biolite) | Thermo | 130188 | |
| Agar (<4% ash) | Sigma-Aldrich | 102218041 | Good quality agar is important for the structural integrity of the culture media, to avoid worm burrowing |
| Agarose | Fisher Scientific | BP1356 | |
| Avanti Centrifuge J-26 XP | Beckman coulter | ||
| Bleach | Honeywell | 425044 | |
| Calcium chloride | Sigma-Aldrich | C5080 | |
| Centrifuge 5415 R | Eppendorf | ||
| Centrifuge 5810 R | Eppendorf | ||
| Cholesterol | Sigma-Aldrich | C8667 | |
| LB agar | Difco | 240110 | |
| LB broth | Invitrogen | 12795084 | |
| LoBind tips | VWR | 732-1488 | Lo-bind reduce worm loss during transfers |
| LoBind tubes | Eppendorf | 22431081 | |
| Magnesium sulfate | Fisher Scientific | M/1100/53 | |
| Plate reader- infinite M nano+ | Tecan | Monochromator setup enables fluorescence tuning but adequate filter-based setups may be used | |
| Plate reader- Spark | Tecan | ||
| Potassium phosphate monobasic | Honeywell | P0662 | |
| Sodium chloride | Sigma-Aldrich | S/3160/63 | |
| Stereomicroscope setup with transillumination base | Leica | MZ6, or M80 | Magnification from 0.6-0.8x up to 40-60x is necessary, as is a good quality transillumination base with a deformable, titable or slidable mirror to adjust contrast |
| t-BHP (tert-Butyl hydroperoxide) | Sigma-Aldrich | 458139 | |
| Transparent adhesive seals Nunc | Fisher Scientific | 101706871 | It is important that it is transparent and that it can tolerate the temperatures involved in the assays. |
| Tryptophan | Sigma-Aldrich | 1278-7099 | |
| Yeast extract | Fisher Scientific | BP1422 |
Referencias
- Krishna, S., et al. Integrating microbiome network: establishing linkages between plants, microbes and human health. The Open Microbiology Journal. 13, 330-342 (2019).
- Amon, P., Sanderson, I. What is the microbiome. Archives of Disease in Childhood - Education & Practice Edition. 102 (5), 257-260 (2017).
- Belkaid, Y., Harrison, O. J. Homeostatic immunity and the microbiota. Immunity. 46 (4), 562-576 (2017).
- Cabreiro, F., Gems, D. Worms need microbes too: microbiota, health and aging in Caenorhabditis elegans. EMBO Molecular Medicine. 5 (9), 1300-1310 (2013).
- Vaga, S., et al. Compositional and functional differences of the mucosal microbiota along the intestine of healthy individuals. Scientific Reports. 10 (1), 14977(2020).
- Nagpal, R., et al. Gut microbiome and aging: Physiological and mechanistic insights. Nutrition and Healthy Aging. 4 (4), 267-285 (2018).
- Mitsuoka, T. Establishment of intestinal bacteriology. Biosci Microbiota Food Health. 33 (3), 99-116 (2014).
- Bonfili, L., et al. Microbiota modulation as preventative and therapeutic approach in Alzheimer's disease. The FEBS Journal. 288 (9), 2836-2855 (2021).
- Vendrik, K. E. W., et al. Fecal microbiota transplantation in neurological disorders. Frontiers in Cellular and Infection Microbiology. 10, 98(2020).
- Wang, Q., et al. The role of gut dysbiosis in Parkinson's disease: mechanistic insights and therapeutic options. Brain. 144 (9), 2571-2593 (2021).
- Zhu, X., et al. The relationship between the gut microbiome and neurodegenerative diseases. Neuroscience Bulletin. 37 (10), 1510-1522 (2021).
- Miller, I. The gut-brain axis: historical reflections. Microbial Ecology in Health and Disease. 29 (1), 1542921(2018).
- Foster, J. A., Rinaman, L., Cryan, J. F. Stress & the gut-brain axis: Regulation by the microbiome. Neurobiology of Stress. 7, 124-136 (2017).
- Coman, V., Vodnar, D. C. Gut microbiota and old age: Modulating factors and interventions for healthy longevity. Experimental Gerontology. 141, 111095(2020).
- Conway, J., Duggal, N. A. Ageing of the gut microbiome: Potential influences on immune senescence and inflammageing. Ageing Research Reviews. 68, 101323(2021).
- Berg, M., et al. Assembly of the Caenorhabditis elegans gut microbiota from diverse soil microbial environments. The ISME Journal. 10 (8), 1998-2009 (2016).
- Dirksen, P., et al. CeMbio - The Caenorhabditis elegans Microbiome Resource. G3: Genes, Genomes, Genetics. 10 (9), 3025-3039 (2020).
- Dirksen, P., et al. The native microbiome of the nematode Caenorhabditis elegans: gateway to a new host-microbiome model. BMC Biology. 14, 38(2016).
- Samuel, B. S., Rowedder, H., Braendle, C., Felix, M. A., Ruvkun, G. Caenorhabditis elegans responses to bacteria from its natural habitats. Proceedings of the National Academy of Sciences of the United States of America. 113 (27), 3941-3949 (2016).
- Zimmermann, J., et al. The functional repertoire contained within the native microbiota of the model nematode Caenorhabditis elegans. The ISME Journal. 14 (1), 26-38 (2020).
- Dinic, M., et al. Host-commensal interaction promotes health and lifespan in Caenorhabditis elegans through the activation of HLH-30/TFEB-mediated autophagy. Aging. 13 (6), 8040-8054 (2021).
- Goya, M. E., et al. Probiotic Bacillus subtilis protects against alpha-Synuclein aggregation in C. elegans. Cell Reports. 30 (2), 367-380 (2020).
- Hacariz, O., Viau, C., Karimian, F., Xia, J. The symbiotic relationship between Caenorhabditis elegans and members of its microbiome contributes to worm fitness and lifespan extension. BMC Genomics. 22 (1), 364(2021).
- Shin, M. G., et al. Bacteria-derived metabolite, methylglyoxal, modulates the longevity of C. elegans through TORC2/SGK-1/DAF-16 signaling. Proceedings of the National Academy of Sciences of the United States of America. 117 (29), 17142-17150 (2020).
- Zhang, F., et al. Natural genetic variation drives microbiome selection in the Caenorhabditis elegans gut. Current Biology. 31 (12), 2603-2618 (2021).
- Zhang, F., et al. High-throughput assessment of changes in the Caenorhabditis elegans gut microbiome. Methods in Molecular Biology. 2144, 131-144 (2020).
- Chan, J. P., et al. Using bacterial transcriptomics to investigate targets of host-bacterial interactions in Caenorhabditis elegans. Scientific Reports. 9 (1), 5545(2019).
- Hartsough, L. A., et al. Optogenetic control of gut bacterial metabolism to promote longevity. Elife. 9, 56849(2020).
- Pryor, R., et al. Host-microbe-drug-nutrient screen identifies bacterial effectors of Metformin therapy. Cell. 178 (6), 1299-1312 (2019).
- Benedetto, A., et al. New label-free automated survival assays reveal unexpected stress resistance patterns during C. elegans aging. Aging Cell. 18 (5), 12998(2019).
- Coburn, C., et al. Anthranilate fluorescence marks a calcium-propagated necrotic wave that promotes organismal death in C. elegans. PLOS Biology. 11 (7), 1001613(2013).
- Porta-de-la-Riva, M., Fontrodona, L., Villanueva, A., Ceron, J. Basic Caenorhabditis elegans methods: synchronization and observation. Journal of Visualized Experiments. (64), e4019(2012).
- Stiernagle, T. Maintenance of C. elegans. WormBook. , 1-11 (2006).
- Naomi, R., et al. Probiotics for Alzheimer's disease: a systematic review. Nutrients. 14 (1), 20(2021).
- Zheng, S. Y., et al. Potential roles of gut microbiota and microbial metabolites in Parkinson's disease. Ageing Research Reviews. 69, 101347(2021).
- Gill, M. S., Olsen, A., Sampayo, J. N., Lithgow, G. J. An automated high-throughput assay for survival of the nematode Caenorhabditis elegans. Free Radical Biology and Medicine. 35 (6), 558-565 (2003).
- Park, H. -E. H., Jung, Y., Lee, S. -J. V. Survival assays using Caenorhabditis elegans. Molecules and Cells. 40 (2), 90-99 (2017).
- Partridge, F. A., et al. An automated high-throughput system for phenotypic screening of chemical libraries on C. elegans and parasitic nematodes. International Journal for Parasitology: Drugs and Drug Resistance. 8 (1), 8-21 (2018).
- Rahman, M., et al. NemaLife chip: a micropillar-based microfluidic culture device optimized for aging studies in crawling C. elegans. Scientific Reports. 10 (1), 16190(2020).
- Stroustrup, N., et al. The Caenorhabditis elegans lifespan machine. Nature Methods. 10 (7), 665-670 (2013).
- Xian, B., et al. WormFarm: a quantitative control and measurement device toward automated Caenorhabditis elegans aging analysis. Aging Cell. 12 (3), 398-409 (2013).
- Brown, A. E., Schafer, W. R. Unrestrained worms bridled by the light. Nature Methods. 8 (2), 129-130 (2011).
- Churgin, M. A., et al. Longitudinal imaging of Caenorhabditis elegans in a microfabricated device reveals variation in behavioral decline during aging. Elife. 6, 26652(2017).
- Jushaj, A., et al. Optimized criteria for locomotion-based healthspan evaluation in C. elegans using the WorMotel system. PLoS One. 15 (3), 0229583(2020).
- Nambyiah, P., Brown, A. E. X. Quantitative behavioural phenotyping to investigate anaesthesia induced neurobehavioural impairment. Scientific Reports. 11 (1), 19398(2021).
- Squiban, B., Belougne, J., Ewbank, J., Zugasti, O. Quantitative and automated high-throughput genome-wide RNAi screens in C. elegans. Journal of Visualized Experiments. (60), e3448(2012).
- Zugasti, O., et al. Activation of a G protein-coupled receptor by its endogenous ligand triggers the innate immune response of Caenorhabditis elegans. Nature Immunology. 15 (9), 833-838 (2014).
- Zugasti, O., et al. A quantitative genome-wide RNAi screen in C. elegans for antifungal innate immunity genes. BMC Biology. 14, 35(2016).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados